基于SRAP分子标记的特早熟荔枝种质资源遗传多样性分析
2021-06-15胡福初吴小波陈哲吴凤芝周文静冯学杰范鸿雁周瑞云王祥和
胡福初 吴小波 陈哲 吴凤芝 周文静 冯学杰 范鸿雁 周瑞云 王祥和


摘 要:以22份特早熟、6份早熟及2份中熟荔枝种质资源为材料,利用SRAP分子标记进行遗传多样性分析,探索特早熟荔枝种质资源的亲缘关系。结果表明:对182对SRAP引物组合进行筛选,获得16对多态性高、条带清晰的SRAP引物组合,共计产生扩增条带182条,其中多态性条带140条,占76.9%。材料间遗传相似系数为0.47~0.99,根据UPGMA聚类分析,在遗传相似系数0.73处可将30份荔枝样本分成4类,其中第Ⅰ类20份,占66.7%,包括绝大多数特早熟荔枝资源;第Ⅱ类4份,占13.3%,包括‘妃子笑与部分杂交早熟资源;第Ⅲ类和第Ⅳ类各3份,分别占10%。以上研究结果为特早熟荔枝种质资源发掘、评价及新品种培育提供理论参考。
关键词:荔枝;SRAP标记;特早熟;遗传多样性
中图分类号:S667.1 文献标识码:A
Genetic Diversity Analysis of Very Early Maturing Litchi Germplasm Resources Based on SRAP Molecular Markers
HU Fuchu, WU Xiaobo, CHEN Zhe, WU Fengzhi, ZHOU Wenjing, FENG Xuejie, FAN Hongyan, ZHOU Ruiyun, WANG Xianghe*
Institute of Tropical Fruit Trees, Hainan Academy of Agricultural Sciences / Key Laboratory of Tropical Fruit Tree Biology of Hainan Province / Haikou Investigation Station of Tropical Fruit Trees, Ministry of Agriculture and Rural Affairs, Haikou, Hainan 571100, China
Abstract: Utilizing the SRAP molecular markers technology, the genetic diversity and relationship of 30 litchi germplasm resources, including 22 extremely early maturity varieties, 6 early maturity varieties and 2 middle maturity varieties, were analyzed in this study. The results showed that 16 pairs SPAP primers with high polymorphism and clear bands were selected in 182 pairs SPAP primers. A total of 182 loci were obtained using 16 pairs SPAP primers and 140 loci were polymorphic. UPGMA cluster analysis and genetic similarity coefficient showed that the similarity coefficient of 30 litchi germplasm resources ranged from 0.47 to 0.99, and 30 resources could be grouped into four distinct families based on similarity of 0.73. There were 20 (66.7%) resources in groupⅠ, including the majority of extremely early maturing resources, 4 (13.3%) resources in group Ⅱ, including ‘Feizixiao and some hybrids, 3 (10.0%) resources respectively in group Ⅲ and group Ⅳ. The results would provide theoretical references for the exploration, evaluation and new variety cultivation of extra-early litchi germplasm resources.
Keywords: litchi; SRAP; very early maturing; genetic diversity
DOI: 10.3969/j.issn.1000-2561.2021.04.002
荔枝(Litchi chinensis Sonn.)为无患子科(Sapindaceae)荔枝属(Litchi Sonn.)多年生常绿乔木,在我国热带亚热带地区广泛栽培,具有十分悠久的种植历史。近年来,我国荔枝产业发展兴旺,总面积稳定保持在5.4×105 hm2,产量超过2.0×106 t,已成为一些区域乡村振兴战略的重要抓手,也是连接“一带一路”战略的一个纽带[1]。在荔枝产业优势区域布局及新品种新技术的推动下,我国荔枝已经形成了早熟、中熟、中晚熟、晚熟、特晚熟的生产格局,果实成熟期从4月中旬延续至8月中下旬[2]。其中,早熟產区包括海南、粤西、桂南及云南部分产区,早熟品种主要有‘妃子笑‘白糖罂‘三月红‘桂早荔等,而‘妃子笑‘白糖罂在早熟品种中占有绝对的主导作用,例如在海南产区,‘妃子笑占种植面积的90%以上。需冷量低的早熟优良品种缺乏,导致早熟产区品种结构过于单一,往往造成果实产期集中,给销售带来较大压力。因此,开展早熟荔枝种质资源的发掘、鉴定评价及新品种培育,对于增加早熟品种的遗传多样化类型、优化荔枝品种结构具有重要意义[3]。
采用分子标记技术对荔枝种质资源遗传多样性分析,前人已经开展了大量的研究。丁晓东等[4]利用RAPD分子标记鉴定了34个荔枝品种的亲缘关系,基于37个多态性RAPD标记将34个品种分为4类。李明芳[5]利用22个荔枝特异性SSR标记对37个荔枝种质和2个龙眼品种进行了遗传多样性分析。彭宏祥等[6]利用AFLP标记对27份广西优稀荔枝资源进行了遗传多样性及分类研究。邓穗生等[7]应用RAPD方法对海南60份野生荔枝进行基因组多态性分析,并通过聚类分析将其分为6类。昝逢刚等[8]采用SRAP标记分析了海南46份荔枝种质的遗传多样性,发现相似系数为0.469~0.838,在0.644处可将46份资源划分为4个类群。向旭等[9]利用EST-SSR分子标记对96份荔枝种质资源进行遗传多样性分析,将96份荔枝种质资源分成了8大类群。刘忠等[10]采用SRAP标记对岷江下游41份荔枝古树和3份合江荔枝进行分析,相似系数为0.5586~0.9279。李焕苓等[11]基于SSR和InDel标记对232份主要来自海南的荔枝种质资源进行遗传多样性分析,在遗传相似系数为0.76处将供试材料分为15类。以上研究表明,采用分子标记技术对荔枝种质资源遗传多样性及亲缘关系进行鉴定,具有较好的准确性和可行性。
然而,关于特早熟荔枝种质资源的遗传多样性却少有研究。近年来,我国特早熟荔枝种质资源调查搜集与鉴定评价工作已经取得了一定的进展,为后续特早熟优良新品种的选育提供了材料。早期的调查显示,广西的崇左、百色和河池等地有较长的早熟荔枝种植历史,并且集中分布着比较丰富的早熟实生种质资源[12]。欧阳若等[13]在广西和广东调查分别发现‘桂红‘早荔果等特早熟古树荔枝,并通过自然授粉和杂交育种等方式获得了一批特早熟的育种中间材料。沈庆庆等[14-15]对桂西南早熟荔枝实生资源进行了系统调查,对部分早熟荔枝实生单株进行了鉴定评价,基于ISSR标记开展了遗传多样性分析,为当地早熟资源发掘、保护和利用提供了依据。朱建华等[16]通过实生选育,获得了特早熟优良品种‘桂早荔,并在海南陵水地区试种成功。此外,在云南‘褐毛荔中也发现了大量的特早熟资源,据报道,‘元阳2号和‘元矮一号都具有特早熟的特性,其中‘元阳2号在元阳当地的成熟期在4月下旬至5月中旬[17]。然而,与丰富的中晚熟荔枝资源相比,我国特早熟荔枝种质资源相对匮乏,且遗传背景比较单一[13]。为了系统地研究特早熟荔枝种质资源遗传多样性和亲缘关系,本研究以国内外收集的30份荔枝种质资源为材料,包括特早熟种质22份,早熟种质6份,中熟种质2份,通过SRAP分子标记确定供试种质资源间的遗传背景差异,为构建特早熟荔枝杂交育种模式及选育优良新品种提供研究基础和理论依据。
1 材料与方法
1.1 材料
供试材料全部定植于海南省农业科学院永发果树科研创新基地,共计30份荔枝种质。以传统的特早熟品种‘三月红、早熟品种‘妃子笑及中熟品种‘桂味的成熟期为参照,在正常开花结果情况下,根据同一基地连续3 a果实成熟期的表现,确定4月中旬或者之前进入成熟期的资源为特早熟,4月下旬至5月下旬进入成熟期的资源为早熟,6月上旬至6月中旬进入成熟期的资源为中熟。以此划分,30份荔枝种质中,其中特早熟资源22份,早熟资源6份,中熟资源2份。供试种质资源的编号与基本信息见表1。分别采集对应种质的幼嫩叶片,经无菌水清洗后存于?80 ℃冰箱备用。
1.2 方法
1.2.1 基因组DNA提取 采用改良CTAB法提取样品基因组DNA[18],通过1.2%的琼脂糖凝胶电泳检测DNA质量,使用核酸蛋白测定仪(Biorad SmartSpec Plus)进一步检测DNA纯度与浓度,检测合格后置于?20 ℃冰箱保存备用。
1.2.2 SRAP-PCR扩增 选取13条正向引物和14条反向引物(表2),一共组成182对SRAP引物组合,筛选出条带清晰、多态性丰富且扩增稳定的引物组合进行PCR扩增。SRAP-PCR反应体系参照昝逢刚等[19]的体系并进行优化,在20 μL的反应体系中,加入2×Taq PCR Master Mix(含染料)10.0 L,20 ng/L的DNA模板2 L,10 mol/L的正反向引物各0.5 L。扩增程序为:94 ℃预变性5 min;94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸1 min,5个循环;94 ℃变性1 min,50 ℃退火1 min,72 ℃延伸1 min,35个循环;最后72 ℃延伸10 min。扩增结束后,使用2.0%的琼脂糖凝胶进行电泳检测。
1.3 数据处理
对30份荔枝样品的SRAP检测结果进行分析,相同电泳位置上有条带记为“1”,无条带记为“0”,依据电泳图谱构建0-1矩阵,通过NTSYSpc-2.1e软件计算30份荔枝种质资源间的遗传系数[20],并采用UPGMA法进行聚类分析,构建树状聚类图。
2 结果与分析
2.1 SRAP标记多态性分析
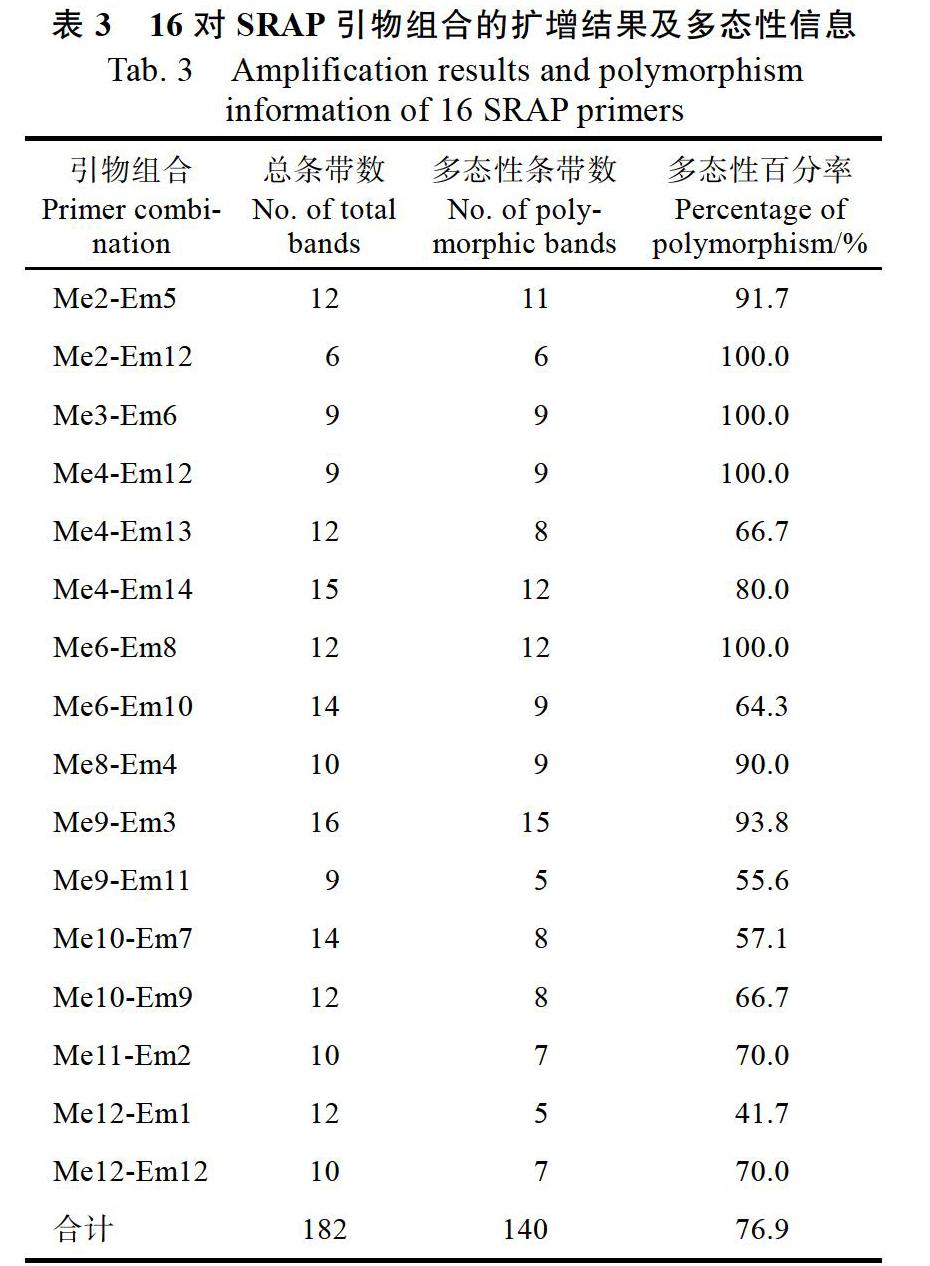
以30份荔枝种质资源的DNA为模板进行SRAP-PCR(图1),从182对引物组合中筛选出条带清晰、多态性好的引物组合16对。由表3可知,16对引物组合共计产生扩增条带182条,其中多态性条带140条,多态性条带比例76.9%。每对SRAP引物组合扩增产生条带6~16條,平均每对引物扩增8.5条,其中有11对引物组合的扩增带达到10条以上,Me4-Em13、Me4-Em14、Me6-Em8、Me6- Em10、Me9-Em3、Me10-Em7、Me10-Em9和Me12-Em1等8对引物扩增条带数在12条以上,Me9-Em3组合的引物扩增条带数达16条。多态性条带比例为41.7%~100%,大多数引物扩增的多态性较高,说明SRAP分子标记可用于特早熟荔枝遗传多样性及亲缘关系分析。
2.2 遗传多样性分析
通过NTSYSpc-2.1e软件计算出30份荔枝种质材料间的遗传相似系数,得到遗传相似系数矩阵表(表4)。遗传相似系数结果显示,供试荔枝种质资源遗传相似系数为0.47~0.99,说明供试荔枝种质间遗传变异较大。其中编号03和17遗传相似系数最大,亲缘关系最近;01和08的遗传相似系数最小,亲缘关系最远。值得注意的是,绝大多数特早熟荔枝种质之间的遗传系数较大,编号01、02、03、05、06、07、09、10、11、13、15、17、19、20、21、26、28、29等18份特早熟种质的亲缘关系几乎都超过了0.85,说明它们之间具有相似的遗传背景,亲缘关系较近。
2.3 聚类分析
根据所得到的遗传相似系数矩阵,利用NTSYSpc-2.1e软件的UPGMA法构建聚类树状图(图2)。聚类分析结果显示,在遗传相似系数0.73处可将供试荔枝材料划分为4个类群。第Ⅰ类群包括‘A20‘99133‘D13‘早优2号‘D14‘早优3号‘99111‘三月红‘B8‘99112‘B7‘B13‘99149‘9918‘A16‘早美人‘D11‘B14‘桂早荔‘褐毛荔等20份种质,这20份材料全部为国内收集的特早熟资源,说明它们具有相似的遗传背景,可能直接或间接来自同一种群;此外,‘D13和‘早优2号相似系数高达0.99,有可能属于同物异名现象。第Ⅱ类群包括‘水东‘妃子笑‘早桂2号‘早糯等4份资源,其中‘早桂2号‘早糯分别是特早熟种质与‘桂味和‘糯米糍的杂交后代,在早熟性状方面具有类似的遗传背景。第Ⅲ类群包括‘KOM‘紫娘喜和‘Kaloka,其中‘KOM和‘Kaloka均来自于泰国,海南本地品种‘紫娘喜与这2个品种有较近的遗传关系,该结果还有待于进一步验证;第Ⅳ类群包括‘桂味‘A4无核荔和‘白糖罂3个品种。来自泰国的特早熟荔枝种质‘KOM和‘Kaloka与国内收集的特早熟荔枝种质亲缘关系较远,遗传相似系数为0.57~0.66。
3 讨论
SRAP分子标记具有操作简单、重复性好、多态性产率高等优点,已经广泛应用于热带果树种质遗传多样性和亲缘关系研究。刘荣等[21]利用SRAP分子标记分析了13份贵州芒果种质资源遗传多样性;张立杰等[22]开展了16份龙眼及1份龙荔种质资源的SRAP遗传多样性分析,鉴定出17份材料间的遗传相似系数范围为0.399~0.871;张颖聪等[23]筛选出28对SRAP引物,完成了20份番木瓜种质资源的亲缘关系和遗传多样性分析。此外,在香蕉[24]、菠萝[25]、荔枝[19]、菠萝蜜[26]、西番莲[27]、澳洲坚果[28]等果树上也有相关报道。以上研究表明,SRAP分子标记技术能较好地反映样本间的遗传变异,可用于开展特早熟荔枝种质资源的遗传多样性分析。
本研究以30份特早熟荔枝为主的荔枝种质资源为材料,包括22份特早熟资源,6份早熟资源和2份中熟资源,分别来自云南、广西、广东、海南等荔枝起源地,其中2份特早熟种质来自泰国本地资源,资源采集的区域具有一定的覆盖范围和代表性。从182对SRAP引物中筛选出引物组合16对用于扩增,获得182条扩增条带,其中多态性条带140条,多态性条带比例76.9%,遗传相似系数为0.47~0.99,0.73处可将供试荔枝材料划分为4个类群;从分子水平来看,遗传距离的变幅越大,表明样本的遗传分化越大,遗传多样性越高,遗传背景越复杂[29],说明供试的30份荔枝材料间具有较丰富的遗传多样性。
聚类结果显示,共有20份特早熟荔枝归属于同一大类,包括‘三月红‘桂早荔‘褐毛荔‘9918等国内起源的所有特早熟种质及部分培育的育种材料。该结果进一步说明了我国特早熟荔枝种质资源相对匮乏,现有种质的特早熟性状可能主要来自云南‘褐毛荔群落,从而导致这些资源具有较为相似的遗传背景,这给特早熟荔枝新品种的选育带来了一定的限制。来自广西的‘桂早荔与‘褐毛荔和‘三月红都具有很高的遗传相似性(0.84和0.82),而朱建华等[16]通过ISSR分子标记鉴定‘桂早荔与‘黑叶亲缘关系最近,遗传相似系数为0.88,意味着‘黑叶与‘褐毛荔可能同样具有较强的亲缘关系。此外,‘水东‘妃子笑‘早桂2号‘早糯等4份早熟种质聚为一类,具有相似的遗传背景,且由于‘早桂2号和‘早糯分别是特早熟‘桂味和‘糯米糍的雜交后代,因而推测‘水东和‘妃子笑也可能是特早熟资源与中晚熟品种通过自然杂交获得的早熟品种。起源于海南的本地品种‘紫娘喜和‘A4无核荔分别聚在第Ⅲ类和第Ⅳ类,二者的遗传相似性为0.73,一定程度上反映了海南荔枝具有较丰富的遗传多样性。来自泰国的特早熟品种‘KOM和‘Kaloka与国内特早熟种质的遗传相似系数较低,为0.57~0.66,说明它们之间存在较明显的基因型差异。根据遗传学研究,在一定范围内,双亲的遗传差异越大,其后代的分离幅度也就越广,从而得到优良后代的机会也就越多[30],预示着这2个泰国品种参与特早熟荔枝新品种的选育,将有助于丰富杂交后代的变异程度。
参考文献
[1]陈厚彬. 荔枝产业发展报告[J]. 现代农业装备, 2018(4): 22-24.
[2]农业部农垦局(南亚办), 中国农垦经济发展中心(南亚中心). 主要热带作物优势区域布局: 2016-2020年[M]. 北京: 中国农业出版社, 2016.
[3]胡福初, 周文静, 陈 哲, 等. 早熟荔枝新品种“桂早荔”在海南陵水的引种表现[J]. 中国南方果树, 2020, 49(1): 89-93.
[4]丁晓东, 吕柳新, 陈晓静, 等. 利用RAPD标记研究荔枝品种的亲缘关系[J]. 热带亚热带植物学报, 2000, 8(1): 49-54.
[5]李明芳. 荔枝SSR标记的研究及其对部分荔枝种质的遗传多样性分析[D]. 儋州: 华南热带农业大学, 2003.
[6]彭宏祥, 曹辉庆, 朱建华, 等. 利用AFLP分子标记对广西荔枝优稀种质遗传多样性及分类研究[J]. 西南农业学报, 2006, 19(1): 108-111.
[7]邓穗生, 陈业渊, 张 欣. 应用RAPD标记研究野生荔枝种质资源[J]. 植物遗传资源学报, 2006, 7(3): 288-291.
[8]昝逢刚, 吴转娣, 曾 淇, 等. 荔枝种质遗传多样性的SRAP分析[J]. 分子植物育种, 2009, 7(3): 562-568.
[9]向 旭, 欧良喜, 陈厚彬, 等. 中国96个荔枝种质资源的EST-SSR遗传多样性分析[J]. 基因组学与应用生物学, 2010, 29(6): 1082-1092.
[10]刘 忠, 李 俊, 杨武云, 等. 利用SRAP分析岷江下游荔枝资源的遗传多样性与亲缘关系[J]. 西南农业学报, 2013, 26(3): 1152-1159.
[11]李焕苓, 田婉莹, 孙进华, 等. 基于SSR和InDel标记的海南荔枝种质资源遗传多样性分析[J]. 分子植物育种, 2018, 16(4): 1343-1356.
[12]吴仁山, 张国辉, 胡友群, 等. 广西荔枝志[M]. 广州: 广东科技出版社, 1986: 100-103.
[13]欧阳若, 胡桂兵, 王泽槐. 我国早熟荔枝种质资源的发掘及研究利用[J]. 中国热带农业, 2005(5): 30-32.
[14]沈庆庆, 朱建华, 彭宏祥, 等. 桂西南早熟荔枝实生资源调查[J]. 中国农学通报, 2011, 27(22): 291-295.
[15]沈庆庆, 朱建华, 彭宏祥, 等. 桂西南早熟荔枝实生资源遗传多样性的ISSR分析[J]. 广西植物, 2013, 33(2): 225-228.
[16]朱建华, 彭宏祥, 曾世江, 等. 早熟荔枝新品种—‘桂早荔的选育[J]. 果树学报, 2014, 31(5): 997-999, 748.
[17]禹崇贵. 早熟高产荔枝良种元阳2号和元矮一号[J]. 云南农业, 1996(9): 12-13.
[18]Puchooa D. A simple, rapid and efficient method for the extraction of genomic DNA from lychee (Litchi chinensis Sonn.)[J]. African Journal of Biotechnology, 2004, 3(4): 253-255.
[19]昝逢刚, 吴转娣, 曾 淇, 等. 荔枝SRAP-PCR反应体系的优化[J]. 基因组学与应用生物学, 2009, 28(1): 132-136.
[20]Rohlf F J. NTSYS-pc: Microcomputer programs for numerical taxonomy and multivariate analysis[J]. American Statistician, 1987, 41(4): 330-330.
[21]刘 荣, 龚德勇, 刘清国, 等. 基于SRAP分子标记的13份贵州芒果种质资源遗传多样性分析[J]. 热带作物学报, 2019, 40(1): 87-91.
[22]张立杰, 陈秀萍, 郑 姗, 等. 龙眼品种SRAP指纹检索系统的开发及遗传多样性研究[J]. 热带作物学报, 2015, 36(02): 219-223.
[23]张颖聪, 任鹏荣, 周常清, 等. 基于SRAP标记的番木瓜种质资源遗传多样性及亲缘关系分析初报[J]. 中国热带农业, 2020(1): 52-59.
[24]漆艳香, 张 欣, 彭 军, 等. 香蕉种质资源的SRAP遗传多样性分析[J]. 分子植物育种, 2017, 15(10): 4220-4227.
[25]竇美安, 邱文武, 吴青松, 等. 菠萝遗传多样性的SRAP分析[J]. 果树学报, 2010, 27(6): 930-937.
[26]吴 刚, 胡丽松, 黄丽芳, 等. 菠萝蜜种质资源遗传多样性的SRAP分析[J]. 热带作物学报, 2015, 36(8): 1385-1391.
[27]夏 玲, 龚家建, 梁昕景, 等. 基于SRAP标记和ITS序列分析西番莲属种质资源遗传多样性[J]. 分子植物育种, 2020, 18(15): 1557-1563.
[28]刘小翠, 范建新, 刘 荣, 等. 澳洲坚果种质资源的SRAP分析及指纹图谱构建[J]. 分子植物育种, 2020, 18(5): 1566-1577.
[29]Martins M, Tenreiro R, Oliveira M M. Genetic relatedness of Portuguese almond cultivars assessed by RAPD and ISSR markers[J]. Plant Cell Reports, 2003, 22(1): 71-78.
[3]刘来福. 作物数量性状的遗传距离及其测定[J]. 遗传学报, 1979, 6(3): 349-355.
责任编辑:谢龙莲
