高山松miR171a及其靶基因的鉴定与表达分析
2021-05-26张雪如王稳利邱宗波曾倩倩
张雪如 王稳利 邱宗波 曾倩倩

摘要:以裸子植物高山松为试验材料,利用笔者所在实验室前期构建的高山松miRNA数据,通过生物信息学方法筛选与高山松生长发育相关的miR171a,并通过基于RNA连接酶的cDNA末端快速扩增法(RLM-5′RACE)验证得出,GRAS家族转录因子(Unigene10015)和肌动蛋白结合蛋白(Unigene83401)的基因为高山松miR171a的靶基因。通过PCR技术克隆得到的高山松miR171a前体序列可形成茎环发夹结构,但其成熟序列的碱基保守性较差。系统进化分析显示,pde-miR171a与裸子植物火炬松的进化关系较近。经qRT-PCR分析发现,pde-miR171a在高山松茎中的相对表达量最高,其次是在针叶中,而靶基因Unigene10015在茎、针叶中的相对表达量较低,暗示pde-miR171a可以通过调控靶基因Unigene10015而参与高山松的生长发育。
关键词:高山松;miR171;靶基因;荧光定量PCR
中图分类号: S718.46文献标志码: A
文章编号:1002-1302(2021)07-0062-05
收稿日期:2020-08-05
基金项目:国家自然科学基金(编号:31500499);河南省高校科技创新人才项目(编号:16HASTIT019)。
作者简介:张雪如(1996—),女,河南洛阳人,硕士研究生,主要从事植物发育生物学方面的研究。E-mail:1365655297@qq.com。
通信作者:邱宗波,博士,教授,主要从事植物分子生物学方面的研究。E-mail:qiuzongbo@126.com。
microRNA(miRNA)是一类由21~24个核苷酸组成的内源性单链小RNA,主要是在转录后水平介导靶mRNA的降解或翻译抑制来调控基因表达[1]。自研究者从秀丽隐杆线虫(Caenorhabditis elegans)中发现第1个 miRNA以来,miRNA一直是研究的热点[2]。近年来,越来越多的研究发现,miRNAs在高山松(Pinus densata)的生长发育[3]、杉木种子的萌发[4]及落叶松体胚的生长发育和形态建成等方面起着重要的调控作用[5]。
miR171是在植物中最早发现的miRNAs家族成员之一[6],在拟南芥中有3个miRNA成员,分别是ath-miR171a、ath-miR171b和ath-miR171c[7]。通过基于RNA连接酶的cDNA末端快速扩增(RNA ligase-mediated 5′ rapid amplification of cDNA ends,简称RLM-5′RACE)试验,研究者发现水稻中的miR171能介导靶基因OsHAM(GRAS家族转录因子)mRNA的剪切降解,从而促进水稻营养生长向生殖生长的过渡及根尖分生组织稳态的形成[8]。杨树miR171通过调控GRAS转录因子而参与杨树的生长发育和光形态建成的调控[9]。张力等研究发现,烟草miR171c通过负调控SCL靶基因TC134811、TC127385,使植物出现顶端优势丧失和茎秆增多等表型[10]。尽管目前关于miR171在多个植物中功能的研究较多,但目前在裸子植物高山松中,miR171的鉴定及其在高山松生长发育过程中的功能尚不清楚。
高山松是一种具有重要生态意义的裸子植物[11]。本研究根据高山松小RNA高通量测序结果获得的miR171a,进行前体序列的克隆与分析。通过在线预测网站获得 miR171a的靶基因,并通过RLM-5′RACE进行验证。同时还分析了miR171a及其靶基因在高山松不同组织部位的表达特性,为揭示miR171a在高山松生长发育中的作用提供一定的理论依据。
1 材料与方法
1.1 试验材料
2月龄的高山松幼苗于2019年5月置于温室中培养(昼—夜生长温度周期为 25 ℃—18 ℃,光—暗周期为16 h—8 h,相对湿度为65%~70%)。
1.2 高山松miR171前体序列的克隆及其二级结构预测
使用植物Concert Plant RNA Reagent (美国Invitrogen公司)从2月龄的高山松幼苗中提取總RNA。从笔者所在实验室得到的高山松miRNA高通量测序数据中获得高山松miR171a的前体序列:5′-AAAGAAUGUGAUGUUGGCUAGGCUCAAUCGGAUUGUAACGCCCACGGAAUUUGGUCUUGUGAUCUGAUUGAGCCGUGCCAAUAUCACAUUCUAAC-3′,用Primer 5.0软件设计特异性引物进行PCR 扩增(表1)。采用Clustalx 2.0软件,以高梁sbi-miR171a、拟南芥ath-miR171a、油菜bna-miR171a、玉米 zma-miR171a、大豆gma-miR171a、火炬松pta-miR171a、毛白杨ppt-miR171a、水稻osa-miR171a、毛果杨ptc-miR171a及卷柏smo-miR171a的成熟序列为模板进行序列比对。用MEGA 7.0构建miR171a前体序列的系统进化树,通过RNAfold web server在线软件预测miR171a前体序列的二级结构。
1.3 高山松miR171a靶基因的预测与切割位点的验证
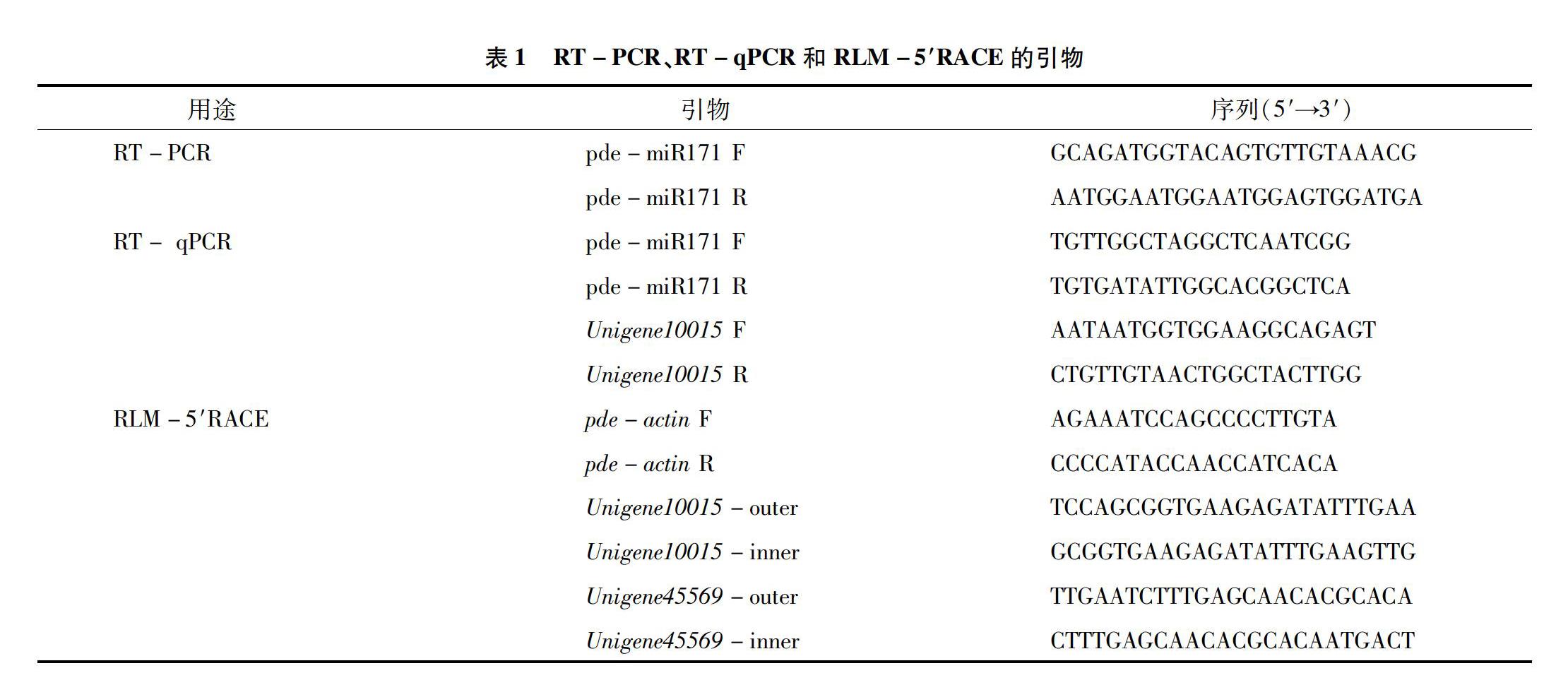
将高山松miR171a的成熟序列提交到靶基因在线预测网站psRNATarget(http://plantgrn.noble.org/psRNATarget/)上,以高山松的转录组数据库作为靶标,设置期望值为2.5,其余参数为默认值。将预测到的靶基因比对到GO(gene ontology)数据库中,确定其功能。依据靶基因的cDNA序列,用Primer 5.0软件设计用于RLM-5′RACE的引物(表1)。RLM-5′RACE试验参照孔雷等的方法[12],扩增出的特异产物后进行克隆测序。
1.4 实时荧光定量PCR
使用植物Concert Plant RNA Reagent (美国Invitrogen公司)分别提取2月龄高山松幼苗根、茎和针叶的总RNA。用Superscript Ⅱ reverse transcriptase (美国Invitrogen公司)进行反转录。荧光定量试验使用Thunderbird SYBR Green PCR Master Mix试剂盒(日本Toyobo公司)。以actin作为miR171a和靶基因Unigene10015的内参基因。利用Rotor-Gene 3000型实时PCR扩增仪检测,每个样品设3次生物学重复。基因相对表达量的测定采用2-ΔΔCT法(C表示循环数;T表示荧光阈值)[13],将基因在根中的表达水平设成1。
2 结果与分析
2.1 pde-miR171a前体序列的扩增及二级结构的预测
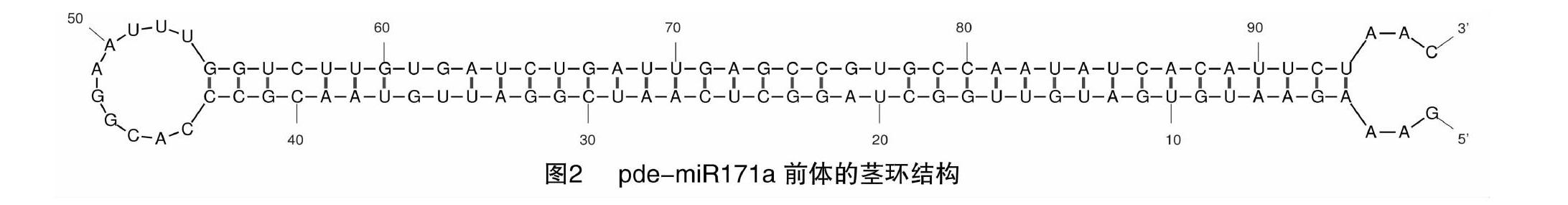
从笔者所在实验室前期得到的高山松miRNA高通量测序数据中筛选得到高山松miR171a的前体序列,进行引物设计,以高山松基因组DNA为模板进行PCR扩增,得到高山松miR171a的前体序列长度为96 bp(图1)。用RNAfolder软件在线分析其二级结构,发现高山松pre-miR171a可折叠成典型的茎环发夹结构(图 2),预测得出其二级结构的最小折叠自由能(minimal folding free energy,简称MFE)为-230.74 kJ/mol,最小折叠自由能指数(minimal folding free energy index,MFEI)为1.28,miR171a的成熟序列(5′-UGAUUGAGCCGUGCCA AUAUC-3′)位于茎环结构的3′端。
2.2 高山松miR171a成熟序列和前体序列的分析
对miRBase数据库中不同植物的miR171a成熟序列与高山松miR171a(pde-miR171a)的成熟序列进行比对,发现除了高梁sbi-miR171a、pde-miR171a的成熟序列完全一致外,其他物种的miR171a如拟南芥ath-miR171a、玉米zma-miR171a、火炬松pta-miR171a、水稻osa-miR171a、油菜bna-miR171a、大豆gma-miR171a、毛白杨ppt-miR171a、毛果杨ptc-miR171a及卷柏smo-miR171a与高山松pde-miR171a的成熟序列间有1个或多个碱基的差异(图3),表明高山松 pde-miR171a的成熟序列在不同物种中的保守性不高。用MEGA 7.0对毛白杨、玉米、卷柏、高梁、拟南芥、火炬松、大豆、水稻、油菜和高山松的MIR171a共25个成员构建系统发育树。结果(图4)显示,不同物种的MIR171前体保守性不高,高山松与火炬松的前体序列聚为一类,二者间的进化关系较近。
2.3 高山松miR171a靶基因的预测及RLM-5′RACE验证以高山松miR171成熟序列为对象、高山松转录组数据库为靶标,用psRNAtarget进行pde-miR171a靶基因预测。如表2所示,pde-miR171a的靶基因分别为GRAS家族转录因子(Unigene10015)、肌动蛋白结合蛋白(Unigene83401) 及未知蛋白 (Unigene84522), 其中pde-miR171a与高山松GRAS家族转录因子Unigene10015)的匹配程度最高。
为了进一步验证高山松miR171a对潜在靶基因是否存在剪切作用,笔者用RLM-5′RACE方法对靶基因Unigene10015、Unigene83401 mRNA的3′ 端剪切產物进行扩增。测序结果表明,高山松 pde-miR171a对GRAS家族转录因子的剪切位点位于第13个至第14个碱基之间,对肌动蛋白结合蛋白的剪切位点位于经典切割位点下游第20个碱基处(图5),表明GRAS家族转录因子(Unigene10015)和肌动蛋白结合蛋白(Unigene83401)的基因确实是miR171a的靶基因,miR171a可以在转录后水平调控GRAS家族转录因子和肌动蛋白结合蛋白基因的表达。
2.4 高山松pde-miR171a及其靶基因Unigene10015在不同组织中的表达分析
通过荧光定量PCR检测pde-miR171a及其靶基因Unigene10015在高山松根、 茎和针叶中的表达情况。结果(图6)显示,pde-miR171a、Unigene10015在高山松不同组织中的表达情况存在差异,pde-miR171a在茎中的相对表达量最高,其次在针叶中,而靶基因Unigene10015在茎、针叶中的相对表达量最低;靶基因Unigene10015的相对表达量与pde-miR171a的相对表达量呈负相关。
3 讨论与结论
随着高通量测序技术的快速发展,对裸子植物进行高通量转录组测序的研究不断增多,有助于通过生物信息学方法对miRNAs的鉴定提供数据基础[14-16]。本研究对鉴定到的 miR171a前体基因Pre-miR171a进行克隆, 得到miR71a的前体序列长度为96 bp,可形成稳定的茎环发夹结构,说明miR171a确实在高山松中存在并表达。最小折叠自由能指数(minimal folding free energy index,简称MFEI)是将miRNA与其他小分子RNA区分开来的重要参数,一般植物的MFEI大于0.85[17]。在本研究中, miR71a前体的MFEI为1.28, 显著高于黑胡椒miR171前体的MFEI(0.80)[6],但与拟南芥[18]、水稻[8]和烟草[10]中miRNA前体的MFEI类似。这些结果为进一步研究高山松miR171a的功能奠定了基础。
植物的成熟miRNA能通过核酸互补指导RNA诱导的沉默复合体(RISC)去切割或者抑制靶基因表达[19]。目前主要采用5′RACE(rapid amplification of cDNA ends)的方法验证miRNA对靶基因mRNA的切割作用[12]。笔者用RLM-5′RACE方法对预测的2个靶基因Unigene10015(GRAS家族转录因子)、Unigene83401(肌动蛋白结合蛋白)进行验证。结果显示,高山松miR171a介导靶基因Unigene10015(GRAS家族转录因子)的mRNA剪切降解,且剪切位点在第13、第14位碱基之间。通过5′RACE试验方法发现,拟南芥中GRAS家族转录因子Scarecrow-Like是miR171的靶基因,切割位点也在第13、第14位碱基之间[7]。另外,高山松miR171a与靶基因Unigene83401(肌动蛋白结合蛋白)的切割位点在典型的miRNA切割位点下游第20个碱基处,这可能是由于siRNA在miRNA切割位点下游第20个核苷酸处发生的第2次切割[20]。以上结果表明,miR71a能够通过识别、结合,然后切割相应的靶基因,从而调控高山松的生长发育。
参考文献:
[1]Bartel D P. MicroRNAs:genomics,biogenesis,mechanism,and function[J]. Cell,2004,116(2):281-297.
[2]Zhang B H,Pan X P,Cobb G P,et al. Plant microRNA:a small regulatory molecule with big impact[J]. Developmental Biology,2006,289(1):3-16.
[3]Wan L C,Zhang H Y,Lu S F,et al. Transcriptome-wide identification and characterization of miRNAs from Pinus densata[J]. BMC Genomics,2012,13:132-142.
[4]Cao D C,Xu H M,Zhao Y Y,et al. Transcriptome and degradome sequencing reveals dormancy mechanisms of Cunninghamia lanceolata seeds[J]. Plant Physiology,2016,172(4):2347-2362.
[5]Li W F,Zhang S G,Han S Y,et al. The post-transcriptional regulation of LaSCL6,by miR171 during maintenance of embryogenic potential in Larix kaempferi(Lamb.)Carr.[J]. Tree Genetics & Genomes,2014,10(1):223-229.
[6]Asha S,Nisha J,Soniya E V. In silico characterisation and phylogenetic analysis of two evolutionarily conserved miRNAs (miR166 and miR171) from black pepper (Piper nigrum L.)[J]. Plant Molecular Biology Reporter,2013,31:707-718.
[7]Llave C,Xie Z X,Kasschau K D,et al. Cleavage of scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA[J]. Science,2002,297(5589):2053-2056.
[8]Fan T,Li X M,Yang W,et al. Rice osa-miR171c mediates phase change from vegetative to reproductive development and shoot apical meristem maintenance by repressing four OsHAM transcription factors[J]. PLoS One,2015,10(5):e0125833.
[9]劉志祥,曾超珍,曾渭贤,等. 杨树MIR171基因家族进化与功能分化研究[J]. 植物遗传资源学报,2014,15(2):313-319.
[10]张 力,沙爱华. 烟草microRNA171c的功能分析[J]. 植物科学学报,2016,34(5):775-780.
[11]Wang B S,Mao J F,Gao J,et al. Colonization of the Tibetan plateau by the homoploid hybrid pine Pinus densata[J]. Molecular Ecology,2011,20(18):3796-3811.
[12]孔 雷,朱向向,王屹玮,等. 茶树miR164a及其靶基因的鉴定与表达分析[J]. 茶叶科学,2018,38(6):547-558.
[13]Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25(4):402-408.
[14]Qiu Z B,Wan L C,Chen T,et al. The regulation of cambial activity in Chinese fir (Cunninghamia lanceolata) involves extensive transcriptome remodelling[J]. New Phytologist,2013,199(3):708-719.
[15]Wang L,Zhao J,Luo K,et al. Deep sequencing discovery and profiling of conserved and novel miRNAs in the ovule of Ginkgo biloba[J]. Trees,2016,30(5):1557-1567.
[16]Wen C H,Lin S S,Chu F H. Transcriptome analysis of a subtropical deciduous tree:autumn leaf senescence gene expression profile of Formosan gum[J]. Plant & Cell Physiology,2015,56(1):163-174.
[17]Zhang B H,Pan X P,Cox S B,et al. Evidence that miRNAs are different from other RNAs[J]. Cellular and Molecular Life Sciences,2006,63(2):246-254.
[18]Wang L,Mai Y X,Zhang Y C,et al. 2010. MicroRNA171c-targeted SCL6-Ⅱ,SCL6-Ⅲ and SCL6-Ⅳ genes regulate shoot branching in Arabidopsis[J]. Molecular Plant,3(5):794-806.
[19]Carthew R W,Sontheimer E J. Origins and mechanisms of miRNAs and siRNAs[J]. Cell,2009,136(4):642-655.
[20]Ronemus M,Vaughn M W,Martienssen R A. MicroRNA-targeted and small interfering RNA-mediated mRNA degradation is regulated by argonaute,dicer,and RNA-dependent RNA polymerase in Arabidopsis[J]. Plant Cell,2006,18(7):1559-1574.
