江苏省植物细胞遗传学研究回顾与展望
2021-05-19王海燕龚志云蒋甲福周宝良娄群峰曹清河席梦利陈佩度顾铭洪张天真陈发棣陈劲枫李宗芸王秀娥
王海燕,龚志云,蒋甲福,周宝良,娄群峰,曹清河,席梦利,陈佩度,顾铭洪,张天真,陈发棣,陈劲枫,李宗芸,王秀娥
江苏省植物细胞遗传学研究回顾与展望
王海燕1,龚志云2,蒋甲福1,周宝良1,娄群峰1,曹清河3,席梦利4,陈佩度1,顾铭洪2,张天真6,陈发棣1,陈劲枫1,李宗芸5,王秀娥1
1. 南京农业大学农学院,作物遗传与种质创新国家重点实验室/JCIC-MCP,南京 210095 2. 扬州大学农学院,江苏省作物遗传生理重点实验室/植物功能基因组学教育部重点实验室/江苏省作物基因组学和分子育种重点实验室,扬州 225009 3. 江苏徐淮地区徐州农业科学研究所,徐州 221131 4. 南京林业大学林学院,南京 210037 5. 江苏师范大学生命科学学院,徐州 221116 6. 浙江大学农业与生物技术学院,杭州 310058
20世纪初“遗传的染色体学说”的提出和证明标志着细胞遗传学交叉学科建立,伴随相关学科的发展,20世纪60年代末期细胞遗传学又与分子遗传学相结合,建立发展了分子细胞遗传学交叉学科。分子细胞遗传学以DNA分子原位杂交技术为核心,不断拓展应用领域,为生命科学研究提供了直观、高效的技术手段。原位杂交技术与基因组、细胞生物学等技术结合,被广泛应用于人类、动物、植物的起源、进化、驯化等基础研究和远缘杂交、染色体工程等应用研究。通过形象地展示DNA、RNA、蛋白质在细胞中的实际位置,揭示DNA序列之间的实际位置和顺序、亲缘物种间的进化关系和结构重排、基因组拼接序列的质量、转录水平RNA和翻译水平蛋白质的位置和数量变化等。江苏省遗传学会会员单位南京农业大学、扬州大学、南京林业大学、江苏师范大学、徐淮地区农科院等自20世纪中期开展细胞遗传学理论技术研究,伴随学科发展不断创新,建立了较完善的分子细胞遗传技术体系,并成功应用于开展植物系统进化、远缘杂交、染色体工程、基因组学等研究,取得了一批研究成果。本文将主要综述江苏省在该领域取得的重要进展,并展望未来发展方向。
江苏省遗传学会;分子细胞遗传学;DNA分子原位杂交;染色体工程;基因组学
20世纪初期,美国遗传学家Sutton和德国生物学家Boveri提出“遗传的染色体学说”,该学说的证明标志着细胞遗传学的诞生和成熟。以美国康奈尔大学为核心的科学家利用玉米作为研究对象,阐明了植物性状遗传变异的细胞学基础,建立和完善了植物细胞遗传的理论和技术体系。日本的Kihara教授和美国的Sears教授以异源六倍体小麦(L.)及其祖先种为研究对象,建立了麦类植物细胞遗传学技术,并将其应用于异源多倍体小麦的基因组分析、非整倍体创制和鉴定、小麦远缘杂交等研究,阐明了小麦3个亚基因组的起源、麦类植物不同亚基因组染色体间的部分同源关系。此后,研究者们在很多植物物种中开展了细胞遗传研究,建立了染色体核型分析、染色体构型分析和染色体分带等经典细胞遗传学核心技术,为物种的起源、系统分类、以及植物遗传改良等提供了基础的细胞学信息。伴随遗传物质的发现、DNA双螺旋结构解析、探针标记技术和显微技术等不断取得突破,分子遗传学理论和技术快速发展,并向细胞遗传学不断渗透,20世纪60年代末期美国耶鲁大学Gall等科学家在动物中建立的DNA分子原位杂交(hybridization, ISH)技术,成为分子细胞遗传学这一分支学科建立的里程碑。该技术很快被用于植物细胞遗传学研究,并得到快速发展和广泛应用,为植物遗传育种、基因组学和分子生物学研究提供了直观有效的研究手段。ISH伴随相关学科的发展不断发展,杂交利用的探针分子可以是DNA、RNA和蛋白抗体;DNA探针包括基因组DNA、重复序列DNA、BAC等人工染色体克隆、单拷贝基因DNA、寡核苷酸探针库等;探针标记物包括放射性分子、半抗原、荧光素等;杂交的对象包括有丝分裂中期染色体、减数分裂粗线期或中期I染色体、间期核DNA纤维。由此建立了genomichybridization (GISH)、multi-color fluorescencehybridization (FISH)、BAC-FISH、fibre-FISH、oligo-FISH、oligo-painting、immuno-staining、seqFISH、rmFISH等新技术,通过形象地展示DNA、RNA、蛋白质在细胞中的实际位置,揭示DNA序列之间的实际位置和顺序、亲缘物种间的进化关系和结构重排、基因组拼接序列的质量、转录水平RNA和翻译水平蛋白质的位置和数量变化等。
江苏省植物细胞遗传学研究起步较早,覆盖面广,应用成效显著。围绕水稻()、小麦(、棉花()等主要作物以及瓜属()、甘薯()、菊属()、杨属()园艺和林木植物,建立了完善的细胞学和分子细胞遗传学研究体系,成功应用于遗传改良、基因组解析和比较基因组、物种起源进化、染色体生物学等研究领域,取得了多项基础和应用研究成果。在江苏遗传40周年之际,本文将回顾江苏省遗传学会会员单位围绕主要粮食作物、园艺作物、林木等物种,在植物细胞遗传学领域的重要研究进展,并展望未来的发展方向。
1 小麦分子细胞遗传学及其应用
普通小麦(2n=6x=42,基因组AABBDD)是异源六倍体。小麦及其近缘物种染色体大,容易观察其形态结构,因此成为早期植物细胞遗传学研究的优良材料。Kihara教授建立基于远缘杂交和细胞遗传学技术的染色体组分析理论,阐明了栽培小麦A、B和D三个亚基因组以及其他小麦族物种的二倍体、四倍体祖先供体。Sears教授是小麦细胞遗传学和染色体工程的先驱,20世纪30年代开始历时40余年,先后培育出小麦单体、三体和缺体–四体及端体等非整倍体系列,将小麦3个染色体组的染色体根据其补偿关系划归到7个部分同源群。Gill教授实验室建立了可识别全部21对小麦染色体的分带技术 (1991)[1];Rayburn和Gill[2~4]最早利用生物素标记的重复序列探针进行ISH,识别小麦染色体;Mukai等[5]建立基于重复序列的双色ISH技术,可识别17对小麦染色体;Zhang等[6]建立以乌拉尔图小麦A组和粗山羊草D组为探针、拟斯卑尔脱山羊草S组为封阻的GISH技术,可以区分普通小麦A、B、D3个亚基因组。近年来,基因组测序技术的快速发展不仅为分子生物学研究提供了丰富的信息,同时也加快了植物分子细胞遗传学的研究,尤其是用于小麦FISH的探针不断发展,出现了寡核苷酸FISH、单拷贝基因FISH和基于单拷贝基因的寡核苷酸探针库。此外,在小麦中还建立了基于流式细胞仪的染色体分拣、基于显微切割的特定染色体区段切割分离等分子细胞遗传学技术,用于结构和功能基因组研究[7~10]。建立的黑麦、大麦等的分子细胞遗传学技术体系,为小麦与近缘物种的远缘杂交、染色体工程和比较基因组研究提供了有效的技术手段。下面简要介绍南京农业大学在小麦细胞遗传学领域的研究进展。
1.1 小麦及近缘物种染色体鉴定技术
染色体身份和染色体结构的准确快速鉴定是开展染色体工程、基因组分析等研究的重要基础。早在20世纪80年代初期,江苏省遗传学会学会前理事长、南京农业大学细胞遗传研究所刘大钧院士带领团队,与Gill教授合作,开展小麦及其近缘物种染色体鉴定技术研究。先后在六倍体普通小麦、四倍体硬粒小麦、簇毛麦、大赖草、鹅观草属等物种中建立了染色体分带技术[11~17];染色体分带与染色体配对的构型分析结合,纠正了小麦4A和4B的身份[11],初步分析我国特有半野生小麦的染色体组成,用于鉴定创制的硬粒小麦–簇毛麦双二倍体等远缘种质[18,19]。20世纪90年代开始分子遗传学研究。建立了以生物素标记基因组DNA为探针的体细胞和花粉母细胞染色体GISH技术,明确了小麦–簇毛麦易位系中小麦和外源染色体的组成[20];从外源物种中克隆基因组特异重复序列,利用在簇毛麦中克隆的特异重复序列进行ISH,特异识别小麦背景中的簇毛麦染色体[21];建立了基于荧光素标记探针的GISH技术,用于鉴定导入小麦背景中的鹅观草属、赖草属、黑麦、偃麦草等物种的染色体或染色体区段[22~29],实验室研究全面进入分子细胞遗传学阶段。
重复序列探针的开发和荧光素标记技术的发展,为基于重复序列和双色/多色FISH的核型分析提供了更快速精确的技术手段。Zhang等[6]利用pSc119.2等4个质粒探针构建了簇毛麦品系91C43的FISH核型,并用于鉴定小麦–簇毛麦异附加系、代换系和易位系中外源染色体的具体身份;为提高FISH效率,Du等[30]开发了基于重复序列的寡核苷酸探针库,并用于鉴定小麦品种、黑麦、偃麦草等染色体结构变异;王艳芝[31]开发了一批寡核苷酸探针用于替代原先的质粒探针,通过ND-FISH或FISH鉴定小麦染色体组成;进一步优化小麦、百萨偃麦草、黑麦等寡核苷酸探针套,建立栽培一粒小麦、硬粒小麦、荆州黑麦、长穗偃麦草等物种的标准核型,可快速鉴定小麦背景中的外源染色体,并构建建国以来我国373个大面积栽培品种及骨干亲本高清核型,揭示其中存在的自发结构变异[30,32~34]。Guo等[35]利用寡核苷酸探针FISH分析了异花授粉黑麦品种染色体演化特征,并细胞学定位荆州黑麦毛颈基因区段。Sun等[36]利用基于簇毛麦基因组序列新开发的寡核苷酸探针,结合利用4个已报道寡核苷酸探针,构建了簇毛麦不同品系的FISH核型,揭示了不同品系间的核型多态性。为解析外源染色体基因组序列,开发更多近缘物种特异重复序列探针和特异分子标记,克隆外源优异基因,与捷克Dolezel教授实验室合作,利用流式细胞仪分拣了簇毛麦6VS和4VS染色体,Xiao等[37]在解析4VS序列基础上,通过生物信息学鉴定出两条反转录转座子重复序列,经FISH分析发现均特异弥散分布于簇毛麦所有染色体上,杂交信号与GISH信号相似;Lei等[38]利用6VS染色体分拣测序获得的基因组序列信息,开发了基于重复序列的7个寡聚核苷酸探针,将其中两个探针相结合,进一步丰富了簇毛麦的FISH核型。
近年来,基因组序列的发布和生物信息分析技术的发展,基于单拷贝序列的染色体或染色体区段特异探针库开发技术得到快速发展,Song等[39]首次报道了小麦4D染色体特异的寡核苷酸探针库,并实现在小麦或染色体工程材料中小麦染色体身份的快速鉴定(图1)。分子细胞遗传技术在外源基因克隆中也发挥了重要作用。Chen等[40]、Yang等[41],Cao等[42]和Xing等[43]利用小片段易位系创制、TAC- FISH等分子细胞遗传学手段,结合染色体分拣、基因芯片、MutRenSeq和NLR富集等技术,将簇毛麦6VS上抗小麦白粉病基因定位于6VS FL0.45- 0.58区段,利用TAC-FISH将定位于特定6VS物理区段,克隆了抗白粉病基因和并验证其功能。
1.2 小麦–远缘新种质创制与利用
小麦族()有300多个物种,小麦近缘植物中含有丰富的抗病、抗逆和抗虫等基因,通过远缘杂交将近缘物种中的优良基因导入普通小麦,是拓宽小麦遗传基础、推进小麦育种取得突破的有效途径。早在1937年,Sears利用远缘杂交把小伞山羊草的抗病基因导入小麦。我国是较早开展远缘杂交的国家之一,且成效显著。早在20世纪中期,鲍文奎先生开展八倍体小黑麦研究,育成了多个小黑麦品种[44];李振声先生在西北植物所和中国科学院遗传与发育生物学研究所期间,带领团队开展小麦–偃麦草远缘杂交研究,育成小偃系列品种大面积推广,获得国家最高科学奖[45];东北师范大学郝水先生、哈尔滨师范大学李集临先生、山西农业大学孙善澄先生等先后开展小麦与冰草、黑麦等物种的远缘杂交研究[46~48]。
自20世纪70年代起,南京农业大学细胞遗传研究所在刘大钧院士的带领下,开始从小麦近缘植物中筛选、发掘可用于小麦抗病育种的基因资源。先后开展小麦与大麦、黑麦草、簇毛麦、大赖草、鹅观草、纤毛鹅观草等物种的远缘杂交研究,构建和完善小麦–远缘种质创制、鉴定、利用的理论和技术体系,并创制了涉及10个外源物种的小麦远缘新种质。

图1 利用染色体涂染技术鉴定小麦–簇毛麦易位系
A1, B1:利用4D染色体特异寡核苷酸探针库FISH鉴定T4VS-4DS·4DL(A1)和T4DS-4VS·4DL(B1)。蓝色:DAPI套染;红色:TAMRA修饰的4D染色体特异探针库。放大图像:4D探针库涂染的易位染色体。A2, B2:利用GISH鉴定T4VS-4DS·4DL(A2)和T4DS-4VS·4DL(B2)。蓝色:DAPI套染;绿色:Fluorescein-12-UTP标记的簇毛麦全基因组探针。放大图像显示簇毛麦全基因组探针在易位染色体上的杂交信号。A3:A1和A2合成图;B3:B1和B2合成图。标尺=2 μm。
最早发现原产于地中海地区的二倍体物种簇毛麦高抗小麦白粉病,获得普通小麦、硬粒小麦与簇毛麦远缘杂种、硬簇麦双二倍体[49];建立染色体分带[12]、ISH[50]、分子标记等簇毛麦染色体鉴定技术[51~57],在培育出双二倍体、异附加系、异代换系和整臂易位系等远缘种质基础上,将抗白粉病位点定位于6VS染色体[58]。以上研究分别于1982年、1986年和1996年获农业部科技进步三等奖、三等奖、二等奖,1997年获国家技术发明三等奖。20世纪80年代中期,又发现鹅观草、纤毛鹅观草和大赖草高抗小麦赤霉病,并通过远缘杂交创制出一批有价值的新种质,为小麦抗赤霉病育种提供了新抗源。“抗小麦赤霉病新种质的搜集、鉴定、转移和利用”曾经获得麦氏基金作物合作研究项目连续三期(1995~2004年)资助。20世纪90年代初,在已经建立的传统细胞遗传学技术基础上,进一步完善了外源染色体精确鉴定的分子细胞遗传学新体系,成果获1998年教育部(基础类)科技进步一等奖。除对簇毛麦、大赖草、鹅观草和纤毛鹅观草进行研究外,还对小麦的其他近缘属约20多个物种进行过研究:如大麦属()、黑麦属()、山羊草属()、偃麦草属()、披碱草属()和旱麦草属()等。
自20世纪中期,创建小麦–近缘物种易位系高效创制和鉴定技术体系,先后利用辐射、杀配子染色体诱导染色体结构变异,结合利用中国春/Ph诱导部分同源染色体配对,创制了涉及簇毛麦、大赖草、黑麦、偃麦草、鹅观草、野大麦等物种的异易位系,建立的双二倍体的花粉辐射创制易位系库和整臂易位系成熟雌配子辐射技术体系,提高了诱导频率[59,60],结合高效分子细胞遗传学鉴定技术,创制了涉及簇毛麦、纤毛鹅观草、大赖草、黑麦和百萨偃麦草不同染色体的结构变异体库,为小麦育种提供了抗病、优质、抗逆等新资源。先后选育出携带抗秆锈病小种Ug99基因的T6AS/ 6VL易位系[61];携带抗菲利普孢囊线虫病基因的6V异附加系和小麦–簇毛麦易位系和缺失系[62];携带具有生育期和组织特异性的抗白粉病基因的5V染色体易位系[63];创制涉及簇毛麦4VS染色体的整臂易位系和易位系库,将4VS上抗黄花叶病基因定位于781 kb的物理区间[64,65]。小麦–簇毛麦T6VS/6AL易位系携基因,对所有白粉菌生理小种免疫,且1B染色体上携带高抗条锈病基因,易位染色体对主要农艺性状和品质性状无不利影响,对每穗粒数和千粒重还有正向效应。以T6VS/6AL易位系为亲本,国内育种单位已育成了约40个小麦新品种,在黄淮、西南、西北和长江中下游麦区大面积推广,在小麦白粉病抗源更替中发挥了重要作用。“小麦–簇毛麦远缘新种质创制及应用”先后获得2011年教育部技术发明奖一等奖和2012年国家技术发明奖二等奖。
2 水稻分子细胞遗传学及其应用
水稻(2n=2x=24)是重要的粮食作物,基因组大小389 Mb[66],是禾本科作物中基因组最小的作物。水稻是最早完成基因组测序的作物,因此也成为作物学研究的模式物种。基因组大小与细胞核内的染色体大小一般呈正相关的关系,水稻染色体在禾本科作物中也属于最小的类型之一,因此水稻细胞遗传学相较于玉米、小麦起步较晚。
2.1 水稻染色体鉴定技术
由于水稻有丝分裂前中期染色体很小,不同染色体的大小十分接近,因此仅仅依靠形态特征很难完全准确辨别出水稻体细胞中的所有染色体。因此,在20世纪90年代以前,有关水稻核型中的染色体相对长度、臂比、核仁染色体等数据,不同研究者的结果不尽相同。后来研究者建立了基于减数分裂粗线期染色体的细胞学分析技术[67],解决了体细胞染色体过短的问题。但传统的粗线期染色体制片技术难度较大,需要发展更简单有效的方法准确识别不同染色体,推进水稻的细胞遗传学研究。
FISH技术为准确识别水稻不同染色体提供了新途径。Cheng等[68]利用不同染色体臂上的单拷贝RFLP 标记序列筛选BAC文库,以筛选获得的阳性BAC克隆为探针进行BAC-FISH,发展出了全套24个水稻染色体臂特异的分子细胞学标记,可以识别和鉴别特定染色体。但这套探针在野生稻其他染色体组中很难显示出较强的杂交信号,限制了其使用范围。Tang等[69]根据水稻全基因组序列上基因分布的不均匀性,重新发展了一套24个富含基因序列的染色体臂特异BAC克隆,在水稻AA、BB、CC组染色体上都会显现明显的杂交信号,具有更高的应用价值。
全套染色体臂特异分子细胞学标记无法一次性在同一个细胞中识别所有不同染色体,FISH技术影响BAC信号的强弱,不同实验室的杂交信号可能强弱不一。近年来发展的oligo-FISH能够更好的保证探针的特异性与重复性。Liu等[70]根据水稻的参考基因组序列,利用生物信息学技术,开发了可以标记成2种颜色的两套寡核苷酸探针,利用这双色寡核苷酸探针,可以在水稻的12对染色体上产生26个不同的信号,这些信号可以作为“条形码”来唯一标记水稻的12对染色体,可以在同一个细胞中把不同12对染色体全部都识别出来。该套探针可以在野生稻AA、BB或CC组的染色体产生与栽培稻相似的特异性强信号;在EE组野生稻的染色体上产生的特异性信号较弱,而FF组没有产生信号。该套双色寡核苷酸探针在研究稻属染色体变异和进化中有重要应用价值(图2A)。
2.2 水稻染色体生物学
染色体鉴定技术和水稻基因组学的发展推进了水稻染色体结构元件的研究进展。端粒是染色体结构的基本元件之一,保护在复制过程中染色体末端的完整性,染色体末端还有一类特殊的亚端粒DNA序列。Os48和TrsA是水稻中的两种亚端粒DNA类型,其中Os48是栽培稻中第一个被分离出来的串联重复序列,每个重复单元长度为355 bp,其在同一物种的不同染色体上分布不同,在籼稻与粳稻染色体中的分布存在显著差异,籼稻拥有更多的拷贝数和位点数[71,72];TrsA是AA基因组水稻所特有的,重复单元同样为355 bp的串联重复序列[73],TrsA在不同水稻品种间的染色体上分布位置和数目有较大差别。Os48和TrsA分布特征表明,亚端粒区域在稻属进化上具有高度活跃的特点。Fan等[74]发现水稻亚端粒区存在高频率基因重组和转座子插入事件,认为其可能是新基因形成的“温床”。因此从某种意义上来说,在保证物种核心基因组稳定的前提下,亚端粒序列的变化可能会促进物种的快速进化。

图2 水稻和黄瓜粗线期染色体双色oligo-FISH
A:水稻粗线期染色体双色oligo-FISH。红色:Digoxin标记的oligos 探针信号;绿色:FAM标记的oligos探针信号。染色体由DAPI复染;B:黄瓜粗线期4号染色体8个连续区段的oligo-FISH。红色:地高辛标记的分别为来源于物理位置0~3 Mb、6~9 Mb、10.5~13.5 Mb和16.5~19.5 Mb区域的oligo探针;绿色:生物素标记的分别为来源于物理位置3~6 Mb、9~10.5 Mb、13.5~16.5 Mb和19.5~23.3 Mb区域的oligo探针。
着丝粒是保证染色体正常运动的重要组件,不同物种着丝粒DNA序列在长短和序列组成上存在很大差别[75]。亚洲栽培稻最主要的功能着丝粒由串联重复序列组成,重复单元为155 bp的CentO[76],CentO在12条非同源染色体上的拷贝数上有很大差别,从65 kb到2 Mb不等[77]。稻属其他物种AA、BB和EE染色体组均拥有相似的串联重复序列CentO,但重复单元在不同染色体组之间可能会有一些序列的变化。而CC和FF染色体组有其特有的着丝粒特异串联重复序列,但GG染色体组并不含有着丝粒串联重复序列[78]。
CRR是插入到CentO串联重复序列中或者分布其两侧的着丝粒特异逆转座序列,其在着丝粒区域高度富集,并且与CentO相间存在。在非同源染色体上CRR的分布也不相同,个别染色体上CRR主要分布在CentO的两侧,而且两侧数量差异较大[77]。除FF染色体组外,稻属不同染色体组之间均存在同源性较高的CRR。FF染色体组含有其特异的与CRR同源性很低的逆转座子[79]。缺乏着丝粒特异串联重复序列的GG染色体组,着丝粒序列主要由型逆转座子组成,与玉米着丝粒特异逆转座序列CRM同源性很高[78]。
与不同物种中着丝粒DNA的快速进化相反,着丝粒区域另外一个重要组成成份着丝粒特异蛋白CENH3在真核生物中相对保守。在水稻双着丝粒染色体中,只有一个着丝粒有CENH3的荧光信号,而失活的着丝粒则没有CENH3荧光信号[80]。Gong等[81]报道了一例在水稻8号染色体着丝粒DNA序列部分缺失的变异,这种缺失导致CENH3结合区域减少,从而导致着丝粒功能不稳定,说明着丝粒的稳定性与CENH3结合范围相关。另外,含有CENH3核小体的总量与基因组大小有必然联系[82],但是着丝粒大小却与染色体长度无关[81]。所以在稻属不同亚种间,CENH3结合总量因基因组的大小不同存在亚种间差异,但是在同一亚种内不同染色体上的着丝粒区域,CENH3的结合区域是基本相同且不变的,与染色体长度没有相关性。
着丝粒功能行使是一个与着丝粒的特殊序列而又不完全决定于该特殊序列的表观遗传调控过程。DNA甲基化是表观遗传调控的重要方式之一。对水稻着丝粒区域DNA的5-甲基胞嘧啶(5-methylcytosine, 5mC)甲基化水平检测发现,部分染色体着丝粒CENH3结合区要比其侧翼H3区域DNA甲基化水平更高,表明水稻中的着丝粒普遍存在着DNA甲基化现象[83]。组蛋白修饰是着丝粒表观调控另一种方式。通用DNA芯片分析技术对水稻着丝粒组蛋白修饰研究发现,4、5、7和8号染色体着丝粒核心区域CENH3呈不连续分布,与H3相间排列。对四种活性组蛋白修饰H3K4me2、H3K4me3、H3K36me3和H3K4K9ac进行ChIP分析,发现H3结合区域内有转录活性的DNA序列均存在这四种组蛋白修饰,而相应的CENH3结合域内没有[84]。暗示通过这些活性组蛋白修饰形成一个标识,防止CENH3定位在H3结合区域内,来维持活性着丝粒的功能。
2.3 稻属远缘新种质创制
野生稻的不同染色体组中带有许多栽培稻所不具备的优良基因,如对褐飞虱等病虫的抗性基因,把这些基因导入到栽培稻中,实现稻属不同染色体组间的基因转移,对水稻育种有重要意义[85,86]。Huang等[87]利用这一技术路线,从药用野生稻染色体附加系的后代中,选育出了抗褐飞虱的栽培稻新品系,实现了外源抗虫基因从野生稻向栽培水稻的转移和利用。颜辉煌等[88]将栽培稻与带有褐飞虱抗性基因的CC组紧穗野生稻杂交与回交,获得了带有部分C组染色体片段的导入系,为抗虫基因的种间转移和利用提供了新材料。
3 甜瓜属分子细胞遗传学及其应用
甜瓜属是葫芦科重要的属,包含52个物种,其中包括两个重要的经济作物,即从大约100亿年以前的一个相同祖先分化而来的黄瓜(L., 2n = 14, 367 Mb)和甜瓜(L., 2n = 24, 450 Mb)[89]。甜瓜属物种染色体相对较小,细胞遗传研究起步较晚。甜瓜属包括许多重要的野生种,具有栽培黄瓜和甜瓜所需的多种优良性状,开展细胞遗传研究,阐明物种间的进化关系,对于挖掘和利用野生种的优良性状基因,推进甜瓜属作物品种改良具有重要意义。
3.1 甜瓜属染色体鉴定技术
甜瓜属中黄瓜细胞遗传学研究相对较多。黄瓜中期染色体较小,传统细胞学技术难以准确描述和识别黄瓜染色体,染色体分带技术在黄瓜染色体鉴定中起到了不可替代的作用,其中以C带在黄瓜中应用最为广泛。Chen等[90]利用改良的染色体制片技术和C带技术建立了黄瓜的有丝分裂中期染色体核型,但黄瓜中期染色体较短,C带分辨率不高,难以准确识别黄瓜每条染色体。钱春桃等[91]采用放线菌酮预处理活体种子根改进制片技术,获得了清晰的黄瓜前中期染色体C带带型。
FISH技术的应用推进了甜瓜属细胞遗传学发展。Zhang等[92]利用五种甜瓜属物种自身基因组DNA为探针在各自中期染色体上进行GISH,揭示重复序列DNA的分布模式;利用其他物种基因组DNA为探针对栽培黄瓜和酸黄瓜进行GISH,发现栽培黄瓜和酸黄瓜具有很高的同源性。Wang等[93]利用酸黄瓜基因组DNA和12条染色体上特异的Fosmid克隆为探针进行双色FISH,可识别出酸黄瓜的每一条染色体。Chen等[94]利用FISH对研究甜瓜属物种18S-5.8S-26S rRNA编码基因的染色体定位,发现黄瓜具有4对的rDNA基因座,定位于1、2、4和7号染色体;甜瓜和酸黄瓜上分别检测到2对和3对rDNA基因座。Zhang等[95]利用FISH研究了20份黄瓜栽培种和野生种的45S和5S rDNA的组成,发现不同材料45S rDNA位点的数量从一个到五个不等,且大部分位于染色体末端,除了sp、spp和具有两对5S rDNA位点外,其他材料中只检测到一对5S rDNA位点。
Lou等[96]首次在甜瓜属物种中开发应用单拷贝基因染色体涂染技术,利用特异性的单拷贝探针成功构建了黄瓜核型。Li等[97]利用rDNA和来自黄瓜的14个单拷贝基因为探针在甜瓜属物种上进行FISH,可以分辨出的每一条染色体。Bi等[98]基于合成寡核苷酸文库的多重聚合酶链式反应开发了一种灵活的染色体涂染技术,通过巧妙的接头设计,可从一个寡核苷酸文库中获得多种不同探针,用于染色体涂染。Zhao等[99]基于oligo-FISH实现了黄瓜全部染色体快速涂染和染色体追踪;设计并合成了黄瓜5和7号染色体的oligo探针库,可鉴别跨物种间部分同源染色体、追踪种间杂交和异源多倍体化后减数分裂单个染色体配对行为(图2B)。
3.2 甜瓜属物种起源与进化
甜瓜属野生资源丰富,包含约52种野生物种,阐明其起源及进化关系对于野生物种优异基因发掘和利用具有重要意义。随着近年来探针的不断地发展和技术体系的不断完善,FISH在越来越多的甜瓜属物种进化研究中得到应用。
Zhang等[95]利用FISH技术研究20个甜瓜属物种的45S和5S rDNA的位点分布,发现5S rDNA位点变异明显大于45S rDNA,二者具有不同的进化模式,5S rDNA有从中间位置向端部位置进化的趋势,但保持了保守的位点数量,而45S rDNA则显示出位点数量增加的趋势,但位置相对保守;Yang等[100]利用5种不同地理来源和染色体数目的四个甜瓜属物种(,,,和)进行重复序列FISH,发现黄瓜、甜瓜和酸黄瓜的亲缘关系较近,和的亲缘关系较近,而与其他三个物种亲缘关系较远。这些研究有助于更全面地认识该属的基因组进化和系统发育关系。
利用黄瓜分子标记筛选出的黄瓜Fosmid克隆做探针进行FISH,比较其在黄瓜与甜瓜染色体的定位,发现黄瓜与甜瓜的着丝粒发生了重新定位[101]。Yang等[102]根据构建的黄瓜高密度遗传图谱和组装的黄瓜基因组草图,筛选出特异的Fosmid克隆为探针进行黄瓜和其野生变种var. hardwickii的粗线期染色体比较涂染,研究其遗传分化,发现两个种群之间在异染色质的数量和分布以及染色体重排方面存在显著差异,研究结果支持这两个黄瓜种群的亚种地位,并表明var. hardwickii是栽培黄瓜的祖先。
3.3 甜瓜属物种种质创新
起源于我国云南的酸黄瓜(Chakr., 2n=2x=24)是黄瓜的近缘野生物种,具有抗线虫、霜霉病、蔓枯病、耐弱光、耐低温等优异特性[103,104]。通过种间杂交将酸黄瓜优良性状转移到栽培黄瓜中是黄瓜品种改良的有效途径。Chen等[105]通过胚拯救获得黄瓜与其近缘野生种酸黄瓜之间种间杂种F1,利用体细胞无性系变异方法诱导杂种加倍,创制了产生可育花粉并形成含种子的果实的异源四倍体,将其命名为Chen & Kirkbride (HHCC, 2n=4x=38)。具有与亲本相似的性状,也有其独特性状,种子形态介于两亲本之间,果实营养价值更高,对一些病虫害具有较强的抗性[104]。Chen等[106]进一步将异源四倍体()与普通二倍体黄瓜()杂交,通过胚胎培养获得异源三倍体。Chen等[107,108]利用异源四倍体与普通二倍体黄瓜连续回交,结合胚胎拯救首次获得两株黄瓜单体异附加系。Li等[109]利用异源四倍体与普通二倍体黄瓜连续回交,结合胚胎拯救获得多份异附加系,并利用oligo-FISH技术结合酸黄瓜染色体特异序列标记,确定了异附加系中外源染色体的组成。
4 棉花分子细胞遗传学及其应用
棉花(, 2n=4x=AADD=52)是重要的纤维作物,也是重要的植物油脂和植物蛋白质来源。目前世界上广为栽培的棉花为异源四倍体种,为纺织工业提供了90%以上的棉纤维。棉属有52个种,包括45个二倍体种,根据亲缘关系分成A-G和K等八个基因组(其中2个为A组栽培种)以及七个由AD基因组组成的异源四倍体种(含两个为栽培种),是改良栽培棉花的重要基因资源,通过远缘杂交转移和利用外源基因对于棉花改良有重要意义。
4.1 棉花染色体鉴定技术
棉花染色体数量多,形态相似且较短小,未建立染色体分带等核型分析技术,因而难以准确识别特定染色体。GISH技术提供了有效鉴定手段。以野生种斯特提棉(Willis)为探针,在陆地棉(L)标准系TM-1体细胞有丝分裂中期染色体上进行GISH,可以鉴定出栽培棉中的外源染色体[110]。以A1基因组的阿非利加草棉(L var.)和C1基因组的斯特提棉两个棉种的DNA同时为探针进行GISH,可以清晰地区分At、Dt和C1基因组染色体,且重复性好,这一棉花多色GISH技术体系成功应用于种间杂种的鉴定[110]。对引自澳大利亚CSIRO的陆地棉与澳洲棉(F.v. M.) (G2基因组)六倍体杂种(基因组组成为AtAtDtDtG2G2)进行的多色GISH,可清晰地区分At、Dt和G2基因组染色体[110]。
Wang 等[111~113]开发了基于棉花连锁群(染色体)的特异BAC克隆,获得第一套多倍体植物四倍体棉染色体特异的BAC克隆,利用BAC-FISH杂交信号可作为染色体特异的细胞学标记,准确地识别出棉花26条染色体。Wang 等[113]利用棉花高密度遗传图谱的SSR分子标记筛选出了20个BAC克隆,与 45S 和5S rDNA相结合,构成了由22个探针混合形成的鸡尾酒式文库,以亚洲棉江陵中棉有丝分裂中期染色体为靶标,一次FISH产生的信号可以同时识别亚洲棉的13对染色体,且与陆地棉A染色体亚组的部分同源染色体一一对应,获得了亚洲棉稳定可靠的标准核型,实现了亚洲棉染色体的准确识别。
4.2 棉花染色体生物学
Wang等[114]和Paterson等[115]各自完成了四倍体棉的D基因组供体种雷蒙德氏棉的基因组测序,Li等[116]完成了四倍体棉的A基因组供体近缘种亚洲棉的基因组测序,Zhang等[117]和Li等[118]分别完成了四倍体栽培种陆地棉的基因组测序,为开展棉花染色体结构元件研究提供了基础。着丝粒区域含有大量的重复序列,对其进行精确定位、克隆、测序难度很大。曹玉洁[119]通过筛选二倍体A组亚洲棉着丝粒反转录转座子,得到了22个候选克隆,利用FISH筛选出两个位于染色体着丝粒区的克隆,测序鉴定出三个着丝粒区特异的反转录转座子元件。基于A组棉种的着丝粒特异引物和D组雷蒙德氏棉全基因组序列信息,通过PCR和FISH验证,获得了三个雷蒙德氏棉着丝粒区特异的串联重复序列。Zhang等[120]鉴定出一个棉花着丝粒特异BAC克隆,经测序和FISH验证,鉴定出棉花着丝粒相关的四个LTR反转录转座子,统称为GhCRs。Fiber-FISH分析显示,GhCRs均位于陆地棉的所有26条粗线期染色体着丝粒异染色质区,呈成簇相间分布在1.8 Mb区间,类似的较强FISH杂交信号只出现在陆地棉D组祖先供体种雷蒙德氏棉,可锚定到D基因组的所有13条染色体,而没有出现在陆地棉A组祖先供体种非洲棉中,因此认为,四倍体棉的着丝粒序列可能来自于D组祖先。这些着丝粒相关序列的发现将促进棉花着丝粒定位、测序和进化研究。
张忠鑫[121]克隆了A组亚洲棉的一个端粒相关序列TAS-1。TAS-1富含A/T碱基(66.6%),含有一个拷贝的TTTAGGG型重复单元、10个串联重复序列、一个未知单元的散布重复序列,具有种属特异性。通过TRF法估测二倍体A组亚洲棉端粒平均长度在10 kb到20 kb之间。FISH表明,TAS-1在亚洲棉的每条染色体末端均有较强信号;在陆地棉一对同源染色体的一端产生较强信号外,其余染色体末端信号较弱;在海岛棉的所有染色体信号均很弱。通过PCR与FISH分析,获得了D组雷蒙德氏棉的10个端粒克隆,序列分析发现染色体末端存在较多的拟南芥型端粒重复单元的变异体,含有TTTAGGG型串联重复的端粒序列在A组、AD组中FISH信号差异较小,而含有其散布重复的端粒序列在A组、AD组染色体中的FISH信号差异较大。
4.3 棉花远缘杂交与新种质创制
通过种间杂交将近缘种优良性状转移到栽培棉可以拓宽棉花品种改良的遗传基础。为克服棉属种间杂交的不亲和性,Chen等[122]将陆地棉(TM-1)× 石系亚1号杂交的3 DPA胚珠再优化棉花胚珠培养基上培养,获得八个正常萌发的杂种胚,将其长成的幼苗嫁接到海岛棉砧木上,获得了染色体数目为2n=3x=39杂种植株。利用秋水仙素对上述杂种F1的30个侧芽芽头浸泡24 h,在成活18株中四株染色体数目为2n=6x=78(AADDAA),通过加倍首次获得了陆地棉–亚洲棉的人工合成六倍体。利用秋水仙素对红星草棉()×澳洲棉()的杂种F1植株的芽头进行加倍处理,也获得了合成双二倍体,GISH证实其中一个染色体组来源于澳洲棉的G2染色体组。
Chen等[123]和Wang等[124]利用形态学、SSR标记和GISH相结合,培育了一套陆地棉–澳洲棉外源异附加系。异常棉()为B基因组棉种,与四倍体棉的A亚基因组同源性较高,Wang等[125]和王琛[126]改进棉花GISH体系,采用异常棉和草棉基因组DNA同时作探针,提高封阻与探针的比例,建立了陆地棉–异常棉外源染色体鉴定GISH技术,从陆地棉与异常棉的回交后代中鉴定出11个单体异附加系。Tang等[127]采用类似方法,鉴定出10个陆地棉–比克氏棉染色体异附加系。
澳洲棉为陆地棉三级基因库,二者染色体重组困难。为将澳洲棉优异基因转入陆地棉,Wang等[124]利用辐射处理陆地棉–澳洲棉倍半二倍体花粉,授予去雄的陆地棉CL-2,利用多色GISH鉴定杂交种子含有澳洲棉染色体或片段,获得了棉花种间的染色体易位系和渐渗系,且发现所有易位事件均发生在澳洲棉与陆地棉的At染色体之间(图3A)。这些远缘种质为研究棉属物种间亲缘关系和棉花遗传改良提供了基础材料。
5 菊花分子细胞遗传学及其应用
菊科(Compositae)春黄菊族(Anthemideae)菊属()的菊属约有43个种和18个变种(亚种),它们以染色体基数9形成一个从二倍体到十倍体的多倍体系列。其中菊花(Ramat.)为多年生草本植物,是我国十大传统名花和世界四大切花之一,观赏和经济价值极高。栽培菊花大多为六倍体及非整倍体,具有高度杂合、自交不亲和及近交衰退等特性。我国是栽培菊花的起源中心,但原有商业主栽品种多为国外引进,抗蚜虫性、耐低/高温性差和种性退化等成为限制我国菊花产业发展的瓶颈。开展菊花分子细胞遗传学研究对于探究菊花起源与演化、菊属种间亲缘关系以及利用远缘杂交拓宽菊花遗传基础具有重大意义。
5.1 菊属染色体鉴定技术
对菊属植物的细胞学研究最早见于Tanaka对日本二倍体和四倍体野生菊花的核型报道[128]。Fedorof统计了广义菊属93种植物的染色体数目,发现多倍体(含种内多倍体)有56种,占60%[129]。20世纪80年代,李懋学[130]、杜冰群等[131]、汪劲武等[132,133]对我国产的部分野生菊属植物和栽培菊花进行细胞学分析,发现栽培菊花大部分为六倍体及非整倍体,染色体数在2n=51~75之间,以2n=54为高峰,提出了菊花是从六倍体的野生种进化而来的观点。从小菊、中菊到大菊的不同类型栽培菊花品种染色体数目存在较大差异,染色体数目与花径存在一定正相关,但同类型品种中,花径与染色体数目无相关性[134]。菊属植物广泛存在B染色体,且野生种出现的概率低于栽培品种,非整倍体出现B染色体的概率高于整倍体[135]。
核型分析发现,菊属植物的染色体以中着丝粒染色体(M)最多,依次分别为近中着丝粒染色体(m)、近端着丝粒染色体(t)和端着丝粒染色体(T)。陈发棣等[136]对三个地理居群野菊核型分析发现,南京野菊和黄山野菊均为四倍体,核型公式分别为2n=4x= 36=24m+10sm+2t、2n=4x=36=26m (2SAT)+6sm+4st,江山野菊为非整倍体,核型公式为2n=4x+1=37= 26m+8sm+3t,指出不同地理居群野菊的遗传基础存在一定差异。Kim等[137]也发现不同居群间或同一居群不同个体间核型分化很大。李畅等[138]对不同株型和用途的17个栽培小菊品种核型分析发现,大部分品种为“2A”型,只有三个地被菊品种为“2B”,认为核型多样性不仅是简单的染色体突变,很可能是染色体重组所致。

图3 FISH技术在棉属、菊属和杨树中的应用
A:澳洲棉–亚洲棉人工合成六倍体(2n=AADDGG=78)的多色FISH鉴定)。红色:Digoxigenin-dUTP标记的澳洲棉(GG) ();Biotin绿色亚洲棉(AA) ()为探针;蓝色:DAPI套染;B:北京植物园野菊() 5S rDNA和45S rDNA的oligo-FISH检测。蓝色:DAPI套染;红色:TAMRA标记的5S rDNA信号;绿色:FAM标记的45S rDNA信号;C:毛果杨中期染色体的oligo-FISH。红色:地高辛标记的1号染色体;绿色:生物素标记的4号染色体,黄色:生物素和地高辛标记的13号染色体。
随着分子细胞遗传学的发展,GISH、FISH技术在菊花上广泛应用。Tang等[139]以芙蓉菊((L.)Makino;2n=2=18)基因组DNA为探针,对大岛野路菊((Kitam.)Kitam;2n=10=90)和芙蓉菊属间远缘杂种F1进行GISH,发现其中45条染色体来自大岛野路菊,9条来自芙蓉菊,还发现不需要封阻DNA就能轻易地将两亲本基因组区分开,证明两属基因组亲缘关系较远。Deng等[140]对菊属、蒿属和芙蓉菊属的三属杂种进行GISH鉴定,发现不同株系间染色体数差异很大(42~ 63),且异常有丝分裂现象发生;杂交后代染色体组成广泛分离,六倍体植株中有9条来自黄金艾蒿、9条来自芙蓉菊,其余36条来自菊属地被菊‘钟山金桂’和大岛野路菊。Qi等[141]利用45S和5S rDNA为探针对12份菊属及其近缘属植物材料进行FISH分析,分别观察到2~10和2~6个杂交信号,其中芙蓉菊有两个45S杂交信号,菊花脑()、异色菊、甘菊、菊蒿有八个杂交信号,细裂亚菊有10个杂交信号,且大部分45S rDNA信号位于染色体末端,5S rDNA信号则位于染色体近末端或末端(图3B)。
5.2 菊属起源与演化
菊花原产中国,距今已有1600多年的栽培历史。据统计,全球品种总数超过两万种,我国选育的菊花品种有4000多个,菊花种类之繁多,变异之丰富,被称为花卉育种史上的一大奇迹。目前关于现代菊花起源的问题仍存在较大争议,主要认为菊花可能是由野菊()、毛华菊()、紫花野菊()、甘菊()、菊花脑等野生种天然杂交并经人工长期选育而来的杂种复合体[142,143],但是其直接的供体基因组并不明确。因此,探究菊花的起源与演化一直是国内外学者关注的焦点。
陈发棣等[144]通过对不同倍性的几种中国野生菊之间杂种F1减数分裂期染色体的配对分析,研究中国野生菊的亲缘关系和演化过程。发现甘菊和菊花脑虽然是两个亲缘关系较近的二倍体,但已发生了某种程度的分化;南京野菊()和尖叶野菊(var.)是含有相同染色体组的异源四倍体;毛华菊为异源六倍体,其染色体在减数分裂中期I(MI)均能较好地配成二价体。尖叶野菊(4x)与菊花脑(2x)、南京野菊(4x)与毛华菊(6x)的F1,染色体配对构型分别接近于9I+9II和9I+18II,表明菊花脑或其近缘种是尖叶野菊染色体组的供体之一,南京野菊或其近缘种是毛华菊两个染色体组的供体。
药用菊在栽培菊花的起源进化过程中可能起一定作用。陈发棣等[145]对原产中国的三个药用菊品种‘杭白菊’、‘滁菊’、‘怀菊’;研究发现,三个药用菊在MI基本配成二价体构型,平均每个花粉母细胞构型为0.20I+17.70II+0.10IV、0.44I+26.66II+0.06IV、1.12I+26.67II+0.09IV。三种药用菊均有部分出现了四价体,后期Ⅰ观察到染色体桥现象,表明某些染色体可能存在易位或倒位等结构变异。药用菊品种的减数分裂行为与菊属野生种相似,说明药用菊品种比观赏菊品种更为原始。除了药用菊,黄色、倍数性较低的栽培观赏小菊品种也较为原始。陈发棣等[146]观察栽培菊中的药用菊品种‘滁菊’(6x)和观赏小菊品种‘小黄菊’(4x)与野菊(4x)和毛华菊(6x)间F1染色体配对构型发现,‘小黄菊’和‘滁菊’的染色体组分别与野菊和毛华菊的染色体组相似,认为四倍体和六倍体栽培菊花极可能直接从野生的四倍体野菊或野生的六倍体毛华菊演化而来。‘滁菊’×野菊、野菊ב滁菊’、‘小黄菊’×毛华菊的F1在MI出现多于18II或出现三价体和四价体构型,推测可能存在染色体组间易位和促进配对或影响同源染色体正常配对的基因。李辛雷和陈发棣[147]用RAPD标记研究菊属野生种、栽培菊及种间杂种亲缘关系,认为九个野生种的进化趋势符合从低倍到高倍异源多倍化的过程。崔娜欣[148]对二倍体菊花脑、甘菊及异色菊进行减数分裂研究,发现异色菊在进化上可能较菊花脑和甘菊更原始,菊花脑、甘菊或其近缘种可能是六倍体药用菊和栽培菊的染色体组供体。
细胞学证据表明菊属植物进化的主要途径是异源多倍化,且多倍体野生种已经明显二倍化,国内其他学者也得到了类似结论[149]。结合利用分子生物学等技术可以为其进化演化提供更多证据。近年来,南京农业大学菊花研究团队与中国中医科学院中药研究所等多家单位联合公布了菊花近缘种菊花脑基因组草图(2.53 Gb)[150],日本研究团队完成了二倍体甘野菊(; 2.72 Gb)的全基因组测序[151],为在基因组水平上研究菊花起源与演化提供了可能。随着基因组和后基因组时代的到来,进一步开展栽培菊花基因组测序,开发oligo-FISH探针,将有助于系统探究菊花的起源与演化关系。
5.3 菊属远缘杂交与种质创新
栽培菊花遗传基础狭窄,种内遗传改良较为困难,而菊花近缘种属植物蕴含栽培菊花不具备的抗蚜虫、耐寒、耐盐等优良性状。因此,远缘杂交是实现优异基因在不同种属间转移的有效手段之一,菊花及其近缘种质资源系统收集、保存、整理与评价,将推动菊花优异近缘种属资源的发掘与育种利用。南京农业大学菊花研究团队建立了“中国菊花种质资源保存中心”,系统开展菊花及其近缘种属植物资源的收集整理、保存与评价工作。共收集保存了包括近缘种属野生种质、品种和育种中间材料的各类种质5000余份,种质数量居世界首位。创建了离体缓慢生长保存技术体系,实现了核心种质的节本高效中长期保存[152]。建立了菊花及其近缘种属植物相关抗性的评价体系,在黄金艾蒿、牡蒿、细裂亚菊等物种中,鉴定出包括抗蚜虫、耐寒、耐盐等各类优异抗性种质。首次发现高抗蚜虫的黄金艾蒿、耐寒性强的细裂亚菊优异种质[153~156]。
开展远缘杂交系列基础理论和技术研究。明确了细胞程序性死亡引起的杂种胚败育是菊花远缘杂交障碍的主要原因,发掘出指示胚胎败育进程的Caspase-like蛋白酶[157];建立了幼胚拯救技术体系,解决了因胚胎败育难以获得远缘杂种的难题[158];创建了基于形态学、细胞学及多色GISH的远缘杂种鉴定技术。成功将外源种属植物的抗性基因导入栽培菊花,创制抗蚜、耐寒、耐盐等远缘种质[159~164]。首次报道了菊属与黄金艾蒿、芙蓉菊、菊蒿、太行菊、白晶菊等七个属间杂种和三属(菊属、蒿属、芙蓉菊属) 4物种新种质,实现了抗蚜性、耐盐性与托桂花型的聚合[165]。育成了系列优质高抗新奇特品种,改变了以往我国菊花商业主栽品种抗蚜和耐寒等抗性差、花色花型单调、依赖进口等状况,使我国自主菊花品种的占有率由原来的10%提高到40%左右,其中选育品种约占现有品种的30%,有力推动了我国菊花品种更新和产业升级。
6 杨树分子细胞遗传学及其应用
杨树(2n=2x=38)为杨属()树种的统称,是北半球绿化、防护林及人工用材林的重要树种,具有重要的生态价值和经济价值。杨树不但分布广泛,生长迅速,且种间杂交可育,易于无性繁殖。杨树基因组较小,遗传转化体系较易建立[166,167]。这一系列的特征,使其成为林木研究的模式树种。2006年报道了毛果杨()的全基因组测序结果[168],杨树成为了第一个完成全基因组测序的多年生木本植物[169]。近年来,胡杨()、新疆杨()的基因组测序结果也相继发表[170,171]。杨树全基因组测序结果为杨树的比较基因组学、基因克隆及分子育种等提供了有效的研究平台。
6.1 杨树分子细胞遗传学技术
杨树染色体不仅数目多,形态十分相似,传统的细胞遗传学方法很难准确辨别每条染色体。传统的杨树细胞遗传学研究主要集中在染色体计数、染色体形态、rDNA 定位等方面[172,173]。已有研究证明杨树的染色体基数为x=19[174];多数杨树是二倍体,只有少数白杨派或种间杂交后代是三倍体或非整倍体[175]。通过对杨树减数分裂及其进程大量研究,建立了部分杨树花序外观形态与减数分裂进程的对应关系,为杨树的倍性育种奠定了基础。康向阳等[176]发现减数分裂过程中,毛白杨()花序的形态和花药颜色变化与其所处的减数分裂的时期密切相关。辛昊阳等[177]研究了美洲黑杨()小孢子母细胞减数分裂中的染色体行为,发现同一花枝上的不同花芽及同一花序中不同小花的发育不同步。兰月等[178]研究发现小叶杨()花粉母细胞减数分裂过程存在核仁数目的动态变化、落后染色体及不同类型的纺锤体,在室外水培的花枝花粉中观察到了2n花粉的存在。ISH技术为杨树染色体准确鉴定提供了有效手段。董凤平等[179]和胡宝全等[180]初步建立了杨树有丝分裂中期染色体的FISH核型。Xin等[181]利用毛果杨参考基因组,开发了一套染色体oligo探针库,不仅可以准确识别毛果杨所有19对染色体,而且也可以在其他杨属物种中通用。通过多轮oligo-FISH技术,在同一细胞中准确识别了杨树所有19对染色体,构建了基于oligo-FISH杨树中期染色体核型图(图3C)。
6.2 杨树性染色体的分子细胞遗传学
杨树为雌雄异株,其性别决定受遗传控制,性别决定区位于19号染色体,然而杨树的性别决定机制及性别决定区在19号染色体上的位置存在争议。对不同杨树种的经典细胞学研究中,没有观察到形态上有差异的性染色体,但是,已构建的不同杨树种的多个遗传连锁图谱均证实,杨树基因组中存在性别决定区。Xin等[182]利用毛果杨19号染色体的oligo探针库,在毛白杨、美洲黑杨和小叶杨的粗线期染色体上进行oligo-FISH,发现19号染色体短臂末端的DNA序列还没有被组装到19号染色体上,在美洲黑杨和毛白杨的22-24%减数分裂细胞中,19号染色体的短臂末端在粗线期阶段出现未配对的现象;利用19号染色体的oligo-FISH及单基因FISH进行比较作图,证明不同杨树的19号染色体保持着很好的共线性,毛白杨和美洲黑杨19号染色体的性别决定区均位于短臂末端,其性别决定机制均为XY型。
7 甘薯分子细胞遗传学及其应用
甘薯((L.)Lam., 2n=6x=90)是属于旋花科(Convolvulaceae)番薯属()的一年生植物,别名番薯、红薯、白薯、地瓜、山芋、红苕等。甘薯是世界卫生组织推荐的最佳食物,可用作鲜食、饲料、加工和菜用等。甘薯属植物约有800~900个种,含有自然二倍体、四倍体和六倍体等不同倍性的物种资源。栽培甘薯属于六倍体无性繁殖作物,自交不亲和且高度杂合,遗传基础狭窄。开展甘薯的分子细胞遗传学研究,一方面可探究甘薯属物种间的亲缘关系,也可为甘薯种质创新和品种改良打下坚实基础。
7.1 甘薯染色体鉴定技术
早在20世纪30年代,Nakajima[183]分析了甘薯属植物的染色体数目,确定了大多数甘薯属物种染色体基数为x=15,但也发现存在x=14和x=12的非整倍体(图4)。核型分析发现,甘薯近缘野生种的3个材料“698001”、“698011”、“P-875-6”的核型公式分别为2n=2x=30=18m(2SAT)+12sm (2SAT);2n=2x=30=17m (2SAT)+13sm (2SAT);2n=6x=90=54m (2SAT)+36sm (2SAT)[184];美国Jacq.的核型公式为2n=2x=30=26m (2SAT)+4sm (2SAT),染色体长度为4.19~8.83 μm[185]。汤佳立等[186]利用银染等技术进行细胞学分析,发现栽培种徐薯18 ()间期核有六对、八对和九对银染点;安婷婷等[187]利用DAPI染色技术,获得中国知名品种徐薯18的核型公式:2n=6x=90=72m+18sm (18SAT),其随体位于第1、3、6染色体上。
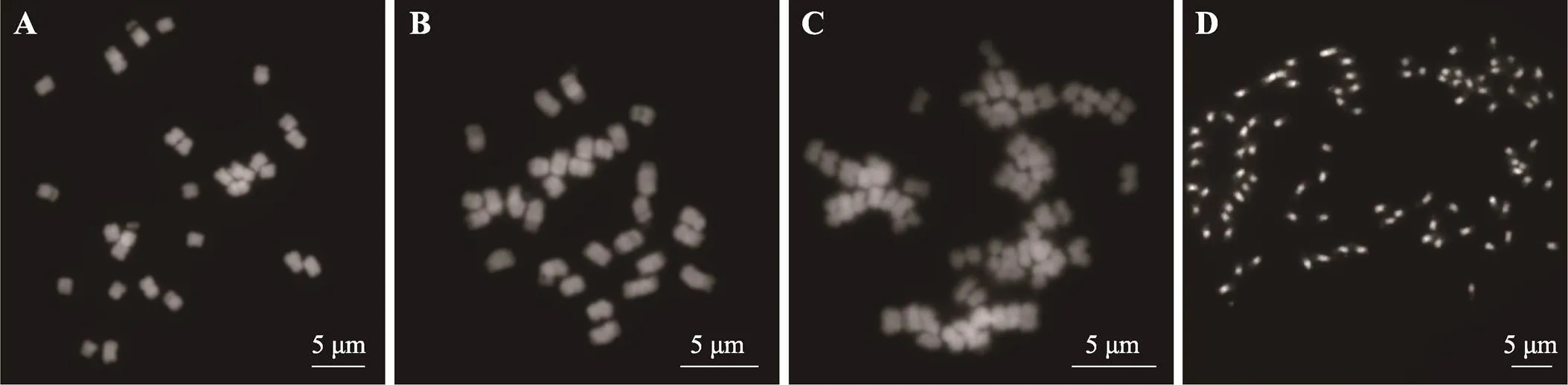
图4 4个甘薯属物种的有丝分裂中期染色体
A:(2n=2x=28); B:(2n=2x=30); C:(2n=4x=60); D:‘Xushu18’(2n=6x=90)。
7.2 甘薯起源与进化
近年来ISH技术被用于研究甘薯的起源与进化关系。向素琼等[188]以甘薯祖先种三浅裂野牵牛(2x)为探针,对(4x) 2个株系“695104”和“697288”进行GISH,发现“695104”几乎在所有染色体全长上都有均匀明亮的信号,推测其为(2x)直接加倍而来;而“697288”虽然各条染色体也均有信号,但信号区域与亮度有差异。汤佳立等[186]和安婷婷等[187]分别利用45S rDNA和5S rDNA为探针对徐薯18进行FISH分析,发现其八对或九对染色体上有强弱不一的45S rDNA信号,三对染色体上有5S rDNA信号,分别位于着丝粒区、亚着丝粒区和染色体端部。
利用测序数据可以开发更多的分子细胞遗传学标记。Sun等[189]利用二代测序技术对甘薯二倍体祖先种进行低通量测序,通过RepeatExplorer分析其中的重复序列组分,鉴定到25个串联重复序列,经进一步筛选,发现两个重复序列可用于识别甘薯及其近缘野生种之间同源染色体。结合利用核糖体DNA序列,建立了甘薯及其五个近缘野生种((4x),(2x),(2x),(2x),I.×(2x))的FISH核型,为进一步探讨甘薯物种基因组间的进化及亲缘关系提供了有效手段。
7.3 甘薯远缘杂交和种质创新
日本是最早利用甘薯近缘野生种进行杂交育种工作的。20世纪70年代初,以Nishiyama为代表的研究者利用K123与甘薯杂交,成功培育出含有野生种(6x)血统的新品种“南丰”,具高产、高淀粉、高抗线虫病等多种优良性状[190]。我国自1977年开展远缘杂交研究,江苏省农科院粮食所在引进和利用近缘野生种,选育出一批(6x)优良系 Y1、Y2、Y3、…Y17,并利用‘华北52-54’和‘Y10’杂交,创制出高产、高淀粉、高抗茎线虫病的种间材料 H11-30。江苏徐州甘薯研究中心先后从国际马铃薯中心(CIP)、美国路易斯安那州立大学、美国国家种质资源库(NPGS)引进了131份近缘野生种[191],曹清河项目组对收集的甘薯野生资源进行抗病性等表型鉴定;开展种间杂交技术研究,发明了“一种成功率高的甘薯远缘杂交育种方法”,在国际上首次获得了五个新的种间杂种[192,193],其中,4个为四倍体,1个为五倍体;发明了一种耐病毒甘薯种间渐渗系的培育方法(申请号201710940343.X),培育了一批耐甘薯病毒病害(Sweet potatovirus diseases, SPVD)的优异渐渗系,通过SSR分子标记对渐渗系群体进行遗传作图,最终关联到了三个耐SPVD的QTLs。这些为甘薯遗传改良提供了重要的基础材料。
8 植物分子细胞遗传学研究展望
分子细胞遗传学的核心技术是DNA分子原位杂交,由于其可以直观显示特定DNA序列在组织或细胞中的空间位置,在植物遗传改良、植物起源与进化、远缘杂交与种质创新、特定染色体的识别、核型构建、外源基因的物理定位和克隆等研究中曾经发挥了重要作用,未来也将在更多的物种中得到更广泛的应用。据不完全统计,目前已经完成400多个物种的基因组测序。伴随测序技术、生物信息学发展,更多的物种基因组序列将被解析。这既给植物分子细胞遗传学带来了挑战,即通过序列比对可以精确揭示染色体水平的结构重排,揭示物种之间的进化关系;但同时也带来了机遇,即海量的组学数据和工具可以提供更多的探针开发资源,可以直观地揭示在DNA、蛋白等水平调控的特征,例如近年来发展的oligo探针库开发和利用,目前已经在水稻、小麦等许多作物上成功开发了染色体或染色体区段特异的探针库,不但大大提高了FISH技术的效率,而且通过优化探针开发流程,可以通过不同细胞分裂时期、不同组织的oligo-painting,开展染色体水平的比较细胞组学、同源染色体或部分同源染色体配对的动态机制研究等[194,195]。
FISH技术进入FISH+阶段,借助和结合相关学科发展的新技术,发展出更多的新技术用于解决更多的生物学问题(表1)。直接分析原位组织和原生环境中转录组和其他基因组信息,对于生物学的许多领域是至关重要的,这也是空间基因组学的重要内容[196~198]。加州理工学院Cai Long团队开发了迭代荧光原位杂交(seqFISH+)技术,可以使用标准共聚焦显微镜在单个细胞中对10,000个基因的mRNA进行成像[196]。单分子荧光原位杂交(smFISH)是一种新的基因表达分析方法,早期的smFISH能报告转录本丰度和空间定位,但还不能实现基因组范围的分析[197]。瑞士苏黎世大学的研究人员报告了一种高通量smFISH策略,该策略通过产生放大的信号,大大增加亮度,提高信噪比,而缩短成像时间,实现了实验过程自动化[198]。FISH+技术的不断发展和完善,必将大大拓展以FISH技术为核心的分子细胞遗传学的应用范围,在生命科学研究中继续发挥其重要作用。近年来不同物种的测序或重测序研究表明,物种进化和驯化过程中广泛存在染色体大片段的结构重排现象,进一步研究发现染色体结构重排对区段内或附近的基因表达和调控有重要影响,进而影响性状变异,对于植物改良影响深远,但这种变异产生的机制还不十分明了。尽管测序技术不断发展,成本也不断下降,但对多数物种来讲仍难以进行大规模深度重测序,分子细胞遗传学与高通量的分子标记技术相结合,可以快速、直观地鉴定不同资源群体中的结构重排,对于评价其在育种中的利用价值、解析其影响基因表达调控机制具有重要意义。

表1 FISH技术及其应用
致谢
首先衷心感谢为以上研究做出重大贡献的各位同仁、研究生!由于杂志要求所限,未能将您们的名字放进本文的作者名单,在此深表歉意!参与本研究的多位中青年老师都曾在美国密西根大学蒋继明教授实验室访学交流,蒋老师为我国分子细胞遗传学培养了一批杰出人才;蒋继明教授出国前也曾经任职江苏省遗传学会秘书,此文的撰写过程中更是得到蒋老师的指导,再次表示衷心感谢!感谢中国科学院遗传与发育研究所程祝宽研究员为本文撰写所提出的宝贵建议!感谢国家重点研发计划(2016YFD0102001-004)的资助。
说明
国内外科学家利用不同的植物物种,广泛开展植物细胞遗传学研究,在基础研究和应用研究领域都取得了重要进展。本综述主要是为纪念江苏省遗传学会成立40周年撰写的,主要展示了江苏省遗传学会从事该领域研究的主要单位在植物细胞遗传和分子细胞遗传方面取得的进展。篇幅所限,未能全面展示国内外兄弟单位的工作,恳请各位同行海涵。
[1] Gill BS, Friebe B, Endo TR. Standard karyotype and nomenclature system for description of chromosome bands and structural aberrations in wheat ()., 1991, 34(5): 830–839.
[2] Rayburn AL, Gill BS. Use of biotin-labeled probes to map specific DNA sequences on wheat chromosomes., 1985, 76(2): 78–81.
[3] Rayburn AL, Gill BS. Molecular identification of the D-genome chromosomes of wheat., 1986, 77(4): 253–255.
[4] Rayburn AL, Gill BS. Molecular analysis of the D-genome of the., 1987, 73(3): 385–388.
[5] Mukai Y, Nakahara Y, Yamamoto M. Simultaneous discrimination of the three genomes in hexaploid wheat by multicolor fluorescence in situ hybridization using total genomic and highly repeated DNA probes., 1993, 36(3): 489–494.
[6] Zhang W, Zhang RQ, Feng YG, Bie TD, Chen PD. Distribution of highly repeated DNA sequences inand its application in the identification of alien chromatin., 2013, 58(8): 890–897.
[7] Dolezel J, Kubalaková M, Paux E, Bartos J, Feuillet C. Chromosome-based genomics in the cereals., 2007, 15(1): 51–66.
[8] Doležel J, Vrána J, Cápal P, Kubaláková M, Burešová V, Simková H. Advances in plant chromosome genomics., 2014, 32(1): 122–136.
[9] Kubaláková M, Valárik M, Barto J, Vrána J, Cíhalíkova J, Molnár-Láng M, Dolezel J. Analysis and sorting of rye (L.) chromosomes using flow cytometry., 2003, 46(5): 893–905.
[10] Hernandez P, Martis M, Dorado G, Pfeifer M, Gálvez S, Schaaf S, Jouve N, Šimková H, Valárik M, Doležel J, Mayer KFX. Next-generation sequencing and syntenic integration of flow-sorted arms of wheat chromosome 4A exposes the chromosome structure and gene content., 2012, 69(3): 377–386.
[11] Chen PD, Gill BS. The Origin of chromosome 4A and genomes B and G of tetraploid wheats., 1984, 10(3): 146–153.陈佩度, Gill BS. 四倍体小麦染色体4A和B, G染色体组的起源. 作物学报, 1984, 10(3): 146–153.
[12] Liu DJ, Chen PD. N-banding inand-amphidiploid., 1984, 11(2): 106–108.刘大钧, 陈佩度. 簇毛麦和硬粒小麦–簇毛麦双二倍体的染色体N-分带. 遗传学报, 1984, 11(2): 106–108.
[13] Dong FG, Chen PD, Liu DJ. An improved C-banding technique forchromosomes., 1991, 18(6): 525–528.董凤高, 陈佩度, 刘大钧. 簇毛麦染色体的改良C-分带. 遗传学报, 1991, 18(6): 525–528.
[14] Yan Y, Liu DJ. Production and cytogenetics of intergeneric hybrid betweenand., 1987, 20(6): 17–21.颜旸, 刘大钧. 纤毛鹅观草与普通小麦属间杂种的产生及其细胞遗传学研究. 中国农业科学, 1987, 20(6): 17–21.
[15] Weng YQ, Liu DJ. Morphology, scab resistance and cytogenetics of inter generic hybrids ofL. withC. Koch (Agropyron) species., 1989, 22(5): 1–7.翁益群, 刘大钧. 鹅观草与普通小麦属间杂种F1的形态—赤霉病抗性与细胞遗传学研究.中国农业科学, 1989, 22(5): 1–7.
[16] Jiang JM, Liu DJ. Morphology and cytogenetics of intergeneric hybrid betweenand., 1990: 373–376.蒋继明, 刘大钧. 鹅观草与大麦属间杂种的形态和细胞遗传学研究. 遗传学报, 1990: 373–376.
[17] Chen PD, Wang ZT, Wang SL, Wang L, Wang YZ, Liu DJ. Transfer of useful germplasm form. to common wheat. III. Development of addition lines with wheat scab resistance., 1995, 22(3): 206–210.
[18] Liu DJ, Chen PD, Wu YL, Wang YN, Qiu BX, Wang SL.amphidiploid., 1986, 12(3): 155–162.刘大钧, 陈佩度, 吴沛良, 王耀南, 邱伯行, 王苏玲. 硬粒小麦–簇毛麦双二倍体. 作物学报, 1986, 12(3): 155–162.
[19] Pei GZ, Chen PD, Liu DJ. A cytogenetic analysis of some powdery mildew resistant strains of the hybrid progeny between wheat and, 1986, 9(1): 1–9.裴广铮, 陈佩度, 刘大钧. 小麦与簇毛麦杂种后代抗白粉病株系的细胞遗传学分析. 南京农业大学学报, 1986, 9(1): 1–9.
[20] Chen PD, Qi LL, Zhou B, Zhang SZ, Liu DJ. Development and molecular cytogenetic analysis of wheat-6VS/6AL translocation lines specifying resistance to powdery mildew., 1995, 91(6–7): 1125–1128.
[21] Li WL, Chen PD, Qi LL, Liu DJ. Isolation, characterization and application of a species-specific repeated sequence from., 1995, 90(3–4): 526–533.
[22] Xu LY, Yang JX, Xuan P, Liu DJ, Yang SH. Analysis the relative relationship of wheat-using the C-banding of pollen mother cell and ditelosomics., 1996, 29(4): 23–28.徐利远, 杨吉秀, 宣朴, 刘大钧, 杨世湖. 用花粉母细胞分带和端体研究小麦与偃麦草的亲缘关系. 中国农业科学, 1996, 29(4): 23–28.
[23] Liu WX, Chen PD, Liu DJ. Development of-translocation lines by irradiating adult plants at meiosis., 1999, 41(5): 463–467.刘文轩, 陈佩度, 刘大钧. 利用减数分裂期成株电离辐射选育小麦–大赖草易位系的研究. 植物学报, 1999, 41(5): 463–467.
[24] Qi ZJ, Liu DJ, Chen PD. Development and identification of--double translocation line 1RS·1BL, 6VS·6ALchromosome C-banding and dual color FISH., 2001, 28(3): 267–273.亓增军, 刘大钧, 陈佩度. 利用染色体C-分带和双色荧光原位杂交技术鉴定普通小麦–黑麦–簇毛麦双重易位系1RS·1BL,·6VS·6AL. 遗传学报, 2001, 28(3): 267–273.
[25] Chen FD, Chen PD, Wang SL. Development of wheat-alien lines with addedchromosomes and 6VS/6AL translocation chromosomes., 2001, 43(4): 359–363.陈发棣, 陈佩度, 王苏玲. 普通小麦–大赖草–簇毛麦异附加、易位系的选育和鉴定. 植物学报, 2001, 43(4): 359–363.
[26] Wang XE, Chen PD, Liu DJ, Zhang P, Zhou B, Friebe B, Gill BS. Molecular cytogenetic characterization ofchromosome additions in common wheat., 2001, 102: 651–657.
[27] Wang XE, Chen PD, Zhou B, Yuan JH, Liu WX, Gill BS, Liu DJ. RFLP analysis of Wheat-translocation lines., 2001, 28(12): 1142–1150.王秀娥, 陈佩度, 周波, 袁建华, 刘文轩, Gill BS, 刘大钧. 小麦–大赖草易位系的RFLP分析. 遗传学报, 2001, 28(12): 1142–1150.
[28] Zhuang LF, Qi ZJ, Ying J, Chen PD, Liu DJ. Development and identification of a set of-disomic alien addition lines., 2003, 30(10): 919–925.庄丽芳, 亓增军, 英加, 陈佩度, 刘大钧. 普通小麦–百萨偃麦草二体异附加系的选育与鉴定. 遗传学报, 2003, 30(10): 919–925.
[29] Chen PD, Liu WX, Yuan JH, Wang XE, Zhou B, Wang SL, Zhang SZ, Feng YG, Yang BJ, Liu GX, Liu DJ, Qi LL, Zhang P, Friebe B, Gill BS. Development and characterization of wheat-translocation lines with resistance to Fusarium head blight., 2005, 111(5): 941–948.
[30] Du P, Zhuang LF, Wang YZ, Yuan L, Wang Q, Wang DR, Dawadondup, Tan LJ, Shen J, Xu HB, Zhao H, Chu CG, Qi ZJ. Development of oligonucleotides and multiplex probes for quick and accurate identification of wheat andchromosomes., 2017, 60(2): 93–103.
[31] Wang YZ. Development and characterization of small segment translocations of Thinopyrum bessarabicum and cytological mapping of interest genes[Dissertation]. Nanjing Agricultural University, 2013.王艳芝. 百萨偃麦草染色体小片段易位的创制、鉴定与基因定位分析[学位论文]. 南京农业大学, 2013.
[32] Yang J, Liu ZT, Chen JY, Wang YZ, Zhuang LF, Qi ZJ. Accurate characterization of wheat-alien chromosome translocations using oligonucleotide multiplex painting combined with genomic in situ hybridization and molecular marker analysis., 2018, 38(3): 253–261.杨骄, 刘志涛, 陈健泳, 王艳芝, 庄丽芳, 亓增军. 寡核苷酸探针套涂染结合基因组原位杂交和分子标记分析准确鉴定小麦–百萨偃麦草异源易位系的研究. 麦类作物学报, 2018, 38(3): 253–261.
[33] Wang DR, Du P, Pei ZY, Zhuang LF, Qi ZJ. Development and application of high resolution karyotypes of wheat “Chinese Spring” aneuploids., 2017, 43(11): 1575–1587.王丹蕊, 杜培, 裴自友, 庄丽芳, 亓增军. 基于寡核苷酸探针套painting的小麦“中国春”非整倍体高清核型及应用. 作物学报, 2017, 43(11): 1575–1587.
[34] Huang XY, Zhu MQ, Zhuang LF, Zhang SY, Wang JJ, Chen XJ, Wang DR, Chen JY, Bao YG, Guo J, Zhang JL, Feng YG, Chu CG, Du P, Qi ZJ, Wang HG, Chen PD. Structural chromosome rearrangements and polymorphisms identified in Chinese wheat cultivars by high- resolution multiplex oligonucleotide FISH., 2018, 131(9): 1967–1986.
[35] Guo JT, Lei YH, Zhang HT, Song DH, Liu X, Cao ZL, Chu CG, Zhuang LF, Qi ZJ. Frequent variations in tandem repeats pSc200 and pSc119.2 cause rapid chromosome evolution of open-pollinated rye., 2019, 39: 133.
[36] Sun HJ, Song JJ, Lei J, Song XY, Dai KL, Xiao J, Yuan CX, An SM, Wang HY, Wang XE. Construction and application of oligo-based FISH karyotype of., 2018, 45(8): 463–466.
[37] Xiao J, Dai KL, Fu L, Vrána J, Kubaláková M, Wan WT, Sun HJ, Zhao J, Yu CY, Wu YF, Abrouk M, Wang HY, Doležel J, Wang XE. Sequencing flow-sorted short arm ofchromosome 4V provides insights into its molecular structure and virtual gene order., 2017, 18(1): 791.
[38] Lei J, Zhou JW, Sun HJ, Wan WT, Xiao J, Yuan CX, Karafiátová M, Doležel J, Wang HY, Wang XE. Development of oligonucleotide probes for FISH karyotyping in, a wild relative of common wheat., 2020, 8(4): 676–681.
[39] Song XY, Song RR, Zhou JW, Yan WK, Zhang T, Sun HJ, Xiao J, Wu YF, Xi ML, Lou QF, Wang HY,Wang XE. Development and application of oligonucleotide- based chromosome painting for chromosome 4D ofL.. 2020, 28(2): 171–182.
[40] Chen PD, You CF, Hu Y, Chen SW, Zhou B, Cao AZ, Wang XE. Radiation-induced translocations with reducedchromatin at thelocus for powdery mildew resistance in wheat., 2013, 31: 477–484.
[41] Yang XM, Cao AZ, Sun YL, Chen PD. Tracing the location of powdery mildew resistance-related geneby FISH with a TAC clone in-alien chromosome lines., 2013, 58: 4084–4091.
[42] Cao AZ, Xing LP, Wang XY, Yang XM, Wang W, Sun YL, Qian C, Ni JL, Chen YP, Liu DJ, Wang XE, Chen PD. Serine/threonine kinase gene, a key member of powdery mildew resistance gene, confers powdery mildew resistance in wheat., 2011, 108(19): 7727–7732.
[43] Xing LP, Hu P, Liu JQ, Witek K, Zhou S, Xu JF, Zhou WH, Gao L, Huang ZP, Zhang RQ, Wang XE, Chen PD, Wang HY, Jones JDG, Karafiátová M, Vrána J, Bartoš J, Doležel J, Tian YC, Wu YF, Cao AZ.fromencodes a CC-NBS-LRR protein conferring powdery mildew resistance in wheat., 2018, 11(6): 874–878.
[44] Bao WK. Breeding of octoploid triticale., 1974, 1(1): 36–49.鲍文奎. 八倍体小黑麦的育种. 遗传学报, 1974, 1(1): 36–49.
[45] Li JY. Selected some academic articles of Zhensheng. 2007, Science Press.李家洋.《李振声论文选集》. 2007, 科学出版社.
[46] Hao S, He MY, Xu ZY, Zhang CS, Zou MQ, Zhang H, Sun P. Cytogenetic studies on five intermediate forms of progeny of×., 1979, 21(3): 259–261.郝水, 何孟元, 徐宗尧, 张传善, 邹明谦, 张汉, 孙平. 小麦×天蓝冰草后代五个中间类型的细胞遗传学研究. 植物学报, 1979, 21(3): 259–261.
[47] Li JL, Xu XL, Zhao LX, Xue X. The cell inheritance ofby induced mutation., 1981, 1: 12–21.李集临, 徐香玲, 赵连喜, 薛玺. 小黑麦()诱发突变的细胞遗传. 中国农业科学, 1981, 1: 12–21.
[48] Sun SC. The approach and methods of breeding new varieties and new species from Agrotriticum hybrids., 1981, 7(l): 51–58.孙善澄. 小偃麦新品种与中间类型的选育途径、程序和方法. 作物学报, 1981, 7(l): 51–58.
[49] Chen PD, Liu DJ. Cytogenetic studies of hybrid progenies betweenand., 1982, 4(4): 1–16.陈佩度, 刘大钧. 普通小麦与簇毛麦杂种后代的细胞遗传学研究. 南京农学院学报, 1982, 4(4): 1–16.
[50] Chen PD, Zhou B, Qi LL, Liu DJ. Identification of wheat-amphiploid, addition, substitution and translocation lines by in situ hybridization using biotin-labelled genomic DNA as a Probe., 1995, 22(5): 380–386.陈佩度, 周波, 齐莉莉, 刘大钧. 用分子原位杂交(GISH)鉴定小麦–簇毛麦双倍体、附加系、代换系和易位系. 遗传学报, 1995, 22(5): 380–386.
[51] Chen YP, Wang HZ, Cao AZ, Wang CM, Chen PD. Cloning of a resistance gene analog from wheat and development of a codominant PCR marker for., 2006, 48(6): 715–721.
[52] Cao AZ, Wang XE, Chen YP, Zou XW, Chen PD. A sequence-specific PCR marker linked withdistinguishes chromosomes 6AS, 6BS, 6DS ofand 6VS of., 2006, 125(3): 201–205.
[53] Wang CM, Bie TD, Chen QZ, Cao AZ, Chen PD. Development and application of molecular markers specific to chromosome 6VS of., 2007, 33(10): 1595–1600.王春梅, 别同德, 陈全战, 曹爱忠, 陈佩度. 簇毛麦6V染色体短臂特异分子标记的开发和应用. 作物学报, 2007, 33(10): 1595–1600.
[54] Zhao RH, Wang HY, Jia Q, Xiao J, Yuan CX, Zhang YJ, Hu QS, Wang XE. Development of EST-PCR markers for the chromosome 4V ofand their application in identification of 4V chromosome structural aberrants., 2014, 13(2): 282–289.
[55] Wang HY, Dai KL, Xiao J, Yuan CX, Zhao RH, Doležel J, Wu YF, Cao AZ, Chen PD, Zhang SZ, Wang XE. Development of intron targeting (It) markers specific for chromosome arm 4VS ofby chromosome sorting and next-generation sequencing., 2017, 18(1): 167.
[56] Zhang XD, Wei X, Xiao J, Yuan CX, Wu YF, Cao AZ, Xing LP, Chen PD, Zhang SZ, Wang XE, Wang HY. Whole genome development of intron targeting (IT) markers specific forchromosomes based on next-generation sequencing technology., 2017, 37: 115.
[57] Sun HJ, Song JJ, Xiao J, Xu T, Wei X, Yuan CX, Cao AZ, Xing LP, Wang HY, Wang XE. Development of EST-PCR markers specific to the long arm of chromosome 6V of, 2018, 17(8): 1720–1726.
[58] Qi LL, Chen PD, Liu DJ, Zhou B, Zhang SZ. New resource of wheat-translocation resistant to wheat powdery mildew., 1994, 2: 52–53.齐莉莉, 陈佩度, 刘大钧, 周波, 张守忠. 小麦白粉病的新抗源—普通小麦簇毛麦易位系. 作物品种资源, 1994, 2: 52–53.
[59] Bie TD, Cao YP, Chen PD. Mass production of intergeneric chromosomal translocations through pollen irradiation ofamphiploid., 2007, 49(11): 1619–1626.
[60] Chen SW, Chen PD, Wang XE. Inducement of chromosome translocation with small alien segments by irradiating mature female gametes of the whole arm translocation line., 2008, 51(4): 346–352.
[61] Qi LL, Pumphrey MO, Friebe B, Zhang P, Qian C, Bowden RL, Rouse MN, Jin Y, Gill BS. A novel robertsonian translocation event leads to transfer of a stem rust resistance gene (Sr52) effective against race Ug99 frominto bread wheat., 2011, 123(1): 159–167.
[62] Zhang RQ, Feng YG, Li HF, Yuan HX, Dai JL, Cao AZ, Xing LP, Li HL. Cereal cyst nematode resistance geneeffective againsttransferred from chromosome 6VL ofto bread wheat., 2016, 36: 122.
[63] Zhang RQ, Sun BX, Chen J, Cao AZ, Xing LP, Feng YG, Lan CX, Chen PD., a developmental-stage and tissue-specific powdery mildew resistance gene introgressed frominto common wheat., 2016, 129(10): 1975–1984.
[64] Zhao RH, Wang HY, Xiao J, Bie TD, Cheng SH, Jia Q, Yuan CX, Zhang RQ, Cao AZ, Chen PD, Wang XE. Induction of 4VS chromosome recombinants using the CSmutant and mapping of the wheat yellow mosaic virus resistance gene from., 2013, 126(12): 2921–2930.
[65] Dai KL, Zhao RH, Shi MM, Xiao J, Yu ZY, Jia Q, Wang ZK, Yuan CX, Sun HJ, Cao AZ, Zhang RQ, Chen PD, Li YB, Wang HY, Wang XE. Dissection and cytological mapping of chromosome arm 4VS by the development of wheat-structural aberration library., 2020, 133(1): 217–226.
[66] Matsumoto TE. International Rice Genome Sequencing Project. The map-based sequence of the rice genome., 2005, 436: 793–800.
[67] Cheng ZK, Gu MH. Karyotype analysis for pachytene chromosome of indica, Japonica Rice and their hybrid., 1994, 21(5): 385–392.程祝宽, 顾铭洪. 籼、粳稻及其杂种粗线期的核型分析. 遗传学报, 1994, 21(5): 385–392.
[68] Cheng Z, Yan H, Yu H, Tang S, Jiang J, Gu M, Zhu L. Development and applications of a complete set of rice telotrisomics., 2001, 157(1): 361–368.
[69] Tang XM, Bao WD, Zhang WL, Cheng ZK. Identification of chromosomes from multiple rice genomes using a universal molecular cytogenetic marker system., 2007, 49(6): 953–960.
[70] Liu XY, Sun S, Wu Y, Zhou Y, Gu SW, Yu HX, Yi CD, Gu MH, Jiang JM, Liu B, Zhang T, Gong ZY. Dual- color oligo-FISH can reveal chromosomal variations and evolution in., 2020, 101(1): 112–121.
[71] Cheng Z, Buell CR, Wing RA, Gu M, Jiang J. Toward a cytological characterization of the rice genome., 2001, 11(12): 2133–2141.
[72] Nakayama S. Molecular cytological diversity in cultivated ricesubspeciesand., 2005, 55: 425–430.
[73] Ohtsubo H, Umeda M, OhtsuboOrganization of DNA sequences highly repeated in tandem in rice genomes., 1991, 66(3): 241–254.
[74] Fan CZ, Zhang Y, Yu Y, Rounsley S, Long MY, Wing RA. The subtelomere ofchromosome 3 short arm as a hot bed of new gene origination in rice., 2008, 1(5): 839–850.
[75] Jiang JM, Birchler JA, Parrott WA, Dawe RK. A molecular view of plant centromeres., 2003, 8(12): 570–575.
[76] Dong F, Miller JT, Jackson SA, Wang GL, Ronald PC, Jiang J. Rice () centromeric regions consist of complex DNA., 1998, 95(14): 8135–8140.
[77] Cheng ZK, Dong FG, Langdon T, Ouyang S, Buell CR, Gu MH, Blattner FR, Jiang JM. Functional rice centromeres are marked by a satellite repeat and a centromere-specific retrotransposon., 2002, 14(8): 1691–1704.
[78] Wu ZG, Fang DM, Yang R, Gao F, An XY, Zhuo XX, Li YF, Yi CD, Zhang T, Liang CZ, Cui P, Cheng ZK, Luo Q. De novo genome assembly ofreveals rapid genome expansion and adaptive evolution., 2018, 1: 84.
[79] Gao DY, Gill N, Kim HR, Walling JG, Zhang WL, Fan CZ, Yu Y, Ma JX, SanMiguel P, Jiang N, Cheng ZK, Wing RA, Jiang JM, Jackson SA. A lineage-specific centromere retrotransposon in., 2009, 60(5): 820–831.
[80] Gong ZY, Xue C, Liu XX, Zhang ML, Zhou Y, Yu HX, Gu MH. Centromere inactivation in a dicentric rice chromosome during sexual reproduction., 2013, 58(36): 4602–4607.
[81] Gong ZY, Yu HX, Huang J, Yi CD, Gu MH. Unstable transmission of rice chromosomes without functional centromeric repeats in asexual propagation., 2009, 17(7): 863–872.
[82] Zhang H, Dawe RK. Total centromere size and genome size are strongly correlated in ten grass species., 2012, 20(4): 403–412.
[83] Yan HH, Kikuchi S, Neumann P, Zhang WL, Wu YF, Chen F, Jiang JM. Genome-wide mapping of cytosine methylation revealed dynamic DNA methylation patterns associated with genes and centromeres in rice., 2010, 63(3): 353–365.
[84] Wu YF, Kikuchi S, Yan HH, Zhang WL, Rosenbaum H, Iniguez AL, Jiang JM. Euchromatic subdomains in rice centromeres are associated with genes and transcription., 2011, 23(11): 4054–4064.
[85] He RF, Wang YY, Shi ZY, Ren X, Zhu LL, Weng QM, He GC. Construction of a genomic library of wild rice and agrobacterium-mediated transformation of large insert DNA linked to BPH resistance locus., 2003, 321: 113–121.
[86] Tan GX, Ren X, Weng QM, Shi ZY, Zhu LL, He GC. Mapping of a new resistance gene to bacterial blight in rice line introgressed fromofficinalis., 2004, 31(7): 724–729.
[87] Huang Z, He G, Shu L, Li X, Zhang Q. Identification and mapping of two brown planthopper resistance genes in rice., 2001, 102: 929–934.
[88] Yan HH, Xiong ZM, Min SK, Hu HY, Zhang ZT, Tian SL, Fu Q. The production and cytogenetical studies of-amphiploid., 1997, 24(1): 30–35.颜辉煌, 熊振民, 闵绍楷, 胡慧英, 张志涛, 田淑兰, 傅强. 栽培稻–紧穗野生稻双二倍体的产生及其细胞遗传学研究. 遗传学报, 1997, 24(1): 30–35.
[89] Sebastian P, Schaefer H, Telford IRH, Renner SS. Cucumber () and melon (C. melo) have numerous wild relatives in Asia and Australia, and the sister species of melon is from Australia., 2010, 107(32): 14269–14273.
[90] Chen JF, Staub JE, Jiang JM. A reevaluation of karyotype in cucumber (L.)., 1998, 45(4): 301–305.
[91] Qian CT, Chen JF, Luo XD, Cao QH, Lei C. C-banding of prometaphase chromosomes in Cucumber and the procedure for slide preparation., 2004, 31(6): 814–816.钱春桃, 陈劲枫, 罗向东, 曹清河, 雷春. 黄瓜前中期染色体C-分带及其制备方法研究. 园艺学报, 2004, 31(6): 814–816.
[92] Zhang YX, Cheng CY, Li J, Yang SQ, Wang YZ, Li ZA, Chen JF, Lou QF. Chromosomal structures and repetitive sequences divergence inspecies revealed by comparative cytogenetic mapping., 2015, 16(1): 730.
[93] Wang YZ, Zhang ZT, Jia L, Li ZA, Li J, Lou QF, Chen JF. Molecular and cytogenetic analyses provide evidence of the introgression of chromosomal segments from the wildhystrix into the cultivated cucumber through the bridge of a synthetic allotetraploid., 2017, 37(7): 89.
[94] Chen JF, Stau JE, Adelberg JW, Jiang JM. Physical mapping of 45S rRNA genes inspecies by fluorescence in situ hybridization., 1999, 77: 389–393.
[95] Zhang ZT, Yang SQ, Li ZA, Zhang YX, Wang YZ, Cheng CY, Li J, Chen JF, Lou QF. Comparative chromosomal localization of 45S and 5S rDNAs and implications for genome evolution in Cucumis., 2016, 59(7): 449–457.
[96] Lou QF, Zhang YX, He YH, Li J, Jia L, Cheng CY, Guan W, Yang SQ, Chen JF. Single-copy gene-based chromosome painting in cucumber and its application for chromosome rearrangement analysis in Cucumis., 2014, 78(1): 169–179.
[97] Li ZA, Bi YF, Wang X, Wang YZ, Yang SQ, Zhang ZT, Chen JF, Lou QF. Chromosome identification in Cucumis anguria revealed by cross-species single-copy gene FISH., 2018, 61(6): 397–404.
[98] Bi YF, Zhao QZ, Yan WK, Li MX, Liu YX, Cheng CY, Lu Z, Yu XQ, Li J, Qian CT, Wu YF, Chen JF, Lou QF. Flexible chromosome painting based on multiplex PCR of oligonucleotides and its application for comparative chromosome analyses in Cucumis., 2020, 102(1): 178–186.
[99] Zhao QZ, Wang YZ, Bi YF, Zhai YF, Yu XQ, Cheng CY, Wang PQ, Li J, Lou QF, Chen JF. Oligo-painting and GISH reveal meiotic chromosome biases and increased meiotic stability in synthetic allotetraploid Cucumis ×hytivus with dysploid parental karyotypes., 2019, 19(1): 471.
[100] Yang SQ, Cheng CY, Qin XD, Yu XQ, Lou QF, Li J, Qian CT, Chen JF. Comparative cyto-molecular analysis of repetitive DNA provides insights into the differential genome structure and evolution of five Cucumis species., 2019, 5(5): 192–204.
[101] Han YH, Zhang ZH, Liu CX, Liu JH, Huang SW, Jiang JM, Jin WW. Centromere repositioning in cucurbit species: implication of the genomic impact from centromere activation and inactivation., 2009, 106(35): 14937–14941.
[102] Yang LM, Koo DH, Li YH, Zhang XJ, Luan FS, Havey MJ, Jiang JM, Weng YQ. Chromosome rearrangements during domestication of cucumber as revealed by high- density genetic mapping and draft genome assembly., 2012, 71(6): 895–906.
[103] Cao QH, Chen JF, Qian CT. Identification and characterization of a Cucumber alien translocation lineCT201 possessing resistance to downy mildew., 2005, 32(6): 1098–1101.曹清河, 陈劲枫, 钱春桃. 黄瓜抗霜霉病异源易位系CT201的筛选与鉴定. 园艺学报, 2005, 32(6): 1098– 1101.
[104] Chen JF, Lin MS, Qian CT, Zhuang FY, Stephen L. Identification of Meloidogyne incognita (kofoid & white) chitwood resistance in CChakr. and the progenies of its interspecific hybrid with cucumber (L)., 2001,1: 21–24.陈劲枫, 林茂松, 钱春桃, 庄飞云, Stephen Lewis. 甜瓜属野生种及其与黄瓜种间杂交后代抗根结线虫初步研究. 南京农业大学学报, 2001, 1: 21–24.
[105] Chen JF, Adelberg J. Interspecific hybridization in Cucumis-progress, problems, and perspectives., 2000, 35(1): 11–15.
[106] Chen J, Staub J, Qian C, Jiang J, Luo X, Zhuang F. Reproduction and cytogenetic characterization of interspecific hybrids derived fromChakr. ×L., 2003, 106(4): 688–695.
[107] Chen JF, Luo XD, Staub JE, Jahn MM, Qian CT, Zhuang FY, Ren G. An allotriploid derived from a amphidiploid × diploid., 2003, 131(2): 235–241.
[108] Chen JF, Luo XD, Qian CT, Jahn MM, Staub JE, Zhuang FY, Lou QF, Ren G. Cucumis monosomic alien addition lines: morphological, cytological, and genotypic analyses., 2004, 108(7): 1343–1348.
[109] Li MX, Zhao QZ, Liu YX, Qin XD, Hu W, Davoudi M, Chen JF, Lou QF. Development of alien addition lines from Cucumis hystrix in Cucumis sativus: cytological and molecular marker analyses., 2020, 63(12): 629–641.
[110] Guan B, Wang K, Zhou BL, Guo WZ, Zhang TZ. Establishment of a multi-color genomic in situ hybridization technique to simultaneously discriminate the three interspecific hybrid genomes in Gossypium., 2008, 50(3): 345–351.
[111] Wang K, Guo WZ, Zhang TZ. Development of one set of chromosome-specific microsatellite-containing BACs and their physical mapping in Gossypium hirsutum L., 2007, 115(5): 675–682.
[112] Wang K, Guan B, Guo WZ, Zhou BL, Hu Y, Zhu YC, Zhang TZ. Completely distinguishing individual A-genome chromosomes and their karyotyping analysis by multiple bacterial artificial chromosome-fluorescence in situ hybridization., 2008, 178(2): 1117– 1122.
[113] Wang K, Song XL, Han ZG, Guo WZ, Yu JZ, Sun J, Pan JJ, Kohel RJ, Zhang TZ. Complete assignment of the chromosomes of Gossypium hirsutum L. by translocation and fluorescence in situ hybridization mapping., 2006, 113(1): 73–80.
[114] Wang KB, Wang ZW, Li FG, Ye WW, Wang JY, Song GL, Yue Z, Cong L, Shang HH, Zhu SL, Zou CS, Li Q, Yuan YL, Lu CR, Wei HL, Gou CY, Zheng ZQ, Yin Y, Zhang XY, Liu K, Wang B, Song C, Shi N, Kohel RJ, Percy RG, Yu JZ, Zhu YX, Wang J, Yu SX. The draft genome of a diploid cotton Gossypium raimondii., 2012, 44(10): 1098–1103.
[115] Paterson AH, Wendel JF, Gundlach H, Guo H, Jenkins J, Jin DC, Llewellyn D, Showmaker KC, Shu SQ, Udall J, Yoo MJ, Byers R, Chen W, Doron-Faigenboim A, Duke MV, Gong L, Grimwood J, Grover C, Grupp K, Hu GJ, Lee TH, Li JP, Lin LF, Liu T, Marler BS, Page JT, Roberts AW, Romanel E, Sanders WS, Szadkowski E, Tan X , Tang HB, Xu CM, Wang JP, Wang ZN, Zhang D, Zhang L, Ashrafi H, Bedon F, Bowers JE, Brubaker CL, Chee PW, Das S, Gingle AR, Haigler CH, Harker D, Hoffmann LV, Hovav R, Jones DC, Lemke C, Mansoor S, ur Rahman M , Rainville LN, Rambani Ai, Reddy UK, Rong JK, Saranga Y, Scheffler BE, Scheffler JA, Stelly DM, Triplett BA, Van Deynze A, Vaslin MFS, Waghmare VN, Walford SA, Wright RJ, Zaki EA, Zhang TZ, Dennis ES, Mayer KFX, Peterson DG, Rokhsar DS, Wang XY, Schmutz J. Repeated polyploidization of Gossypium genomes and the evolution of spinnable cotton fibres., 2012, 492(7429): 423–427.
[116] Li FG, Fan GY, Wang KB, Sun FM, Yuan YL, Song GL, Li Q, Ma ZY, Lu CR, Zou CS, Chen WB, Liang XM, Shang HH, Liu WQ, Shi CC, Xiao GH, Gou CY, Ye WW, Xu X, Zhang XY, Wei HL, Li ZF, Zhang GY, Wang JY, Liu K, Kohel RJ, Percy RG, Yu JZ, Zhu YX, Yu SX. Genome sequence of the cultivated cotton Gossypium arboreum., 2014, 46(6): 567–572.
[117] Zhang TZ, Hu Y, Jiang WK, Fang L, Guan XY, Chen JD, Zhang JB, Saski CA, Scheffler BE, Stelly DM, Hulse-Kemp AM, Wan Q, Liu BL, Liu CX, Wang S, Pan MQ, Wang YK, Wang DW, Ye WX, Chang LJ, Zhang WP, Song QX, Kirkbride RC, Chen XY, Dennis E, Llewellyn DJ, Peterson DG, Thaxton P, Jones DC, Wang Q, Xu XY, Zhang H, Wu HT, Zhou L, Mei GF, Chen SQ, Tian Y, Xiang D, Li XH, Ding J, Zuo QY, Tao LN, Liu YC, Li J, Lin Y, Hui YY, Cao ZS, Cai CP, Zhu XF, Jiang Z, Zhou BL, Guo WZ, Li RQ, Chen ZJ. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement., 2015, 33(5): 531–537.
[118] Li FG, Fan GY, Lu CR, Xiao GH, Zou CS, Kohel RJ, Ma ZY, Shang HH, Ma XF, Wu JY, Liang XM, Huang G, Percy RG, Liu K, Yang WH, Chen WB, Du XG, Shi CC, Yuan YL, Ye WW, Liu X, Zhang XY, Liu WQ, Wei HL, Wei SJ, Huang GD, Zhang XL, Zhu SJ, Zhang H, Sun FM, Wang XF, Liang J, Wang JH, He Q, Huang LH, Wang J, Cui JJ, Song GL, Wang KB, Xu X, Yu JZ, Zhu YX, Yu SX. Genome sequence of cultivated upland cotton (Gossypium hirsutum TM-1) provides insights into genome evolution., 2015, 33(5): 524–530.
[119] Cao YJ. Studies of centromere-specific sequences in cotton[Dissertation]. Nanjing Agricultural University, 2013.曹玉洁. 棉花着丝粒区特异序列的发掘与分析[学位论文]. 南京农业大学, 2013.
[120] Zhang WP, Cao YJ, Wang K, Zhao T, Chen JD, Pan MQ, Wang Q, Feng SL, Guo WZ, Zhou BL, Zhang TZ. Identification of centromeric regions on the linkage map of cotton using centromere-related repeats., 2014, 104 (6 Pt B): 587–593.
[121] Zhang ZX. The isolation of telomeric DNA sequencesand[Dissertation]. Nanjing Agricultural University, 2013.张忠鑫. 亚洲棉及雷蒙德氏棉端粒DNA序列的发掘与分析[学位论文]. 南京农业大学, 2013.
[122] Chen Y, Wang YY, Zhao T, Yang JW, Feng SL, Nazeer W, Zhang TZ, Zhou BL. A new synthetic amphiploid (AADDAA) betweenandlays the foundation for transferring resistances to verticillium and drought., 2015, 10(6): e0128981.
[123] Chen Yu, Wang YY, Wang K, Zhu XF, Guo WZ, Zhang TZ, Zhou BL. Construction of a complete set of alien chromosome addition lines fromin: morphological, cytological, and genotypic characterization., 2014, 127(5): 1105–1121.
[124] Wang YY, Feng SL, Li S, Tang D, Chen Y, Chen Y, Zhou BL. Inducement and identification of chromosome introgression and translocation ofon., 2018, 19(1): 15.
[125] Wang XX, Wang YY, Wang C, Chen Y, Chen Y, Feng SL, Zhao T, Zhou BL. Characterization of eleven monosomic alien addition lines added fromtousing improved GISH and SSR markers., 2016, 16(1): 218.
[126] Wang C. Development of alien addition lines of-and evaluation of its excellent characters[Dissertation]. Nanjing Agricultural University, 2018.王琛. 陆地棉–异常棉异附加系创制与优异性状鉴定[学位论文]. 南京农业大学, 2018.
[127] Tang D, Feng SL, Li S, Chen Y, Zhou BL. Ten alien chromosome additions ofdeveloped by integrative uses of GISH and species-specific SSR markers., 2018, 293(4): 945–955.
[128] Tanaka R. On the speciation and karyotypes in diploid and tetraploid species of Chrysanthemum., 1960, 25(1): 43–58.
[129] Fedorov AV, Fedorov AA, Fedorov YA. Chromosome numbers of flowering plants. West Genabt: Otto Koeltz Science Publishers, 1974.
[130] Li MX, Zhang XF, Chen JY. Cytological studies on some Chinese wild dendranthema species and chrysanthemum cultivars., 1983, (3): 199–206.李懋学, 张斅方, 陈俊愉. 我国某些野生和栽培菊花的细胞学研究. 园艺学报, 1983, (3): 199–206.
[131] Du BQ, Liu QH, Zhu CY, Ke SQ. Karyotype studies of two species on dendranthema., 1989, (3): 293–296.杜冰群, 刘启宏, 朱翠英, 柯善强. 两种菊属植物的核型研究. 武汉植物学研究, 1989, (3): 293–296.
[132] Wang JW, Yang J, Li MX. Karyotype study of five species of chinese dendranthema., 1991, (4): 411–416.汪劲武, 杨继, 李懋学. 国产五种菊属植物的核型研究. 云南植物研究, 1991, (4): 411–416.
[133] Wang JW, Yang J, Li MX. The morphological variation and the karyotypical characters ofand., 1993, 31(2): 140–146.汪劲武, 杨继, 李懋学. 野菊和甘菊的形态变异及其核型特征. 植物分类学报, 1993, 31(2): 140–146.
[134] Endo N. The chromosome survey on the cultivated chrysanthemums,Ram., 1969, 38(3): 267–274.
[135] Endo M. On the occurrence of B chromosome in the garden chrysanthemum,Ramat., 1990, 59(3): 613–620.
[136] Chen FD, Zhao HB, Li C, Chen SM, Fang WM. Advances in cytology and molecular cytogenetics of the genus Dendranthema., 2008, 31(1): 118–126.陈发棣, 赵宏波, 李畅, 陈素梅, 房伟民. 菊属植物细胞学与分子细胞遗传学研究进展. 南京农业大学学报, 2008, 31(1): 118–126.
[137] Kim JS, Pak JH, Seo BB, Tobe H. Karyotypes of metaphase chromosomes in diploid populations of Dendranthema zawadskii and related species () from Korea: diversity and evolutionary implications., 2003, 116(1): 47–54.
[138] Li C, Chen FD, Zhao HB, Chen SM. Karyotype diversity of 17 chrysanthemum cultivars with small inflorescences., 2008, 35(1): 71–80.李畅, 陈发棣, 赵宏波, 陈素梅. 栽培小菊17个品种的核型多样性. 园艺学报, 2008, 35(1): 71–80.
[139] Tang FP, Chen FD, Chen SM, Teng NJ, Fang WM. Intergeneric hybridization and relationship of genera within the tribe Anthemideae Cass. (I.(Kitam.) Kitam. ×(L.) Makino)., 2009, 169: 133–140.
[140] Deng YM, Chen SM, Lu AM, Chen FD, Tang FP, Guan ZY, Teng NJ. Production and characterisation of the intergeneric hybrids betweenandexhibiting enhanced resistance to()., 2010, 231(3): 693–703.
[141] Qi XY, Zhang F, Guan ZY, Wang HB, Jiang JF, Chen SM, Chen FD. Localization of 45S and 5S rDNA sites and karyotype ofand its related genera by fluorescenthybridization., 2015, 62: 164–172.
[142] Chen JY. Studies on the origin of Chinese florist’s chrysanthemum., 1985, 167: 349–361.
[143] Dai SL, Wang WK, Huang JP. Advances of researches on phylogeny ofand origin of chrysanthemum., 2002, 24(5/6): 230–234.戴思兰, 王文奎, 黄家平. 菊属系统学及菊花起源的研究进展. 北京林业大学学报, 2002, 24(5/6): 230– 234.
[144] Chen FD, Chen PD, Li HJ. Genome analysis and their phylogenetic relationships of several wild species ofin China., 1996, 23(1): 67–72.陈发棣, 陈佩度, 李鸿渐. 几种中国野生菊的染色体组分析及亲缘关系初步研究. 园艺学报, 1996, 23(1): 67–72.
[145] Chen FD, Chen PD, Fang WM, Li HJ. Cytological studies of three varieties of medicinal chrysanthemum., 1997, 20(2): 28–31.陈发棣, 陈佩度, 房伟民, 李鸿渐. 3个药用菊品种的细胞学研究. 南京农业大学学报, 1997, 20(2): 28–31.
[146] Chen FD, Chen XL, Fang WM. On cytology of three geocotypes of., 1998, 693–696.陈发棣, 陈秀兰, 房伟民. 3个地理居群野菊的细胞学研究. 园艺学进展(第2辑), 1998, 693–696.
[147] Li XL, Chen FD. RAPD analysis of wild species, cultivars and interspecific hybrids in., 2004, 27(3): 29–33.李辛雷, 陈发棣. 菊属野生种、栽培菊花及种间杂种的RAPD分析. 南京农业大学学报, 2004, 27(3): 29–33.
[148] Cui NX. Studies on meiosis behaviors and genetic relationships of severalspecies and their hybrids[Dissertation]. Nanjing Agricultural University, 2004.崔娜欣. 几种菊属植物及其杂种减数分裂行为与亲缘关系研究[学位论文]. 南京农业大学, 2004.
[149] Chen JY. The origin of garden chrysanthemum. Hefei: Anhui Science and Technology Press. 2012, 67–74.陈俊愉. 菊花起源. 合肥: 安徽科学技术出版社. 2012, 67–74.
[150] Song C, Liu YF, Song AP, Dong, GQ, Zhao HB, Sun W, Ramakrishnan S, Wang Y, Wang SB, Li TZ, Niu Y, Jiang JF, Dong B, Xia Y, Chen SM, Hu ZG, Chen FD, Chen SL. Thegenome provides insights into the evolution and diversification of chrysanthemum flowers and medicinal traits., 2018, 11(12): 1482–1491.
[151] Hirakawa H, Sumitomo K, Hisamatsu T, Nagano S, Shirasawa K, Higuchi Y, Kusaba M, Koshioka M, Nakano Y, Yagi M, Yamaguchi H, Taniguchi K, Nakano M, Isobe SN. De novo whole-genome assembly in, a model species of, and its application to genetic and gene discovery analysis., 2019, 26(3): 195–203.
[152] Lan W, Chen SM, Yin DM, Chen FD. Studies on in vitro conservation of., 2010, 37(12): 2007–2016.兰伟, 陈素梅, 尹冬梅, 陈发棣. 那贺川野菊的离体保存. 园艺学报, 2010, 37(12): 2007–2016.
[153] Xu Y, Chen FD. The LT50 and cold tolerance adaptability ofduring a natural drop in temperature., 2008, 35 (4): 559–564.许瑛, 陈发棣. 菊花8个品种的低温半致死温度及其抗寒适应性. 园艺学报, 2008, 35 (4): 559–564.
[154] Li N, Fang WM, Chen FD, Chen SM, Chen Y. Physiological indexes in florets of two winter cut chrysanthemum cultivars under low temperature and their cold tolerance., 2010, 30(4): 741–746.李娜, 房伟民, 陈发棣, 陈素梅, 陈煜. 切花寒菊小花对低温胁迫的生理响应及其抗寒性分析. 西北植物学报, 2010, 30(4): 741–746.
[155] He JP, Chen FD, Chen SM, Fang WM. Aphid-resistance of chrysanthemum cultivars., 2010, 29(7): 1382–1386.何俊平, 陈发棣, 陈素梅, 房伟民. 不同菊花品种抗蚜虫性鉴定. 生态学杂志, 2010, 29(7): 1382–1386.
[156] Sun Y. Studies on aphid resistance and mechanisms in chrysanthemum and it’s related species at seedling stage [Dissertation]. Nanjing Agricultural University, 2011.孙娅. 菊花近缘种属植物苗期抗蚜虫性鉴定与抗蚜机理研究[学位论文]. 南京农业大学, 2011.
[157] Zhang FJ, Wang ZQ, Dong W, Sun CQ, Wang HB, Song AP, He LZ, Fang WM, Chen FD, Teng NJ. Transcriptomic and proteomic analysis reveals mechanisms of embryo abortion during chrysanthemum cross breeding., 2014, 4: 6536.
[158] Deng YM, Chen SM, Chen FD, Cheng X, Zhang F. The embryo rescue derived intergeneric hybrid between chrysanthemum andshows enhanced cold tolerance., 2011, 30(12): 2177– 2186.
[159] Cheng X, Chen SM, Chen FD, Fang WM, Deng YM, She LF. Interspecific hybrids between(Ramat.) Kitamura and(Nakai) Tzvel. achieved using ovary rescue and their cold tolerance characteristics., 2010, 172: 101–108.
[160] Deng YM, Jiang JF, Chen SM, Huang CB, Fang WM, Chen FD. Drought tolerance of intergeneric hybrids betweenand., 2012, 148(4): 17–22.
[161] Sun CQ, Chen FD, Teng NJ, Liu ZL, Fang WM, Hou XL. Interspecific hybrids between(Ramat.) Kitamura and(L.) Des Moul. and their drought tolerance evaluation., 2010, 174: 51–60.
[162] Tang FP, Wang HB, Chen SM, Chen FD, Teng NJ, Liu ZL. First intergeneric hybrids within the tribe Anthemideae Cass.III.L. Des Moul. ×(Ling) Shih., 2012, 43: 87–92.
[163] Zhu WY, Jiang JF, Chen SM, Wang L, Xu LL, Wang HB, Li PL, Guan ZY, Chen FD. Intergeneric hybrid between×andachieved via embryo rescue shows salt tolerance., 2013, 191: 109–119.
[164] Zhu WY, Zhang F, Chen SM, Xu LL, Wang L, Wang HB, Qi XY, Li HY, Chen FD. Intergeneric hybrids between‘Nannongxiaoli’ and‘Variegata’ show enhanced resistance against both aphids and alternaria leaf spot., 2014, 197: 399–408.
[165] Chen FD, Chen SM, Fang WM, Zhang F, Jiang JF, Teng NJ, Guan ZY, Wang HB, Song AP, Zhao S. Discovery of excellent chrysanthemum germplasms and germplasm enhancement., 2016, 30(2): 112–115.陈发棣, 陈素梅, 房伟民, 张飞, 蒋甲福, 滕年军, 管志勇, 王海滨, 宋爱萍, 赵爽. 菊花优异种质资源挖掘与种质创新研究. 中国科学基金, 2016, 30(2): 112–115.
[166] Bradshaw HD, Ceulemans R, Davis J, Stettler R. Emerging model systems in plant biology: poplar (Populus) as a model forest tree., 2000, 19(3): 306–313.
[167] Zhang Y, Zhang SG, Qi LW, Chen XQ, Chen RY, Song WQ. Poplar as a model for forest tree in genome research., 2006, 23(3): 286–293.张勇, 张守攻, 齐力旺, 陈小强, 陈瑞阳, 宋文芹. 杨树——林木基因组学研究的模式物种. 植物学报, 2006, 23(3): 286–293.
[168] Tuskan GA, Difazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A, Schein J, Sterck L, Aerts A, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W, Brun A, Brunner A, Busov V, Campbell M, Carlson J, Chalot M, Chapman J, Chen GL, Cooper D, Coutinho PM, Couturier J, Covert S, Cronk Q, Cunningham R, Davis J, Degroeve S, Déjardin A, Depamphilis C, Detter J, Dirks B, Dubchak I, Duplessis S, Ehlting J, Ellis B, Gendler K, Goodstein D, Gribskov M, Grimwood J, Groover A, Gunter L, Hamberger B, Heinze B, Helariutta Y, Henrissat B, Holligan D, Holt R, Huang W, Islam-Faridi N, Jones S, Jones-Rhoades M, Jorgensen R, Joshi C, Kangasjärvi J, Karlsson J, Kelleher C, Kirkpatrick R, Kirst M, Kohler A, Kalluri U, Larimer F, Leebens-Mack J, Leplé JC, Locascio P, Lou Y, Lucas S, Martin F, Montanini B, Napoli C, Nelson DR, Nelson C, Nieminen K, Nilsson O, Pereda V, Peter G, Philippe R, Pilate G, Poliakov A, Razumovskaya J, Richardson P, Rinaldi C, Ritland K, Rouzé P, Ryaboy D, Schmutz J, Schrader J, Segerman B, Shin H, Siddiqui A, Sterky F, Terry A, Tsai CJ, Uberbacher E, Unneberg P, Vahala J, Wall K, Wessler S, Yang G, Yin T, Douglas C, Marra M, Sandberg G, Van de Peer Y, Rokhsar D. The genome of black cottonwood,(Torr. & Gray)., 2006, 313(5793): 1596–1604.
[169] Yin TM. A review of the genomic and gene cloning studies in trees., 2010, 32(7): 677– 684.尹佟明. 林木基因组及功能基因克隆研究概述. 遗传, 2010, 32(7): 677–684.
[170] Ma T, Wang JY, Zhou GK, Yue Z, Hu QJ, Chen Y, Liu BB, Qiu Q, Wang Z, Zhang J, Wang K, Jiang DC, Gou CY, Yu LL, Zhan DL, Zhou R, Luo WC, Ma H, Yang YZ, Pan SK, Fang DM, Luo YD, Wang X, Wang GN, Wang, J, Wang Q, Lu X, Chen Z, Liu JC, Lu Y, Yin Y, Yang HM, Abbott RJ, Wu YX, Wan DS, Li J, Yin TM, Lascoux M, Difazio SP, Tuskan GA, Wang J, Liu JQ. Genomic insights into salt adaptation in a desert poplar., 2013, 4: 2797.
[171] Ma JC, Wan DS, Duan BB, Bai XT, Bai QX, Chen NN, Ma T. Genome sequence and genetic transformation of a widely distributed and cultivated poplar., 2019, 17(2): 451–460.
[172] Ribeiro T, Barão A, Viegas W, Morais-Cecíli L. Molecular cytogenetics of forest trees., 2008, 120(3–4): 220–227.
[173] Islam-Faridi MN, Nelson CD, Difazio SP, Gunter LE, Tuskan GA. Cytogenetic analysis of Populus trichocarpa--ribosomal DNA, telomere repeat sequence, and marker-selected BACs., 2009, 125(1): 74–80.
[174] Blackburn KB, Harrison JWH. A preliminary account of the chromosomes and chromosome behaviour in the Salicaceae., 1924, 38(2): 361–378.
[175] Einspahr DW, Van Buijtenen JP, Peckham JR. Natural variation and heritability in triploid aspe., 1963, 12(2): 51–58.
[176] Kang XY, Zhu ZT, Zhang ZY. Cytogenetic studies on the origin of chinese white poplar., 1999, 21(6): 6–10.康向阳, 朱之悌, 张志毅. 毛白杨起源的细胞遗传学研究. 北京林业大学学报, 1999, 21(6): 6–10.
[177] Xin HY, Wang S, Liu GX, Zhen Y, Shi JS, Kuang HL, Xi ML. Relationship between the meiosis processes of microsporocytes and morphology of male flower buds and anthers in Populus deltoides Marsh., 2016, (2): 48–52.辛昊阳, 王帅, 刘光欣, 甄艳, 施季森, 匡华琳, 席梦利. 美洲黑杨小孢子母细胞减数分裂进程与花芽及花药外观形态相关性研究. 南京林业大学学报(自然科学版), 2016, (2): 48–52.
[178] Lan Y, Xin HY, Zhao YL, Sun ZQ, Dai X, Xie ZW, Xi ML. Study on meiosis of microspore mother cells and germination rate of pollen grains in Populus simonii Carr., 2017, (6): 177–180.兰月, 辛昊阳, 赵乙琏, 孙志强, 戴筱, 解志伟, 席梦利. 小叶杨花粉母细胞减数分裂及花粉萌发力观测. 南京林业大学学报(自然科学版), 2017, (6): 177–180.
[179] Dong FP, Han SY, Zhang SG, Qi LW, Liu B, Li XL, Chen CB. Physical mapping of 25S rDNA on metaphase chromosomes of(Salicaceae) in five sections by fluorescence in situ Hybridization., 2007, 29(4): 423–428.董凤平, 韩素英, 张守攻, 齐力旺, 刘博, 李秀兰, 陈成彬. 25S rDNA在杨属植物染色体上的定位. 云南植物研究, 2007, 29(4): 423–428.
[180] Hu BQ, Dong FP, Wang CG, Qi LW, Song WQ, Chen CB. Multicolor fluorescence in situ Hybridization of sevenspecies-ribosomal DNA and telomere repeat sequence., 2012, 45(1): 58–64.胡宝全, 董凤平, 王春国, 齐力旺, 宋文芹, 陈成彬. 五派杨树多色荧光原位杂交分析(英文). 南开大学学报(自然科学版), 2012, 45(1): 58–64.
[181] Xin HY, Zhang T, Wu YF, Zhang WL, Zhang PD, Xi ML, Jiang JM. An extraordinarily stable karyotype of the woodyspecies revealed by chromosome painting., 2020, 101(2): 253–264.
[182] Xin HY, Zhang T, Han YH, Wu YF, Shi JS, Xi ML, Jiang JM. Chromosome painting and comparative physical mapping of the sex chromosomes inand., 2018, 127(3): 313–321.
[183] Nakajima G. Chromosome numbers in some crops and wild angiosperms., 1931, 12: 211–217.
[184] Liang GL, Xiang SQ, Wang WX, Yan Y, Li XL, Pei Y, Zhang DP. Karyotype analysis of the wild(2x, 6x) complex closely related to the sweet potato., 2001, 1: 404–406.梁国鲁, 向素琼, 汪卫星, 闫勇, 李晓林, 裴炎, 张大鹏. 甘薯近缘野生种Ipomoea trifida (2X、6X)的核型分析. Advances in Chromosome Sciences, 2001, 1: 404–406.
[185] Cao QH, Ma DF, Zhang A. Chromosome karyotype and pollen ultrastructure of a related species of sweet potato., 2008, 28(8): 1610–1613.曹清河, 马代夫, 张安. 甘薯近缘种染色体核型及花粉粒超微结构分析. 西北植物学报, 2008, 28(8): 1610–1613.
[186] Tang JL, Qi DS, Zhang Y, Liu HJ, Sun JY, Cao QH, Ma DF, Li ZY. FISH analysis of chromosomes of sweet potato (cv. Xushu No.18)., 2010, 32(2): 177–182.汤佳立, 戚大石, 张俞, 刘慧娟, 孙健英, 曹清河, 马代夫, 李宗芸. 荧光原位杂交技术分析栽培种甘薯(Ipomoea batatas cv. Xushuno.18)染色体. 遗传, 2010, 32(2): 177–182.
[187] An TT, Tang JL, Sun JY, Cao QH, Ma DF, Li ZY. rDNA-FISH analysis and DAPI-karyotype ofcv.andJacq., 2012, 32(4): 682–687.安婷婷, 汤佳立, 孙健英, 曹清河, 马代夫, 李宗芸. 甘薯栽培种及其近缘野生种的DAPI核型及rDNA- FISH分析. 西北植物学报, 2012, 32(4): 682–687.
[188] Xiang SQ, Wang WX, Li XL, Chen Y, Guo QG, He Q, Liang GL. GISH analysis of sweet potato wild relative(4x)., 2008, 34(2): 341–343.向素琼, 汪卫星, 李晓林, 陈瑶, 郭启高, 何桥, 梁国鲁. 甘薯近缘野生种Ipomoea trifida (4x) GISH分析. 作物学报, 2008, 34(2): 341–343.
[189] Sun JY, Yu LX, Cai ZX, Zhang A, Jin WW, Han YH, Li ZY. Comparative karyotype analysis among sixspecies based on two newly identified repetitive sequences., 2019, 62(4): 243–252.
[190] Shen JQ. A simple technique for the determination of cross incompatibility groups in sweet potato varieties., 1984, 8: 12-13+2.沈稼青. 甘薯品种杂交不亲和群的简易测定技术, 农业科技通讯, 1984, 8: 12-13+2.
[191] Li ZY, Chen FY, Jiang JJ, Yu LX, Sun JY, Han YH. Cytogenetics research progress of sweet potato., 2016, 35(4): 34–39.李宗芸, 陈孚尧, 蒋姣姣, 俞立璇, 孙健英, 韩永华. 甘薯细胞遗传学研究进展. 中南民族大学学报(自然科学版), 2016, 35(4): 34–39.
[192] Cao QH, Zhang A, Ma DF, Li HM, Li Q, Li P. Novel interspecific hybridization between sweetpotato ((L.) Lam.) and its two diploid wild relatives., 2009, 169(3): 345–352.
[193] Cao QH, Tang J, Li A, Gruneberg W, Huamani K, Ma DF. Ploidy Level and Molecular Phylogenic Relationship among Novel Ipomoea Interspecific Hybrids., 2014, 50(1): 32–38.
[194] do Vale Martins L, Yu Fan, Zhao HN, Dennison T, Lauter N, Wang HY, Deng ZH, Thompson A, Semrau K, Rouillard JM, Birchler JA, Jiang JM. Meiotic crossovers characterized by haplotype specific chromosome painting in maize., 2019, 10(1): 4064.
[195] Koo DH, Molin WT, Saski CA, Jiang JM, Putta K, Jugulam M, Friebe M, Gill BS. Extrachromosomal circular DNA-based amplification and transmission of herbicide resistance in crop weed., 2018, 115 (13): 3332–3337.
[196] Eng CHL, Lawson M, Zhu Q, Dries R, Koulena N, Takei Y, Yun J, Cronin C, Karp C, Yuan GC, Cai L. Transcriptome-scale super-resolved imaging in tissues by RNA seqFISH., 2019, 568(7751): 235–239.
[197] Battich N, Stoeger T, Pelkmans L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution., 2013, 10(11): 1127–1133.
[198] Lubeck E, Cai L. Single-cell systems biology by super-resolution imaging and combinatorial labeling., 2012, 9(7): 743–748.
[199] Feng C, Yuan J, Bai H, Liu YL, Su HD, Liu Y, Shi LD, Gao Z, Birchler JA, Han FP. The deposition of CENH3 in maize is stringently regulated., 2020, 102(1): 6–17.
Research progress of plant cytogenetics in Jiangsu province
Haiyan Wang1, Zhiyun Gong2, Jiafu Jiang1, Baoliang Zhou1, Qunfeng Lou1, Qinghe Cao3, Mengli Xi4, Peidu Chen1, Minghong Gu2, Tianzhen Zhang6, Fadi Chen1, Jinfeng Chen1, Zongyun Li5, Xiue Wang1
Cytogenetics was established based on the “Chromosome theory of inheritance”, proposed by Boveri and Sutton and evidenced by Morgan’s lab in early stage of the 20thcentrary. With rapid development of related research areas, especially molecular genetics, cytogenetics developed from traditional into a new era, molecular cytogenetics in late 1960s. Featured by an established technique named DNAhybridization (ISH), molecular cytogenetics has been applied in various research areas. ISH provids vivid and straightforward figures showing the virtual presence of DNA, RNA or proteins. In combination with genomics and cell biology tools, ISH and derived techniques have been widely used in studies of the origin, evolution, domestication of human, animal and plant, as well as wide hybridization and chromosome engineering. The physical location and order of DNA sequences revealed by ISH enables the detection of chromosomal re-arrangments among related species and gaps of assembled genome sequences. In addition, ISH using RNA or protein probes can reveal the location and quantification of transcripted RNA or translated protein. Since the 1970s, scientists from universities or institutes belonging to the Jiangsu Society of Genetics have initiated cytogenetics researches using various plant species. In recent years, research platforms for molecular cytogenetics have also been well established in Nanjing Agricultural University, Yangzhou University, Nanjing Forestry University, Jiangsu Xuhuai Academy of Agricultural Sciences, and Jiangsu Normal University. The application of molecular cytogenetics in plant evolution, wide hybridization, chromosome engineering, chromosome biology, genomics has been successful. Significant progresses have been achieved, both in basic and applied researches. In this paper, we will review main research progresses of plant cytogenetics in Jiangsu province, and discuss the potential development of this research area.
Jiangsu Genetics Society; molecular and cytogenetics; DNAhybridization; chromosome engineering; geneomics
2020-12-29;
2021-02-25
国家重点研发计划(编号:2016YFD0102001)项目,国家自然科学基金国际(地区)合作与交流项目(编号:31661143005),国家自然科学基金项目(编号:31771782)资助[supported by the National Key Research and Development Program of China (No. 2016YFD0102001), International Cooperation and Exchange Programme of the National Natural Science Foundation of China (No. 31661143005), and the National Natural Science Foundation of China (No. 31771782)
王海燕,博士,教授,研究方向:植物分子细胞遗传学。E-mail: hywang@njau.edu.cn
王秀娥,博士,教授,研究方向:小麦遗传育种。E-mail: xiuew@njau.edu.cn
10.16288/j.yczz.20-448
2021/3/19 11:02:50
URI: https://kns.cnki.net/kcms/detail/11.1913.r.20210317.1347.002.html
(责任编委: 孔令让)
