芝麻过敏原Ses i 3同源建模及B细胞线性抗原表位预测
2021-05-15马秀丽张九凯孙劲旅黄文胜韩建勋葛毅强
马秀丽,张九凯,孙劲旅,李 宏,黄文胜,韩建勋,葛毅强,4*,陈 颖*
(1 中国检验检疫科学研究院 北京100176 2 中国农业大学食品科学与营养工程学院 北京100083 3 北京协和医院变态反应科 北京100730 4 中国农村技术开发中心 北京100045)
芝麻(Sesamum indicum)是重要的油料、蛋白基料、香料化工、医药保健用品等常用加工原料,广泛添加于糕点、菜肴和饮品中。同时,芝麻也是常见的食物过敏原之一,因其致敏的发病率逐年升高,故引发了越来越多的关注[1-3]。
研究表明,芝麻主要过敏原包括:2 种2S 白蛋白Ses i 1(9~14 ku)和Ses i 2(7 ku),7S 类豌豆球蛋白Ses i 3 (45 ku),2 种油脂蛋白Sesi 4(17 ku)和Ses i 5(15 ku),2 种11S 球蛋白Ses i 6(52.2 ku)和Ses i 7(56.6 ku)[4-8],其中,芝麻7S类豌豆球蛋白中含量最丰富的是Ses i 3,它是芝麻的主要过敏原之一,能被75%以上的芝麻过敏患者血清IgE 所识别[6]。分子质量为45 ku(SDSPAGE),缺乏二硫键,属于Cupin 超家族(AF045)。由cDNA 推导的Ses i 3 前体共有585 个氨基酸[9],202~354 aa 和395~560 aa 是其2 个结构域,2 个cupin 串联排列,与β-伴大豆球蛋白cupin 结构相似。在Ara h 1 中已知的IgE 结合抗原表位(TPGQFEDFFP) 上,Ses i 3 和Ara h 1 相应的区域有高达80%的同源性,因此二者的IgE 抗体之间有交叉反应[6]。另外,Ses i 3 与该家族中的开心果、榛子、花生中的7S 豌豆球蛋白过敏原之间分别有47%,42%,36%的同源性[10]。抗原表位是过敏原分子引发食物过敏反应的免疫学物质基础,是能够参与结合抗体的重要组成部分。通过对抗原表位的预测和比对,能够进一步判断芝麻过敏原抗原表位以及与其它过敏原之间的交叉反应情况。B 细胞表位预测多是线性表位预测,是表位预测的重要环节。预测多是根据蛋白质的氨基酸序列,涉及抗原蛋白组成氨基酸的理化性质、统计显著性、结构性质等特征属性而衍生出一系列的算法进行表位预测。其中,包括亲水性、表面可及性、柔韧性、抗原指数等方案以及二级结构等预测算法。在预测过程中需要联用多种方案,以提高预测的准确性[11]。蛋白质三级结构是深入了解过敏原的一个重要方面。然而其解析仍是一个难点。目前PDB(Protein data bank)数据库是全球唯一的收录大型生物分子(蛋白质和核苷酸)3D 结构的数据库。结合目前迅速发展的生物信息学软件,如SWISS-MODEL 等,可实现对蛋白质三级结构的预测和模拟。通过生物信息学软件预测蛋白性质、结构等,有助于提高表位等免疫试验设计上的合理性[12]。本研究将分子生物学技术与生物信息学技术相结合,使用多参数软件预测芝麻过敏原Ses i 3 的抗原表位,并对其三维结构进行模拟,为深入了解芝麻过敏原,进而为芝麻过敏原的精准检测和诊断提供参考。
1 材料与方法
1.1 芝麻过敏原Ses i 3 的氨基酸序列
在Uniprot 数据库中获得Ses i 3 的氨基酸序列,登录号:Q9AUD0,共585 个氨基酸,序列如下:MSCGGRLCLVLFALLLASAVVASESKDPELKQCK HQCKAQQQISKEQKEACIQACKEYIRQKHQGEH GRGGGDILEEEVWNRKSPIERLRECSRGCEQQH GEQREECLRRCQEEYQREKGRQDDDNPTDPEKQ YQQCRLQCRRQGEGGGFSREHCERRREEKYREQ QGREGGRGEMYEGREREEEQEEQGRGRIPYVFE DQHFITGFRTQHGRMRVLQKFTDRSELLRGIENY RVAILEAEPQTFIVPNHWDAESVVFVAKGRGTISL VRQDRRESLNIKQGDILKINAGTTAYLINRDNNER LVLAKLLQPVSTPGEFELFFGAGGENPESFFKSFS DEILEAAFNTRRDRLQRIFGQQRQGVIVKASEEQ VRAMSRHEEGGIWPFGGESKGTINIYQQRPTHSN QYGQLHEVDASQYRQLRDLDLTVSLANITQGAMT APHYNSKATKIALVVDGEGYFEMACPHMSRSRG SYQGETRGRPSYQRVASRLTRGTVVIIPAGHPFVA VASSNQNLQVLCFEVNANNNEKFPLAGRRNVMN QLEREAKELAFGMPAREVEEVSRSQQEEFFFKGP RQQQQGRADA。
1.2 芝麻过敏原Ses i 3 蛋白理化性质分析
Expert Protein Analysis System (ExPASy)Proteomics tools(http://www.Expasy.ch/tools/)所提供的Prot-Param 在线软件,预测其氨基酸组成、等电点、分子质量、亲水/疏水性等基本理化性质。DNAStar 生物分析软件中的子程序Protean,其Jameson-Wolf 方案预测抗原指数,Hopp-Woods 和Kyte-Doolittle 方案预测亲水性,Emini 方案预测分析蛋白表面可及性,Karplus-Schulz 方案预测分析蛋白柔韧性[13-16]。
1.3 芝麻过敏原Ses i 3 二级结构预测
DNAStar 生物分析软件中的子程序Protean,选择Chou-Fasman 方案、Gamier-Robson 方案以及Deleage-Roux 方案对α-螺旋、β-折叠、β-转角和无规则卷的预测。生物信息分析软件SOPMA 网络服务器 (http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)包括Garnier-Gibrat-Robson (GOR)方法、双重预测方法、Levin 同源预测方法、PHD 方法、CNRS 的SOPMA方法,可实现二级结构的预测,预测结果为5 种方法的合并结果[17]。将Ses i 3 序列提交至SOPMA网络服务器,“Number of conformational states”设置 为“4(Helix,Sheet,Turn,coil)”,“Similarity threshold”设置为“8”,“Window width”设置为“17”[17]。
1.4 芝麻过敏原Ses i 3 抗原表位预测
1)BepiPred 预测 BepiPred 1.0 Server 使用隐马尔可夫模型(Hidden Markov Model,HMM)和亲水性参数评分来预测线性B 细胞结合表位[18-19]。登录BepiPred 1.0 Server 网络服务器,提交Ses i 3 序列,设置选择默认值。
2)综合预测 将蛋白的特性(亲水性、柔韧性、表面可及性等)与二级结构预测汇总分析。预测序列不同区段的亲水性、表位可及性、柔韧性和抗原指数,筛选出亲水性、抗原指数同为正值,表面可及性大于1,有柔韧性的合适区段,作为预测蛋白的B 细胞线性表位。
1.5 芝麻过敏原Ses i 3 蛋白质三级结构预测
Swiss-Model[20](http://www.swissmodel.expasy.org/)网络服务器,运用结构仿真模拟(ProMod II程序)和能量最小化分析(GROMOS96 程序)构建目标序列的结构,根据同源蛋白质的晶体结构预测出目标蛋白的三级结构模型。登录在线软件Swiss-Model,上传Ses i 3.fasta 序列文件,选择自动模式。从搜索结果中选择同源性最高的蛋白作为模板(Template),下载模板序列后,同时提交Ses i 3 和模板序列至Swiss-Model 服务器进行自动建模,获得蛋白的三级结构预测结果。利用Structure Assessment 模块对预测的Ses i 3 蛋白三维结构进行稳定性评估分析。
2 结果与分析
2.1 Ses i 3 蛋白B 细胞线性表位预测
2.1.1 Ses i 3 蛋白的基本理化性质 利用Prot-Param 预测 Ses i 3 蛋白得到分子式为C2882H4570N904O901S24,原子数为9 281,分子质量为67.1 ku,理论等电点为4.64,在氨基酸的分布上:Glu(E)含量最高,共计67 个,占11.5%,其次是Arg(R),共计62 个,占10.6%。该蛋白的理论等电点为(pI)为7.55,平均亲水系数 (Grand average of hydropathicity,GRAVY)为:-0.902,预测为亲水性蛋白,说明蛋白整体的亲水性较好。
2.1.2 DNAStar 预测Ses i 3 蛋白B 细胞线性表位结果 抗原的线性表位一般都暴露在过敏原分子的外部,位于柔韧性区域内,由极性氨基酸残基组成,线性表位所含氨基酸一般大于3 个[21]。DNAStar 软件预测Ses i 3 的抗原指数、亲水性、表面可及性和柔韧性如图1所示。从图中可以看出,柔韧性指数中有矩形覆盖的部分为抗原表位的优势区域,Ses i 3 中柔韧性好的区域分布相对平均;有20 多个区域的抗原指数较高;亲水性较好的区域主要集中在序列前半部分;有近20 个区域的表面可及性好。其中,亲水性、抗原指数为正值,表面可及性大于1,柔韧性预测结果中有矩形覆盖的合适区段,作为DNAStar 预测Ses i 3 蛋白的B 细胞线性表位的一类指标。另外,蛋白质的二级结构是抗原表位预测另一个重要参数。α-螺旋和β-折叠结构不易变形,难以与抗体结合,处于这2 个区域内的氨基酸序列成为抗原表位的可能性较小。β-转角和无规则卷曲由于其突出的结构,极易与抗体结合,这2 个区域内的氨基酸序列极有可能成为抗原表位。DNAStar 的子程序能够预测蛋白质二级结构。通过Chou-Fasman 方案、Gamier-Robson 方案以及Deleage-Roux 方案预测Ses i 3 蛋白质的二级结构结果如图2所示,β-转角(T)或无规则卷曲(C)出现的区域在整个氨基酸序列分布均匀,预示这些区域出现线性表位可能性较高。

图1 Ses i 3 蛋白柔韧性、抗原指数、亲水性、表面可及性分析Fig.1 Prediction of antigenicity,hydrophilicity,surface probability and flexibility of Ses i 3
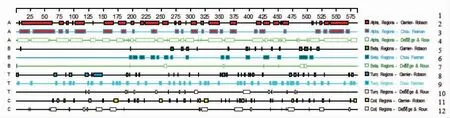
图2 DNAStar 预测Ses i 3 蛋白二级结构结果Fig.2 Prediction of secondary structure of protein Ses i 3 using DNAStar
综合考虑DNAstar 预测的亲水性指数、抗原指数、表面可及性指数、柔韧性指数以及二级结构的结果,预测的线性表位如表1所示(加粗部分)。

表1 DNAStar 预测B 细胞线性表位预测结果Table 1 Prediction of B-cell epitopes using DNAStar
2.1.3 SOMPA 预测Ses i 3 蛋白B 细胞线性表位结果 利用SOPMA 网络服务器对该蛋白的二级结构进行预测,结果如图3所示,其中构成α-螺旋的氨基酸占比为36.24%,构成β-折叠的氨基酸占比为19.32%,构成β-转角的氨基酸占比为10.09%,构成无规则卷曲的氨基酸占比为34.36%,详细的统计结果如表2所示。可以看出,近一半的氨基酸残基构成了β-转角和无规则卷曲,这部分是抗原表位的优势区域,结合2.1 节中预测的氨基酸特性,由此预测出Ses i 3 的线性表位如表3所示。
2.1.4 BepiPred 1.0 预测Ses i 3 蛋白B 细胞线性表位结果 将Ses i 3 蛋白提交至Bepi Pred网络服务器后,通过Markov 模型和规模化方法结合来预测B 细胞线性表位。BepiPred 具有生物序列比对、同源性分析、基因识别、蛋白质二级结构预测、信号肽预测、抗原表位预测等功能[22],广泛的应用于生物信息学领域,是最佳的单一模式预测方法,其预测结果如表3所示。
将上述3 种方法所预测得到区域进行比对,发现3 种预测结果大体一致。根据文献[23]介绍的方式进行多种软件结合,筛选最终的预测肽段。根据表3的预测结果,本研究选择同时涵盖2 种及以上的预测区域作为最终的线性表位区域,既提高了预测结果的准确性,也能保证更多的线性表位优势区域可供选择,预测的表位肽段信息详见表4。图4为Ses i 3 所有预测到的B 细胞线性表位的空间分布情况,所展现的是不同角度的观测结果,可以看出预测表位多分布在无规则卷曲处。可以发现,预测的Ses i 3 线性表位在整个序列中分布比较均匀,整个蛋白序列的前、中、后段都有预测的表位区域分布。并且短肽段的表位所占比例较多,其中表位4 和表位10 的序列相同,这里作为同种表位。Ses i 3 与Cupin 超家族中的其它过敏原有较高的同源性,并且在报道的已知表位上存在着部分区域氨基酸序列高度相似的情况,也存在某些表位关键氨基酸相同的情况。将预测的表位与该家族中有已知表位的7S 豌豆球蛋白过敏原(腰果中的Ana o 1、英国胡桃中的Jug r 2、花生中的Ara h 1、扁豆中的Len c 1)序列进行比对[24-28],将有重叠的部分进行展示,结果如图5所示,黄色区域为已知线性抗原表位,绿色区域为Ses i 3 预测表位。在预测的表位中,其中有10 个肽段的位置与已经报道的表位区域存在部分重合的现象,说明生物信息学方法在预测过敏原蛋白抗原表位时有较高的准确度。

图3 SOPMA 预测Ses i 3 二级结构Fig.3 Prediction of secondary structure of Ses i 3 using SOPMA

表2 SOPMA 预测Ses i 3 二级结构Table 2 Prediction of secondary structures of Ses i 3 using SOPMA

表3 3 种信息学软件预测结果Table 3 Prediction by three bioinformation tools
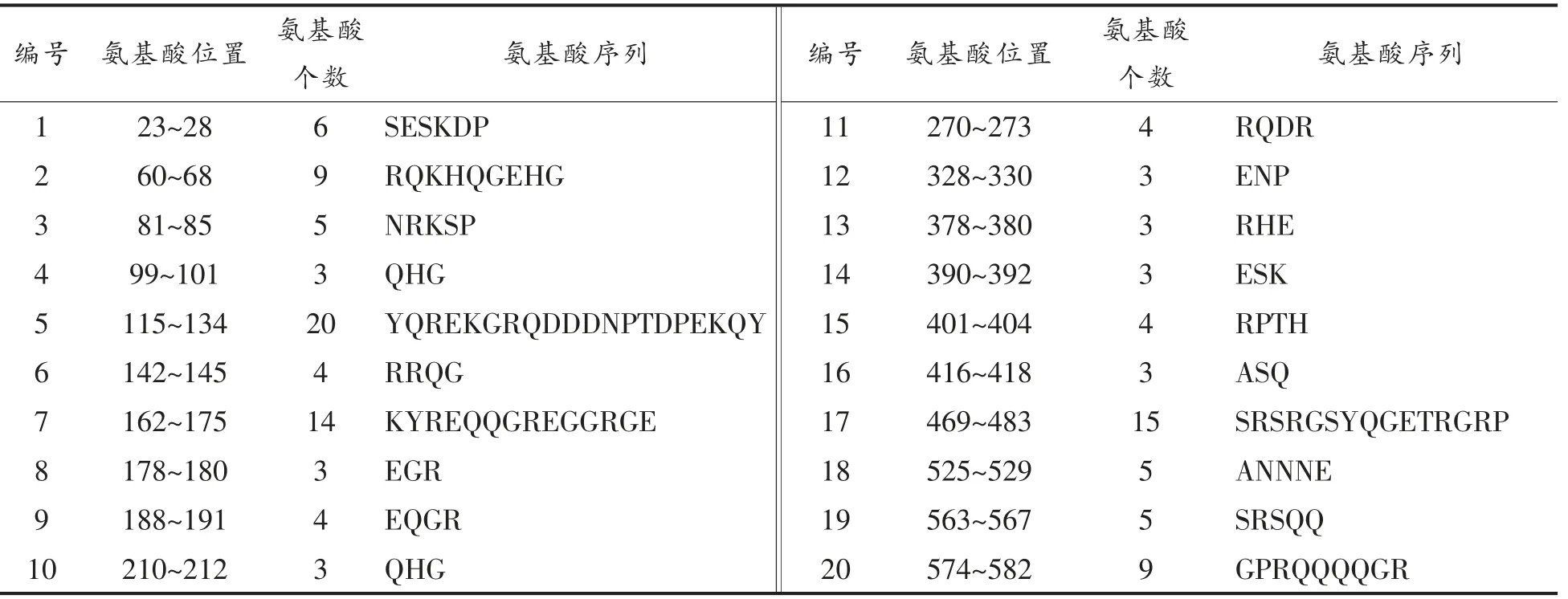
表4 B 细胞线性表位最终预测结果Table 4 Final prediction of B-cell liner peptides
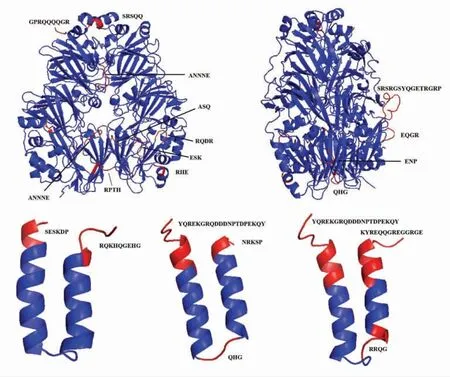
图4 预测芝麻过敏原蛋白Ses i 3 B 细胞线性抗原表位空间构象Fig.4 Spatial conformation of predicted B cell liner antigenic epitopes in sesame allergen Ses i 3

图5 芝麻过敏原Ses i 3 与Cupin 超家族过敏原序列比对结果[24-28]Fig.5 Sequence alignment of sesame allergen Ses i 3 and Cupin allergens[24-28]
2.2 Ses i 3 蛋白的三级结构预测
使用Swiss-Model 服务器预测蛋白三维结构时,软件首先会选择一系列被PBD(http://www.rcsb.org/pdb/)数据库收录且结构已知的同源性蛋白质。从筛选结果中选择相似性较高的作为模板,建立模型。经过评估后选择合适的模型作为最终的预测结果。经过SWISS-MODEL 分析,与Ses i 3 同源性最高的是碧根果中新发现的一种7S 类豌豆球蛋白过敏原[29],命名为Car i 2,与Ses i 3同属于Cupin 超家族,在PDB 数据库中编号为5e1r.2。比对后发现两者同源性达到了48.74%,通常当目标蛋白与模板蛋白的同源性达到30%以上时,建立的模型才有可靠性,因此可选择5e1r.2 建模,结果如图6所示。通过软件所带的“Structure Assessment”(结构评价)功能,评价预测蛋白结构的稳定性,得到图7所示的反映立体化学质量参数Ramachandran 图。Ramachandran 图表示的是α碳的两面角,在实际中要保持蛋白质构象的稳定,主链就需要具备合理φ 角和ψ 角。骨架的集合需要分布在Ramachandran 图的可接受区域,否则将形成空间结构空间位阻[30]。通过分析ψ 角和φ 角分布情况,显示高达93.96%骨架的φ 角和ψ 角位于分布在Ramachandran Favoured,即可接受区域内。如图7所示深绿色和浅绿色部分,是最理想的ψ 角和φ 角分布区域,只有极少数(1.28%)分布在白色区域Ramachandran Outliers,即不合理区域。因此,以5e1r.2 为模板预测的Ses i 3 蛋白的三级结构是稳定可靠的,是高质量的模型结构。
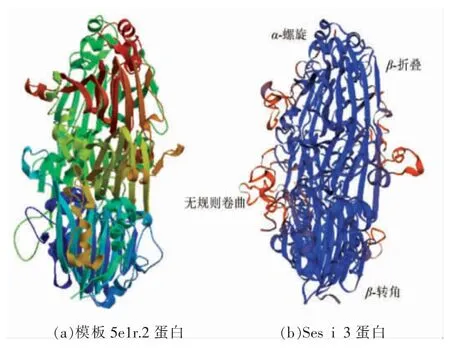
图6 蛋白空间结构Fig.6 Steric structure of protein
3 结论
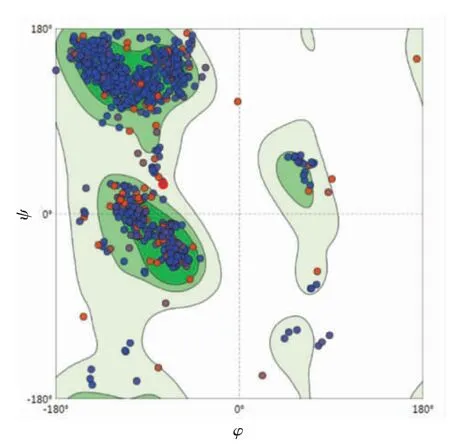
图7 Ses i 3 蛋白三级结构的拉氏构象Fig.7 Ramachandran plot analysis of Ses i 3 three-dimensional model
对于B 细胞线性表位的预测有多种方式,然而主要都是借助于多种参数,如二级结构、亲水性、表面可及性、抗原性以及柔韧性等。其中,亲水性和表面可及性在形成表位时十分关键,然而也离不开二级级结构等其它因素的综合影响[31]。蛋白质高级结构的维持主要依赖于高化学键能的α-螺旋和β-折叠,它们通常位于蛋白质内部,与抗体嵌合困难,难以成为抗原表位;相反,β-转角及无规则卷曲柔韧性好,结构松散,易扭曲,多位于蛋白质表面,易与抗体嵌合,容易成为抗原表位。
本研究根据蛋白的氨基酸序列,利用生物信息学软件及服务器DNAStar、SOPMA 和BepiPred-1.0 3 种方法结合蛋白质特性和二级结构,预测分析了Ses i 3 的B 细胞线性表位,利用Swiss-Model 进行同源建模并预测了三级结构,对预测结构的稳定性进行评估,提高了预测的准确性。结果表明,Ses i 3 蛋白存在多个可能的抗原表位,SESKDP,RQKHQGEHG,NRKSP,QHG,YQREK GRQDDDNPTDPEKQY,RRQG,KYREQQGREG GRGE,EGR,EQGR,QHG,RQDR,ENP,RHE,ESK,RPTH,ASQ,SRSRGSYQGETRGRP,ANNNE,SRSQQ,GPRQQQQGR 最有可能成为其线性表位,在整个氨基酸序列中分布均匀,并且与同家族已知的过敏原线性表位上存在着部分区域氨基酸序列高度相似的情况。本研究的表位预测结果增加了对芝麻过敏原的认识,为定位表位缩小了范围,为抗体制备,免疫检测提供了一定的理论基础。探究芝麻过敏原Ses i 3 的三级结构对过敏原的空间表位筛选,以及过敏原识别和检测具有重要的参考和借鉴作用。
