基于TCGA数据库的肝细胞癌自噬相关基因预后风险模型的建立
2021-05-08许杨尹侃李霄霞狄国虎侯琳赵春华
许杨 尹侃 李霄霞 狄国虎 侯琳 赵春华
[摘要]目的基于自噬相关基因(ATGs)构建肝细胞癌病人预后风险模型。方法TCGA数据库下载374例肝细胞癌及50例正常肝组织的转录组数据和临床信息,首先筛选出差异表达基因(DEGs),然后从中筛选出差异表达的ATGs(DEATGs),最终利用单因素Cox回归分析、LASSO回归分析以及多因素Cox回归分析构建预后风险模型,并应用ROC曲线评估模型对于肝细胞癌病人预后的预测能力。结果肝细胞癌组织与正常肝组织筛选得到12 471个DEGs,其中包含62个DEATGs。单因素Cox回归分析得到40个预后相关DEATGs,LASSO回归分析以及多因素Cox回归分析得到7个风险自噬基因(RAB7A、FOS、ATG9A、HSPA8、PRKN、TUSC1和GAPDH)用于構建预后风险模型。Kaplan-Meier分析显示,高风险组病人的总体生存期显著低于低风险组(χ2=36.972,P<0.05)。多因素Cox回归分析表明,预后风险模型可以作为一个独立预后因素(HR=2.574,P<0.01)。结论基于ATGs构建的预后风险模型是一个独立的预后因素,可有效预测肝细胞癌病人的预后。
[关键词]癌,肝细胞;自噬;基因;预后;计算生物学
[中图分类号]R730.7[文献标志码]A[文章编号]2096-5532(2021)01-0001-07
[ABSTRACT]ObjectiveTo establish a prognostic risk model for patients with hepatocellular carcinoma (HCC) based on autophagy-related genes (ATGs). MethodsThe transcriptome data and clinical information of 374 HCC samples and 50 normal liver tissue samples were downloaded from TCGA database. Firstly, differentially expressed genes (DEGs) were screened out, then differentially expressed ATGs (DEATGs) were screened out, and finally, the univariate Cox regression analysis, the LASSO regression analysis, and the multivariate Cox regression analysis were used to establish a prognostic risk model and the receiver ope-rating characteristic curve was used to evaluate the value of this model in predicting the prognosis of HCC patients. ResultsA total of 12 471 DEGs were screened out from HCC tissue and normal liver tissue, including 62 DEATGs. The univariate Cox regression analysis obtained 40 DEATGs associated with prognosis, and the LASSO regression analysis and the multivariate Cox regression analysis obtained 7 risk autophagy genes (RAB7A, FOS, ATG9A, HSPA8, PRKN, TUSC1, and GAPDH) which were used to establish the prognostic risk model. The Kaplan-Meier analysis showed that the patients in the high-risk group had a significantly lower overall survival than those in the low-risk group (χ2=36.972,P<0.05). The multivariate Cox regression analysis showed that the prognostic risk model could be used as an independent prognostic factor (HR=2.574,P<0.01). ConclusionThe risk prognostic model based on ATGs is an independent prognostic factor and can effectively predict the prognosis of HCC patients.
[KEY WORDS]carcinoma, hepatocellular; autophagy; genes; prognosis; computational biology
肝细胞癌(HCC)是癌症死亡的主要原因,其发病率逐年递增[1]。HCC的治疗主要包括手术切除、分子靶向治疗和肝移植等[2]。然而,由于其具有恶性程度高以及术后易复发等特点,许多国家HCC的发病率和死亡率还在持续上升[3]。尽管多项研究提出了包括病人的基本特征(如年龄和性别)和肿瘤相关因素(如肿瘤分级)等在内的预后因素,可用于预测HCC病人的生存状态[4-5],但是仍然缺乏有效的预后因素。自噬是一种高度保守的溶酶体降解途径[6],由一系列高度协同的信号通路调控,发生在所有细胞的基础水平,在维持细胞内环境稳定方面起着重要作用[7-8]。研究结果显示,自噬在多种疾病如癌症、心血管和神经系统疾病等病理过程中起关键作用[9]。自噬参与调控HCC的进程[10-11]。癌症基因组图谱(TCGA)计划是通过大规模测序的基因组分析技术来绘制人类肿瘤的基因组图谱[12],其中包含33种癌症[13-14]。本研究基于TCGA公共数据库中HCC转录组数据和临床信息进行分析,构建预后风险模型并将其应用于HCC的预后预测。现将结果报告如下。
1资料和方法
1.1数据资料收集及处理
研究涉及的数据资源下载于TCGA(https://portal.gdc.cancer.gov/),其中包括所有HCC病人RNA-seq数据及相关的临床数据。从人类自噬数据库(HADb,http://www.autophagy.lu/)下载232个自噬相关基因(ATGs)。根据病人的ID号码将转录组数据与病人的临床信息进行匹配,最终从TCGA数据库中获得了365例病人完整的生存信息和基因表达谱数据。
1.2差异表达的ATGs(DEATGs)的鉴定
利用edgeR函数包对数据进行分析,以FDR<0.05和|fold change(FC)|>1为条件,筛选HCC组织和正常组织样本中的差异表达基因(DEGs)以及DEATGs。
1.3预后风险模型的构建
使用Survival函数包对DEATGs进行单因素Cox回归分析,以P<0.05为条件筛选影响预后的DEATGs,使用LASSO回归分析降维,纳入多因素Cox回归分析得到了用于HCC预后模型的风险ATGs。建立预后风险模型评分公式如下:风险评分=∑nj=1Coefj×Xj,其中Coef为ATGs的多变量回归分析系数,X为各ATGs的相对表达水平。根据公式为每个病人生成风险评分,并以风险评分中位数为界值,将病人分为高风险组和低风险组,高风险组提示病人预后差。采用Kaplan-Meier分析和log-rank检验评估两组病人的总体生存期,并利用ROC曲线及曲线下面积(AUC)评估该预后模型的准确性。
1.4统计学方法
使用R3.6.1软件(https://www.R-project.org/)进行统计学分析及图形绘制。采用Cox回归分析风险比(HR)及其95%可信区间(CI)评价ATGs表达与预后的关联。使用Cox回归分析和LASSO回归分析降维筛选模型相关风险ATGs,构建多基因预后风险模型。利用ROC曲线和AUC评估预后风险模型的预测能力。采用单因素和多因素Cox回归分析确定具有独立预后价值的因素。利用Wilcoxon-signedrank检验和Kruskal检验分析临床变量与ATGs的关系。以P<0.05为差异有统计学意义。
2结果
2.1DEATGs的筛选和富集分析
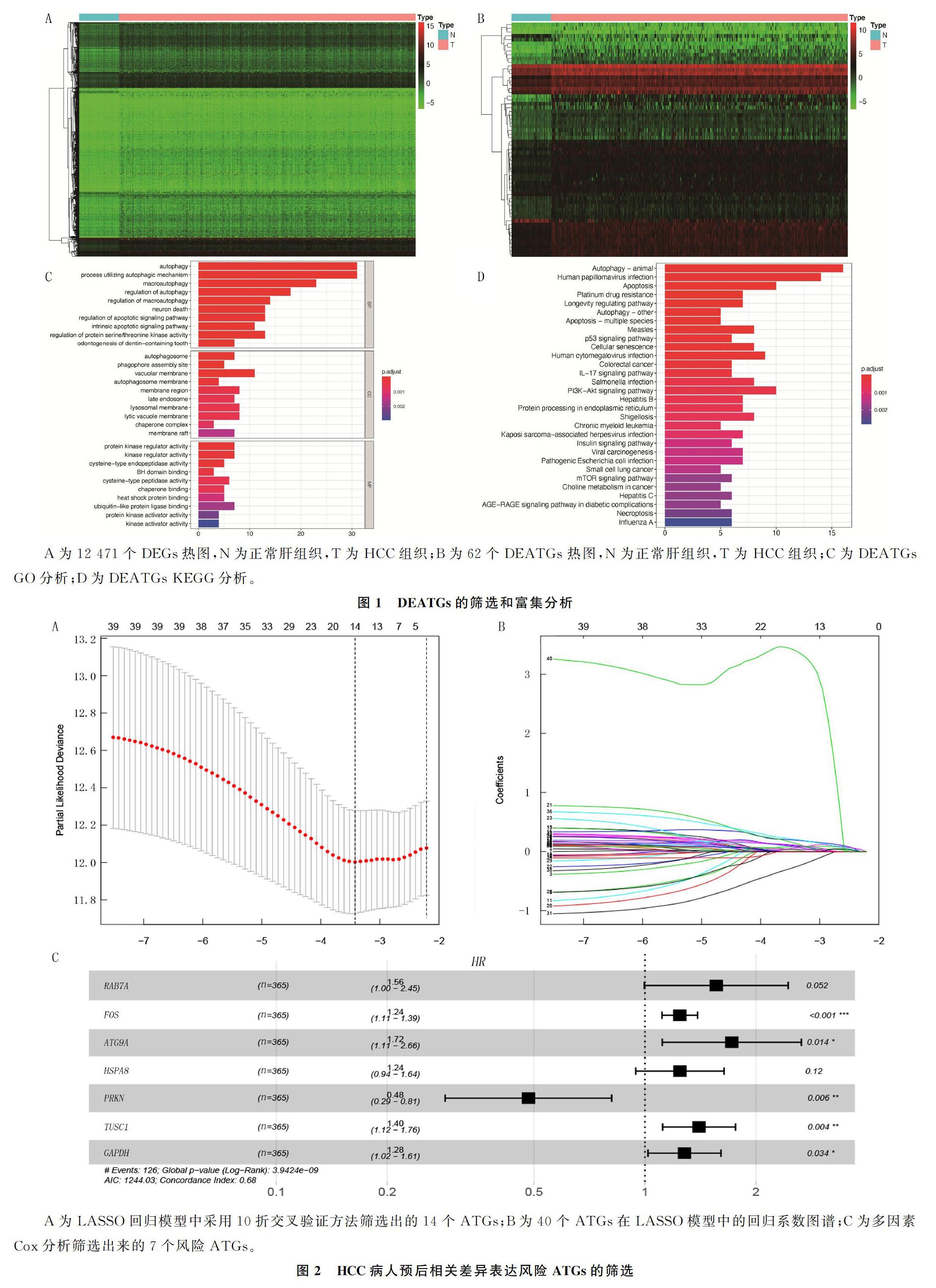
共筛选出12 471个DEGs(图1A),并进一步从中筛选出62个DEATGs(图1B)。对DEATGs进行Gene Ontology(GO)分析,包括生物过程(BP)、细胞成分(CC)和分子功能(MF)。DEATGs在BP组中自噬和利用自噬机制的过程等方面显著富集、CC组中空泡膜和膜区域等方面显著富集、MF组中蛋白激酶调节活性和激酶调节活性等方面显著富集(图1C)。此外,KEGG通路分析显示,DEATGs主要富集于自噬、细胞凋亡和人乳头瘤病毒感染等方面(图1D)。
2.2预后风险模型风险ATGs的构建
对62个DEATGs进行单因素Cox回归分析,共有40个与HCC预后显著相关的ATGs纳入后续的分析。经过LASSO回归分析降维,再使用多因素Cox回归分析,最终得到了7个与HCC预后相关的风险ATGs(RAB7A、GAPDH、ATG9A、HSPA8、PRKN、TUSC1和FOS),风险ATGs与HCC预后的关联见图2。利用风险评分函数构建预后风险模型,风险评分=(RAB7A表达量×0.445 444 289)+(FOS表达量×0.217 687 513)+(ATG9A的表达量×0.542 642 682)+(HSPA8的表达量×0.218 555 915)+(PRKN的表达量×-0.727 657 489)+(TUSC1表达量×0.337 901 906)+(GAPDH表达量×0.246 856 663)。
2.3预后风险模型的评价
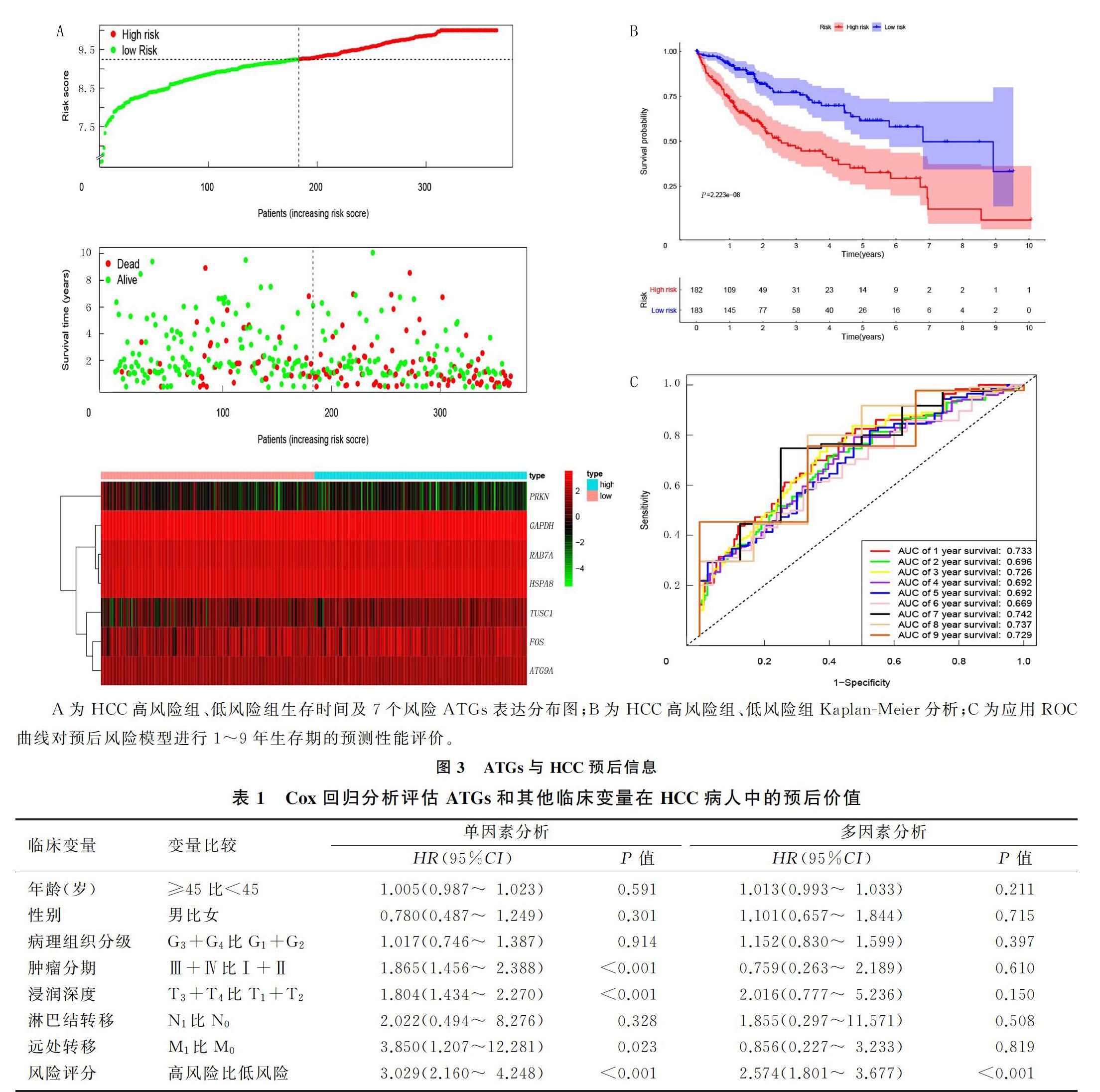
基于上述风险评分函数生成每个病人的风险评分,高风险组 182例,低风险组183例。随着风险评分的升高,病人的生存期缩短且死亡例数增加(图3A)。Kaplan-Meier分析显示,高风险组病人的总体生存期较低(χ2=36.972,P<0.05)。ROC曲线分析显示,风险评分模型对HCC病人1~9年总生存期预测的AUC值分别为0.733、0.696、0.726、0.692、0.692、0.669、0.742、0.737和0.729(图3B、C)。
1期许杨,等. 基于TCGA数据库的肝细胞癌自噬相关基因预后风险模型的建立3
A为12 471个DEGs热图,N为正常肝组织,T为HCC组织;B为62个DEATGs热图,N为正常肝组织,T为HCC组织;C为DEATGs GO分析;D为DEATGs KEGG分析。
A为LASSO回归模型中采用10折交叉验证方法筛选出的14个ATGs;B为40个ATGs在LASSO模型中的回归系数图谱;C为多因素Cox分析筛选出来的7个风险ATGs。
2.4ATGs预后风险模型的独立预后价值
单因素Cox回归分析显示,病人的肿瘤分期(HR=1.865,95%CI=1.456~2.388,P<0.05)、肿瘤浸润深度(HR=1.804,95%CI=1.434~2.270,P<0.05)、肿瘤远处转移(HR=3.850,95%CI=1.207~12.281,P<0.05)和风险评分模型(HR=3.029,95%CI=2.160~4.248,P<0.05)为预后预测因素。多因素Cox回归分析显示,风险评分是HCC病人的独立预后因素(HR=2.574,95%CI=1.801~3.677,P<0.05)。見表1。
A为HCC高风险组、低风险组生存时间及7个风险ATGs表达分布图;B为HCC高风险组、低风险组Kaplan-Meier分析;C为应用ROC曲线对预后风险模型进行1~9年生存期的预测性能评价。
2.5预后风险模型的临床应用
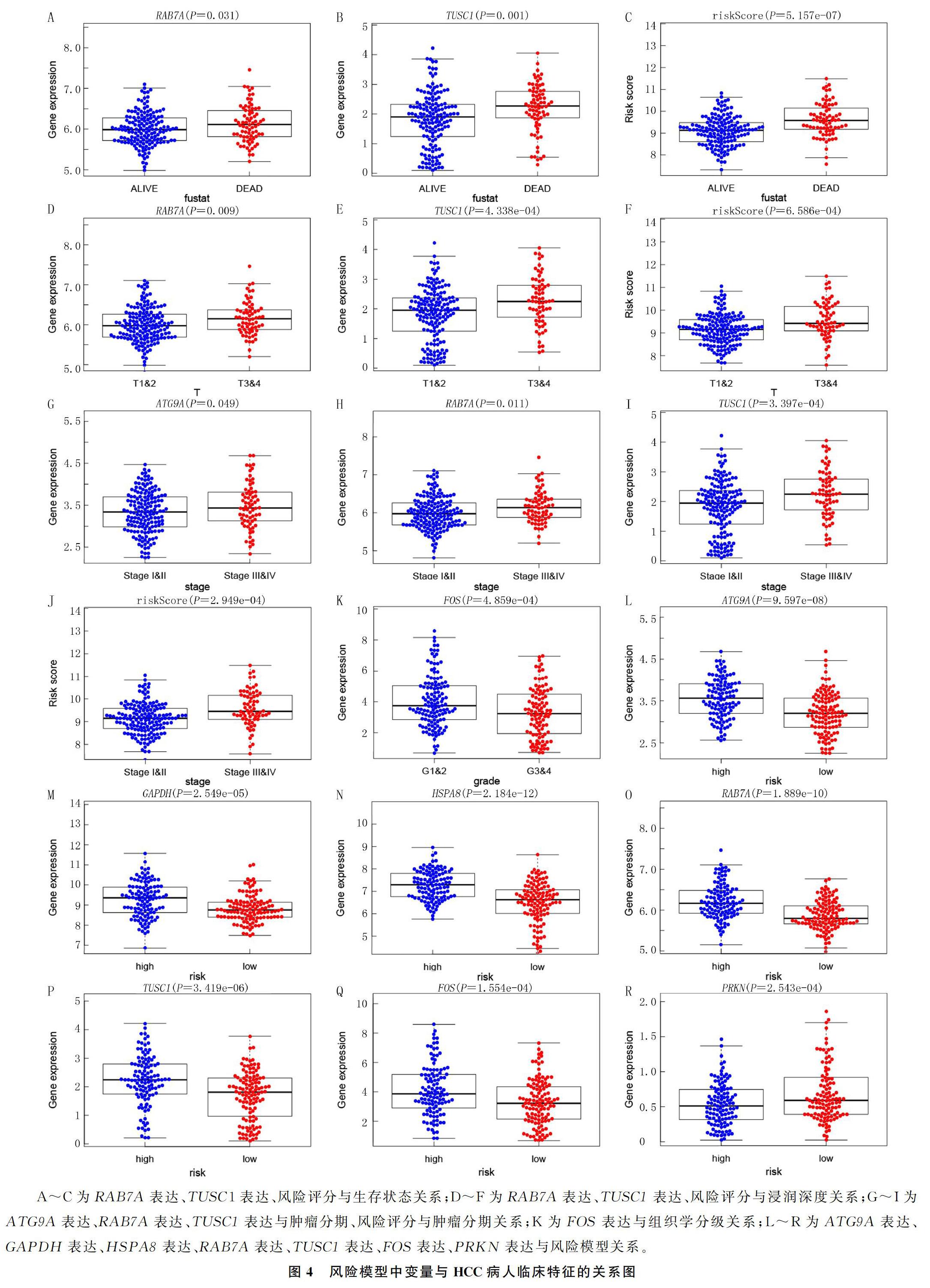
为了检验预后风险模型预测HCC进展的能力,分析了风险因素(风险评分和风险基因)与临床变量(生存状态、年龄、性别、病理组织分级和病理TNM分期)之间的关系。结果显示,随着RAB7A、TUSC1和风险评分因素的增加,HCC病人生存状态变差(t=-5.265~-2.175,P<0.05),肿瘤浸润深度增加(t=-3.613~-2.644,P<0.05)(图4A~F);随着RAB7A、ATG9A、TUSC1和风险评分的增加,HCC病人的病理分期增加(t=-3.737~-1.992,P<0.05)(图4H~J)。高风险组病人自噬基因RAB7A、FOS、ATG9A、HSPA8、TUSC1和5GAPDH表达高于低风险组(t=3.848~7.467,P<0.05)(图4L~Q),PRKN基因表达低于低风险组(t=-3.741,P<0.05)(图4R)。随着FOS基因表达的增加,HCC病人病理组织分级明显降低(t=3.542,P<0.05)(图4K)。
3讨论
HCC病人术后高复发率和化疗药物耐药性的产生是肝癌病人病死率高的主要因素。因此,预测HCC预后的可靠分子标志物对于指导病人的预后具有重要意义。业已证实,ATGs参与多种肿瘤的进展和预后,例如结直肠癌、膀胱癌和小细胞性肺癌等[15-17]。本研究结果显示,ATGs与HCC的临床病理特征和预后密切相关。本文通过对HCC肿瘤组织和正常组织DEGs表达的分析,筛选出62个DEATGs。GO分析显示,DEATGs在BP组中自噬和利用自噬机制的过程等方面显著富集、CC组中空泡膜和膜区域等方面显著富集、MF组中蛋白激酶调节活性和激酶调节活性等方面显著富集。KEGG富集分析显示,DEATGs主要富集在自噬、细胞凋亡和人乳頭瘤病毒感染等方面。本文应用单因素Cox回归、LASSO回归分析和多因素Cox回归分析,确定7个风险ATGs(RAB7A、GAPDH、ATG9A、HSPA8、PRKN、TUSC1和FOS)构建的预后风险模型;Kaplan-Meier分析显示,高风险组的生存期明显低于低风险组。本文还分析了风险评分模型中的因素与某些临床特征的关系,研究结果显示模型中的一些因素(如基因RAB7A、FOS、ATG9A、HSPA8、TUSC1和GAPDH表达)的增加与HCC的病情进展呈正相关。提示本文构建的模型在预测HCC发展、演化等方面具有较高的临床适用性,并为ATGs在HCC发生发展中的作用机制研究提供了信息。
FOS是一种核原癌基因,编码c-Fos,参与细胞增殖和凋亡的调控。有研究显示,HCC组织中c-Fos高表达[18]。免疫组化研究显示,HCC肿瘤组织中c-Fos的表达明显高于非肿瘤组织[19]。本文研究结果表明,FOS与HCC病人预后相关,并随着病理组织分级的增高,其表达量下降,提示FOS在HCC的进展中可能发挥保护性作用,为进一步研究提供了更多的信息。ATG9A是一种跨膜蛋白,可以促进自噬体的形成[20],有研究表明ATG9A在HepG2细胞中高表达[21],而ATG9A基因的沉默则抑制胶质瘤生长[22]。本文研究结果显示,ATG9A与HCC病人预后相关,并随着肿瘤分期的增加,其表达量升高,提示ATG9A是HCC发生发展的一个危险因素。HSPA8是应激蛋白,也是肝癌的早期生物标志物,HSPA8高表达提示HCC复发[23]。本文研究结果显示,HSPA8在高危组中的表达高于低危组,提示HSPA8是HCC发展过程中的危险因素。SHIMIZU等[24]研究显示,TUSC1基因内高甲基化病人的疾病特异性生存期明显短于未发生基因内高甲基化者。本文研究结果显示,TUSC1与HCC病人预后有关,随着TUSC1表达的增加,HCC肿瘤分期升高、肿瘤浸润深度增加,该结果提示TUSC1是HCC进程中的一个危险因素。另外有研究显示,GAPDH在HCC病人中表达上调,GAPDH通过与3-BrPA或GAPDH shRNA的分子靶点作用阻断肿瘤的进展[25]。本文研究显示,GAPDH在高危组中表达高于低危组,多因素Cox回归分析显示GAPDH是HCC的危险因素。XIE等[26]研究发现,RAB7A影响体内乳癌细胞增殖和迁移。PRKN通过抑制线粒体铁介导的慢性炎症和免疫功能障碍,显著抑制KRAS驱动的胰腺肿瘤的发生[27]。DUAN等[28]研究发现,PRKN通过EGFR/AKT/mTOR通路抑制肺癌的生长和转移。但RAB7A和PRKN在HCC中的作用机制研究较少。本文研究单因素、多因素Cox回归分析显示,RAB7A和PRKN对于HCC病人的预后具有一定的预测作用,提示RAB7A和PRKN可能是HCC治疗的潜在靶点,但这一结论需要大规模、多中心的基础实验以及临床试验进一步验证。
综上,本研究通过ATGs表达谱分析,筛选出了7个(RAB7A、FOS、ATG9A、HSPA8、PRKN、TUSC1、GAPDH)与HCC病人的预后显著相关的风险ATGs。基于这7个风险ATGs组成的风险模型能够很好地识别预后不良高风险的HCC病人,并且可以作为一个独立的预后因素预测HCC病人的预后。基于此建立的预后风险模型对判断HCC预后显示出较高的准确率。
[参考文献]
[1]KULIK L, EL-SERAG H B. Epidemiology and management of hepatocellular carcinoma[J]. Gastroenterology, 2019,156(2):477-491.e1.
[2]EL-SERAG H B, MARRERO J A, RUDOLPH L, et al. Diagnosis and treatment of hepatocellular carcinoma[J]. Gastroenterology, 2008,134(6):1752-1763.
[3]Global Burden of Disease Cancer Collaboration, FITZMAURICE C, AKINYEMIJU T F, et al. Global, regional, and national cancer incidence, mortality, years of life lost, years lived with disability, and disability-adjusted life-years for 29 cancer groups, 1990 to 2016: a systematic analysis for the global burden of disease study[J]. JAMA Oncology, 2018,4(11):1553-1568.
[4]ROAYAIE S, BLUME I N, THUNG S N, et al. A system of classifying microvascular invasion to predict outcome after resection in patients with hepatocellular carcinoma[J]. Gastroenterology, 2009,137(3):850-855.
[5]CUCCHETTI A, VIVARELLI M, PISCAGLIA F, et al. Tumor doubling time predicts recurrence after surgery and describes the histological pattern of hepatocellular carcinoma on cirrhosis[J]. Journal of Hepatology, 2005,43(2):310-316.
[6]BOGA J A, CABALLERO B, POTES Y, et al. Therapeutic potential of melatonin related to its role as an autophagy regulator: a review[J]. Journal of Pineal Research, 2019,66(1):e12534. doi:10.1111/jpi.12534.
[7]TOWERS C G, THORBURN A. Therapeutic targeting of autophagy[J]. EBio Medicine, 2016,14:15-23.
[8]WHITE E. The role for autophagy in cancer[J]. J Clin Investig, 2015,125(1):42-46.
[9]CHENG Y, REN X C, HAIT W N, et al. Therapeutic targeting of autophagy in disease:biology and pharmacology[J]. Pharmacological Reviews, 2013,65(4):1162-1197.
[10]ZHANG K, CHEN J, ZHOU H, et al. PU.1/microRNA-142-3p targets ATG5/ATG16L1 to inactivate autophagy and sensitize hepatocellular carcinoma cells to sorafenib[J]. Cell Death & Disease, 2018,9(3):312.
[11]ZHAO Z Z, WU J Y, LIU X D, et al. Insufficient radiofrequency ablation promotes proliferation of residual hepatocellular carcinoma via autophagy[J]. Cancer Letters, 2018,421:73-81.
[12]TOMCZAK K, CZERWISKA P, WIZNEROWICZ M. The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge[J]. Contemporary Oncology (Poznan,Poland), 2015,19(1A):A68-A77.
[13]DING L, BAILEY M H, PORTA-PARDO E, et al. Perspective on oncogenic processes at the end of the beginning of can-cer genomics[J]. Cell, 2018,173(2):305-320.
[14]SANCHEZ-VEGA F, MINA M, ARMENIA J, et al. Oncogenic signaling pathways in the cancer genome atlas[J]. Cell, 2018,173(2):321-337.
[15]BEDNARCZYK M, MUC-WIERZGO M, WANICZEK D, et al. Autophagy-related gene expression in colorectal cancer patients[J]. Journal of Biological Regulators and Homeostatic Agents, 2017,31(4):923-927.
[16]EISSA S, MATBOLI M, AWAD N, et al. Identification and validation of a novel autophagy gene expression signature for human bladder cancer patients[J]. Tumour Biology: the Journal of the International Society for Oncodevelopmental Biology and Medicine, 2017,39(4):1010428317698360.
[17]LIU Y, WU L G, AO H J, et al. Prognostic implications of autophagy-associated gene signatures in non-small cell lung cancer[J]. Aging, 2019,11(23):11440-11462.
[18]LEE J S, HEO J, LIBBRECHT L, et al. A novel prognostic subtype of human hepatocellular carcinoma derived from hepatic progenitor cells[J]. Nature Medicine, 2006,12(4):410-416.
[19]YUEN M F, WU P C, LAI V C, et al. Expression of c-Myc, c-Fos, and c-Jun in hepatocellular carcinoma[J]. Cancer, 2001,91(1):106-112.
[20]ORSI A, RAZI M, DOOLEY H C, et al. Dynamic and tran-sient interactions of Atg9 with autophagosomes, but not membrane integration, are required for autophagy[J]. Molecular Biology of the Cell, 2012,23(10):1860-1873.
[21]KUNANOPPARAT A, KIMKONG I, PALAGA T, et al. Increased ATG5-ATG12 in hepatitis B virus-associated hepatocellular carcinoma and their role in apoptosis[J]. World Journal of Gastroenterology, 2016,22(37):8361-8374.
[22]ABDUL RAHIM S A, DIRKSE A, OUDIN A, et al. Regulation of hypoxia-induced autophagy in glioblastoma involves ATG9A[J]. British Journal of Cancer, 2017,117(6):813-825.
[23]XIANG X, YOU X M, LI L Q. Expression of HSP90AA1/HSPA8 in hepatocellular carcinoma patients with depression[J]. OncoTargets and Therapy, 2018,11:3013-3023.
[24]SHIMIZU D, KANDA M, NOMOTO S, et al. Identification of intragenic methylation in the TUSC1 gene as a novel prognostic marker of hepatocellular carcinoma[J]. Oncology Reports, 2014,31(3):1305-1313.
[25]GANAPATHY-KANNIAPPAN S, KUNJITHAPATHAM R, TORBENSON M S, et al. Human hepatocellular carcinoma in a mouse model: assessment of tumor response to percutaneous ablation by using glyceraldehyde-3-phosphate dehydrogenase antagonists[J]. Radiology, 2012,262(3):834-845.
[26]XIE J M, YAN Y, LIU F, et al. Knockdown of Rab7a suppresses the proliferation, migration, and xenograft tumor growth of breast cancer cells[J]. Bioscience Reports, 2019,39(2):BSR20180480.
[27]KANG R, XIE Y C, ZEH H J, et al. Mitochondrial quality control mediated by PINK1 and PRKN: links to iron metabolism and tumor immunity[J]. Autophagy, 2019,15(1):172-173.
[28]DUAN H J, LEI Z, XU F, et al. PARK2 suppresses prolife-ration and tumorigenicity in non-small cell lung cancer[J]. Frontiers in Oncology, 2019,9:790.
(本文編辑 黄建乡)
