人丝氨酸蛋白酶抑制剂B3基因的生物信息学分析
2021-05-08李靖飞许威张顺李峰
李靖飞, 许威, 张顺, 李峰
人丝氨酸蛋白酶抑制剂B3基因的生物信息学分析
李靖飞, 许威, 张顺, 李峰
(湖南文理学院 生命与环境科学学院, 湖南 常德, 415000)
人丝氨酸蛋白酶抑制剂B3 SERPINB3能抑制半胱氨酸蛋白酶, 具有抗细胞调亡、促进细胞增殖和迁移作用, 且与许多肿瘤的发生与发展密切相关。通过基因电子克隆(in silico cloning)获得SERPINB3基因全长cDNA序列(1 779 bp), 编码由390个氨基酸组成的蛋白质。核酸序列分析表明: SERPINB3基因位于人体18号染色体长臂(18q21.33), 基因组跨越80 373 285 bp, 含有个16外显子, 15个内含子, 其cDNA序列上总共有45个酶切位点。SERPINB基因核酸序列同源性分析显示, 人类与黑猩猩的亲缘关系较近, 与白眉猴和猕猴的亲缘关系是最远。SERPINB3蛋白序列同源性分析显示, 人类和黑猩猩亲缘关系较近, 和苍疣猴亲缘关系较远。SERPINB3相互作用蛋白的生物信息学分析表明: 与SERPINB3相互作用的蛋白中, 以丝氨酸蛋白酶家族成员居多, 且大多在调节炎症反应、细胞增殖分化以及凋亡等方面起着关键作用。本论文通过对SERPINB3基因的生物信息学分析, 为SERPINB3的功能和分子进化研究提供更多的生物信息学参考。
SERPINB3; 基因; 生物信息学分析
人丝氨酸蛋白酶抑制剂B3(SERPINB3)能抑制半胱氨酸蛋白酶如组织蛋白酶K、L、S和V, 具有抗细胞调亡、促进细胞增殖和迁移作用, 与许多肿瘤的发生发展及侵袭迁移密切相关[1]。SERPINB4能抑制糜蛋白酶样丝氨酸蛋白酶, 如组织蛋白酶G和肥大细胞糜酶, 通过p38MAPK信号通路抑制肿瘤细胞凋亡[2]。此外还有研究发现SERPINB3和SERPINB4均高表达于宫颈癌、肺癌、食管癌和头颈部肿瘤等鳞状细胞癌[3]。SERPINB3和Serpin B亚家族的其他成员有2种作用方式, 一种是在细胞内通过自杀式底物机制抑制丝氨酸蛋白酶而发挥作用; 另外一种则是分泌到细胞外发挥作用[1]。本论文通过对SERPINB3基因的生物信息学分析, 为SERPINB3的功能和分子进化研究提供更多的生物信息学参考。
1 材料与方法
1.1 SERPINB3基因的核酸序列分析
1.1.1 SERPINB3基因核酸序列的获取
在http://www.ncbi.nlm.nih.gov/网站中输入SERPINB3的GenBank登录NM_006919, 得到人SERPINB3基因的mRNA序列, 对该序列阅读框架进行分析。若未获得基因的全长cDNA序列, 则应用BLAST 检索非冗余数据库或者EST数据库, 进行核酸序列拼接, 调用Human genomics+transcript数据库进行核酸序列校对, 以获得其全长cDNA序列。
1.1.2 SERPINB3基因内含子和外显子范围的界定分析和基因染色体的电子定位分析
利用UCSC基因组数据库(http://genome.ucsc.edu/)进行基因的内含子外显子分析。通过在线软件https://www.ncbi.nlm.nih.gov/genome/gdv进行基因染色体的电子定位。
1.1.3 SERPINB3基因上游的CpG岛分析和cDNA序列的限制性酶切位点分析
通过http://www.ebi.ac.uk/Tools/emboss/cpgplot/index.html在线软件对该基因的CpG岛进行分析。将获得的SERPINB3基因的cDNA序列输入到酶切位点在线分析软件(NEB cutter)或者调用DNAMAN软件, 获得该基因的酶切位点分析结果。
1.1.4 SERPINB3基因核酸序列的同源性分析
用BLAST选出与SERPINB3基因具有较高同源性基因的6个序列(人、大猩猩、黑猩猩、金丝猴、猕猴、白眉猴), 利用DNAMAN进行同源性分析。打开DNAMAN, 依次点击Sequence→Alignment→ Multiple Sequence Alignment进行多序列比对, 再输出同源树、遗传树状图等。
1.2 SERPINB3基因的蛋白质序列分析
1.2.1 SERPINB3蛋白质序列的获得和一级结构预测
将通过核酸序列拼接和校对后的SERPINB3的cDNA序列, 输入http://au.expasy.org/tools /#translate对话框中, 根据6种不同的阅读方式我们得到了6种不同的翻译结果, 选择最合适的序列, 获得SERPINB3蛋白质序列。利用在线分析软件http://web.expasy.org/cgi-bin/protparam/protparam, 预测分析蛋白质相对分子量、理论pI值、氨基酸构成和原子组成性等重要基本信息。
1.2.2 SERPINB3蛋白质二级和三级结构的预测
蛋白质二级结构是指多条肽链在一级结构的基础上进一步盘旋折叠形成的有规律的构型。访问网址http://npsa-pbil.ibcp.fr/cgi-bin/npsa _automat.pl?page=npsa_gor4.html, 将SERPINB3蛋白的氨基酸序列输入空白框, 选择运算条件, 运行程序, 获得SERPINB3蛋白的二级结构信息。访问SWISS-MODEL服务器https://swissmodel.expasy.org/interactive建立同源模型来进行蛋白质三级结构的预测,将氨基酸序列输入空白框, 选择运算条件, 运行程序, 获得获得SERPINB3蛋白的三级结构信息。
1.2.3 SERPINB3蛋白质跨膜结构、疏水性和卷曲螺旋预测
Tmpred是利用跨膜结构区段的数量、位置以及侧翼信息, 通过加权打分来进行预测。登陆http://www.ch.embnet.org/software/TMPRED_form.html, 将氨基酸序列输入对话框, 选择所需要的运算条件后运行程序,分析跨膜结构。访问服务器http://www.expasy.org/tools/protscale.html/,分析蛋白质的疏水性。访问COILS服务器http://www.ch.embnet.org/software/COILS_form.html,将氨基酸序列输入对话框, 运行程序, 选择所需要的运算条件运行程序进行蛋白质卷曲螺旋的分析与预测。
1.2.4 SERPINB3蛋白质的信号肽预测和亚细胞定位
TargetP服务器是一个预测真核蛋白亚细胞定位的软件, 主要对叶绿体转运肽, 线粒体转运肽和分泌通路信号肽的N端预测, 登陆http://www.cbs.dtu.dk/services/TargetP/对该蛋白质进行亚细胞定位。
1.2.5 利用SMART进行蛋白质结构域与功能位点的预测
打开SMART蛋白质结构域预测工具http://smart.embl-heidelberg.de/ , 选择常规模式(NORMAL), 蛋白质序列分析(Sequence Analysis)处输入SERPINB3蛋白质序列, 勾选PFAM domains(结构域家族数据库)后运行“Sequence SMART”得到SERPINB3蛋白质结构域(domains)和基序(motifs)的预测结果。
1.2.6 利用DNAMAN进行SERPINB3基因蛋白质序列的同源性分析
登录http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi, 将氨基酸序列按指定格式输入对话框, 选择所需要的运算条件并运行程序选取不同物种的序列选中后点击Download中的FASTA格式下载, 并保存为TXT格式。利用DNAMAN进行同源性分析, 打开DNAMAN, 依次点击Sequence→Alignment→ Multiple Sequence Alignment进行多序列比对, 输出同源树、遗传树状图。
1.3 SERPINB3相互作用蛋白的生物信息学分析
1.3.1 利用STRING检索SERPINB3相互作用蛋白的实验与预测数据
进入STRING数据库https://string-db.org/, 选择默认的“Protein by name”搜索模式, 输入蛋白质名称(SERPINB3)和物种名称(Homo sapiens)后进行搜索以得到SERPINB3蛋白质相互作用信息, 通过设置栏的调整可分别对实验数据和预测数据进行筛选。
1.3.2 SERPINB3相互作用蛋白的基因GO分析
运用DAVID在线工具进行SERPINB3相互作用蛋白的基因GO分析时, 输入文件为一个Gene List(基因列表), 即一群基因。如果输入文件(Gene List)的基因个数较少, 那么丧失了统计学检测效力, 在小的样本集下, DAVID只能被用来做探索性筛查。本文通过STRING数据库筛选了所有评分(score)大于等于0.4的SERPINB3互作蛋白(共165个), 将这些互作蛋白的名称生成一个Gene List 作为输入文件, 再利用DAVID在线工具对这些互作蛋白进行基因GO分析, 下载分析结果利用Microsoft Excel工作表处理并绘制富集分析结果。
1.3.3 SERPINB3蛋白质相互作用网络的分析
利用STRING数据库构建SERPINB3蛋白质相互作用网络, 进入STRING数据库https://string-db. org/, 选择默认的“Protein by name”搜索模式, 输入蛋白质名称(SERPINB3)和物种名称(Homo sapiens)后进行搜索以得到SERPINB3蛋白质相互作用网络图。所得到的默认的SERPINB3蛋白互作网络是通过置信度(Confidence score)最高的10种SERPINB3互作蛋白构建的, 置信度(Confidence score)是根据证据来源, 即实验数据、预测数据、文本挖掘、数据库导入等信息通过权重记分系统计算所得的。STRING提供了BioGRID和IntAct等互作蛋白数据库的信息, 并有相关的PubMed文献链接, 利用这些文献资料分别对SERPINB3的这10个相互作用蛋白进行简要分析[4–5]。
2 结果与分析
2.1 SERPINB3核酸序列的生物信息学分析
调用在线分析软件https://web.expasy.org/translate/对该基因cDNA序列进行开放阅读框架分析, 发现在cDNA序列的起始密码子前面, 同一阅读框架内未见终止密码子, 说明该基因的cDNA序列不完整。应用BLAST 检索非冗余数据库或者EST数据库, 以NM_006919为种子序列, 进行核酸序列拼接(Homo sapiens)。经过几次的拼接, 并通过应用BLAST 调Human genomics+transcrip数据库进行核酸序列校对, 最终得到了一段1 779 bp完整的cDNA序列。此过程又称基因的电子克隆(in silico cloning)。SERPINB3基因编码的蛋白质全长390 aa, 基因的cDNA的5’端具有Kozak(-3A/+4A)序列, 3’端具有加尾信号(attaaa)。使用在线软件http:// www.ncbi.nlm.nih.gov/genome/gdv/进行SERPINB3基因的染色体电子定位分析, 显示SERPINB3基因定位于人体18号染色体长臂上(18q21.33)。利用UCSC基因组数据库进行SERPINB3基因内含子和外显子范围的界定分析, 发现SERPINB3基因跨越80 373 285 bp, 含有16个外显子, 15个内含子, 内含子是以gt开头, ag结尾, 符合gt-ag法则(表1)。
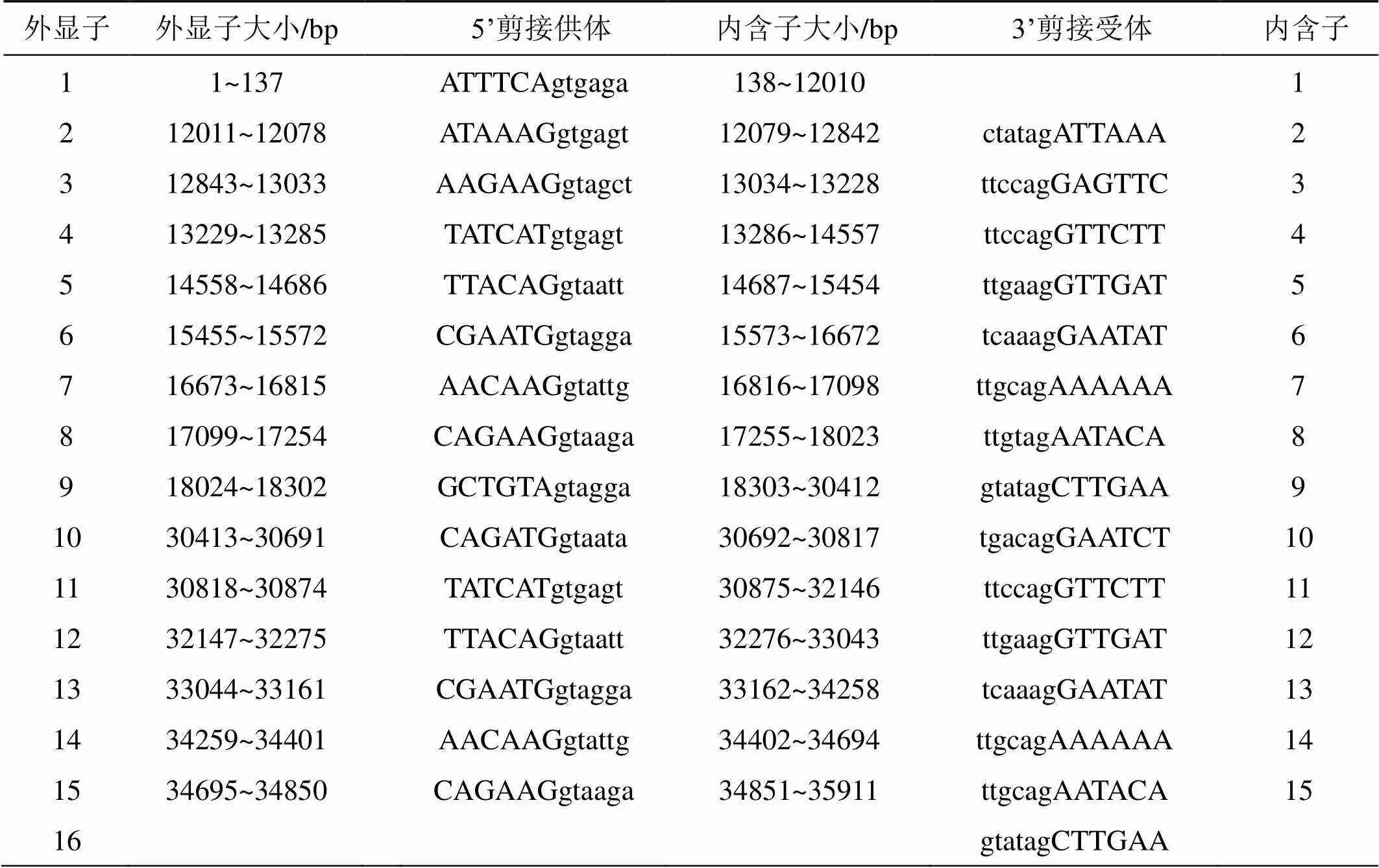
表1 SERPINB3 基因内含子和外显子范围的界定
注: 大写与小写字母分别代表外显子与内含子序列; 粗体代表保守的二核苷酸剪切位点。
利用在线工具http://www.ebi.ac.uk/Tools/emboss/cpgplot/index.html进行SERPINB3基因CpG岛分析, 虽观察值/预测值比例(Obs/Exp,Y值)大于0.6、GC含量大于50%且长度大于200 bp, 但并没有达到所期望的阈值, 因此SERPINB3不存在CPG岛。
限制性酶切位点分析通过用NEB cutter和DNAMAN的分析结果表明, SERPINB3基因的cDNA序列包含45种限制性酶切位点, 并得出了位点名称、序列、长度、出现频率和酶切位点位置。这些信息为设计引物, 基因克隆鉴定和亚克隆提供了重要依据。
利用BLAST找出与SERPINB3有较高同源性的6种生物的基因序列(人类、大猩猩、金丝猴、猕猴、白眉猴、黑猩猩), 再利用DANMAN进行多序列比对和同源性分析, 建立同源树、系统发生树。多序列比对发现6种生物基因的匹配度达92.67%; 对6种不同的生物进行同源性分析, 可得出这6个物种之间的亲缘关系的远近, 依次建立蛋白质序列同源性系统发育树, 结果表明人类与黑猩猩的亲缘关系较近, 白眉猴和猕猴的亲缘关系是最远(图1, 图2)。
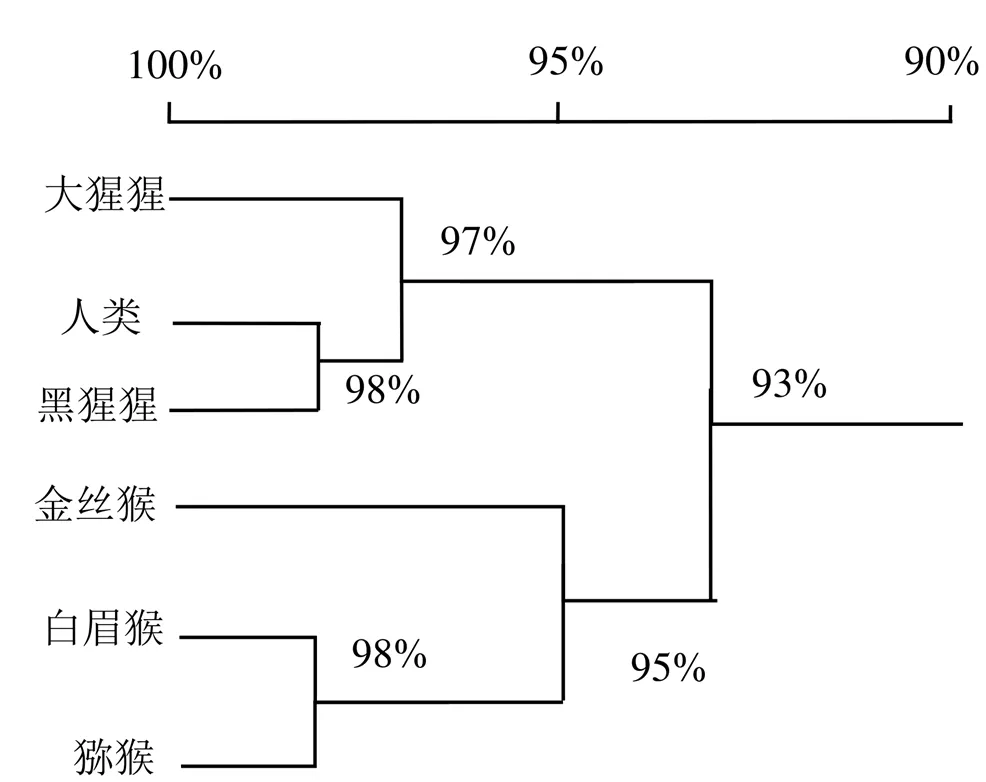
图1 核酸序列同源性分析的Homology tree
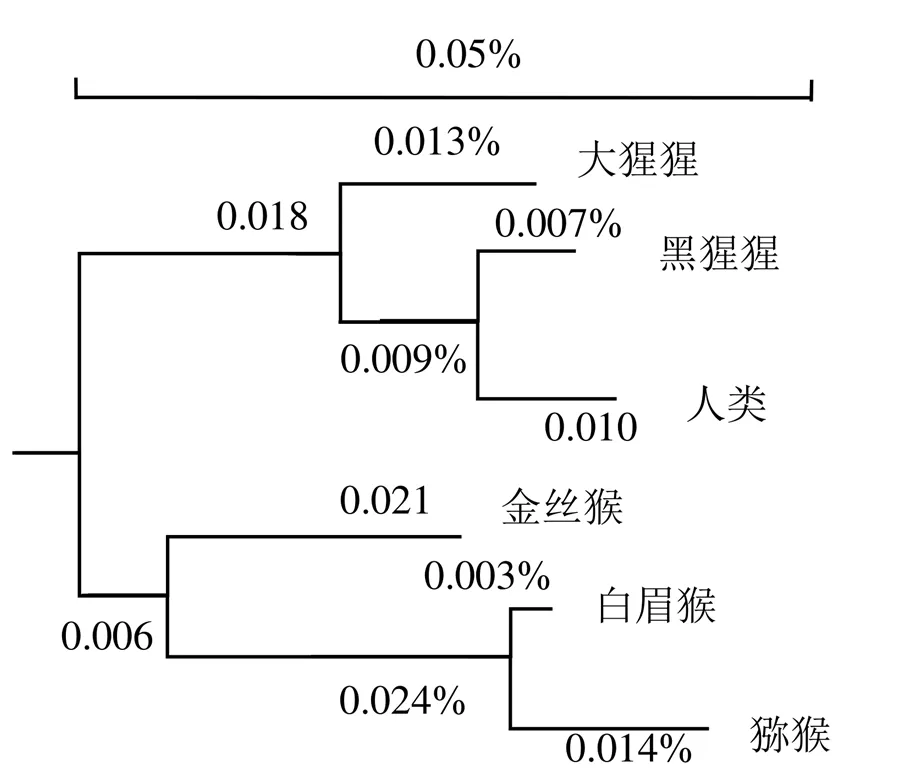
图2 核酸序列同源性分析的Phylogenetic tree
2.2 SERPINB3蛋白质序列的生物信息学分析
使用ExPASy的ProtParam工具进行分析, 结果显示, SERPINB3由390个氨基酸组成, 分子质量为44 564.60, 理论pI为6.35, 原子总数为6 252, 分子式为C2003H310N524O603S12, 且氨基酸序列的N端是 M(Met); 蛋白质不稳定系数(Instability index)为29.55(<40), 可以推测该蛋白质是不稳定蛋白, 脂肪族指数(Aliphatic index)是76.26; 该蛋白的总平均亲水性(Grand average of hydropathicity )为-0.461。
使用GOR服务器分析SERPINB3的二级结构(C表示无规则卷曲, H表示α-螺旋, E表示的是延伸链), 发现SERPINB3的二级结构由43.08%α-螺旋, 18.46%延伸链和38.46%的无规则卷曲组成, 无β-转角。其中含有较多的α-螺旋与无规则卷曲。
利用SWISS-MODEL同源建模进行三级结构的预测, 第2~390位氨基酸残基参与建模, 模板为2zv6.2.A, 两者之间的一致性可达到100%。预测结果表明, 在SERPINB3的三级结构中无规卷曲、α-螺旋分布较多, 这与二级结构中预测的结构元件分布大致相同(图3)。
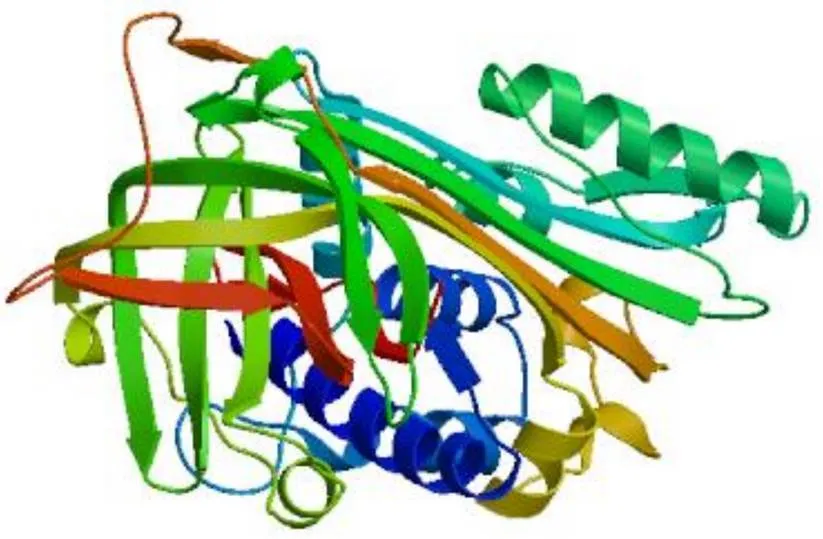
图3 SERPINB3蛋白质三级结构的预测结果及结构示意图
使用Tmpred预测蛋白质的跨膜区段和取向, TMHMM服务器预测蛋白跨膜螺旋。结果表明, SERPINB3蛋白序列的TMHs预期值为12.568, 该蛋白质存在跨膜螺旋区域。
应用COILS服务器预测serpinB3蛋白卷曲螺旋结构, 大部分含有卷曲螺旋结构的蛋白质具有非常重要的生物学功能。结果发现在250~270之间的COILS值超过0.5, 该蛋白质具有卷曲螺旋结构。
应用SMART进行结构域预测, 结果显示, SERPINB3蛋白的第6~390位之间是一个高度保守的结构功能域—SERPIN, 即Serpin蛋白家族成员所共有的结构域(决定Serpin蛋白质家族成员类似保守功能的区域), 该结构域包含一个反应位点环(RCL), 不同的RCL序列决定了Serpin家族蛋白质不同的功能; 第332~352是Low-complexity region(低复杂度区域, LCR), 即SERPINB3序列中某一特定氨基酸或少量不同氨基酸(丙氨酸A、颉氨酸V、谷氨酸E)富集的区域。
使用BLASTp分析SERPINB3蛋白序列的同源性, 再使用DNAMAN软件建立同源树。对人、黑猩猩、苏门答腊猩猩、金丝猴和苍疣猴的同源性匹配分析显示, 这5种生物的氨基酸序列的匹配率达87.29%; 对5种不同的生物进行同源性分析, 可得出这5个物种之间的亲缘关系的远近, 由图4可知人类与黑猩猩的亲缘关系较近, 而人类与苍疣猴的亲缘关系较远; 通过对4种动物的蛋白质同源性分析, 建立蛋白质序列同源性系统发育树可以得出, 人类和黑猩猩亲缘关系较近, 和苍疣猴亲缘关系较远, 人和黑猩猩属于同一类, 金丝猴和苍疣猴属于同一类, 苏门答腊猩猩单独一类, 但5种物种最终还是同归于同一大类, 这说明5个物种都是具有共同的祖先(图4)。
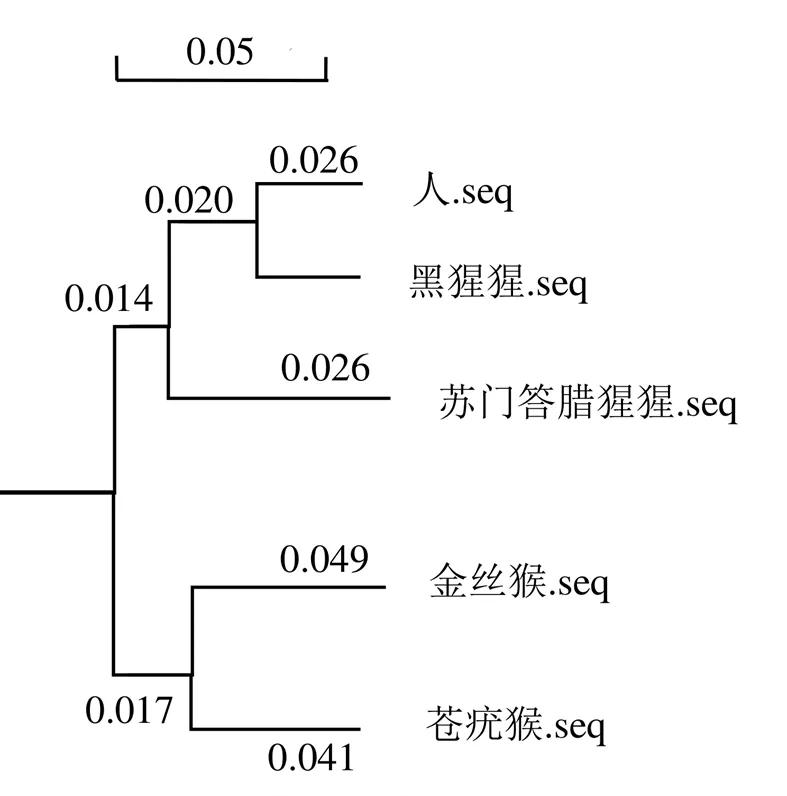
图4 SERPINB3蛋白序列同源性分析的Homology tree
2.3 SERPINB3相互作用蛋白质的生物信息学分析
2.3.1 SERPINB3相互作用蛋白的实验数据
通过STRING数据库检索, 设置数据信息来源为Experi- ments可分别在阅读栏、说明栏查看BioGRID、IntAct等数据库中记载的关于SERPINB3互作蛋白的实验证据及相关文献资料; 以同样方法调整设置, 可查看SERPINB3互作蛋白的预测数据。是通过STRING数据库“Setting”栏调整设置后, 筛选出的置信度较高的20种SERPINB3相互作用蛋白, 分别是SERPINB4、C11orf54、 PKD2L2、 FCRL5、GPR18、SH3GLB1、SUSD3、CTSS、CTSL、CTSK、EIF2B1、TPI1、MAPK8、UCP2、C16orf70、KIR3DL1、RASSF7、ASCC1、CBLB和WDR37。
利用Viewers栏的查看工具, 可查看预测的20种互作蛋白与SERPINB3的预测数据, 以下分别挑选了Gene cooccurence(基因共发生)和Gene coexpression(基因共表达)两项预测数据进行分析。
Gene cooccurence预测数据显示, SERPINB3与SERPINB4保守序列比例较高, 这表明它们在蛋白质序列上高度一致, 可能具有功能上的相似, 或者存在于同一信号或代谢通路中。
Gene coexpression三角矩阵记录了各基因间的共表达评分, 该共表达评分是基于RNA表达模式和ProteomeHD(蛋白质共调控分析工具)提供的蛋白质共调控信息计算所得。色彩强度代表共表达评分的高低, 也代表了两种蛋白质具有相关的功能的置信水平(the level of confidence)。即颜色越深, 两种蛋白质共表达评分越高, 越有可能具有功能上的相关性。左边的三角矩阵图中可以看出人SERPINB3的互作蛋白中, SERPINB4对应的颜色最深, 其下依次是S100A7和C3, 代表在人体中SERPINB3与SERPINB4、C3蛋白可能具有功能上的相关性。结果显示, 在其它物种中SERPINB3与SERPINB4、TTR、LYZ、ARG1、NPC2存在共表达。
2.3.2 SERPINB3相互作用蛋白的基因GO分析
为保证统计学检测效力, 我们利用STRING筛选了165个评分较高(≥0.4)的SERPINB3互作蛋白, 将这些互作蛋白的名称生成一个Gene List作为输入文件, 再用DAVID进行基因GO分析, 分析结果经Excel的筛选与处理后, 制作可视化图表。
Biological Process、Cellular Component、Molecular Function富集分析结果(表2、表3、表4), 结果表明, SERPINB3的互作蛋白:
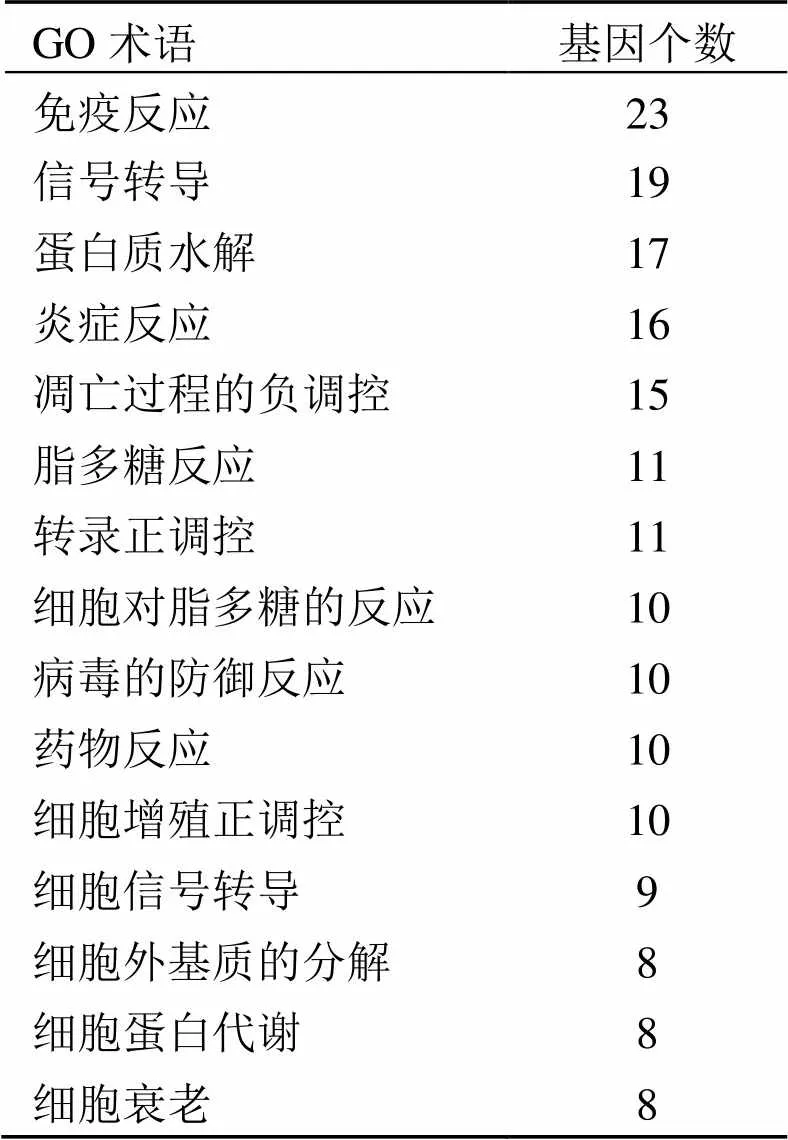
表2 SERPINB3相互作用蛋白的Biological Process(BP)分析结果
(1) 主要参与的生物学过程有免疫反应、信号转导、蛋白质水解、炎症反应、细胞凋亡过程的负调控等;
(2) 主要定位于胞外体、细胞质、细胞外间隙、细胞质基质等处;
(3) 主要行使的分子功能有蛋白质结合作用、丝氨酸型肽链内切酶活性、细胞活素活性以及酶结合作用等。
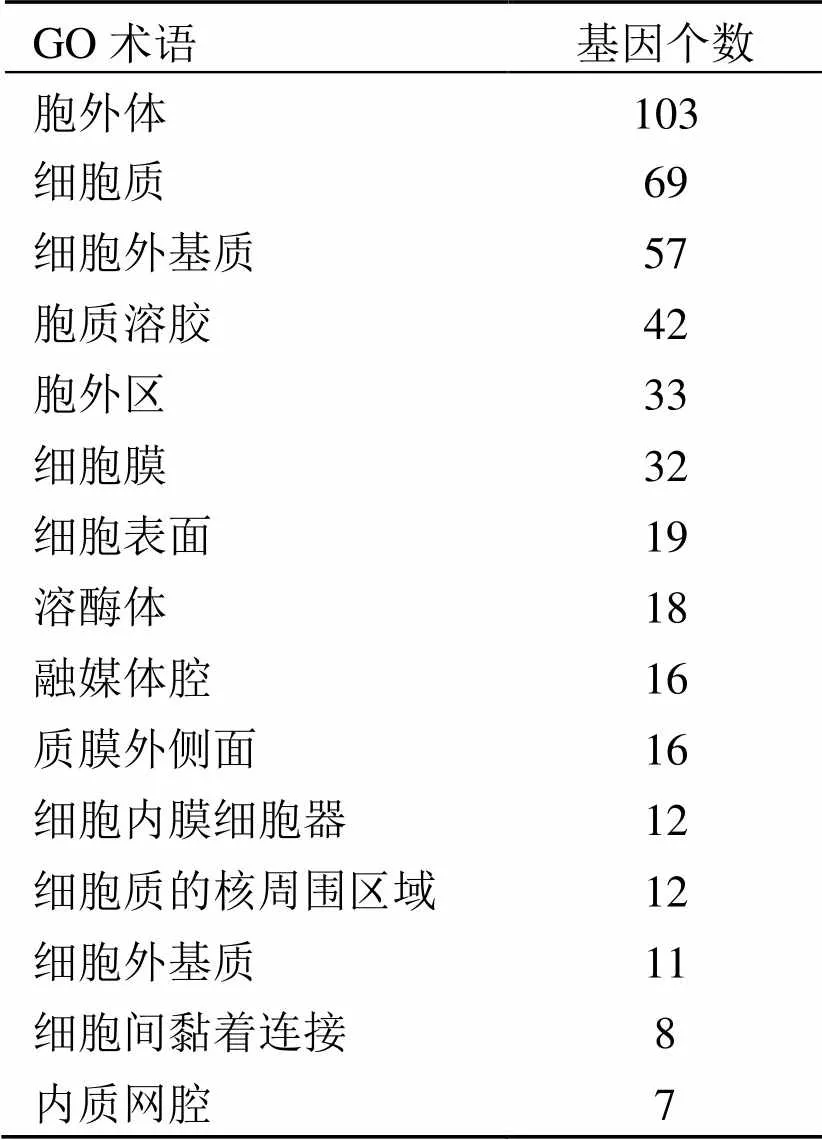
表3 SERPINB3相互作用蛋白的Cellular Component(CC)分析结果
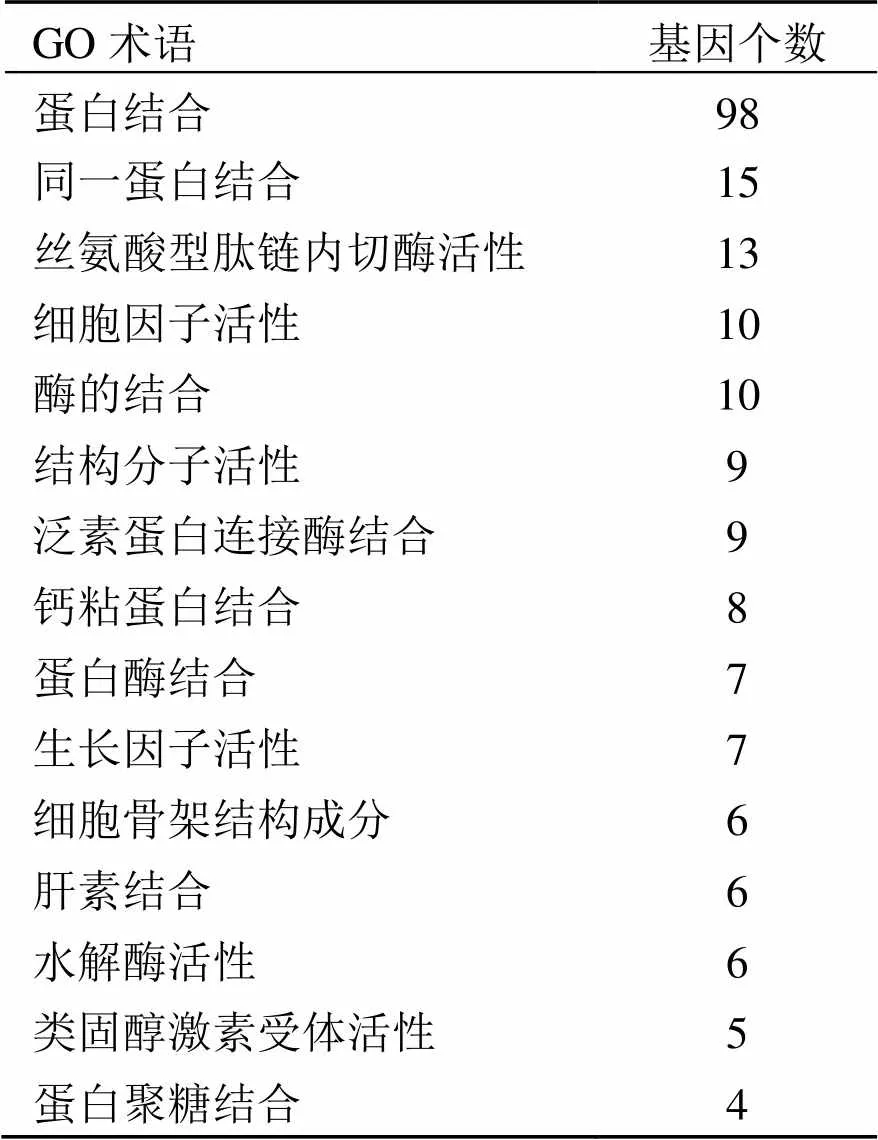
表4 SERPINB3相互作用蛋白的Molecular Function(MF)分析结果
2.3.3 SERPINB3相互作用蛋白网络的构建与分析
利用STRING数据库构建SERPINB3蛋白质相互作用网络, 所得到的默认的SERPINB3蛋白互作网络是通过置信度(Confidence score)最高的10种SERPINB3互作蛋白构建的(图5)。STRING提供了BioGRID和IntAct等互作蛋白数据库的信息, 并有相关的PubMed文献链接, 利用这些文献资料分别对SERPINB3的这10个相互作用蛋白进行简要分析。结果发现, SERPINB3的互作蛋白中, 丝氨酸蛋白酶家族成员居多, 且大多在调节炎症反应、细胞增殖分化以及凋亡等方面起着关键作用。
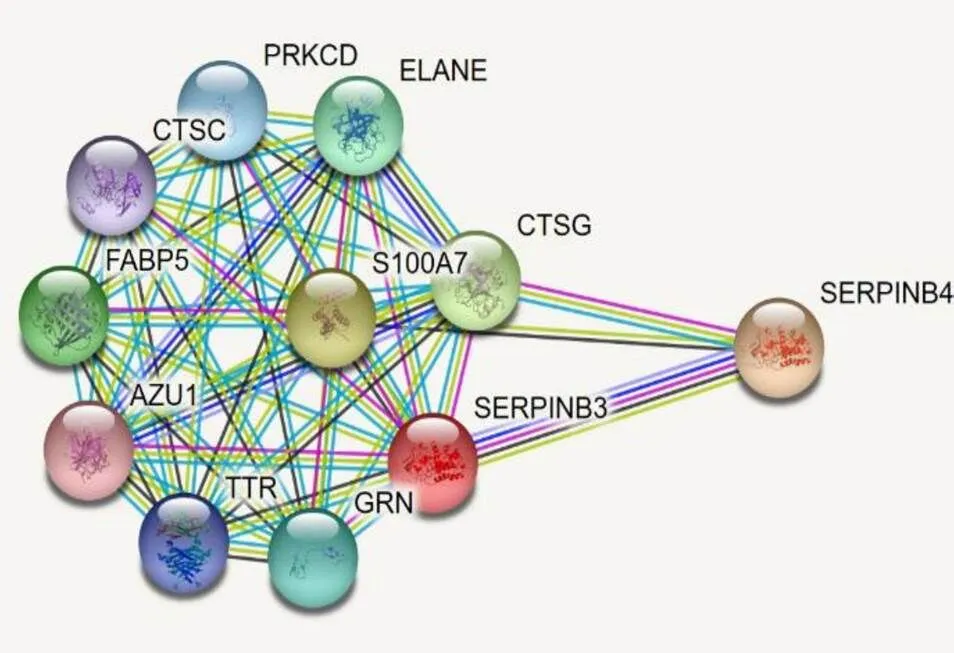
图5 SERPINB3蛋白质相互作用网络
3 讨论
早期国内外针对SERPINB3的研究多为对其本身功能的探究, 关于SERPINB3生物信息学分析的资料较少,本论文通过对SERPINB3基因的生物信息学分析, 为SERPINB3的功能和分子进化研究提供更多的生物信息学参考。我们通过基因的电子克隆得到全长1 779 bp的SERPINB3的cDNA 序列。通过DNAMAN分析出该基因包含45种酶可识别的碱基序列, 这些信息为设计引物、基因克隆鉴定和亚克隆提供了重要依据。SERPINB3核酸序列的同源性分析显示人与黑猩猩和大猩猩的亲缘关系较近, 与猕猴和白眉猴的亲缘关系最远。SERPINB3蛋白序列同源性分析显示, 人类和黑猩猩亲缘关系较近, 和苍疣猴亲缘关系较远。核酸序列同源性分析结果与蛋白质序列同源性分析结果一致。
此外,本文还预测了SERPINB3的互作蛋白, 以这些互作蛋白为研究对象进行了基因GO富集分析,筛选了部分置信度较高的互作蛋白建立SERPINB3相互作用网络, 最后针对这些互作蛋白的结构和功能进行简要分析。结果发现SERPINB3的互作蛋白大多具有蛋白质结合作用、丝氨酸型肽链内切酶活性、细胞活素活性以及酶结合作用等分子功能, 大多都参与了免疫反应、信号转导、蛋白质水解、炎症反应、细胞凋亡过程的负调控等生物学过程。SERPINB3互作蛋白网络的分析发现, SERPINB3的互作蛋白中, 丝氨酸蛋白酶家族成员居多, 且大多在调节炎症反应、细胞增殖分化以及凋亡等方面起着关键作用。SERPINB3作为一种人丝氨酸蛋白酶抑制剂[6–7], 与这些丝氨酸蛋白的相互作用, 恰恰体现出了SERPINB3的某些重要生物学意义。
[1] 刘燕华. Serpin B家族在消化系统肿瘤中的研究进展[J]. 临床荟萃杂志, 2017, 32(7): 633–636.
[2] Silverman GA, Whisstock JC, Askew DJ, etal. Human clade B Serpins (ov-Serpins) belong to acohort of evolutionarily dispersed intracellular proteinase inhibitor clades that protect cells from promiscuous proteolysis [J]. Cell Mol Life Sci, 2004, 61(3): 301–325.
[3] Sun Yu, Namratha Sheshadri, Zong Weixing. SERPINB3 and B4: from biochemistry to biology [J]. Semin Cell Dev Biol. 2017, 62: 170–177.
[4] 陈铭. 生物信息学[M]. 3版. 北京: 科学出版社, 2018.
[5] 吴祖建, 沈建国. 生物信息学分析实践[M]. 北京: 科学出版社, 2010.
[6] 樊静. 丝氨酸蛋白酶抑制剂B亚家族[J]. 生命的化学杂志, 2003, 23(4): 275–276.
[7] SERPINB3 (serpin peptidase inhibitor, clade B (ovalbumin), member 3) [J]. Atlas Genet Cytogenet Oncol Haematol, 2015; 19(3): 202–209.
Bioinformatics analysis of SERPINB3 gene
Li Jingfei, Xu Wei, Zhang Shun, Li Feng
(Biology and Environmental Science College, Hunan University of Arts and Science, Changde 415000, China)
SERPINB3 can inhibit cysteine protease, has anti-apoptosis, promote cell proliferation and migration, and is closely related to the occurrence and development of many tumors. The full-length cDNA sequence (1 779 bp) of the SERPINB3 gene was obtained by in silico cloning, which encodes a protein composed of 390 amino acids. Nucleic acid sequence analysis showed that the SERPINB3 gene is located on the long arm of human chromosome 18 (18q21.33), the genome spans 80 373 285 bp, contains 16 exons, 15 introns, and a total of 45 restriction sites on the cDNA sequence point. The homology analysis showed that humans are closely related to chimpanzees, and are the farthest to humans and white-browed monkeys and macaques. The sequence homology analysis of SERPINB3 protein showed that humans are closely related to chimpanzees, and farther related to colobus monkeys. The bioinformatics analysis of SERPINB3 interacting proteins showed that among the proteins that interact with SERPINB3, members of the serine protease family are the majority, and most of them play a key role in regulating inflammation, cell proliferation, differentiation, and apoptosis. This thesis provides more bioinformatics references for the study of the function and molecular evolution of SERPINB3 through the bioinformatics analysis of the SERPINB3 gene.
SERPINB3; gene; bioinformatics analysis
Q 785
A
1672–6146(2021)02–0031–07
10.3969/j.issn.1672–6146.2021.02.007
李峰, youquanli@126.com。
2020–6–10
湖南文理学院大学生研究性学习和创新性实验计划项目(校办通[2017]19号)。
(责任编校: 郭冬生)
