级联稀疏卷积与决策树集成的病理图像细胞核分割方法
2021-04-18宋杰肖亮练智超
宋杰 肖亮 练智超
复杂背景病理图像个体细胞分割是在成千上万个体细胞汇集的图像中逐个分割出具有相对完整边界的细胞,为后续细胞形态计算和病理特征提供定量分析.传统的病理切片分割方法,需要经过专门训练的病理医师在图像中逐个寻找感兴趣区域(Regions of interest,RoI),而后根据专业知识分析诊断.一张病理切片通常包含成百上千个细胞或细胞核,这给医师带来很大的工作负担,疲劳阅片现象时有发生[1].虽然目前已有很多病理细胞图像处理方法,但这些方法大多只关注于特定类型或单一器官的细胞或细胞核分割.因此,临床和医学研究迫切需要能够进行多个器官和疾病状态的细胞核病理图像高精度分割方法[2].然而,如图1所示,个体分割具有如下挑战:一方面,对于病理状态(例如增生或某种癌症亚型)图像而言,由于细胞核增大,并呈现染质浓集贴边,即核內染质较浅,而边缘附近染质较深;而着色较深的核仁也大量出现在核内.另一方面,由于病理图像中往往细胞密度高、细胞间出现重叠和成团等突出问题,加剧了个体细胞分割的难度.目前方法体系主要分两类:传统基于人工特征的图像分割方法和基于表示特征学习的图像分割方法.

图1 病理细胞核图像分割的挑战与人工分割结果Fig.1 Challenges in nuclear segmentation and associated ground truth
传统图像分割方法包括:水平集方法、图论方法和分水岭方法等.水平集算法是目前比较流行的分割方法.其基本模型包括两类:基于边缘的水平集模型[3]和基于区域的水平集模型[4].近年来,基于后者,很多改进的方法[5]被提出来广泛应用于病理图像的细胞分割中.在现有方法中,基于图论的分割方法是病理图像分析中应用最为广泛的技术之一.最常用算法是最小割算法[6],除了主流的图割方法,其他类型的图模型也已经应用到个体细胞核的分割,例如属性关系图模型[7].另一类基于图像分析的代表性方法是分水岭算法,分水岭算法通常期望目标细胞核内灰度分布均匀,并且目标与背景具有明显灰度或颜色差异.图像中的噪声、目标内部灰度变化,都会产生过度分割的现象.为此,人们提出若干标记控制的分水岭及其变种算法[8−10].传统图像分割方法为了得到精确的个体分割结果,通常需要分析RoI特有属性来设计额外的后处理步骤,从而导致算法可迁移性较差.
虽然深度学习方法能较好地处理病理图像细胞核表观多样性的分割问题,但由于网络架构、网络复杂性以及超参数的影响,使其分割性能受到限制.针对病理图像细胞核表观多样性的分割问题及深度神经网络的局限性,国内外研究者提出了系列具有较小参数规模的快速浅层分割学习模型.与现有深度学习模型相比,浅层学习模型无需非线性激活和后向传播计算,且学习模型参数规模较小.传统浅层分割学习方法[11−12]为了得到密集预测结果,通常使用像素周围的一个图像块作为模型的输入用于训练和预测.由于只能提取一些局部的特征,从而导致分割的性能受到限制.而基于卷积操作的浅层学习模型[13−18]则是从抽象的特征中恢复出每个像素所属的类别,从而可以实现比传统模型更加精确的分割结果.然而,这类浅层分割学习模型没有充分考虑像素间的局部依赖关系,因此对图像细节特征快速捕获和紧致表达能力有待加强.此外,在传统集成决策树学习算法中最先学习得到的一两棵树对预测结果的影响最为显著,这使得整个模块对这些树所做出的决策过于敏感,容易产生过拟合现象.
为了解决这些问题,本文的主要贡献是:
1)提出了级联稀疏卷积与决策树集成(Cascade sparse convolution and decision tree ensemble,CscDTE)学习模型,该模型没有非线性激活和后向传播计算,且学习模型参数规模较小,其特征学习过程具有一种替代深度神经网络的新型学习机制;
2)采取多层稀疏可分离卷积特征学习捕获图像上下文特征;采取秩-1张量分解[19]的可分离卷积加速特征学习过程;
3)建立集成决策树学习的正则化回归模型,采取局部二阶近似逼近优化决策树,提高分类回归泛化性能;
4)在乳腺、前列腺、肾脏、胃和膀胱等多组病理图像的分割实验表明该模型优于目前CNN2、CNN3和U-Net等深度学习方法,对于病理图像分割具有较好应用前景.
1 相关工作
1.1 浅层特征学习方法
浅层特征学习分割方法中代表性方法包括稀疏编码方法[11,13]以及多层稀疏卷积特征学习方法[15−18].相比标准的卷积滤波器组来说,稀疏可分离卷积滤波器组能够在不影响其性能的情况下大大降低计算复杂度.例如Sironi等[13−15]通过学习一组可分离卷积滤波器有效提取图像中曲线状奇异边缘结构.具体地,稀疏卷积滤波器学习方法是利用一组样本,通过利用稀疏编码方法[20−21]学习J个卷积滤波器
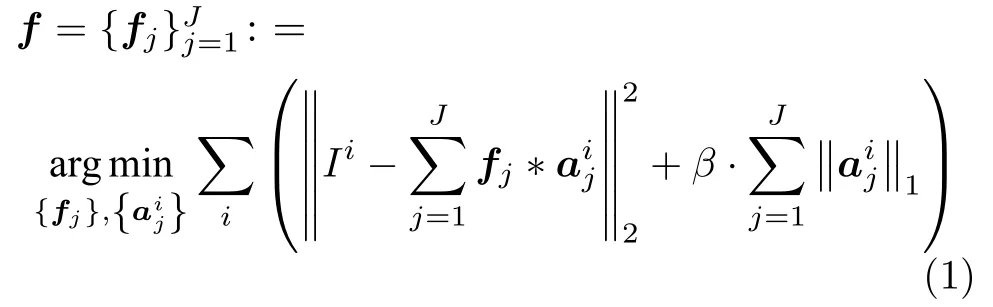
1.2 深层特征学习方法
随着深度学习的兴起,研究者提出了系列深层特征表示学习的病理图像分割方法[23−27].代表性网络包括卷积神经网络(Convolutional neural networks,CNN)[24−25]和全卷积网络(Fully convolutional network,FCN)[26−27].深度学习方法为解决细胞核表观多样性的图像分割问题提供了有效途径.通常,CNN网络在卷积层之后会接上若干个全连接层,将卷积层产生的特征图映射成一个固定长度的特征向量,以实现图像级的分割任务[28](见图2).例如,Xing 等[24]针对组织病理细胞核图像,构建了两个卷积—池化层对和两个全连接层构成的CNN2模型,实现了端到端的模型训练,进而对测试图像的细胞核进行分割.在此基础上,Kumar等[25]提出了更深的CNN3模型,实现了更广泛病理图像的分割.与CNN不同,FCN可以接受任意尺寸的输入图像,采用反卷积层对最后卷积层的特征图进行上采样,使它恢复到输入图像相同的尺寸,从而可实现逐像素预测,并可保留原始输入图像的空间信息,最后在上采样的特征图上进行逐像素分类[29],完成像素级的分割.例如,Zhang 等[26]通过结合FCN和基于图论的方法,提出了一种宫颈癌细胞核分割新方法.最近,Ronneberger等训练了一种特殊的UNet网络结构[27](见图3),通过在收缩路径上捕获全局特征和在扩展路径上实现精确定位,较好解决了复杂神经元结构的分割问题.然而,深度学习方法需要较大规模的训练数据集以经验拟合深层网络参数.
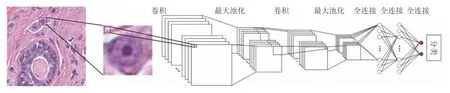
图2 用于病理图像分割的CNN体系结构Fig.2 The CNN-style architecture for pathology image segmentation

图3 用于病理图像分割的U-Net体系结构Fig.3 The U-Net-style architecture for pathology image segmentation
目前,Zhou等提出了一种多粒度级联森林方法,称为gcForest[12].这是一种采取非神经网络,而采取决策树集成的方法,性能较之深度神经网络有很强的竞争力.深度神经网络需要花大力气调参,相比之下,gcForest方法更容易训练得多.实际上,在几乎完全一样的超参数设置下,gcForest在处理不同领域的不同数据时,也能达到极佳的性能.
2 本文方法
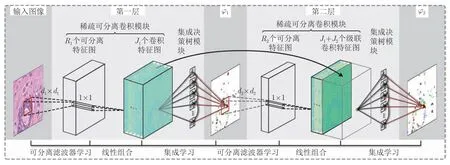
针对病理图像细胞核表观多样性的分割问题以及深度神经网络的局限性,本文提出了一种复杂病理细胞核分割的级联稀疏卷积与决策树集成(CscDTE)学习模型,如图4所示.假设(m,y)表示训练样本集,其中,是从输入图像I提取的特征图集,f1是第一层学习得到的卷积滤波器组,y是尝试预测的目标.CscDTE框架旨在渐进地提供一种非线性模型φh(m)拟合数据样本,其中,h表示模型参数.
2.1 稀疏可分离卷积特征学习
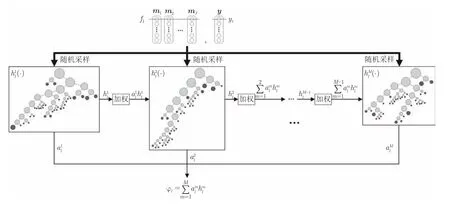
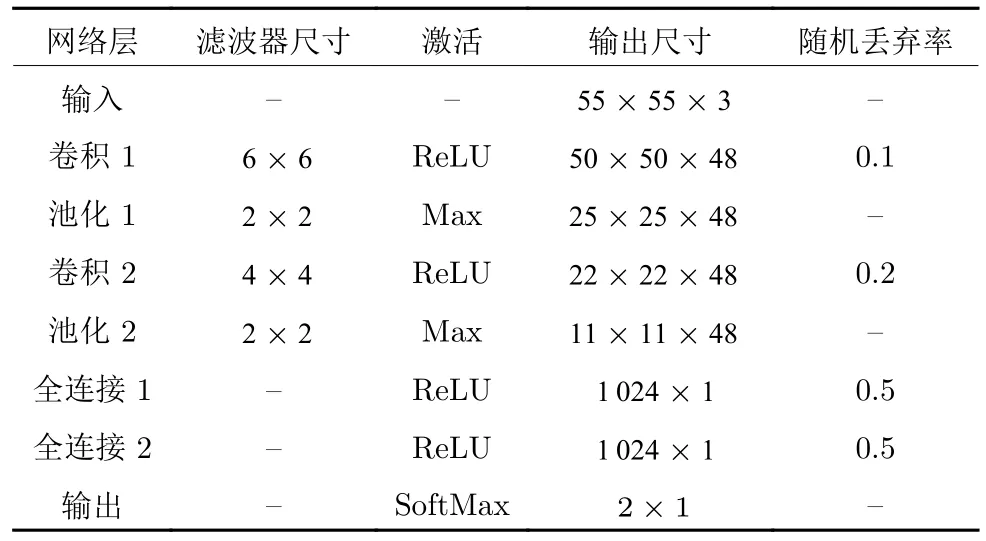
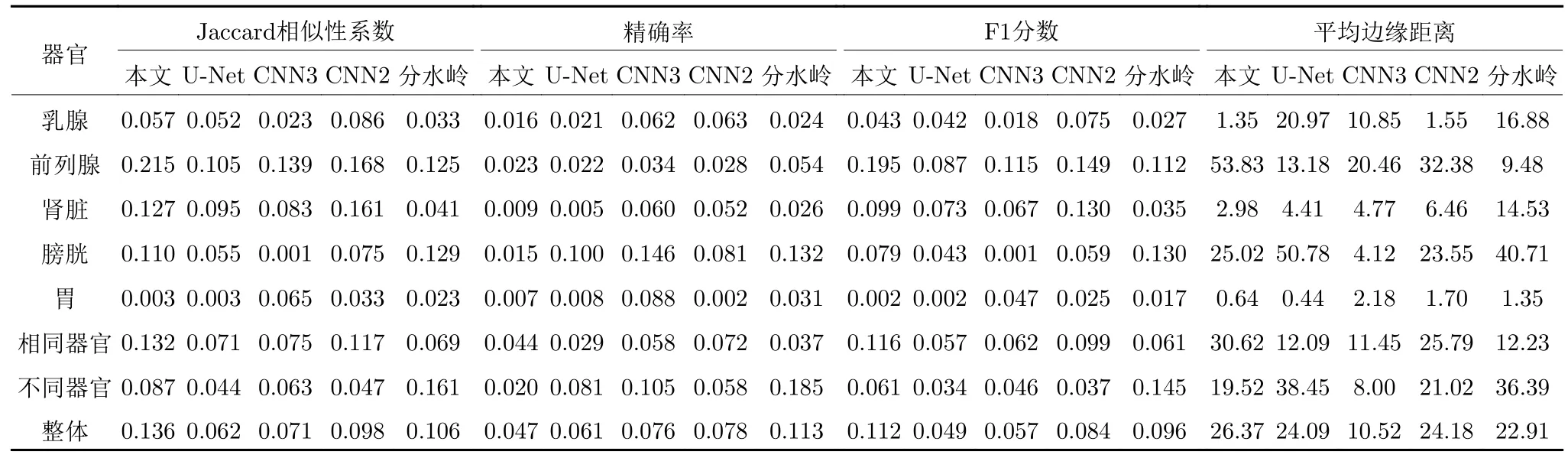
一般而言,fj是满秩的,当卷积较大图像时,计算耗时.如果将这些卷积滤波器堆叠成一个三维张量Z∈Rd×d×J,其中,Z的第j个切片对应第j个卷积滤波器fj,可发现该张量存在大量冗余特征.为此,为了学习可分离核,本文进一步引入低秩张量分解技术[19],利用一组秩为1(秩-1)张量的线性组合来近似Z,从而实现可分离卷积核,加速特征计算.该方法基于如下假设:一个秩为R的滤波器fj总能由一组秩-1可分离核:线性表示:为标量权重.此时所有的fj共享同一组秩-1可分离核,只有权重系数因不同的fj而改变,且R 其中,ar和br均是长度为d的向量,cr是一个长度为J的向量,符号◦表示张量积.式(2)的求解可通过使用共轭梯度下降法[30]来实现.如图5所示,可分离核kr可由典范多元分解(Canonical polyadic decomposit ion,CPD)成分ar和br给出,即kr=ar◦br.用于重构卷积滤波器fj的权重则由向量cr的第j个成分给出.为了捕获不同类型的图像特征,本文相应学习多个可分离核组.归于可分离卷积核的学习机制,每个kr都能分解成一个水平滤波器和一个垂直滤波器.因此,为了获得m,首先使用对图像I进行快速卷积,生成一组可分离特征图.然后,针对每个像素位置,将这些R维向量映射到一个J维向量.这等价于在可分离特征图上应用J个空间维度为1×1的滤波器[31]. 这样,基于秩-1张量分解学习机制,本文实现了病理细胞核的多尺度方向分布式抽象特征的分层抽取.模型的每层始于稀疏可分离卷积核组的训练及相应卷积特征图的计算.然后以卷积特征作为样本输入,通过集成决策树学习的正则化回归模块生成得分图预测结果,如图4所示. 图4 两层CscDTE学习模型示例Fig.4 Example of two-layer CscDTE architecture 图5 基于张量分解技术学习一组秩-1可分离核Fig.5 Tensor decomposition for learning rank-1 separable kernels 令fi=f(xi,I)∈RJ表示输入图像I中像素点xi对应的特征向量,则从给定的样本集(m,y)中随机采样N对训练样本集CscDTE框架中的集成决策树学习模块的目标是学习M棵既有预测能力又相对简单的决策树(见图6):并使用这M棵决策树的组合来预测每层的输出: 其中,第l层第m棵决策树(·)定义如下: 图6 CscDTE框架中的集成决策树学习模块的训练过程Fig.6 Flowchart of the training procedure for the decision tree ensemble learning module of CscDTE framework 所构建的CscDTE框架由L−1个隐层和1个最终输出层构成,且每层包含一个稀疏可分离卷积模块和一个集成决策树学习的正则化回归模块.不妨将第l层的输出定义为φl=[φl,1,φl,2,···,φl,n]T,其中,n为图像的像素个数.CscDTE模型将模块对进行堆叠,这样上层集成决策树学习模块的得分图输出为下层稀疏可分离卷积模块的输入.对于给定参数集合hl,CscDTE模型的前向生成过程为: 其中,nl表示从第l层输出结果φl提取的特征图集,且作为第l+1层集成决策树学习模块φl+1(·)的输入. 如图7 所示,由于低级图像表观特征占主导作用,较浅层对背景杂斑和细胞核内不均匀的染质较为敏感.因此,本文进一步引入高级上下文特征[35].具体地,从得分图中训练一组新的稀疏可分离卷积核并获取新的上下文卷积特征图集nl.为了实现更好的预测,采用级联卷积特征[m,nl]代替nl作为新层集成决策树学习模块的样本输入.重复这一过程L次,最终获得一个最大程度近似目标区域的输出(见图7(c)).同时,由于卷积特征的级联将产生分布式冗余特征,为此,本文采取了如下两个策略:1)在每层训练之前,从输入样本中随机采样新的像素位置以构建新的训练集;2)随机采样这些像素对应的卷积特征. 图7 基于本文提出的CscDTE模型的分割改进Fig.7 Improvement obtained by our CscDTE model 本节首先介绍用于测试分割方法的病理图像数据集,并在此基础上,给出所提出的CscDTE模型的最优超参数设置.然后,我们描述评估指标,并对比标记控制的分水岭分割方法[10]、CNN 2[24]、CNN3[25]、U-Net技术[27]以及CscDTE模型的分割性能及参数规模. 癌症基因组图谱(The cancer genome atlas,TCGA)是被广泛用于细胞核分割的病理数据集,因为它覆盖了多家医院、多位病人、不同器官以及疾病状态信息.为了最大化细胞核表观多样性,本文使用Kumar等采集并标注的TCGA全切片图像(Whole slide images,WSIs)集[25]作为算法的验证和比较.其中,每张图像大小为1 000×1 000.为了便于算法的验证和比较,采用Kumar等的方法将整个数据集拆分成三个部分.第一部分用于训练和验证,对应12位病人,3个器官,包括4 张乳腺病理细胞核图像、4张前列腺病理细胞核以及4 张肾脏病理细胞核图像,总共有11 460个细胞核.因此,不管是基于图像块级分类的分割方法还是基于像素级分类的分割方法,即使不对数据进行扩充,这个数目也足以使它们训练出相应的学习系统,并产生预测的分割结果.第二部分用于相同器官的测试,对应不同病人,相同三个器官,图像总数为6幅.大多数已有的细胞核分割学习算法只局限于同一器官的训练与测试,因此,这一部分能够对模型的泛化能力进行有效的评估.第三部分用于更具挑战性的不同器官的测试,对应2个器官,包括2 张胃病理细胞核图像和2张膀胱病理细胞核图像. 为了进一步验证本文方法的有效性,本文也采用肾细胞癌(KIdney renal cell carcinoma,KIRC)病理数据集[36]来训练相应CscDTE模型,并分析分割性能.来自TCGA数据门户网站的KIRC病理数据集包含了不同类型的WSIs,涵盖了一定范围的KIRC 病理分级.实验中,18 幅图像用于训练,20 幅图像用于测试,且平均每幅图像具有96个细胞核,大小为400×400. 基于验证集中的样本,采用留一交叉验证方法[37]来确定CscDTE模型关键超参数的最优值,包括训练样本数量、集成决策树学习模块尺寸、CscDTE模型尺寸以及稀疏可分离卷积滤波器尺寸和数目.表1列出了在TCGA WSIs病理数据集上所提出CscDTE模型的最优超参数值.在训练过程中,随机采样四分之一的像素位置用于集成决策树学习模块的训练,在计算每棵树的响应之后,进而使用所有的位置样本来学习式(3)中的权重 为了与深度学习技术进行对比,本文研究了三种不同的网络体系:CNN2[24]、CNN3[25]和U-Net[27],并在NVIDIA GeForce GTX 1080 Ti®图形处理单元上使用Tensorflow 框架训练所有的网络模型. 表2和表3分别给出了用于复杂TCGA WSIs病理图像分割的CNN2和CNN3体系结构,包括连续的卷积—池化层对以及若干个全连接层.最后一层是Softmax层,旨在基于学习到的特征预测输入图像块属于每一类的概率.Xing 等[24]采取两个卷积层和两个全连接层,而Kumar等[25]则采取三个卷积层和两个全连接层,因为加入更多的层并不会显著提高网络的分割精确度,反而会增加计算时间.为了获得最好的分割性能,经验设置其他的超参数值,包括卷积层中的滤波器尺寸和数量、隐层节点数目、以及输入输出尺寸等.图3给出了用于复杂TCGA WSIs病理图像分割的U-Net体系结构[27].该体系结构由编码器和解码器组成.编码器包含由修正线性单元(Rectified linear unit,ReLU)实现的填充卷积操作,共两层.每两个卷积层之后,有一个步长为2的最大池化操作.每次最大池化层下采样之后,特征通道数加倍.在解码器中,每两个卷积层之前,有一个 2×2 的上采样操作,且其输出与来自相应编码器部分的特征相结合.最后两层分别是1×1卷积操作和像素级Softmax 输出,其中,1×1卷积层用于将特征图的通道数降至所需的类别数.在本文的U-Net网络实现中,代替数据扩充,只是随机提取了图像块,不过仍然实现了令人满意的分割性能.本文使用Tensorflow深度学习框架在TCGA WSIs病理数据集的所有样本上对CNN2、CNN3和U-Net网络均训练了95次(Epochs).针对每个网络,分别从12幅训练图像和3幅验证图像中提取了158 400个图像块和32 000个图像块用于训练和验证,其中包括相等数目的以细胞核像素为中心的正样本块和以非细胞核像素为中心的负样本块.另外,基于TCGA WSIs病理数据集,比较实验分析了标记控制的分水岭分割方法[10]. 图8和图9显示了在5个器官和疾病状态下,病理细胞核图像的分割算法对比.图8和图9中第(a)~ (g)列分别是原始图像、人工分割结果、CscDTE模型分割结果、U-Net网络分割结果、CNN3网络分割结果、CNN2网络分割结果和标记控制的分水岭分割结果.需要注意的是,考虑到不同类型病理图像颜色的异质性,在分割模型训练之前,将原始图像从RGB颜色空间转换到CIE LUV 均匀颜色空间并作线性归一化.可以看出,所提出CscDTE模型能有效应对细胞核大小、形状和方向的变化,并最大程度地检测正确细胞核的数目.另外,针对存在染色质稀疏、重叠和复杂背景杂斑的情形,例如,部分前列腺病理图像和膀胱病理图像,CscDTE模型也表现出了良好的鲁棒性(见图10).相比之下,虽然U-Net通过融入上下文信息能较好地避免复杂背景杂斑的干扰,但由于网络深度的影响,仍然对染色质稀疏的细胞核内细节不够敏感.CNN2利用各向同性区域生长从网络输出中获取细胞核种子标记,因而易受细胞核形状的影响,尤其是带分割目标存在重叠和成团的情况,产生欠分割的现象.CNN3使用各向异性区域生长代替CNN2中的各向同行区域生长,虽然增加了对重叠、成团分割的鲁棒性,但其受限于图像块级的分类.由于只能提取局部特征,导致其分割精度相比于CscDTE模型较低.作为传统图像分析的方法,分水岭在分割灰度均匀的孤立细胞核时,表现得相当优异,例如图8中的肾脏细胞核分割.然而,对于Kumar病理数据中复杂背景图像,其分割性能遭受大幅度的下降.提出的CscDTE模型在训练和预测时间上也优于基于CNN和FCN的体系结构,训练所有的细胞核仅花费了两个小时左右. 表1 提出的CscDTE模型的最优参数值.像素位置样本总数为800 000Table 1 The optimal hyper-parameter values of our CscDTE model.The total number of pixel samples is 800 000 表2 用于病理图像分割的CNN2体系结构Table 2 The CNN2 architecture for pathology image segmentation 表3 用于病理图像分割的CNN3体系结构Table 3 The CNN3 architecture for pathology image segmentation 图8 Kumar 病理细胞核相同器官图像的分割比较Fig.8 Comparative segmentation results on the Kumar same-organs testing pathology images 图9 Kumar 病理细胞核不同器官图像的分割比较Fig.9 Comparative segmentation results on the Kumar different-organs testing pathology images 图10 不同算法在具有重叠、形状及大小不规则的病理图像上的轮廓可视化分割结果图Fig.10 Visualizing segmented contours of different algorithms on the pathology image with shape and color variations and overlapping regions 图11显示了通过使用KIRC数据集对比细胞核分割算法的可视化结果.图中第1,5、2,6、3,7和4,8行分别是原始图像、人工分割结果、本文方法分割结果以及U-Net网络分割结果.提出的Csc DTE模型提供了一种定义复杂形式假设φ(·)的级联方式,并使用其最优参数拟合病理数据.本文方法使得模型在训练过程中能够获取足够多的上下文信息.如图11所示,相比U-Net,CscDTE模型驱动的拟合重构能够起到信号正则化的作用.因此,即使当细胞核出现密集重叠以及形状、大小等变化时,提出的分割方法也仍然有效. 为了定量评估分割算法的性能,本文分别使用Jaccard相似性系数、精确率、F1分数和平均边缘距离(ABD1ABD是基于像素级边缘的评价方法,表示自动分割与人工分割目标边缘间的平均距离,且漏分割和误分割会造成ABD值过大.)三个指标来比较算法输出的掩模P与Ground Truth 掩模R,计算如下 图11 本文方法与U-Net方法在KIRC数据集上的分割比较Fig.11 Comparative segmentation using ours and U-Net on the KIRC dataset 表4 本文方法与其他方法在Kumar 病理数据集上的分割性能(均值)比较Table 4 Performance comparison of other methods with our CscDTE(For each metric,the mean are listed) 表5 本文方法与其他方法在Kumar 病理数据集上的分割性能(标准差)比较Table 5 Performance comparison of other methods with our CscDTE (For each metric,the standard deviation are listed) 其中,PB和RB分别对应P和R的边缘.表4和表5给出了在TCGA WSIs病理数据集上不同算法实现的定量分割结果.可以看出,相比其他方法,提出的CscDTE模型在不同器官图像和整个数据集上对三个指标值的改进明显.由于本文方法更加有效地级联低级图像表观特征和高级上下文特征,并结合集成决策树学习模块的使用,提出方法在Jaccard 相似性系数和F1分数整体值上分别超出表现最好的U-Net和CNN3方法4.4 %和2.7 %,同时在平均边缘距离值上相比U-Net减少了大约4.8个像素.另外,定量分割结果也反映出U-Net和CNN3在相同器官和不同器官测试方面均强于CNN2,而在前列腺病理细胞核图像上,所提出方法略逊于U-Net和CNN3.需要注意的是:1)虽然标记控制的分水岭分割算法在相同器官测试上,超过CNN2的性能,但它们的分割精度较差,造成了大量的误分割结果;2)本文算法无需非线性激活和后向传播计算,虽然性能较之深度神经网络有很强的竞争力,但由于端到端训练的简易性,因而当染质稀疏较为严重时,分割结果会有较大差异.为了便于模型的显著性差异分析,本文进一步采用双样本t-检验统计指标[38]来对不同方法进行比较.基于F1分数,虽然提出的CscDTE模型在整个Kumar数据集上与U-Net模型、CNN3模型、CNN2模型、分水岭模型之间没有显著性差异发生的概率(p值)分别为0.491、0.493、0.197、0.110,但在不同器官图像上却有本质区别,概率分别为0.001、0.141、0.018、0.021.表6给出了本文方法和U-Net方法在KIRC测试图像上的各指标均值和标准差.可以看出,CscDTE模型的分割性能明显优于表现最好的UNet模型,且具有更好的可靠性. 对于逐像素分割任务而言,学习模型的参数规模是另一个重要的性能评估指标.学习模型中带参数的层主要有卷积层、批归一化层以及全连接层等,而激活函数、池化层和上采样层旨在提供一种非线性变换,无需学习.本文采取秩-1可分离卷积核代替卷积滤波器,且无需全连接层特征映射,参数规模较之传统深度学习方法较小,如表7 所示. 本文提出了一个级联稀疏卷积与决策树集成学习(CscDTE)的细胞核分割模型.常用的分水岭分割方法和深度学习网络对算法初始化和病理图像数据的多样性较为敏感.与之相比,CscDTE模型具有四个方面的优势: 1)为了实现鲁棒特征的提取和CscDTE模型算法的加速,基于秩-1张量分解学习机制,CscDTE模型在各层引入稀疏可分离卷积模块,分层抽取细胞核的多尺度方向分布式抽象特征; 2)为了提升模型的分割性能,充分考虑像素间的关系,CscDTE模型使用级联卷积特征作为样本输入,迭代有监督地训练集成决策树模块直到收敛; 3)为了增强集成决策树模块对数据复杂性和可变性的自适应能力,Csc DTE模型采取随机采样、树剪枝以及正则化回归机制提升逐像素分布式特征集成回归分类能力; 4)不同器官和疾病状态的复杂病理数据集上的大量试验表明了所提出的CscDTE方法相比于已有最先进技术的优越性. 表6 本文方法与U-Net在KIRC数据集上的分割性能(均值,标准差)比较Table 6 Performance comparison of U-Net with our CscDTE(For each metric,the mean and standard deviation are listed) 表7 不同算法在病理数据集上的参数规模Table 7 Number of parameters of different algorithms on the pathology dataset 在现有框架的基础上,将利用更先进的预测模型对集成决策树学习模块进行替换,以实现更好的分割性能;将结合语义分割,在应用层面对CscDTE其进行拓展;将探索管状显微结构的提取与重构,例如血管、神经突等,并拓展至三维医学图像应用.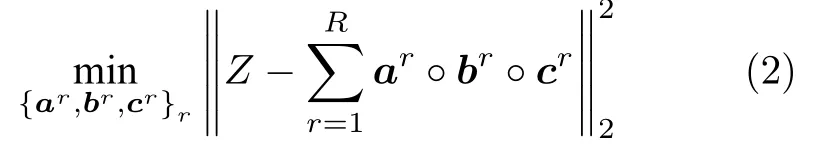
2.2 集成决策树学习的正则化回归模型



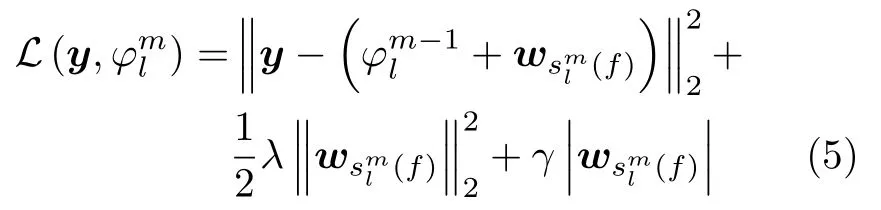
2.3 局部二阶近似优化



2.4 CscDTE学习模型
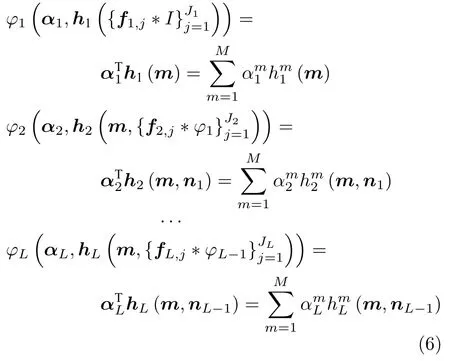

3 实验结果及分析
3.1 数据集与最优超参数设置
3.2 分割比较

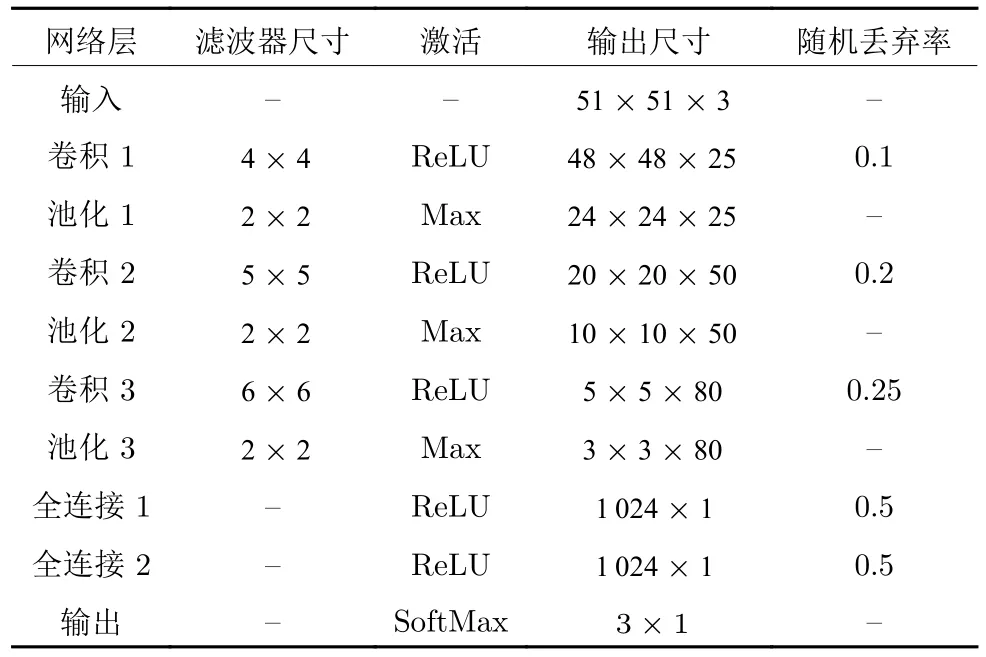

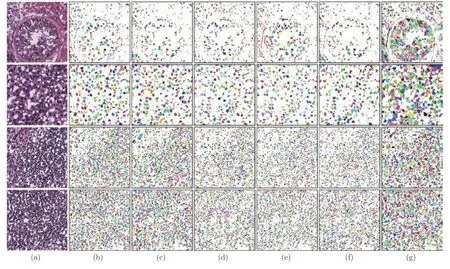
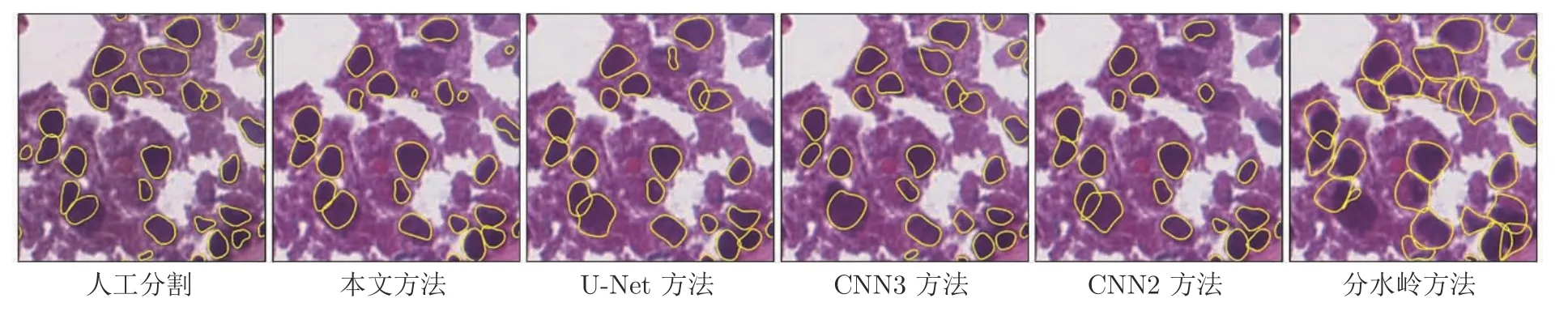
3.3 定量分析
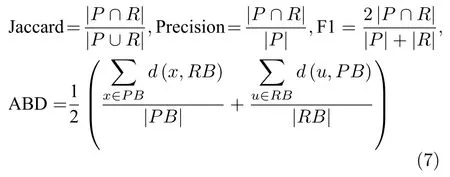


4 结论及后续工作


