环状RNA hsa_circ_0006689在系统性红斑狼疮中的分子机制的初步研究*
2021-04-17李文宇范润哥温斯健林有坤
韦 悦,李文宇,范润哥,侯 伟,温斯健△,林有坤△
(1.广西医科大学第一附属医院皮肤性病科,南宁 530021;2.中国医学科学院地中海贫血防治研究重点实验室,南宁 530021;3.广西地中海贫血防治重点实验室,南宁 530021)
系统性红斑狼疮(systemic lupus erythematosus,SLE)是一种以自身抗体产生及多器官受累为主要特点的自身免疫性疾病[1],其发病机制尚未完全明确,既往研究认为其与遗传、免疫、环境、激素水平等有关,而近来,表观遗传学在SLE 发病中的作用越来越受到重视[2-4]。功能性的非编码RNA 对基因的调控作为表观遗传学中的重要机制之一,在SLE的发生发展中发挥着重要作用[5-6]。环状RNA 是一类环状非编码RNA 分子,可通过“miRNA 海绵”等机制调节基因表达[6]。本课题组前期通过高通量测序技术检测了SLE 患者及健康志愿者外周血单个核细胞(PBMC)中环状RNA的表达,通过进一步验证筛选出了hsa_circ_0006689。本研究以该前期工作为基础,扩大样本进一步探究hsa_circ_0006689在SLE中的表达并验证其生物学特性,并初步探讨其在SLE中可能的作用机制,为寻找新的SLE生物标记物提供科学依据,为SLE的发病机制研究提供新的视角及思路。
1 对象与方法
1.1 研究对象
选取广西医科大学第一附属医院皮肤性病科2019年3月至2020年9月收治的30例SLE患者(观察组)和34例健康志愿者(对照组)作为研究对象。观察组中女27例,男3例,年龄12~59 岁,平 均(31.8±13.6)岁,均符合美国风湿病协会(ACR)1997年制定的SLE诊断标准,且已排除严重感染、妊娠、恶性肿瘤、糖尿病及其他自身免疫性疾病[7]。对照组均为女性,年龄18~26岁,平均(22.76±2.28)岁,满足以下几个条件:(1)非SLE患者,且无其他自身免疫疾病;(2)无重大疾病;(3)近3 个月来无使用激素、免疫抑制剂等药物。依据系统性红斑狼疮疾病活动性指数(systemic lupus erythematosus activity index,SLEDAI)[8]对观察组进行评分并分组,将观察组中处于活动期的24例患者(SLEDAI 评分≥5)设为活动组,6例稳定期患者(SLEDAI 评分<5)设为非活动组。本研究经广西医科大学伦理委员会批准(NO.伦审[2017-KY-国基-081]),受试者均签署知情同意书。
1.2 主要试剂和仪器
磷酸盐缓冲液(PBS)、人外周血单个核细胞分离液(北京索莱宝公司);Trizol RNA分离试剂、lipofectamine TM2000(美国invitrogen 公司);逆转录试剂盒(日本TAKARA 公司);2×Universal SYBR Green Fast qPCR mix(武汉爱博泰克公司);hsa_circ_0006689、ACTB 引物(上海生工生物工程公司);RNase R(美国epicentre 公司),胎牛血清、DMEM、Opti-MEM(美国GIBCO公司),Dual-Luciferase Reporter Assay System(美国Promega 公司)。Lightcycle 96 荧光定量PCR 仪(美国Roche 公司),荧光显微镜(德国LEICA 公司),超净工作台(苏州净化设备公司),酶标仪(TECAN公司)。
1.3 实验方法
1.3.1 PBMC提取 根据人全血单个核细胞分离液的操作说明,分离收集3 mL新鲜全血中的PBMC用于后续RNA提取。
1.3.2 RNA 提取与逆转录 使用Trizol 试剂提取PBMC 中的总RNA,用分光光度计测量RNA 的浓度及纯度,随后根据逆转录试剂操作说明在T100 Thermal cycler PCR仪上合成cDNA。
1.3.3 实时荧光定量PCR(qPCR)检测 通过circ-Base 网站(http://www.circbase.org/)获取hsa_circ_0006689 的序列,将序列头尾相接,使用NCBI 网站(https://www.ncbi.nlm.nih.gov/)设计引物,扩增产物须包含环化位点,以ACTB为内参,引物序列见表1,qPCR 反应体系,见表2。反应条件:95 ℃,3 min 预变性;95 ℃10 s,60 ℃60 s,共循环40 次;每间隔1 ℃收集1次荧光信号,根据溶解曲线分析扩增产物的特异性;每个样本进行3 次技术重复,取3 次CT值平均值,采用2-ΔΔCT法,以ACTB 为内参,计算hsa_circ_0006689的相对表达量。
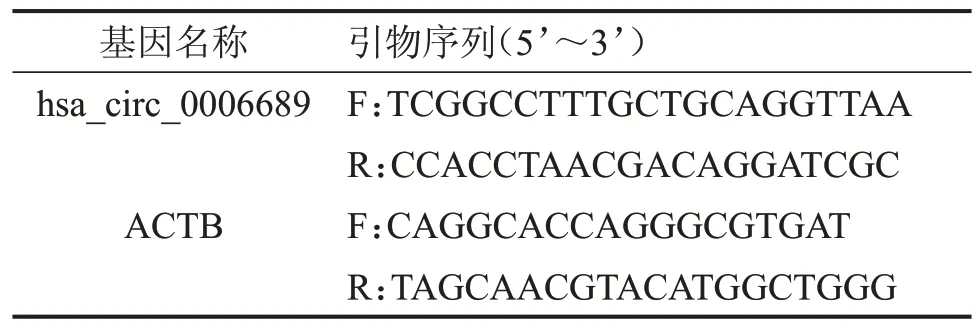
表1 基因引物序列

表2 qPCR反应体系
1.4 hsa_circ_0006689的耐酶消化特性
取一份上述RNA 平均分为2 份,分别加入RNsae R和等体积无酶水,反应体系见表3,混匀后置于普通PCR仪中孵育,37 ℃,20 min,随后将消化后的产物进行常规逆转录,进行qPCR检测,具体步骤同前。以RNase R(-)组的CT 值为参照,计算RNase R(+)组circRNA及mRNA的相对表达量。

表3 RNsae R反应体系
1.5 双荧光素酶报告基因实验
使用circBank(http://www.circbank.cn/)网站预测hsa_circ_0006689 可能结合的miRNA,筛选出可能结合的miRNA。将circ-0006689及其突变序列克隆至报告基因luciferase 下游构建表达载体;培养293T 细胞,当细胞生长至70%时,使用lipofectamine TM2000 将circ_0006689-Wt/Mut 与miRNA mimics 或阴性对照(NC)共转染至293T 细胞,培养48h后使用双荧光素酶报告基因检测试剂盒检测荧光素酶活性。
1.6 hsa_circ_0006689的调控网络分析
分别使用TargetScan(http://www.targetscan.org/)、miRWalk(http://mirwalk.umm.uni-heidelberg.de/)网站来预测上述所得miRNA 可能靶向的mRNA,取能被两个网站共同预测到的mRNA 进行下一步分析。将交集所得的靶基因利用David v6.8(https://david.ncifcrf.gov/)进 行GO(gene ontology)和KEGG pathway分析。
1.7 统计学方法
采用SPSS 23.0、GraphPad Prism 8.0.2、Rstudio软件进行数据分析及绘图。首先将数据进行正态性检验,若数据符合正态分布则以均数±标准差()表示,两样本比较采用t检验;若不符合正态分布则采用中位数(四分位数间距)[M(P25~P75)]表示,两样本比较采用非参数检验中的Mann-Whitney检验,以P<0.05为差异有统计学意义。
2 结果
2.1 两组hsa_circ_0006689相对表达量比较
观察组PBMC 中hsa_circ_0006689 的相对表达量明显低于对照组(P<0.01),见图1。进一步对观察组中活动组与非活动组的hsa_circ_0006689 表达水平进行比较,差异无统计学意义(P=0.3),见图2。
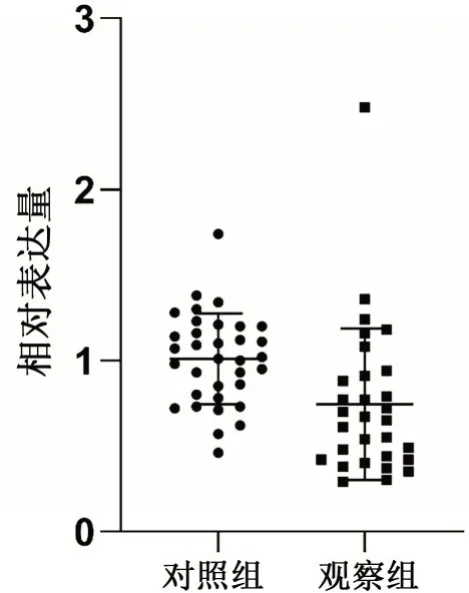
图1 两组hsa_circ_0006689相对表达量比较
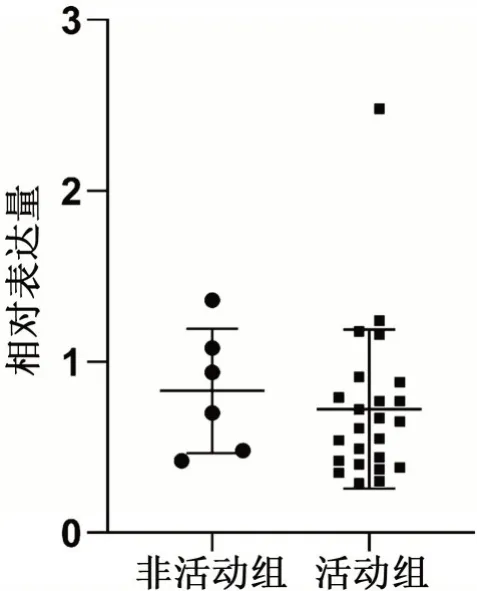
图2 观察组中活动组和非活动组hsa_circ_0006689相对表达量比较
2.2 hsa_circ_0006689可耐RNase R消化
经RNase R消化处理后,hsa_circ_0006689的相对表达量有轻度升高,而作为mRNA的ACTB的相对表达量出现显著降低,见图3。表明hsa_circ_0006689具有较强的耐受RNase R酶消化的能力。
2.3 双荧光素酶报告基因实验
通过circBank(http://www.circbank.cn/)网站发现hsa_circ_0006689 可能靶向65个miRNA,见图4。其中hsa-mir-1255a 碱基序列的种子区与hsa_circ_0006689存在2处结合位点,见图5。双荧光素酶报告基因实验结果证实mir-1255a mimics可以显著抑制circ_0006689-Wt 质粒的荧光强度(P<0.05),而circ_0006689-Mut组质粒的荧光强度比较,差异无统计学意义(P>0.05),表明hsa_circ_0006689 可以靶向结合hsa-mir-1255a,见图6。
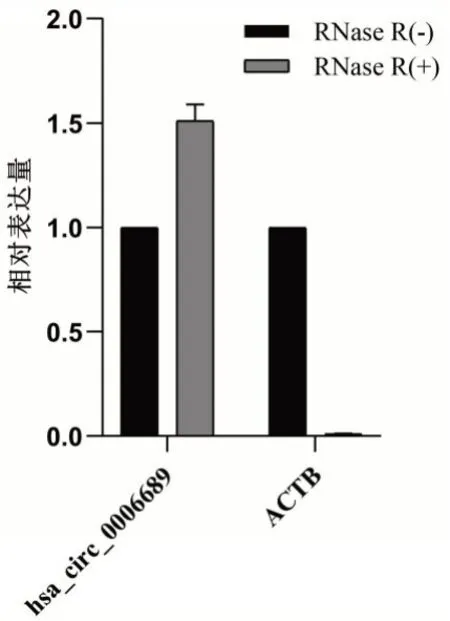
图3 RNase R 处理后hsa_circ_0006689 与ACTB 相对表达量比较
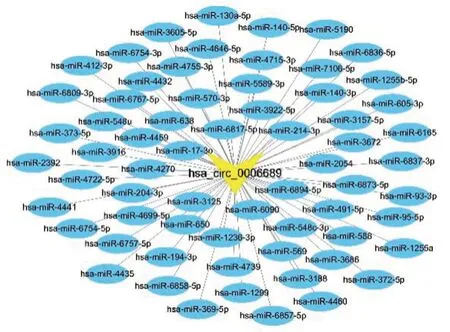
图4 hsa_circ_0006689-miRNA网络图

图5 hsa_circ_0006689、hsa-mir-1255a、circ_0006689-Mut 三者碱基结合位点
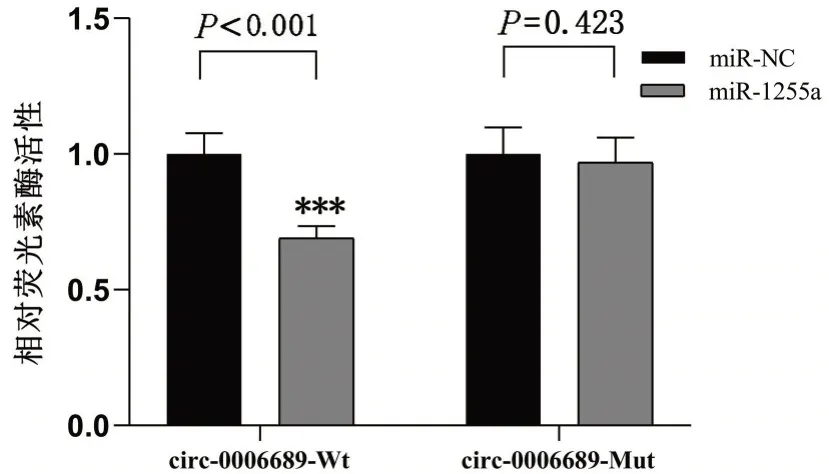
图6 hsa_circ_0006689靶向结合hsa-mir-1255a
2.4 hsa-mir-1255a调控网络分析
将hsa-miR-1255a 输入TargetScan、miRWalk 网站来预测可能的靶基因,选择能被两个网站共同预测到的靶基因,共获得1 050 个靶基[9];RNA-seq 中下调的基因共8 196个差异基因,将预测的1 050个靶基因与RNA-seq中的差异基因取交集得到525个基因,见图7,将上述525 个基因输入,将交集所得525 个靶基因利用David v6.8(https://david.ncifcrf.gov/)进行GO和KEGG pathway分析。

图7 hsa-mir-1255a靶基因与RNA-seq的交集
2.5 差异靶基因的GO分析
将上述525个靶基因进行GO分析,分别从分子功能(molecular function,MF)、细胞组分(cellular component,CC)、生物过程(biological process,BP)3个方面分析这些基因功能的富集情况,探究hsa_circ_0006689 在SLE 中潜在的作用机制。MF的结果显示,靶基因主要富集在metal ion binding、receptor activity、transcription factor activity、sequence-specific DNA binding、GTP binding retinoic acid receptor binding、transmembrane receptor protein tyrosine kinase activity、SMAD binding GTPase activity、transcription factor binding 等分子功能中富集。CC 结果提示,靶基因主要分布在cytoplasmic vesicle、intracellular、endoplasmic reticulum lumen、cytoplasm、cell junction、plasma membrane、endoplasmic reticulum membrane、neuromuscular junction、cytosol 等细胞组分,主要富集在胞质中。BP 结果提示主要在transmembrane receptor protein tyrosine kinase signaling pathway、positive regulation of transcription,DNA-templated、signal transduction by protein phosphorylation、post-embryonic development、negative regulation of transcription from RNA polymerase II promoter、regulation of small GTPase mediated signal transduction、transcription,DNA-templated、positive regulation of mesenchymal cell proliferation、positive regulation of osteoblast different、activation of cysteine-type endopeptidase activity involved in apoptotic process等生物学过程,这些生物过程主要与转录调控和蛋白质磷酸化有关,见图8。
2.6 靶基因的KEGG pathway分析
通过KEGG pathway 分析发现,靶基因富集在Signaling pathways regulating pluripotency of stem cells、TGF-beta signaling pathway、Hippo signaling pathway、ErbB signaling pathway、Cell adhesion molecules(CAMs)、Amphetamine addiction、Chronic myeloid leukemia 等信号通路中。主要与信号转导相关,其中TGF-beta signaling pathway 为免疫相关通路,见图9。

图8 靶基因的GO富集结果

图9 靶基因KEGG pathway富集结果
3 讨论
环状RNA是一种由反向剪接、共价连接而形成的闭环非编码RNA,它缺少常规mRNA 的5’帽子结构及3’poly A尾[10],这个特点使得环状RNA可以对耐受RNase R 的消化,其在组织中表达稳定的特性,使其具有潜力成为SLE的生物标记物。已经有学者报道,SLE 合并肾损害患者外周血中has_circ_0082688、has_circ_0008675 水平升高,可作为诊断和治疗的潜在生物标志物[11]。Guo 等[12]研究发现hsa_circ_0000479 有可能成为诊断SLE 的一种新的生物标志物。本研究中,笔者所选择的hsa_circ_0006689 经RNase R 消化处理后,线性结构的ACTB mRNA 相对表达量出现显著降低,而环状RNAhsa_circ_0006689 的相对表达量则出现轻度升高,这种升高考虑为线性RNA被消化后、环状RNA相对富集所致,该结果一方面说明了hsa_circ_0006689 具有耐受RNase R 酶消化的特性,另一方面也从侧面印证了hsa_circ_0006689 的环状结构。此外,笔者也扩大样本进一步验证了hsa_circ_0006689 在SLE 患者中的表达较正常人下降(P<0.05),结合本课题组前期已经发现hsa_circ_0006689 的表达与SLE 的活动度具有相关性,表明hsa_circ_0006689 生物学性质稳定,具有成为SLE生物标志物的巨大潜力。
目前已报道的环状RNA 功能主要有:(1)作为miRNA的“海绵”;(2)充当蛋白质的“海绵”,增强蛋白质的功能;(3)充当支架来介导酶—底物复合物的形成并将蛋白质募集到特定的位置;(4)不依赖帽的翻译功能;(5)与线性剪接发生竞争,影响其宿主基因的表达[6]。在肿瘤、心血管疾病、糖尿病、类风湿性关节炎等疾病中,环状RNA被发现可以通过上述功能对这些疾病的发生发展起到调节作用。已有研究表明,环状RNA 可以影响SLE 的发生发展,其中环状RNA 作为“miRNA 海绵”作用的研究较多。研究发现,CircIBTK 在SLE 中表达下调,可能通过与miR-29b 结合来调节SLE 中DNA 去甲基化和AKT信号通路从而影响SLE的发生发展,并且可作为SLE 的生物标志物和治疗靶点[13]。Zhang等[14]发现,hsa_circ_0123190 可能通过海绵吸附hasmiR-483-3p 在狼疮性肾炎中充当ceRNA 来调节APLNR 的表达。Zhang 等[15]发现hsa_circ_0012919在SLE CD4+T 细胞下调,并且可以靶向结合miR-125a-3p 从而促进CD4+T 细胞中CD11a 和CD70 的DNA甲基化。
为进一步探讨hsa_circ_0006689 作用的分子机制,本研究通过生物信息学分析发现,hsa_circ_0006689序列中存在两个hsa-mir-1255a种子区的合位点,通过双荧光素酶报告基因实验证实了hsa_circ_0006689可以靶向结合hsa-miR-1255a。这一结果说明hsa_circ_0006689 可能通过与靶基因竞争性结合hsa-mir-1255a 从而调节靶基因的表达。使用TargetScan、miRWalk 网站预测hsa-mir-1255a的靶基因,构建hsa_circ_0006689-hsa-miR-1255amRNA 互作网络,分析hsa_circ_0006689 可能的作用机制;通过预测共获得525 个可能与hsa-miR-1255a 靶向结合的mRNA。对以上525 个靶基因进行GO、KEGG pathway分析后发现部分靶基因富集在跨膜受体蛋白酪氨酸激酶信号转导途径上,且与TGF-β 信号通路高度相关。目前多个研究表明TGF-β 信号通路、酪氨酸激酶信号转导途径与SLE的发生发展密切相关,可能通过影响免疫细胞的增殖、分化和激活而影响SLE 的发生发展[16]。研究报道,脾酪氨酸激酶(Sky)在SLE患者的T细胞中过表达,在免疫细胞中参与膜介导的信号转导。抑制Sky表达可以改善(NZB x NZW)狼疮易感小鼠T细胞的功能,减缓皮肤及肾脏病变的发展[17]。酪氨酸激酶2的基因多态性与SLE相关[18]。
综 上,hsa_circ_0006689 在SLE 患 者PBMC 中明显下调,且生物学性质稳定,可以耐受RNase R酶的消化;生物信息学分析发现hsa_circ_0006689 通过竞争性结合hsa-mir-1255a 从而调控靶基因的表达,这些靶基因与跨膜受体蛋白酪氨酸激酶信号转导途径及TGF-β 信号通路等免疫相关通路有关。我们有理由推测,hsa_circ_0006689 可能通过靶向结合hsa-mir-1255a 来调节跨膜受体蛋白酪氨酸激酶信号转导途径及TGF-β 信号通路,从而影响了SLE 的发生发展,并且有潜力成为帮助SLE 诊断和评估病情的生物标志物。
