基于RNA 结合蛋白构建膀胱癌预后模型及验证
2021-04-14储昭阳朱向明鲁柯兵龚儒杰马平川徐凯慧
储昭阳, 朱向明, 鲁柯兵, 龚儒杰, 马平川, 徐凯慧
(1.皖南医学院研究生学院, 安徽 芜湖241002; 2.皖南医学院弋矶山医院超声医学科; 3.台州市黄岩区妇幼保健院超声医学科)
膀胱癌(bladder cancer, BC) 2018 年全球新发患者近54.9 万人, 相关死亡20 万人[1], 患者的5 年存活率持平[2]。 开发新的诊断和治疗BC 的方法至关重要, 而识别新的与BC 诊断和预后相关的生物标志物也同样重要。 RNA 结合蛋白(RNA binding proteins, RBPs) 是一类与多种类型的RNA相互作用的蛋白质, 包括rRNAs、 ncRNAs、 snRNAs、miRNAs、 mRNAs、 tRNAs 和snoRNAs。 迄今为止,在人类基因组中已经发现了超过1 500 个RBPs 基因[3]。 这些RBPs 在维持细胞的生理平衡方面发挥着重要作用, 尤其是在发育过程和应激反应中。RBPs 可以以一种结构或序列依赖的方式与目标RNA 结合, 形成核糖核酸蛋白复合物, 在转录后水平调控mRNA 的稳定性、 RNA 加工、 剪接、 定位、 输出和翻译[4]。 在过去的几十年里, 许多研究揭示了RBPs 在肿瘤中表达异常, 影响了mRNA向蛋白的转化, 并参与肿瘤的发生[5-7]。 其中, 只有少数RBPs 被深入研究并发现在人类癌症中发挥关键作用[8-12]。 RBPs 的系统功能研究将有助于我们全面了解其在肿瘤中的作用。 因此, 本研究从癌症基因组图谱 (Cancer Genome Atlas, TCGA)数据库下载了BC 的RNA 测序和临床病理数据。随后通过高通量生物信息学分析鉴定了BC 组织和正常膀胱组织间异常表达的RBPs, 并系统地探索了它们的潜在功能和分子机制。 本研究确定了一些可能影响BC 预后的RBPs, 促进了对BC 进展的分子机制的理解。 这些RBPs 可能为诊断和预后提供潜在的生物标志物。
1 材料与方法
1.1 数据处理 从TCGA (https: / /portal.gdc.cancer.gov/) 数据库下载19 例正常膀胱组织和411 例BC 组织的RNA 测序数据集以及相应的临床数据, 其中数据的筛选为包含组织的病理分期以及生存期。 为了鉴定正常膀胱组织和BC 组织之间的差异表达基因, 所有原始数据使用LIMMA( http: / /www.bioconductor.org/packages/release/bioc/html/limma.html) 软 件 包 进 行 预 处 理, 根据| logFC (fold change)| ≥1 和伪发现率(FDR)<0.05 标准, 进行Wilcox 检验筛选出RBPs 相关的差异基因。
1.2 KEGG 途径与GO 富集分析 通过GO 富集和京都基因与基因组百科全书(KEGG) 途径分析,综合检测这些差异表达的RBPs 的生物学功能。GO 分析术语包括细胞成分 (CC)、 分子功能(MF) 和生物过程(BP)。 所有富集分析均利用org.Hs.eg.db、 DOSE、 clusterProfiler、 enrichplot 软件包完成, P 和FDR 值均<0.05 为差异有统计学意义。
1.3 PPI 网络的构建和模块筛选 将差异表达的RBPs 提交到STRING 数据库(http: / /www.string-db.org/)[13], 以识别蛋白质-蛋白质相互作用信息。 使用Cytoscape 3.8.0 软件进一步构建PPI 网络并进行可视化。 使用聚类构建功能模块(MCODE)插件得到PPI 网络中的重要模块和基因, MCODE得分和节点数均大于5。 P<0.05 为差异有统计学意义。
1.4 BC 患者RBPs 筛选及预后生存模型构建 使用Survival 软件包对TCGA 数据集差异表达的RBPs进行单因素Cox 回归分析。 采用对数秩检验进一步筛选有意义的候选基因。 随后, 在初步筛选出有意义的候选基因的基础上, 构建了多因素Cox风险回归模型, 并计算风险分数来评估患者的预后。 每个样本的风险评分公式: Risk score =β1×Exp1+β2×Exp2 +βi×Expi。 其中β 表示系数值,Exp 表示基因表达水平。 根据中位风险评分生存分析, 将BC 患者分为低危组和高危组。 采用对数秩检验比较2 个亚组之间的总生存期(OS) 差异。此外, 使用Survival ROC 软件包进行ROC 曲线分析以评估上述模型的预测能力。 将TCGA 数据随机分为2 组, 一组作为train 组分析, 另一组作为test 组进行模型验证, 以证实该预测模型的预测能力。 最后, 使用RMS 软件包绘制列线图, 预测患者的生存期。
1.5 验证预后模型RBPs 的预测价值 分别对train 组及test 组进行单因素和多因素独立预后分析。 单因素独立预后分析是将每个因素跟生存时间和生存状态进行比较, 观察与患者生存是否相关; 多因素独立预后分析是将这里面的因素一起输入, 跟生存时间和生存状态进行比较, 并考虑这些因素之间的一个相关性。 如果train 组及test组分析结果P 值均<0.05, 则说明模型可以独立于其他的临床现状作为独立的预后因子。 之后分别探讨预后模型中的各个RBPs 在BC 中的预后价值, 使用Kaplan-Meier Plotter Server (https: / /kmplot.com/analysis/) 在线工具绘图仪来验证预后模型中每个RBPs 与OS 间的关系。
1.6 统计学方法 使用Perl 5.30.2 进行数据表达矩阵的处理, 应用R4.0.0 软件进行统计学分析及相应图形绘制, 采用Wilcox 检验筛选差异基因,采用ggplot 包进行图形绘制, Survival 包进行单因素和多因素Cox 比例回归模型筛选, 并建立多基因预后模型。 使用Survival ROC 包计算ROC 曲线评价模型的有效性, 并计算曲线下面积。 P<0.05为差异有统计学意义。
2 结果
2.1 筛选得到差异表达的BC 组织中RBPs 将TCGA 得到的数据通过perl 和R 语言处理后, 处理数据共纳入差异表达的RBPs 有1 495 个[3], 其中109 个符合本研究筛选标准, 其中包括52 个上调的RBPs 和57 个下调的RBPs。 这些差异表达的RBPs 的分布用热图和火山图来表示。
2.2 差异表达的BC 组织中RBPs 的GO 和KEGG通路富集分析 为研究所鉴定的RBPs 的功能和机制, 将这些不同表达的RBPs 分为2 组: 表达上调组和表达下调组。 然后, 将这些差异表达的RBPs进行富集分析。 结果表明, 上调差异的RBPs 的GO 富集在催化活性、 核糖核酸作用活性、 核苷酰转移 酶 活 性、 mRNA 3′ 端- 非 编 码 区 结 合 区(UTR) 等分子功能中; 就细胞组成而言, 上调差异的RBPs 的GO 富集在细胞质核糖核蛋白颗粒、核糖核蛋白颗粒; 生物过程的富集主要包括大分子甲基化、 甲基化、 RNA 分解过程等(图1A);下调差异的RBPs 的GO 富集分析中分子功能主要包含mRNA 3′端-UTR 结合区、 mRNA 3′端-UTR富含AU 的结合区、 富含AU 的元素结合区等; 细胞组成主要包含细胞质核糖核蛋白颗粒、 核糖核蛋白颗粒、 P-颗粒体等结构; 生物过程主要包括调节mRNA 代谢过程、 调节RNA 剪接、 调节翻译等(图1B)。 上调差异的RBPs 相关基因的KEGG通路分析结果表明, DEGs 主要参与癌症中的miR⁃NAs、 mRNA 监测途 径、 DNA 复制等信号通路(图1C); 下调差异的RBPs 相关基因的KEGG 通路分析结果表明, DEGs 相关基因主要参与孕酮介导的卵母细胞成熟、 卵母细胞减数分裂、 mRNA监测途径等信号通路(图1D)。

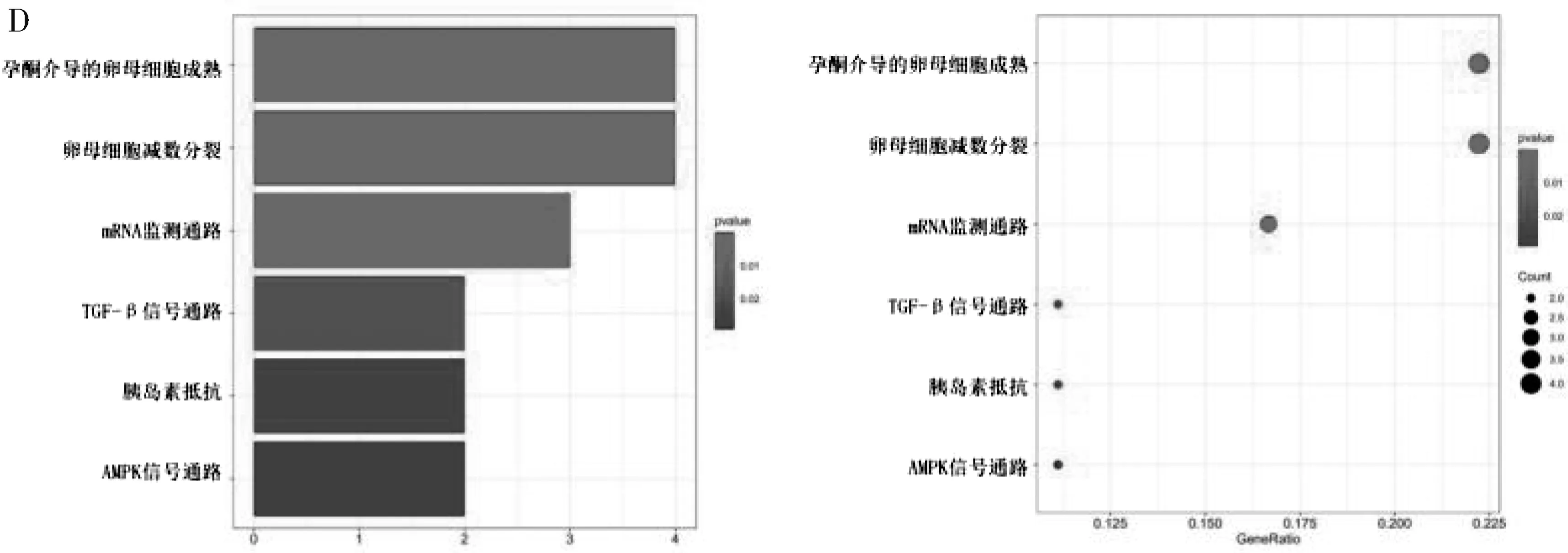
图1 差异表达RBPs 的GO 富集分析(A、 B) 和KEGG 途径(C、 D)
2.3 蛋白工作网络及核心模块 Cytoscape 得出88个RBPs (45 个上调, 43 个下调) 对应的166 个蛋白互作的关系。 使用MCODE 模块构建出7 个子网络, 并得出16 个基因是与BC 相关的关键RBPs(这些RBPs 在DNA 烷基化、 DNA 甲基化或去甲基化、 翻译的负调控等方面都有显著的富集)。
2.4 预后RBPs 筛选及生存模型构建 通过筛选得到11 个与预后相关的RBPs 相关基因(图2A),通过COX 回归降维分析构建预后模型(图2B),由2 个高风险的RBPs 基因(TRIM71、 DARS2) 及3 个低风险的RBPs 基因(APOBEC3H、 PABPC3、MTG1) 组成; 模型在train 组及test 组中ROC 曲线下面积分别为0.701、 0.664 (图2C、 D), 反映了模型预测BC 患者的生存具有良好的诊断效能,train 组及test 组风险曲线(图2E、 F) 也验证了模型预测BC 患者的生存的准确性。 绘制train 组及test 组的生存曲线(图2G、 H), 可以看出在train 组及test 组中, 高低风险2 组生存率的差异有统计学意义, 说明得到的模型可以预测BC 患者的生存。
2.5 列线图 根据5 个RBPs 绘制出列线图(图3), 可以根据列线图去预测患者的生存期。
2.6 独立预后分析 将train 组及test 组通过多元回归分析结果绘制森林图(图4), 得出预后模型P 值均<0.05, 说明模型可以独立于其他的临床现状作为独立的预后因子。
2.7 模型RBPs 的预测价值的验证 Kaplan-Meier Plotter Server 鉴定了预后模型中的5 个(TRIM71、DARS2、 APOBEC3H、 PABPC3 和MTG1) RBPs 与BC 患者的总生存期显著相关(图5)。
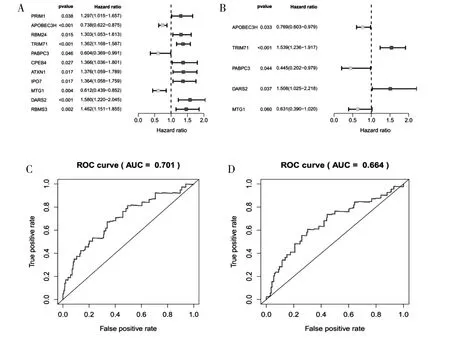

图2 A: 单变量Cox 回归分析中与BC 预后相关的RBPs; B: 多变量Cox 回归分析中预后相关的RBPs; C、 D: 基于风险评分预测OS 的ROC 曲线; E、 F: 风险评分分布、 表达热图和生存状态; G、 H: 低风险和高风险亚组的生存曲线
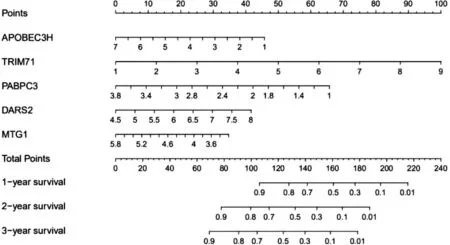
图3 TCGA 队列中预测BC 患者1 年、 2 年和3年OS 的列线图
3 讨论
RBPs 失调已被报道在各种恶性肿瘤[6-13]。 然而, 只有一部分RBPs 被深入研究, 部分证实它们参与了癌症[9-13]的发生和发展。 本研究基于TCGA的BC 数据确定了肿瘤组织和正常膀胱组织之间的109 个不同表达的RBPs, 系统分析了相关的生物学途径, 构建了这些RBPs 的PPI 网络, 对异常表达RBPs 进行单变量Cox 回归分析、 生存分析、 多元逐步Cox 回归分析和ROC 分析, 进一步探讨其生物学功能和临床意义。 本研究构建了一个基于5个预后相关的RBPs 基因预测BC 预后的风险模型。 这些发现可能有助于开发新的BC 患者诊断和预后的生物标志物。
表达的功能通路富集分析显示, 不同RBPs 在翻译的调控、 RNA 加工、 RNA 代谢中具有较高富集程度。 已有研究证明翻译的调控、 RNA 加工、RNA 代谢与多种人类疾病的发生和发展有关[14-17]。 RNA 稳定性的转录后调控是基因表达过程中的一个重要环节。 RBPs 可以与RNA 相互作用形成核糖核酸蛋白复合物, 从而增加靶mRNA的稳定性, 促进基因表达, 在各种疾病的进展中发挥关键作用。 此外, 我们还建立了这些不同表达的RBPs 的蛋白-蛋白相互作用网络, 接下来利用TCGA 队列进行分析, 通过多元逐步COX 回归分析, 建立预测BC 预后的风险模型。 ROC 曲线分析显示, 这5 个RBPs 具有较好的诊断能力, 可以选择预后较差的BC 患者。 在预后模型的5 个RBPs中, 许多已被证明在肿瘤的发展和进展中发挥重要作用[18-21]。 APOBEC3H 结构揭示了一种与双链RNA 相互作用的异常机制[18]; TRIM71 通过降解突变体p53 抑制卵巢肿瘤发生[19], 并且TRIM71通过调节Lin28B-let-7-HMGA2 信号传导抑制肿瘤发生[20]; PABPC3 在滤泡性甲状腺癌细胞系外显子组中被认为是反复突变的癌症驱动基因[21];乙型肝炎病毒(HBV) 对DARS2 的上调通过miR-30e-5p、 MAPK、 NFAT5 途径促进肝癌发生[22];人类心肌细胞以及发育中的斑马鱼的MTG1 沉默揭示了早期的心血管损伤[23]。 然而, 这5 种RBPs的分子机制对BC 的发生仍知之甚少, 进一步探索其可能的机制可能是有价值的。 随后, 本研究建立了一个列线图来帮助更直观地预测BC 患者1年、 2 年和3 年的OS, 并使用Kaplan-Meier Plotter检测了5 个RBPs 编码基因的预后价值, 结果与TCGA 队列的预后分析结果基本一致。 提示5 个RBPs 的预后模型对BC 患者治疗方案的调整具有一定的参考价值。 这可能有利于临床医师为BC 的治疗制定临床治疗决策。

图4 train 组(A) 和test 组(B) 单因素独立分析(左侧) 和多因素独立分析(右侧) 结果

图5 Kaplan-Meier Plotter Server 对BC 中预后模型RBPs 预后价值的验证
总体而言, 本研究基于5 个RBPs 构建的预后模型, 显著降低测序成本, 更有利于临床应用。此外, 5 个RBPs 预测模型在BC 患者的生存预测中有更好的表现。 此外, RBPs 相关基因也显示出重要的生物学功能, 这表明它们有可能用于临床辅助治疗。 尽管如此, 本研究仍有一些局限性:(1) 预后模型仅基于TCGA 数据库的数据, 未在临床患者队列等数据库中得到验证; (2) 本研究是在回顾性分析的基础上设计的, 未来应进行前瞻性研究来验证结果; (3) 数据集临床信息不够完善, 可能会降低多元逐步Cox 回归分析的统计效度和信度。
综上所述, 本研究通过一系列生物信息学分析系统地探讨了不同表达的RBPs 在BC 中的表达及预后价值。 这些RBPs 在BC 的发生、 发展中发挥着重要作用。 通过构建5 个RBPs 编码基因的预后模型, 对未来BC 治疗和预后有着积极指导意义。 目前还鲜见关于RBPs 相关的BC 预后模型的报道, 本研究结果对揭示BC 的发病机制, 开发新的诊断思路、 寻找新的治疗靶点以及预后分子标志物提供一定证据。
