黄曲霉中pyrG筛选标记循环利用的方法
2021-04-08聂鑫怡王银春丁霞飞汪世华
聂鑫怡, 薛 杨, 王银春, 丁霞飞, 汪世华
(1.福建农林大学生命科学学院;2.福建省病原真菌与真菌毒素重点实验室,福建 福州 350002)
乳清酸核苷-5′-磷酸脱羧酶基因URA3/pyrG是一类常用的真菌遗传转化营养缺陷筛选标记,其编码产物是真菌尿嘧啶核苷酸合成过程中的关键酶,该基因突变或敲除会使乳清酸核苷-5′-磷酸脱羧酶失活或缺失,表现为尿嘧啶营养缺陷,只能通过外源添加尿嘧啶或尿嘧啶核苷(尿苷)才能在培养基上生长.URA3筛选标记最早在酿酒酵母(Saccharomycescerevisiae)转化中应用,后来应用范围扩大到其他酵母科真菌,如白念珠菌(Candidaalbicans)[1-4].由于尿嘧啶生物合成途径非常保守,在丝状真菌中也有相似的合成过程.黄曲霉(Aspergillusflavus)、烟曲霉(A.fumigatus)、构巢曲霉(A.nidulans)、黑曲霉(A.niger)、米曲霉(A.oryzae)和土曲霉(A.terreus)等真菌中的pyrG基因都与酿酒酵母URA3基因同源,在遗传转化中也得到了广泛应用[5-9].
除了URA3/pyrG筛选标记外,还有一些常见的筛选标记,如氮源和碳源营养缺陷标记基因、药物抗性标记基因等.在比较菌株性状时,携带不同的筛选标记会造成菌株间遗传背景的不同和性状的差异,从而影响研究结果的准确性.URA3/pyrG环出系统的出现解决了这一难题.该系统仅用URA3/pyrG一个筛选标记,配合5-氟乳清酸(5-FOA)的负筛选,使URA3/pyrG筛选标记插入真菌细胞的基因组-环出-再插入-再环出,如此循环,进行多次遗传操作[10-11].但URA3/pyrG环出系统也存在缺陷,如使用该系统前必须要在URA3/pyrG基因两端分别插入一段同向重复序列(direct repeat),该操作过程繁琐、耗时,且由于存在两段同向重复序列,PCR扩增基因重组片段时特别容易造成引物的错配,出现非特异条带.此外,URA3/pyrG环出后会在基因组上残留一段重复序列,该重复序列是否会影响菌株的性状尚未可知.针对这些问题,本研究在黄曲霉中建立了一种新的pyrG筛选标记循环利用的方法.
1 材料与方法
1.1 材料
黄曲霉WT(Δku70)菌株和CA14PTs(Δku70ΔpyrG)菌株由美国农业部南方研究中心Perng-Kuang Chang教授惠赠,黄曲霉GHS(pyrG+)菌株由本研究构建,构巢曲霉FGSC A4菌株(ATCC 38163)和烟曲霉AF293菌株(ATCC MYA-4609)由福建省病原真菌与真菌毒素重点实验室购买保存.
质粒pUC18-HA-SumO上携带的639 bp的HA-sumOCDS-3′-UTR核苷酸片段由通用生物系统(安徽)公司全基因合成.
高保真DNA聚合酶、rTaq DNA聚合酶和未染色蛋白质分子质量标准购自上海翊圣生物科技公司,DNA分子质量标准购自东盛生物科技公司,凝胶回收试剂盒为OMEGA公司产品,酵母提取物和琼脂糖为奥赛公司产品,RIPA裂解液购自上海碧云天生物技术公司,预染蛋白质分子量标准和HA标签抗体购自赛默飞公司,Actin抗体为CST公司产品,5-氟乳清酸、尿嘧啶和尿苷均购自上海生工公司.
1 000×微量元素母液(100 mL):ZnSO4·7H2O 2.2 g,H3BO31.1 g,MnCl2·4H2O 0.5 g,FeSO4·7H2O 0.16 g,CoCl2·5H2O 0.16 g,CuSO4·5H2O 0.16 g, (NH4)6Mo7O24·4H2O 0.11 g,Na4EDTA 5 g,溶于去离子水,过滤除菌.
YGT固体培养基(1 L):酵母提取物 5 g,葡萄糖 20 g,1 000×微量元素母液1 mL,琼脂粉20 g.在YGT培养基中分别加入1 mg·L-1尿嘧啶和尿苷即为YGTUU液体培养基.
本研究所用引物见表1.

表1 本研究所用引物Table 1 Primers used in this study
1.2 GHS(pyrG+)重组菌株的构建
以尿嘧啶营养缺陷型黄曲霉CA14PTs(Δku70ΔpyrG)菌株为出发菌,构建GHS(pyrG+)重组菌株,构建策略如图1所示.步骤如下:以黄曲霉CA14PTs(Δku70ΔpyrG)菌株基因组DNA为模板,用引物P1和P2扩增长度为1 171 bp的sumO基因上游非转录区5′-UTR片段;利用引物P3和P4从烟曲霉AF293菌株基因组DNA中扩增长度为1 890 bp的pyrG表达元件;利用引物P5和P6从构巢曲霉FGSC A4菌株基因组DNA中扩增长度为1 510 bp的3′-磷酸甘油醛脱氢酶基因启动子gpdA(p)片段;利用引物P7和P8从质粒pUC-18-HA-SumO中扩增长度为639 bp的片段HA-sumOCDS-3′-UTR;将上述各片段用重叠PCR法连接到一起,以巢式引物P9和P10扩增长度为5 146 bp的融合片段5′-UTR-pyrG-gpdA(p)-HA-sumOCDS-3′-UTR,转化黄曲霉CA14PTs(Δku70ΔpyrG)菌株原生质体,通过DNA同源重组使融合片段整合到基因组的sumO基因座上替换掉内源的sumO基因,在不含尿嘧啶和尿苷的复苏培养基上37 ℃培养3 d后筛选、鉴定,获得GHS(pyrG+)重组菌株.

CA14PTs(Δku70ΔpyrG)为出发菌株,GHS(pyrG+)为按常规方法以pyrG作为遗传筛选标记对黄曲霉sumO基因位点改造后的重组菌株,GHS(pyrG-)为pyrG筛选标记被剔除后的黄曲霉sumO基因位点改造重组菌株.“5′-UTR”表示黄曲霉sumO基因上游非转录区DNA片段,“pyrG”表示来源于烟曲霉的表达元件,“gpdA(p)”表示来源于构巢曲霉的3′-磷酸甘油醛脱氢酶基因启动子序列,“HA”表示来源于流感病毒的红细胞凝集素表面抗原决定簇HA标签蛋白编码序列,“sumO CDS”表示来源于黄曲霉的翻译产物为SumO蛋白的编码核苷酸序列,“3′-UTR”表示黄曲霉sumO基因下游非翻译区DNA片段.图1 GHS(pyrG+)和GHS(pyrG-)重组菌株构建策略示意图Fig.1 Schematic diagram of the strategy for the construction of GHS(pyrG+) and GHS(pyrG-) strains
1.3 pyrG遗传筛选标记的剔除
以GHS(pyrG+)重组菌株为受体菌,剔除pyrG筛选标记,构建GHS(pyrG-)重组菌株,构建策略如图1所示.步骤如下:利用引物P1和P11从黄曲霉GHS(pyrG+)重组菌株基因组中扩增长度为1 171 bp的sumO基因上游非转录区5′-UTR片段;利用P12和P6从黄曲霉GHS(pyrG+)重组菌株基因组中扩增长度为1 510 bp的3′-磷酸甘油醛脱氢酶基因启动子gpdA(p)片段;将上述两个片段用重叠PCR法连接到一起,用巢式引物P9和P13扩增长度为2 372 bp的融合片段5′-UTR-gpdA(p),转化黄曲霉GHS(pyrG+)重组菌株原生质体,在含有2 mg·mL-15-氟乳清酸、1 mg·L-1尿苷和1 mg·L-1尿嘧啶的复苏培养基上37 ℃培养3 d.抽提转化子基因组DNA,用引物P3和P4扩增pyrG片段进行PCR鉴定,用引物P9和P13扩增5′-UTR-gpdA(p)片段并用引物P9进行测序分析.
1.4 营养缺陷表型观察
取103个黄曲霉GHS(pyrG-)重组菌株阳性转化子的新鲜孢子,分别接种于YGT和YGTUU固体培养基平板的中心点,37 ℃持续培养3 d,用佳能IXUS相机(16.1 MEGA PIXELS)观察并拍照.
1.5 免疫杂交分析
黄曲霉总蛋白的提取、电泳和免疫杂交方法参考文献[5].
2 结果与分析
2.1 表达HA-SumO的黄曲霉重组菌株GHS(pyrG+)的构建
构建GHS(pyrG+)重组菌株的过程如图2A所示.将5′-UTR-pyrG-gpdA(p)-HA-sumOCDS-3′-UTR融合片段转化黄曲霉CA14PTs(Δku70ΔpyrG)菌株原生质体,并筛选、鉴定GHS(pyrG+)阳性转化子,抽提总蛋白并用HA标签抗体和Actin抗体进行Western blot免疫杂交,分析黄曲霉GHS(pyrG+)重组菌株中的HA-SumO的表达水平和SUMO化修饰状态,结果如图2B所示.GHS(pyrG+)重组菌株中可以检测到HA-SumO蛋白单体的条带,被SUMO化修饰的多种靶标蛋白条带也能被检测到,呈现阶梯状分布,而对照CA14PTs(Δku70ΔpyrG)菌株和WT(Δku70)菌株中都检测不到特异性杂交条带.

A.构建5′-UTR-pyrG-gpdA(p)-HA-sumO CDS-3′-UTR融合片段的琼脂糖凝胶电泳图谱(M为DS10 000 DNA分子质量标准,泳道1为长度约1.2 kb的sumO基因上游非转录区5′-UTR片段,泳道2为长度约1.9 kb的pyrG表达元件,泳道3为长度约1.5 kb的gpdA(p)片段,泳道4为长度约0.6 kb的HA-sumO CDS-3′-UTR片段,泳道5为长度约5.2 kb的5′-UTR-pyrG-gpdA(p)-HA-sumO CDS-3′-UTR融合片段);B.免疫杂交检测GHS(pyrG+)重组菌体内的SUMO化修饰状态[泳道1为CA14PTs(Δku70ΔpyrG)菌株总蛋白,泳道2为WT(Δku70)菌株总蛋白,泳道3为GHS(pyrG+)重组菌株总蛋白].图2 GHS(pyrG+)重组菌株的构建和验证Fig.2 Construction and identification of the GHS(pyrG+) strain
2.2 GHS(pyrG+)重组菌株中pyrG遗传筛选标记的剔除
分别从黄曲霉GHS(pyrG+)重组菌株基因组中扩增长度约1.2 kb的sumO基因上游非转录区5′-UTR片段和1.5 kb的3′-磷酸甘油醛脱氢酶基因启动子gpdA(p)片段(图3A),并用重叠PCR法连接到一起,以巢式引物P9和P13扩增长度约2.4 kb的融合片段5′-UTR-gpdA(p)(图3B),转化黄曲霉GHS(pyrG+)原生质体,并筛选、鉴定GHS(pyrG-)重组菌株阳性转化子.经鉴定,挑取的12个转化子中有3个为阳性转化子,阳性率为25%.如图3C所示,用引物P3和P4进行PCR扩增,GHS(pyrG+)基因组DNA中可扩增出约1.9 kb的pyrG片段,而T3#、T7#和T11#转化子的基因组和阴性对照中则无法扩增pyrG片段;同时,利用引物P9对转化子T3#、T7#和T11#中扩增出的5′-UTR-gpdA(p)片段进行测序分析表明,阳性转化子的5′-UTR-gpdA(p)片段的实际序列与理论序列一致(图3D).这些结果说明:GHS(pyrG+)基因组上整合的pyrG筛选标记已被剔除,且剔除过程不依赖pyrG筛选标记两端的同向重复序列,剔除pyrG筛选标记后GHS(pyrG-)阳性转化子基因组上也未残留重复序列;除了pyrG筛选标记外,GHS(pyrG-)阳性转化子与GHS(pyrG+)菌株的遗传背景一致.

A.PCR扩增5′-UTR片段(泳道1)和gpdA(p)片段(泳道2)的琼脂糖凝胶电泳图谱(M为DS2 000 DNA分子质量标准);B.重叠PCR扩增>5′-UTR-gpdA(p)融合片段(泳道1、2)的琼脂糖凝胶电泳图谱(M为DS5 000 DNA分子质量标准);C.GHS(pyrG-)重组菌株阳性转化子的PCR鉴定[M为DS2 000 DNA分子质量标准,泳道1的模板为蒸馏水,泳道2和3的模板为CA14PTs(Δku70ΔpyrG)菌株基因组,泳道4的模板为GHS(pyrG+)重组菌株基因组,泳道5、6和7的模板分别为GHS(pyrG-)重组菌株T3#、T7#和T11#转化子基因组];D.GHS(pyrG-)重组菌株阳性转化子基因组扩增5′-UTR-gpdA(p)产物测序结果的序列比对.图3 GHS(pyrG-)重组菌株的构建和筛选Fig.3 Construction and identification of the GHS(pyrG-)strain
2.3 GHS(pyrG-)重组菌株的pyrG营养缺陷表型
转化子T3#、T7#和T11#在YGTUU固体培养基上能够正常生长出细密的菌丝,而在YGT固体培养基上则不能正常生长,呈现尿嘧啶营养缺陷表型(图4A).这一结果证明,GHS(pyrG-)阳性转化子T3#、T7#和T11#的营养缺陷型是正确的.

A.GHS(pyrG-)重组菌株阳性转化子的尿嘧啶营养缺陷表型鉴定;B.免疫杂交分析GHS(pyrG-)重组菌株阳性转化子的SUMO化修饰状态[泳道1为蛋白分子质量标准,泳道2为GHS(pyrG+)重组菌株总蛋白,泳道3为WT(Δku70)菌株总蛋白,泳道4、5和6分别为GHS(pyrG-)重组菌株转化子T3#、T7#和T11#的总蛋白].图4 GHS(pyrG-)重组菌株的性状表型和SUMO化修饰状态Fig.4 Phenotype and SUMO modification status of the GHS(pyrG-) strain
2.4 GHS(pyrG-)重组菌株的HA-SumO的表达水平
分别提取WT(Δku70)菌株、GHS(pyrG+)重组菌株以及GHS(pyrG-)阳性转化子T3#、T7#和T11#的总蛋白,利用HA标签抗体进行免疫杂交分析,从蛋白水平上比较pyrG筛选标记剔除前后SUMO化修饰状态是否有差异,并以SDS-PAGE考马斯亮蓝染色结果作为上样量对照.从试验结果来看,WT(Δku70)菌株检测不到特异性杂交条带,而GHS(pyrG+)重组菌株和GHS(pyrG-)阳性转化子T3#、T7#和T11#都特异性地表达了HA-SumO单体蛋白,体内还有许多被SUMO化修饰的不同分子质量的靶标蛋白,呈现阶梯式的分布,且GHS(pyrG+)重组菌株和GHS(pyrG-)各个阳性转化子SumO的表达量和表达模式之间没有差异(图4B).这些结果说明,按本研究的方法剔除pyrG筛选标记不影响菌株的蛋白表达模式.
3 讨论
本研究建立了一种新的乳清酸核苷-5′-磷酸脱羧酶基因pyrG遗传筛选标记循环利用的方法,仅需一个pyrG遗传筛选标记的循环利用即可实现多次遗传转化操作,避免了不同遗传筛选标记的差异对真菌遗传背景和性状的影响;同时,该方法在剔除基因组中的pyrG遗传筛选标记时不需要依赖同向重复序列和在URA/pyrG两端插入重复序列,操作简单易行,且剔除pyrG筛选标记后在基因组上不会残留重复序列.
由于许多真菌都以URA3/pyrG作为遗传筛选标记,本方法除了用于黄曲霉外,也可应用到以乳清酸核苷-5′-磷酸脱羧酶基因URA3/pyrG表达元件作为遗传筛选标记的其他真菌中(应用方法如图5所示),使得重组菌株重新获得尿嘧啶营养缺陷,再次作为出发菌株循环利用URA3/pyrG筛选标记进行遗传转化,为多基因、多位点的真菌遗传改造提供支持.
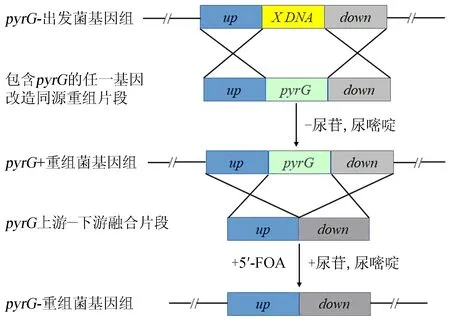
“X DNA”表示真菌基因组上需要利用pyrG进行改造的任一DNA片段,“up”和“down”分别表示pyrG整合位点上游和下游DNA片段.图5 pyrG筛选标记循环利用示意图Fig.5 Schematic diagram for the pyrG selectable marker recycling system
