大豆抗倒伏相关性状全基因组关联分析
2021-04-06李文滨张翔超郭志文郑立娜韩英鹏滕卫丽
李文滨,吴 航,刘 冀,张翔超,郭志文,郑立娜,赵 雪,韩英鹏,滕卫丽
(东北农业大学大豆研究所,大豆生物学教育部重点实验室,农业农村部东北大豆生物学与遗传育种重点实验室,哈尔滨150030)
大豆是重要经济和粮食作物,其营养价值高,用途广泛。倒伏是限制大豆高产、稳产和优质重要因素之一,大豆品种抗倒伏性是获得高产稳产重要保证[1-2]。倒伏是受遗传、栽培、气候和生态等多种因素共同制约的复杂性状。以往研究表明作物倒伏与植株地上部性状(如株高、茎粗、茎秆强度)[3-4]和地下部性状(如根重、根长、根体积等)有关[5-6]。在关于大豆倒伏及其相关性状研究中,周蓉和钟开珍等认为大豆倒伏性与茎秆性状(株高、主茎节数、节间长度、分枝数等)和产量性状(单株荚数、单株粒数、单株产量和百粒重)显著相关[7-8]。由于大豆倒伏性与该性状密切相关,且目前对于解析大豆抗倒性遗传基础研究报道相对较少,利用分子标记技术探索大豆倒伏性遗传特性成为大豆科研工作者研究热点。通过QTL定位方法已发掘出许多与大豆倒伏性相关位点,但利用QTL定位通常需构建作图群体,研究时间较长、定位精度低[9-10],而基于自然群体的关联分析具有研究时间短,检测效率高、定位精度高等优势[11]。
诸多学者已对大豆主要农艺性状开展GWAS(Genome-wide association analysis)分析,例如,Jing等通过GWAS检测到14个与株高相关SNP位点,预测6个可能与株高相关新基因[12]。Chang等对368份大豆种质资源作GWAS分析,检测到45个与株高相关位点和43个与主茎节数相关位点[13]。张军等对190份大豆种质多个农艺性状作GWAS分析,累计检测到136个位点与目标性状显著关联,其中14个位点与株高关联,13个位点与倒伏性关联[14]。张友谊利用224份大豆微核心种质和1 749个SNP标记作GWAS分析,共检测到36个SNPs位点,其中与株高关联的13个位点,主茎节数4个,茎粗9个[15]。王自力对由573份种质建立的江淮地区大豆育种群体作GWAS分析,两年重复检测到与倒伏性关联SNP标记2个,株高130个,主茎节数92个[16]。关联分析不仅可找到与大豆倒伏性状有关遗传位点,还可预测与目标性状相关新基因。因此利用大豆自然群体作关联分析研究抗倒伏性遗传特性,对实现大豆高产稳产优质等目标具有重要意义。
本研究利用150个大豆种质资源组成的自然群体开展多年多点试验,通过RTM-GWAS方法结合SNP位点对株高、主茎节数等性状作全基因组关联分析,挖掘候选基因,为分子标记辅助大豆抗倒伏育种提供依据。
1 材料与方法
1.1 试验材料
供试材料选用150份不同大豆种质资源构成的自然群体。其中国外品种(系)3份、北京品种(系)8份、黑龙江品种(系)93份、吉林品种(系)20份、辽宁品种(系)3份、内蒙古品种(系)15份、新疆品种(系)2份、河北品种(系)4份、山东品种(系)1份和甘肃品种(系)1份。
1.2 试验设计
试验于2018~2019年在向阳(A)、呼兰(B)、阿城(C)3个试验地点开展,采用随机区组设计,3次重复,行长2 m、垄宽0.65 m,株距0.06 m。成熟期收获并室内考种,田间管理同大田。
1.3 方法
1.3.1 性状调查
根据周蓉等方法于成熟期调查倒伏性[17],田间倒伏分级标准:1级为小区植株全部直立;2级为轻度倒伏(植株倾斜<15°);3级为重倒伏(植株倾斜15°~44°);4级为严重倒伏(植株倾斜>45°)。从自然群体每个资源中选择具有代表性连续5株测定株高、主茎节数、节间长度、茎粗、重心高度、茎秆强度、单株荚数、单株粒数、单株粒重、百粒重、茎秆抗倒指数等性状,3次重复,以平均值为观测值。其中重心高度和茎秆抗倒指数测定均参照陈喜凤等方法[2],其他室内考种项目及标准均参照邱丽娟等方法[18]。
1.3.2 基因型检测与分析
采用特异性位点扩增片段测序(SLAF-seq)技术,利用Illumina基因组分析仪Ⅱ获得大豆染色体均匀分布高质量单核苷酸多态性(SNPs)。提取150份大豆种质资源基因型信息,使用Seqtk(https://github.com/lh3/seqtk)过滤,最终得到最小等位基因频率(Minor allele frequency,MAF)>0.05的23 309个标记用于进一步关联分析。标记覆盖所有染色体区段,标记之间平均距离为28 kb。
1.3.3 全基因组关联分析
利用RTM-GWAS方法采用两阶段策略对大豆倒伏相关性状表型值作关联分析。
第一阶段模型公式如下:
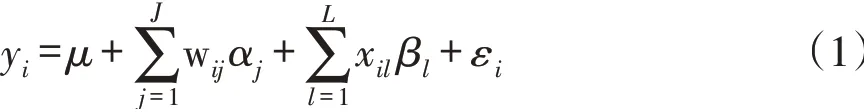
式中,yi表示个体i表型观测值;μ表示总体平均数;J表示用于群体结构矫正的特征向量个数,wij表示遗传相似系数矩阵第j个特征向量在个体i上的系数,αj表示第j个特征向量效应;L表示测验标记位点等位基因数目,xil表示测验标记位点第l个等位基因对于个体i基因型指示变量,取值0或1;βl表示第l个等位基因效应;εi表示假定服从正态分布残差效应。
第二阶段模型公式如下:

式中,K表示总QTL数目;Lk表示第k个位点等位基因数目,xikl表示第k个位点第l个等位基因在个体i上基因型指示变量,取值0或1;βkl表示第k个位点第l个等位基因效应。其他符号含义与模型(1)相同。
1.4 数据处理与分析
采用Microsoft Office Excel 11.1.0.10314、SPSS 3.0软件分析表型数据和作图。基于Phytozome数据库(https://phytozome.org/)和Soybase数据库(https://www.Soybase.Org/)中基因功能注释预测与大豆抗倒伏相关候选基因。在eFP数据库(https://bar.Utoronto.ca)查询候选基因表达量数据,利用R软件CMplot程序包以及pheatmap程序包绘制曼哈顿图和候选基因组织表达热图。
2 结果与分析
2.1 大豆倒伏相关性状表型数据分析
分析多环境下大豆自然群体各农艺性状相关性(见表1)。结果表明,在不同环境下倒伏级别与各性状相关性表现不同,除2019年呼兰点外倒伏级别与株高均表现显著相关,相关系数为0.243~0.527;两年阿城点倒伏级别与主茎节数均表现显著相关,相关系数为0.171~0.221;2018年呼兰点倒伏级别与节间长度表现显著相关,相关系数为0.275;2019年向阳点倒伏级别与茎粗均表现显著相关,相关系数为0.290;除2018年阿城点外倒伏级别与重心高度均表现显著相关,相关系数为-0.209~0.345;两年呼兰点倒伏级别与抗倒指数均表现显著相关,相关系数为-0.254~0.178,表明倒伏受植株株高、主茎节数、节间长度、茎粗、重心高度以及抗倒指数等性状影响显著,该性状均为倒伏显著相关性状。
分析多环境下大豆自然群体6个倒伏显著相关性状表型(见表2)。结果表明,不同环境下,大豆自然群体倒伏相关性状表现均不同。各倒伏相关性状在6个环境下偏度和峰度绝对值多数<1,且具有广泛变异,其中株高变异系数为0.13~0.29;主茎节数变异系数为0.11~0.23;节间长度变异系数为0.15~0.22;茎粗变异系数为0.14~0.23;重心高度变异系数为0.14~0.23;抗倒指数变异系数为0.18~0.44。

表1 大豆自然群体倒伏级别与不同农艺性状相关系数Table 1 Correlation coefficients between lodging grade and agronomic characters in soybean natural population

表2 大豆自然群体倒伏相关性状表型分析Table 2 Phenotypic analysis of lodging traits in soybean natural population
大豆自然群体6个倒伏显著相关性状在6个环境下频数分布情况见图1。结果表明,该群体各倒伏相关性状呈连续性变异,在群体中均呈正态分布,表明此性状为多基因控制数量性状,适合于关联分析。
2.2 大豆倒伏相关性状全基因组关联分析
利用23 309个单核苷酸多态性位点基因分型信息,通过RTM-GWAS程序对自然群体6个倒伏相关性状作多环境关联分析(见图2)。

图1 不同环境下大豆自然群体倒伏相关性状分布Fig.1 Distribution of lodging traits of soybean natural population in different environments
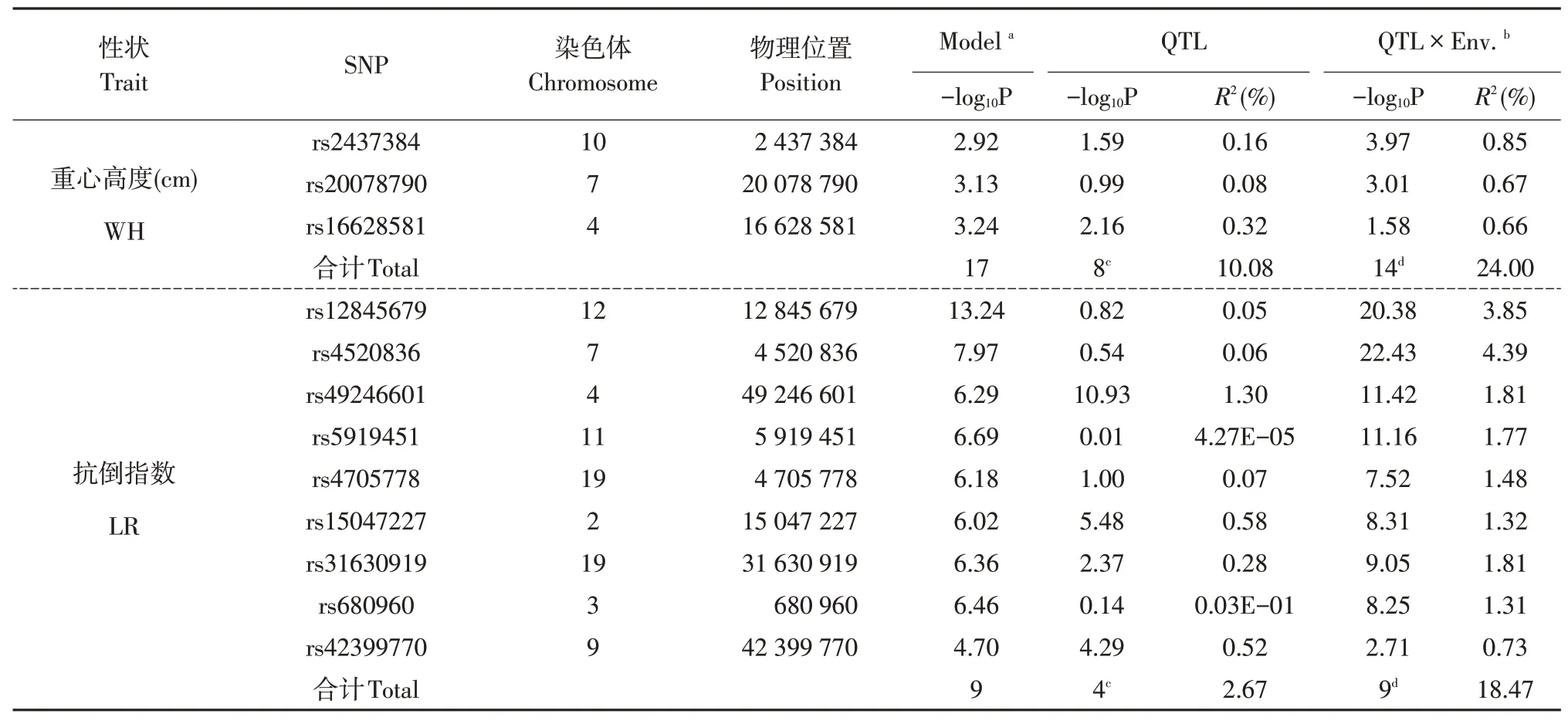
结果表明,在P<0.01显著性水平上,共检测到99个显著关联SNP位点,且这些显著关联SNP位点在大豆染色体上呈不均匀分布(见表3)。检测到16个与株高显著相关SNP位点,分布于1号、2号、3号、4号、6号、7号、8号、9号、13号、15号和18号染色体上,其中有9个效应显著位点总表型变异解释率为13.98%,14个与环境互作效应显著位点总表型变异解释率为19.47%,其中表型变异解释率高于1%位点5个。检测到19个与主茎节数显著相关SNP位点,分布于3号、4号、7号、9号、12号、13号、15号、18号、19号和20号染色体上,其中有10个效应显著位点总表型变异解释率为14.54%,16个与环境互作效应显著位点总表型变异解释率为34.45%,表型变异解释率高于1%位点7个。检测到18个与节间长度显著相关SNP位点,分布于2号、3号、4号、5号、10号、14号、15号、17号、18号、19号和20号染色体上,其中有7个效应显著位点总表型变异解释率为5.92%,18个与环境互作效应显著位点总表型变异解释率为37.81%,表型变异解释率高于1%位点1个。检测到20个与茎粗显著相关SNP位点,分布于2号、3号、4号、6号、8号、10号、11号、15号、16号和19号染色体上,其中有10个效应显著位点总表型变异解释率为7.73%,18个与环境互作效应显著位点总表型变异解释率为19.45%,表型变异解释率高于1%位点3个。检测到17个与重心高度显著相关SNP位点,分布于1号、4号、7号、8号、9号、10号、14号、15号、17号和20号染色体上,其中有8个效应显著位点总表型变异解释率为10.08%,14个与环境互作效应显著位点总表型变异解释率为24.00%,表型变异解释率高于1%位点4个。检测到9个与抗倒指数显著相关SNP位 点,分 布 于2号、3号、4号、7号、9号、11号、12号和19号染色体上,其中有4个效应显著位点总表型变异解释率为2.67%,9个与环境互作效应显著位点总变异解释率为18.47%,表型变异解释率高于1%位点1个。
2.3 候选基因预测
将以上检测到的99个显著SNP位点作候选基因挖掘分析,在Phytozome和Soybase数据库中共检索到22个有功能注释基因(见表4)。根据注释信息,该基因在大豆中主要与转运蛋白、各种激酶、氧化还原酶等相关。
研究表明倒伏相关基因主要在作物茎、叶、根及顶芽等部位表达丰富[19-20]。本研究检索Bar.utoronto.ca数据库中已收录大豆基因表达量数据,发现上述22个候选基因中20个基因在大豆10个组织中表达(见图3),其中与株高关联Glyma.15G124700基因在叶中高水平表达;与节间长度关联Glyma.04G244100基因在根瘤中较高水平表达,Glyma.10G144600基因在茎尖、叶、根、根尖及根毛中高水平表达,Glyma.18G149700基因在茎尖和根尖中高水平表达,Glyma.18G299500基因在茎尖、根及根尖中高水平表达;与茎粗关联Glyma.18G011400基因在根尖中表达水平相对较高;与抗倒指数关联Glyma.09G199600基因在根瘤中高水平表达,Glyma.11G078600基因在大豆10个组织中均低水平表达。根据基因表达量和基因功能注释推测,3个基因Glyma.04G244100、Glyma.18G149700、Glyma.18G011400为大豆倒伏性状相关重要候选基因。

图2 大豆倒伏相关性状全基因组关联分析曼哈顿图Fig.2 Manhattan plots of genome wide association analysis of lodging-related traits in soybean

表3 与大豆倒伏性状显著相关位点Table 3 Loci significantly associated with soybean lodging traits(P<0.01)

续表3
续表3

表4 大豆倒伏性状相关联候选基因信息Table 4 Information on candidate genes associated with lodging traits in soybean

图3 候选基因组织表达热图Fig.3 Heatmap profiles of the candidate genes in tissues
3 讨论
大豆具有极高营养和经济价值,从大豆种质资源中挖掘与大豆倒伏相关功能标记,通过标记辅助选择育种方法加快培育抗倒性强大豆品种是一种经济又有效的手段。近年来,随大豆基因序列公布和基因组学发展[21-22],促进大豆重要数量性状遗传解析进程[23]。RTM-GWAS是由He等提出的限制性两阶段多位点全基因组关联分析方法[24],与以往方法相比更适于自然群体(包括种质资源群体),可降低假阳性,使检测更为精准[25]。本研究利用RTM-GWAS对6个环境下大豆自然群体抗倒伏性状作遗传解析,以期发掘与大豆倒伏性相关重要标记和遗传位点,预测候选基因,为通过分子育种技术改良大豆抗倒性状奠定基础。
本研究共检测到99个与大豆倒伏相关性状显著关联SNP位点,均是与茎秆性状显著关联位点;共检索出22个有功能注释基因,其中2个基因与前人研究结果一致,如与重心高度相关联Glyma.15G223700在Kim、张彦威等研究中被定位到[26-27];与抗倒指数相关联Glyma.11G078600在Espinosa等研究中被定位到[28]。其中3个基因在不同植物中出现相关报道,与节间长度相关联UDP糖基转移酶超家族蛋白Glyma.18G149700,糖基转移酶基因家族主要功能是参与调节植物激素平衡、内外源物质解毒、防御反应和次生代谢产物修饰[29],细胞壁合成主要由不同类型糖基转移酶完成,糖基转移酶影响细胞壁组成成分,进而改变细胞壁理化结构与组成结构[30],以应对各种胁迫,根据Glyma.18G149700在茎尖和根尖中较高表达量,可将其作为与倒伏相关首要候选基因。如与节间长度相关细胞壁关联蛋白激酶2Glyma.04G244100,细胞壁关联蛋白激酶(WAK)具有信号转导、细胞扩大与伸长、生殖生长、防御病原菌和机械伤害、金属离子胁迫等生物学功能,在植物生长发育过程中发挥重要作用,Kanneganti和Wagner等研究推测WAK家族基因可能通过参与植物细胞壁元件相互作用调控细胞生长从而改变植株生长[31-32],且Glyma.04G244100在根瘤中具有较高表达量,可作为与倒伏相关重要候选基因之一。与茎粗相关联G蛋白偶联受体Glyma.18G011400,G蛋白偶联受体(GPCRs)主要负责生物细胞信号传导[33],参与植物激素和抗逆反应,在植物胁迫响应中发挥重要作用[34]。Dymock等发现拟南芥GCR1基因失活将导致植株根、茎对细胞分裂素不敏感,根、子叶和叶片伸展受到抑制[35]。Chakraborty等研究表明拟南芥GCR1突变体植株中胁迫相关基因表达量较野生型差异明显,GCR1影响胁迫基因表达[36]。由此可知,G蛋白偶联受体基因通过调控植物细胞对多种激素信号和外界胁迫响应过程抵抗逆境[34];Glyma.18G011400在根尖中具有相对较高表达量,也可作为与倒伏相关重要候选基因之一。上述基因可在逆境中发挥作用,通过参与调控植物体细胞建成、信号传导、生长发育等多种生理活动过程抵抗逆境胁迫,当植株发生倒伏时,基因具体调控机制尚不明确。
此外,本研究中还有4个分别在叶、根瘤、根及根毛、根尖中高表达基因(Glyma.15G124700、Glyma.09G199600、Glyma.10G144600、Glyma.18G299500)首次被定位到,但目前在这些基因相关研究中尚未发现其与倒伏之间联系,还需进一步研究。
综合基因表达量分析和基因功能注释,可将Glyma.18G149700、Glyma.04G244100、Glyma.18G011400等3个基因依次作为研究与大豆倒伏性状相关重要候选基因。
4 结论
a.在大豆自然群体中共检测到99个显著关联SNP位点,在大豆染色体上呈不均匀分布,其中48个主效显著位点,89个与环境互作显著位点;其中21个位点表型变异解释率高于1%。
b.检测到与株高显著关联SNP位点16个,与主茎节数显著关联SNP位点19个,与节间长度显著关联SNP位点18个,与茎粗显著关联SNP位点20个,与重心高度显著关联SNP位点17个,与抗倒指数显著关联SNP位点9个。
c.共检索出22个有功能注释基因,通过20个候选基因在大豆10个组织中均表达,根据基因表达量分析和基因功能注释,推测3个基因(Glyma.18G149700、Glyma.04G244100、Glyma.18G011400)可作为大豆倒伏性状相关候选基因。
