基于细胞色素C氧化酶亚基Ⅰ序列的DNA微条形码技术鉴别11种生鲜肉制品掺假的研究
2021-03-20励炯吴琼扈明洁金朦娜邱红钰
励炯,吴琼,扈明洁,金朦娜,邱红钰
(杭州市食品药品检验研究院,杭州310017)
为预防人畜共患的传染病,国内许多地区已经禁止市场交易活禽类,消费者只能购买宰杀后的生鲜肉或者生鲜肉制品来满足日常的饮食需求[1]。一般情况下,消费者在整只或者整块切割购买生鲜肉时,可以通过感官,从肉的肌理以及色、香、味等指标对其进行鉴别。但是很多生鲜肉制品,比如肉干、肉丸、肉糜、肉卷等经过加工后,破坏了原来的一些性状,导致消费者很难用感官来辨别,所以他们就只能通过参考外包装上的标签信息来购买所需的生鲜肉制品[2-3]。这就导致一部分不法商家在生鲜肉制品中掺假掺杂一些低经济价值的肉,以获得更大的利润[4]。一些文献报道显示,在墨西哥、土耳其以及南非等地区,包括肉肠、碎肉制品、肉丸以及肉干中的掺假率为20%~70%[5-7]。南非的一份研究揭露了南非地区销售的生鲜肉制品中,高达68%的样品中含有标签中没有标示的肉品种。在2013年欧洲发生的肉类掺假丑闻中,很多标识纯牛肉的产品中掺有马肉。英国食品标准局(Food Standards Agency,FSA)开展的一项牛肉千层面掺假的调查中,发现有60%的产品掺有马肉。由爱尔兰食品安全署(Food Safety Authority of Ireland,FSAI)开展的一项针对牛肉汉堡、碎牛肉制品以及牛肉肠的掺假调查中,发现有37%的产品掺有马肉,85%的产品掺有猪肉[8-9]。自从出现大规模的掺假肉制品事件后,欧洲各国已经采取各种积极措施,以避免各种肉类掺假事件的发生,从而保障消费者的权益。在美国,虽然各州政府部门都严令禁止销售掺假肉制品,但早在1995 年前的一项研究表明,虽然未发现以整块销售的肉的掺假问题,但是有近17%的碎肉制品中的种类与标签标示的不一致[10]。
在国内的食品安全事件中,“掺假”问题一直是消费者投诉的焦点和社会关注的热点。一些不法商贩和企业因利益驱使将猪、鸭等低成本原料掺入牛、羊等高价肉及肉制品中,如果原料未经检验检疫,还将涉及疫病的传播[11-12]。这些行为不仅侵害了消费者的利益,还可能因清真食品中含有非清真成分而引起纠纷,不利于维护社会安定[13]。
对经过加工处理的畜禽类肉制品中的掺假进行鉴别,一般采用DNA或者基于蛋白质的分子生物学方法[14-15],包括酶联免疫吸附测定(enzyme-linked immunosorbent assay, ELISA)[16]、聚合酶链式反应(polymerase chain reaction, PCR)[17-18]、限制性内切酶片段长度多态性(restriction fragment length polymorphism,RFLP)[19]技术和DNA测序[20]等方法。ELISA和PCR法的优点是能快速、准确地鉴定一些深加工的产品,包括掺混等现象[21]。尽管PCR 在肉类和禽肉类掺混鉴别中能同时鉴定出多种不同种类的肉,而且灵敏度高,但缺点是每一类品种都需要不同的引物,实验操作比较烦琐[14]。RFLR 相对PCR法的优势是不需要特定引物,通用型的引物就可以满足实验要求,但缺点是步骤烦琐,需要比较长的DNA目标物,而且RFLR的结果分析也比较烦琐,需要大量的酶来进行不同品种的肉类和禽肉类鉴定[14]。近年来,用质谱手段分析蛋白质和多肽的方法,成功解决了分子生物学的一些应用问题,但是由于实施质谱手段需要昂贵的设备以及复杂的操作技术,因此,并未被广泛应用于肉类掺假鉴别中[22-23]。
DNA条形码(DNA barcoding)技术是基于DNA序列测定的技术,该技术被广泛应用于物种鉴别上[24]。该技术已经被美国食品药品管理局(Food and Drug Administration, FDA)应用于水产品的鉴定上,并被一些研究用于肉类和禽肉类产品中的掺混鉴别[25]。DNA条形码技术基于标准的基因片段,是利用标准的、有足够变异的、易扩增且相对较短的DNA片段而创建的一种新的身份识别系统,且该DNA 片段在物种内具有足够的特异性和种间多样性,可以快速、有效地鉴别动物、植物和真菌类物种。一般采用全片段DNA 条形码(full-length DNA barcoding)技术来对保存完好的新鲜样本进行鉴别,比如肉类鉴别中采用的大约650 bp大小的细胞色素C 氧化酶亚基Ⅰ(cytochrome C oxidase subunitⅠ,COⅠ)序列基因片段[26]。但是,在实际研究中发现,受环境等外来因素影响,部分DNA 可能会被破坏分解,运用全片段DNA条形码技术进行物种鉴别非常困难,所以MEUSNIER等[27]设计了专门针对全片段DNA 条形码中部分小片段DNA 的通用型引物,也就是DNA 微条形码(mini-DNA barcoding)技术。他们发现,利用该通用型“微条形码引物”,测试的92%物种的DNA 小片段能够进行扩增,包括哺乳动物、鱼类、禽鸟类以及昆虫类。因此,本文采用基于COⅠ序列的DNA微条形码技术对生鲜肉制品中11种肉类进行了掺假鉴别研究。
1 材料与方法
1.1 主要仪器与试剂
高速离心机(德国Sigma公司),干式恒温器(杭州奥盛仪器有限公司),Veriti 96 孔梯度PCR 仪(美国ABI 公司),EYELA FDU-1100 真空冷冻干燥器(日本东京理化公司),PowerPac Basic电泳仪(美国Bio-Rad 公司),Gel Doc XR+凝胶成像系统(美国Bio-Rad 公司),NanoDrop 1000 微量核酸蛋白测定仪(美国Thermo公司)。
血液与组织DNA 提取试剂盒(德国Qiagen 公司),PCR 产物及DNA 片段回收试剂盒、T4 连接试剂盒、DH5α感受态细胞[天根生化科技(北京)有限公司],即用PCR扩增试剂盒[生工生物工程(上海)股份有限公司]。
1.2 方法
1.2.1 样品采集与预处理
所用的11种肉类(包括猪肉、牛肉、羊肉、鸡肉、鸭肉、鸽子肉、马肉、驴肉、鹅肉、兔肉、鼠肉等)为未加工过的完整生鲜肉,作为本研究用的纯肉。所用的样品部分来自于2019 年杭州市场监督管理部门抽样,部分采购于超市,包括牛肉、羊肉、鸽子肉、鹅肉、兔肉等共5种生鲜肉制品,共计25批次,包括肉糜、肉条、鲜肉丸、风干肉、肉排、肉卷等。将11种纯肉以及25批次生鲜肉制品分别用料理机打匀,肉末放于-40 ℃冰箱中预冷4 h,将预冷后的样品放在真空冷冻干燥箱中,在气压低于1×10-4Pa、-50 ℃条件下干燥24 h,随后将样品经中药粉碎机粉碎,置于-20 ℃条件下保存,备用。
1.2.2 DNA 提取
取真空冷冻干燥后的样品粉末0.5 g,置于10 mL聚氟乙烯离心管中,加入2 mL ATL组织裂解缓冲溶液和200 μL 蛋白酶K,漩涡振荡30 s 后,经56 ℃裂解3 h(每隔半小时进行漩涡振荡一次)。取裂解液250 μL,采用柱膜法提取试剂盒(DNeasy 血液与组织提取试剂盒)进行提取和净化DNA。用37 ℃预热的三羟甲基氨基甲烷-乙酸-乙二胺四乙酸(TAE)缓冲液洗脱DNA,并将其储存在-20 ℃条件下。同时,设置空白对照。
1.2.3 引物的选择
DNA微条形码技术一般是选用100~300 bp大小的片段进行扩增,理想的微条形码片段需要具备以下特征:1)标准的DNA 片段,其目标片段便于提取和扩增;2)包括足够的系统进化信息,具有足够的遗传变异性和一定的分化性;3)便于设计通用引物。以往的研究发现,细胞色素C氧化酶Ⅰ(COⅠ)是动物界中最合适的DNA条形码标准基因。所以,本文最终选用了MEUSNIER 等[27]公开发表的一对引物(COⅠ-A),且为了方便DNA测序,在每对引物上连接M13,由杭州擎科梓熙生物技术有限公司合成。
1.2.4 PCR 扩增
对每批样品中提取的DNA 模板进行微型片段的PCR 扩增。每个PCR 扩增体系(25 μL)如下:PCR 扩增试剂12.5 μL,ddH2O 9.5 μL,10 μmol/L 正向引物0.5 μL,10 μmol/L 反向引物0.5 μL,DNA 模板2 μL。DNA 微条形码片段扩增程序:95 ℃预变性2 min;95 ℃变性1 min,46 ℃退火1 min,72 ℃延伸30 s,循环5次;95 ℃变性1 min,53 ℃退火1 min,72 ℃延伸30 s,循环35 次;最后,72 ℃延伸10 min。扩增产物于-20 ℃条件下保存。
1.2.5 PCR 扩增片段的检验及DNA 测序
采用2.0%琼脂糖凝胶电泳对PCR 扩增片段进行检验。每一个凝胶孔内加入3 μL上样缓冲液(日本TaKaRa公司)和6 μL PCR扩增产物,电泳20 min,同时设置DNA 标志物和阴性空白对照。PCR 扩增产物经分离后,在上述凝胶成像系统中进行确认分析。然后,将PCR扩增产物送至杭州擎科梓熙生物技术有限公司进行测序,所得的测序结果经删除两端引物序列,获得最终的扩增序列。
1.2.6 PCR 产物克隆测序
PCR 产物经纯化后直接测序,若出现杂峰,则说明该扩增DNA 条带不止一个物种。针对多种肉制品掺混的情况,采用克隆测序,从PCR 产物中挑选不同阳性克隆进行测序分析。纯化后的PCR 产物与pGEM-T 载体连接后,导入DH5α 感受态细胞上,于36 ℃培养箱中培养12 h,经过蓝-白筛选后,挑取10 个不同的白色菌落接种到LB(Luria-Bertani)液体培养基上,并于36 ℃条件下,在摇床上以150 r/min 培养过夜;取菌液1 mL,提取质粒DNA,并送至杭州擎科梓熙生物技术有限公司进行测序。测序结果提交到GenBank数据库(http://blast.ncbi.nlm.nih.gov/Blast.cgi)中进行Blast比对。
2 结果与分析
2.1 DNA 提取方式优化
DNA 的一般提取方法是用一次性镊子直接取适量的组织,然后进行裂解提取,但是本实验需要考察肉的掺假情况,所以样品的均一性对于实验结果的影响非常大,如果直接取或者简单用料理机搅拌处理后取适量,会导致漏检。针对上述情况,本研究采用真空冷冻干燥法对样品进行预处理。取一定量代表性的样品组织,冷冻干燥后,将样品用粉碎机粉碎均匀。为验证不同预处理的可靠性,采用牛肉掺猪肉模型[掺假(猪肉)比例为10%],分别为经料理机搅拌处理的掺假样品A 和经冷冻干燥处理后的掺假样品B,取A和B样品各10份,前处理和分析参照1.2节。试验结果发现,在样品A中,检出猪肉成分的比例为20%,而B 中的猪肉检出比例为100%。所以,本研究最终选择冷冻干燥法对样品进行预处理。
样品的取样量也会决定样品取样的代表性,按照DNeasy 血液与组织提取试剂盒的要求,一般纯的组织样品只需要取10~25 mg 即可满足试验要求,但是如果掺假样品的取样量太少,则会影响取样的代表性,造成漏检。且在肉类加工中,如果没有对加工设备进行完全清洁,则可能在加工下一批次其他种类的肉的时候带入上一批次的肉(一般量非常少,我们可以认为这种行为不属于掺假),这时如果取样少、没有代表性,则可能会造成对试验结果的误判。所以,本研究采用牛肉掺猪肉模型验证样品取样量的可靠性。样品经料理机搅拌处理后,对25 mg 和0.5 g 取样量进行考察,分别取10 份样,前处理和分析参照1.2节。试验结果发现:当取样量为25 mg时,样品中检出猪肉成分的比例为30%,而当取样量为0.5 g 时,样品中检出猪肉成分的比例为100%。所以,本研究最终采用0.5 g 的取样量进行DNA提取。
对样品的预处理方式和取样量进行优化后,本研究继续对DNA 提取方式进行选择和优化。动物组织DNA一般采用快速提取法和柱膜法进行提取。快速提取法的优点是简单、快捷、高效,缺点是没有进行纯化,DNA样品中带入的杂质会影响后续PCR的效率;柱膜法的优点是提取的DNA纯度高、干净,基本去除了样品中带入的杂质,缺点是步骤烦琐,成本高。本研究分别采用快速提取盒和柱膜法提取试剂盒(DNeasy 血液与组织提取试剂盒)提取11种肉的DNA,提取后的DNA 经PCR 扩增和凝胶电泳确认。试验结果发现:经柱膜法提取净化的DNA条带比较清晰;而经快速提取法提取的DNA 中,牛肉、羊肉、马肉没有相应的清晰条带,同时柱膜法的条带亮度明显高于快速提取法。所以,本研究最终采用柱膜法对肉类的DNA进行提取净化。
2.2 DNA 微条形码序列片段扩增条件的优化
11 种肉样品DNA 经优化的预处理和提取后,分别以通用引物COⅠ-A、COⅠ-B和COⅠ-C(具体引物序列见表1)进行PCR 扩增,扩增产物用2%琼脂糖凝胶电泳进行检测。结果表明:引物COⅠ-B扩增的目标片段大小为250 bp左右,不能扩增出鼠的PCR 产物,而在250 bp 左右有颜色较浅的条带;引物COⅠ-C 扩增的目标片段大小为200 bp 左右,不能扩增出羊、马和驴的PCR产物;引物COⅠ-A扩增的目标片段大小为130 bp左右,能全部扩增出11种肉的PCR 产物。所以,本研究采用COⅠ-A 作为生鲜肉制品中11种肉掺混研究的引物。

表1 引物序列Table 1 Primer sequence
同时,本研究对PCR 扩增条件进行优化,包括PCR扩增体系和扩增程序的优化。采用25 μL扩增体系对DNA 模板浓度进行考察,当DNA 模板浓度过低或者过高时都会抑制扩增效率。本研究分别采用1、2、3 μL 的DNA 模板量,当DNA 模板量为1和3 μL 时,11 种肉类的DNA 扩增条带颜色较DNA模板量为2 μL 的时候淡,所以本研究采用的DNA模板量为2 μL。同时,我们对扩增程序进行了优化。在PCR扩增程序中,退火温度对扩增效率影响较大。本研究采用50、53和57 ℃这3个退火温度分别对11 种肉的DNA 扩增效率进行考察。结果发现:当退火温度为53 ℃时,11种肉的DNA扩增效率最高,电泳条带最清楚,而且在该扩增条件下,低经济价值的肉类(猪肉、鸡肉、鸭肉、马肉、驴肉、鼠肉)的DNA扩增效率较高,能满足本研究掺假模型的建立以及目标掺假肉类的检测。
11 种肉的PCR 扩增产物电泳结果如图1 所示。11种肉在100~200 bp之间均有一条较明显的扩增条带。

图1 11种纯肉的DNA微条形码PCR电泳图Fig.1 Electrophoresis images of DNA mini-barcoding fragments from 11 species of meats amplified by PCR
将11 种肉的PCR 扩增产物送至杭州擎科梓熙生物技术有限公司进行克隆测序,测序鉴定结果如表2所示。
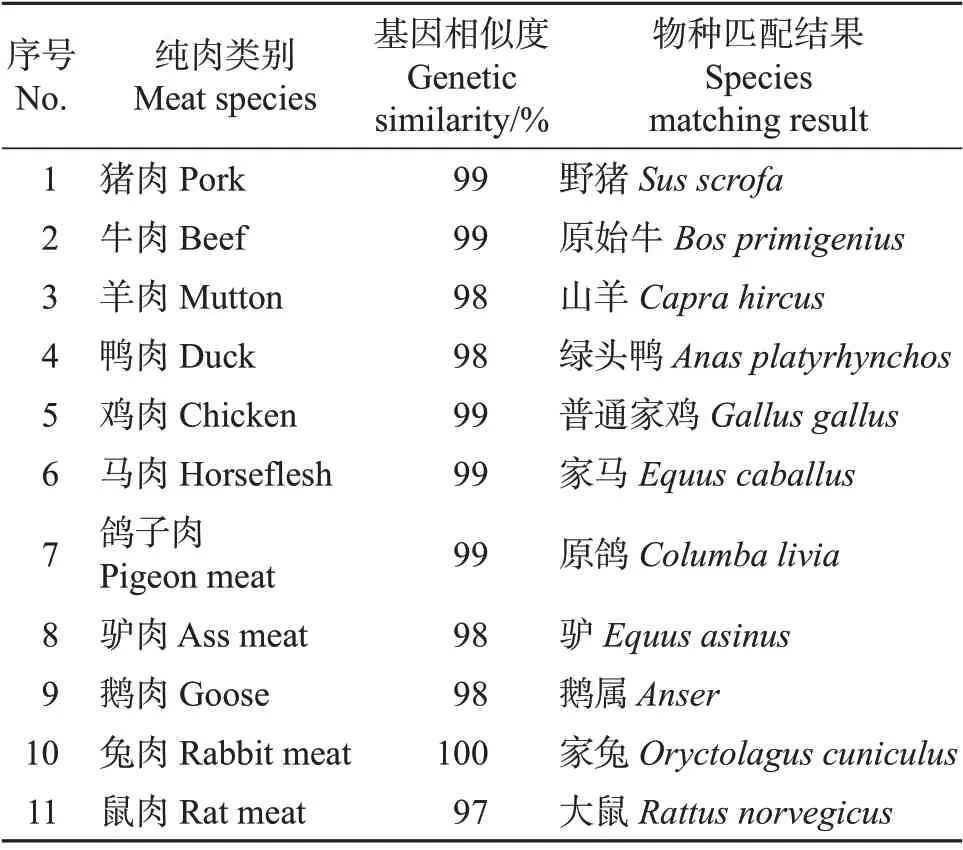
表2 11种纯肉的DNA微条形码检测结果Table 2 DNA mini-barcoding testing results for 11 species of meats
2.3 生鲜肉掺假模型的建立
本研究主要设置18种肉的掺假模型,采用高经济价值的肉类(牛肉、羊肉、鸽子肉、鹅肉、兔肉)与低经济价值的肉类(猪肉、鸡肉、鸭肉、马肉、驴肉、鼠肉)的掺混模式,具体见表3。

表3 肉类掺假模型Table 3 Meat adulteration model
在生鲜肉制品加工过程中,除了破坏其正常的感官性状之外,还可能加入一些香精、香料、色素等添加剂,以掩饰掺入的其他低经济价值的肉类。基于此,本研究考察了5 种高经济价值肉类掺假的可能性,一共建立18 个掺假模型,然后在每个掺假模型中分别加入5%、10%、15%和20%的掺假比例来考察本方法的最低检出比例(检测灵敏度)。试验结果发现:本方法对牛肉和羊肉生鲜肉制品中掺假的检出灵敏度较高,6 种掺假肉类(猪肉、马肉、驴肉、鸭肉、鸡肉、鼠肉)的最低检出比例为5%,而在鸽子肉、鹅肉、兔肉的掺假模型中,各种肉类的最低检出(掺假)比例均为10%,表明本方法能满足日常的检测要求。
2.4 实际样品检测
为了进一步验证建立的检测方法的适用范围,在实际样品检测中,尽量选择不同性状的生鲜肉制品,包括肉糜、肉条、鲜肉丸、风干肉、肉排、肉卷等。
利用建立的检测方法,25 批次生鲜肉制品经提取DNA 模板和扩增之后,均能得到较清晰的条带。阴性对照组无扩增条带,证明整个体系均未被污染。将扩增的PCR 产物送至杭州擎科梓熙生物技术有限公司进行克隆测序,结果如表4 所示。从中可以看出:11 批次的生鲜牛肉制品中有4 批次检出掺假其他肉类,其中3 批次检出猪肉成分,1 批次检出鸭肉成分,掺假比例为36%,主要集中在牛肉卷、牛肉丸、牛肉糜以及牛肉条中;9 批次的生鲜羊肉制品中有2 批次检出掺假其他肉类,其中1 批次生羊肉串检出鸭肉成分,1 批次羊肉丸检出猪肉成分,掺假比例为22%;2 批次的生鲜鹅肉制品、2 批次的生鲜兔肉制品和1 批次的鸽子肉均未发现有掺假现象。25 批次的生鲜肉制品中一共有6 批次出现掺假问题,掺假率高达24%。其中,有4 批次的生鲜肉制品中检出猪肉成分,2 批次的生鲜肉制品中检出鸭肉成分。同时,为了验证本检测方法的可靠性,我们对上述6 批次的掺假样品采用农业行业标准《肉类源性成分鉴定 实时荧光定性PCR法》(NY/T 3309—2018)进行检测,本方法与此结果一致。从上述结果可以看出,2 类高经济价值生鲜肉类制品的掺假率比较高,会被掺入猪肉和鸭肉成分。虽然从理论上说这些掺假肉在感官、性状等方面与被掺假的肉不一样,但是在实际生产过程中一些不法商家会加入一些添加剂(色素、香精、香料等)来改变生鲜肉制品原有的性状,使得消费者很难用肉眼来辨别真假,也导致市售生鲜肉制品的掺假率居高不下。

表4 实际样品检测结果Table 4 Detecting results of practical samples
3 小结
本文建立了基于COⅠ的DNA 微条形码技术,以对生鲜肉制品中的11种肉掺假情况进行鉴定;同时,优化了样品前处理、DNA提取和PCR扩增条件。与其他检测技术相比,该DNA微条形码技术检出灵敏度较高,简便、经济、高效,适合正常的生鲜肉制品掺假鉴别,又不易出现误判,能够满足日常大批量生鲜肉制品质量监督检测的需求。
