盐胁迫下四个水稻类受体蛋白激酶的功能分析
2021-03-17路凯陈涛姚姝梁文化魏晓东张亚东王才林
路凯 陈涛 姚姝 梁文化 魏晓东 张亚东 王才林
盐胁迫下四个水稻类受体蛋白激酶的功能分析
路凯 陈涛 姚姝 梁文化 魏晓东 张亚东*王才林*
(江苏省农业科学院 粮食作物研究所/江苏省优质水稻工程技术研究中心/国家水稻改良中心南京分中心,南京 210014;*通信联系人,E-mail:zhangyd@jaas.ac.cn;clwang@jaas.ac.cn)
盐胁迫是制约水稻生长和产量主要逆境之一,研究盐胁迫响应基因对于了解植物耐盐机理和培育耐盐水稻品种具有重要意义。类受体蛋白激酶RLK(receptor-like protein kinases)广泛参与调控植物细胞信号转导和对逆境胁迫的响应过程。本研究的目的是分析盐胁迫下四个RLK基因的表达模式和生物学功能。通过荧光定量PCR检测4个基因在NaCl处理下的表达变化以及在不同组织器官中的表达情况,同时利用CRISPR/Cas9对4个基因分别进行编辑。4个基因的转录均受NaCl诱导或抑制,其中基因和基因主要在根中表达;基因主要在叶片中表达;基因在根、茎、叶、叶鞘中均有表达。通过测序分别筛选到4个基因的功能缺失突变体,耐盐性实验结果表明四个基因的突变体对NaCl的敏感程度与野生型一致。鉴定的4个基因的转录受NaCl调控且表达具有组织特异性,突变单个基因不影响水稻的耐盐性。为进一步揭示盐胁迫下基因的功能和作用机制奠定了基础。
类受体蛋白激酶;盐胁迫;基因编辑;粳稻
水稻是世界上最主要的粮食作物之一,也是我国种植面积最大的农作物,而盐胁迫是制约其生长和产量的主要逆境之一[1-2]。水稻栽培面积的20%受到不同程度的盐害威胁,这些盐碱地分布于沿海及部分内陆地区[3-4]。盐胁迫严重影响水稻的生长发育和代谢,高浓度的Na+可以直接对细胞膜造成伤害,而Na+的间接作用常导致渗透胁迫,使植物根系吸水困难,从而造成水分和营养的亏缺[5-7]。
研究表明类受体蛋白激酶RLK(receptor-like protein kinases)广泛参与到细胞信号转导和植物对逆境胁迫的响应过程[8-9]。近年来,已有报道表明RLK蛋白在植物响应盐胁迫过程中起着非常重要的作用[10]。水稻()基因编码一个典型的LRR型RLK蛋白,其表达受到NaCl、干旱和H2O2的诱导。高表达的转基因水稻对盐和干旱胁迫的抗性均比对照增强,而基因的两个功能缺失突变体和和RNAi株系均对盐和干旱敏感[10]。水稻()基因编码凝集素样类受体蛋白激酶LecRLK,其表达受NaCl的诱导。SIT1的RNAi株系对盐胁迫的抗性明显增强,而且体外和体内磷酸化的结果表明在NaCl存在条件下SIT1可以直接磷酸化MPK3和MPK6[11]。在拟南芥中高表达基因可以使植物对盐的敏感性增强[12]。以上研究结果表明,类受体蛋白激酶通过细胞信号转导调节下游基因表达来影响植物对盐胁迫的耐性。
RLK家族是植物最大的膜受体家族,它在拟南芥成员数达610多个,水稻中达1100多个[8-9]。典型的类受体蛋白激酶由胞外结构域(extracellular domain)、单次跨膜结构域(transmembrane domain)和胞内激酶结构域(cytoplasmic kinase domain) 三部分组成。其中,胞外结构域主要用来感受信号或外界的刺激;胞内激酶结构域比较保守,通过磷酸化下游底物来传递信号[13-14]。RLK的胞质激酶结构域有一个非常保守的赖氨酸,当把赖氨酸突变成谷氨酸后,RLK的激酶活性丧失,从而不能磷酸化下游底物和传递信号[23-24]。根据胞外结构域的结构以及胞内激酶结构域氨基酸序列的差异,RLK蛋白可以分为不同的亚家族:富含半胱氨酸的类受体蛋白激酶CRK(cysteine-rich repeat RLKs)、凝集素样类受体蛋白激酶LecRLK(lectin receptor kinase)、富含亮氨酸的类受体蛋白激酶LRR-RLK(leucine-rich repeat RLKs)、S结构域的类受体蛋白激酶(S-domain RLKs)等[14, 17]。
本研究以优良食味水稻品种南粳9108作为野生型植株,利用CRISPR/Cas9技术分别敲除四个新的响应盐胁迫的类受体蛋白激酶编码基因(、、、),并分析了四个基因的表达模式,以期对这些基因在水稻遭受盐胁迫时的生物学功能进行阐释。
1 材料与方法
1.1 试验材料
以南粳9108为野生型(wild-type, WT)对照并将其作为转化受体材料,基因编辑相关载体(pYLCRISPR/Cas9PUbi-H和pYLsgRNA-OsU6a/U3)由华南农业大学刘耀光院士馈赠[18]。实验所用引物序列见表1,引物合成以及DNA测序工作在南京擎科生物技术有限公司完成。
1.2 基因表达模式分析
将野生型南粳9108种子置于1/4 MS培养液中培养10 d,然后将幼苗分别置于含有0 mmol/L NaCl、200 mmol/L NaCl、100 μmol/L ABA和200 mmol/L甘露醇(D-Mannitol)的1/4 MS培养液中培养6 h。收材料提取总RNA并且反转录cDNA。以作为内参,实验重复3次,结果一致。提取幼苗以及灌浆期水稻的根、茎、叶、叶鞘的RNA。收材料提取总RNA并且反转录cDNA。荧光定量PCR检测各组织中基因的表达。
1.3 基因编辑靶位点选择
登录NCBI(National Center for Biotechnology Information)网站(https://www.ncbi.nlm.nih.gov/),根据4个基因登录号(、、、)搜索其序列。参考CRISPR-P2.0(http://crispr.hzau. edu.cn/ CRISPR2/)网站推荐的靶位点,在靠近基因编码蛋白的N端,挑选两个特异性较高的位点(PAM序列前19/20 bp)序列作为sgRNA结合的靶位点。
1.4 CRISPR/Cas9敲除载体的构建
CRISPR/Cas9敲除载体的构建参考刘耀光院士发表论文所用方法,即先将靶位点构建到入门载体pYLsgRNA-OsU6a/U3,然后利用Golden Gate连接方法将两个sgRNA表达盒片段进行高效组装[18]。首先,构建OsU6a/U3-Target1-sgRNA和OsU6a/U3-Target2-sgRNA两个入门载体。两个靶点接头的正反向引物(Target-1F+Target-1R,Target-2F+Target-2R)退火形成双链DNA片段Target-1和Target-2,采用边切边连的方法构建入门载体。反应体系(10 μL)包括1 μL pYLsgRNA-OsU6a/U3(约10 ng),1 μL接头,1 μL 10× T4 DNA 连接酶缓冲液,0.2 μL T4 DNA 连接酶,0.5 μLⅠ,1 μL CutSmart缓冲液,5.3 μL dd H2O。然后,经过两轮巢式PCR扩增sgRNA表达盒,琼脂糖凝胶回收PCR产物。最后,利用Golden Gate连接方法将两个sgRNA表达盒构建到表达载体pYLCRISPR/Cas9PUbi-H。反应体系(10 μL)包括1 μL Target1-sgRNA(约20 ng),1 μL Target2-sgRNA(约20 ng),1 μL pYLCRISPR/Cas9PUbi-H(约50 ng),1 μL 10×T4 DNA连接酶缓冲液,0.2 μL T4 DNA 连接酶,0.5 μLⅠ,1 μL CutSmart缓冲液,4.3 μL dd H2O。将连接后的产物转化大肠杆菌,对阳性菌落利用引物对(Target-1F+Target-2R)进行PCR鉴定排除假阳性。阳性克隆送公司进行测序鉴定,确保CRISPR/Cas9敲除载体构建成功。构建pYLCRISPR /Cas9PUbi-RLKs-sgRNA载体转化农杆菌EHA105,对阳性克隆经PCR鉴定正确后备用。
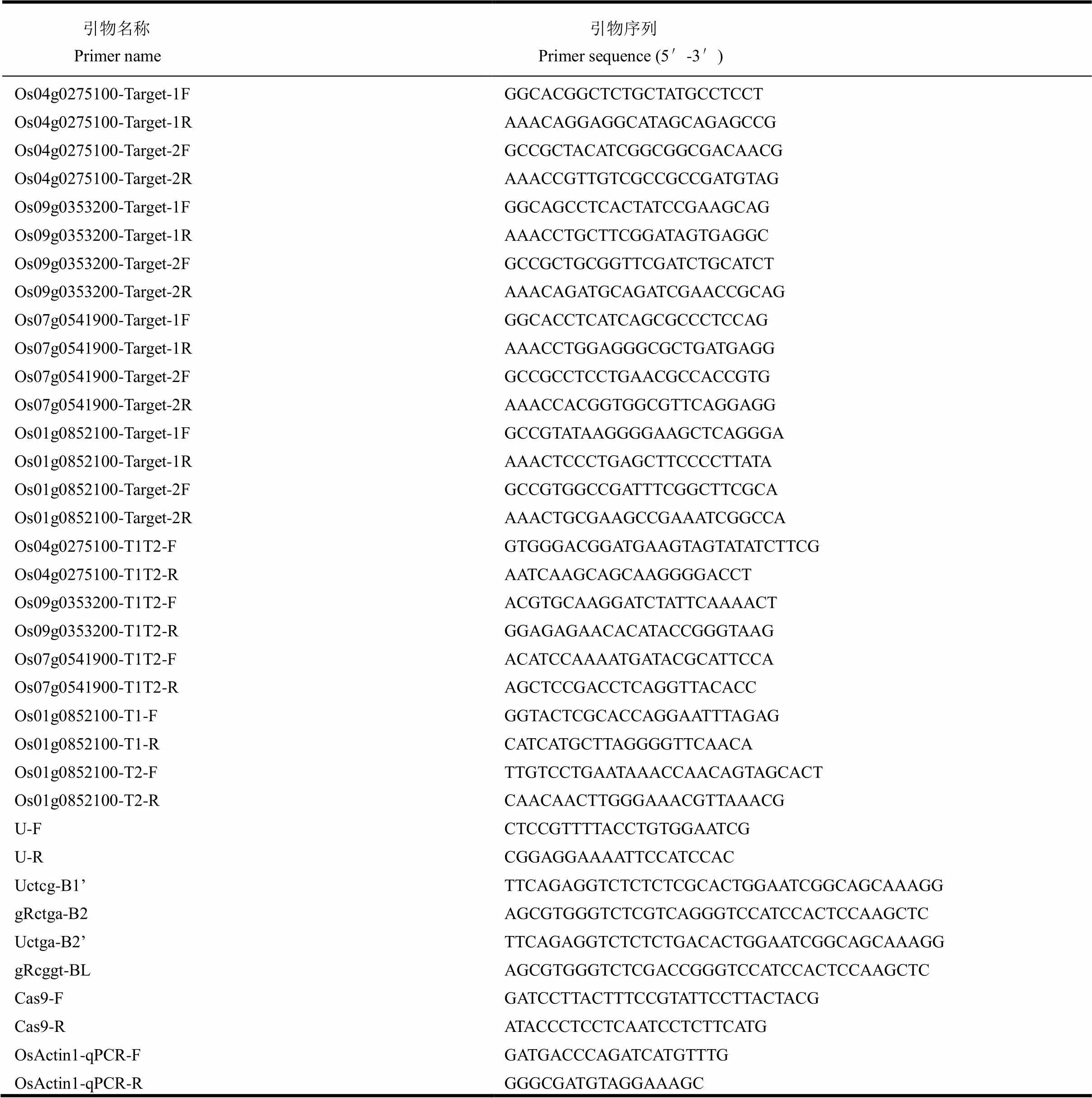
表1 本研究所用引物序列
1.5 转基因阳性苗的获得
利用含有pYLCRISPR/Cas9PUbi-RLKs-sgRNA载体的农杆菌对南粳9108愈伤组织进行转化,用潮霉素对转化后的愈伤组织进行两次抗性筛选。诱导阳性愈伤分化成苗,最后诱导生根。将生长至合适大小的植株转移至土壤中继续生长,取叶片提取DNA,并利用引物对(Cas9-F和Cas9-R)进行PCR扩增,进一步对阳性苗进行鉴定。
1.6 靶位点突变检测
以1.5中检测呈阳性的叶片DNA为模板,在两个靶位点的上游和下游设计一对引物(RLKs-T1T2-F+RLKs-T1T2-R,或者RLKs-T1-F+ RLKs-T1-R,RLKs-T2-F+RLKs-T2-R)对靶位点进行扩增。利用上游扩增引物对扩增产物进行测序分析。此外,以野生型南粳9108的基因组DNA为模板进行扩增和测序,通过比较野生型和突变体的测序峰图判断靶位点的突变类型。
1.7 盐胁迫下的表型分析
突变体和野生型种子在37℃黑暗条件下萌发2 d,选择长势一致的幼苗在1/2MS培养液中培养10 d,培养条件为温度30℃、16 h光照/8 h黑暗。然后将其转移至含有120 mmol/L NaCl的1/2MS培养液中培养8 d,拍照记录幼苗长势。继续在不含NaCl的1/2 MS培养液中恢复培养一周,拍照记录幼苗长势并统计存活率。
2 结果与分析
2.1 盐胁迫下四个类受体蛋白激酶的表达模式分析
为鉴定水稻中响应盐胁迫的类受体蛋白激酶,我们前期通过RNA-seq的方法筛选了水稻中受NaCl诱导或者抑制的基因。然后再进一步利用荧光定量PCR的对RNA-seq的结果进行验证,筛选到4个转录水平受NaCl调节的,其中1个基因()转录受诱导、3个基因(、、)转录受抑制(图1)。此外,还检测了ABA和甘露醇对表达水平的影响。结果表明基因受甘露醇诱导,基因转录受ABA和甘露醇抑制,基因转录受ABA抑制,基因转录受ABA诱导(图1)。这些结果反映了这4个基因有可能参与到植物对盐和非生物胁迫的响应过程。
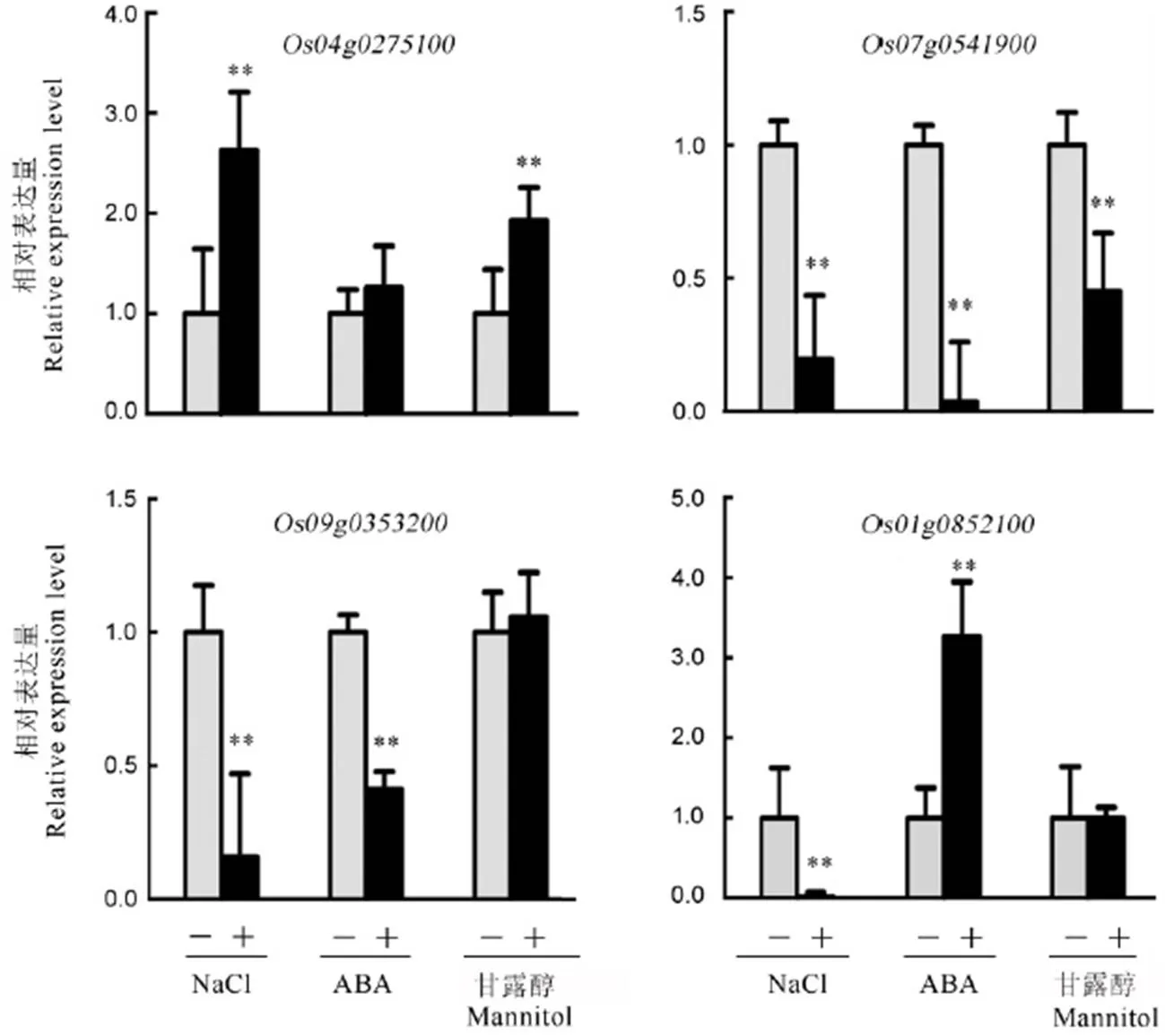
荧光定量检测RLK基因的相对表达水平。**代表处理间差异达显著水平(P<0.01)。

qPCR检测RLK基因在灌浆期水稻的根、茎、叶、剑叶、叶鞘中的相对表达水平。
2.2 四个RLK基因在不同组织器官中表达分析
为了分析4个基因在不同组织器官中的表达情况,我们利用荧光定量PCR检测了抽穗期这4个基因在水稻的根、茎、叶、剑叶和叶鞘中的相对表达水平。研究结果表明,和基因主要在根中表达,而在茎、叶、剑叶和叶鞘中的表达水平较低;基因主要在叶片中表达,而在根、茎、叶鞘中的相对表达水平较低;基因在根、茎、叶、叶鞘中均有表达,在剑叶中表达水平较高(图2)。以上结果说明在水稻不同组织器官中的表达情况不同,具有组织特异性。
2.3 RLK基因编辑载体的构建
为了揭示这4个盐胁迫响应基因的生物学功能,利用基因编辑技术对其进行定点敲除。根据CRISPR/Cas9编辑原理,参考生信预测网站CRISPR-P2.0(http://crispr.hzau.edu.cn/CRISPR2/)推荐的靶位点,在基因外显子区设计两个特异性较高的靶位点TS1(Target site 1)和TS2(Target site 2),作为sgRNA结合的靶位点(图3-A)。两靶点的正反向引物分别退火,形成的双链DNA采用边切边连的方法(Ⅰ酶切,T4 DNA连接酶连接)构建至入门载体pYLsgRNA-OsU6a/U3,形成两个重组载体OsU6a-TS1-sgRNA和OsU6a-TS2- sgRNA。经过两轮巢式PCR分别扩增两个sgRNA表达盒,并采用边切边连的方法将两个sgRNA表达盒构建到表达载体形成pYLCRISPR/Cas9PUbi- TS1-sgRNA-TS2-sgRNA,的表达受启动子的驱动,两个sgRNA受或启动子驱动(图3-B)。通过农杆菌介导的转化方法转化南粳9108的愈伤组织,分化出苗。
2.4 转基因植株的鉴定及靶位点突变检测
为了进一步对转基因阳性植株进行鉴定,提取T0代植株的基因组DNA,利用基因的特异性引物进行扩增,获得了4个的转基因阳性植株(图4)。然后对各转基因阳性植株的变异位点进行PCR扩增以分析其突变类型。本研究选取基因的功能缺失突变体以完成后续表型鉴定,突变体的Target 1位点发生双等位突变,两条染色体分别缺失1个碱基,Target 2位点为纯合突变,缺失1个碱基;突变体的Target 1位点发生双等位突变,两条染色体分别插入1个碱基,Target 2位点为野生型;突变体的Target 1位点与Target 2位点之间发生345个碱基缺失;突变体的Target 1位点为纯合突变,缺失4个碱基,Target 2位点为插入和碱基替换(图5)。上述鉴定突变体的相应基因位点均发生了移码突变,造成基因功能缺失。

A−RLK基因靶位点; B−pYLCRISPR/Cas9PUbi-RLKs-sgRNA载体。
2.5 突变体耐盐性鉴定
为了测试4个基因突变对水稻耐盐性的影响,采用水培法培养野生型和突变体幼苗,将生长10 d(2叶1心期)的幼苗用120 mmol/L NaCl处理。结果表明,NaCl处理后幼苗生长受到明显抑制,叶片萎蔫发黄,植株大部分干枯,且突变体的生长状态与野生型没有明显差异(图6-A)。存活率统计结果表明盐处理条件下突变体与野生型的存活率差异不显著(图6-B)。以上研究结果表明这4个基因缺失对水稻的耐盐性没有明显影响。
3 讨论
耐盐水稻新品种选育的主要方式是利用耐盐种质与主推品种杂交和回交,然后通过多代的耐盐表型鉴定结合分子标记辅助选择选育耐盐且农艺性状优良的水稻新品种[19]。基因编辑可以快速高效地对单基因或多基因进行编辑。近年来,通过该技术已经实现了对水稻多个农艺性状调控基因的编辑,包括产量、育性、粒型等调控基因以及抗稻瘟病、抗除草剂等抗性基因[20-21]。利用基因编辑技术对水稻的耐盐性进行快速定向改良将是创制耐盐水稻新种质的重要方式。南粳9108是江苏省农业科学院选育并在江苏省大面积推广的优良食味迟熟中粳品种,在0.3% NaCl胁迫条件下表现良好,适宜在沿海滩涂种植[19, 22]。本研究以南粳9108作为受体植株,利用CRISPR/Cas9对4个新的响应盐胁迫的基因进行编辑,揭示在调控植物耐盐性中的作用。
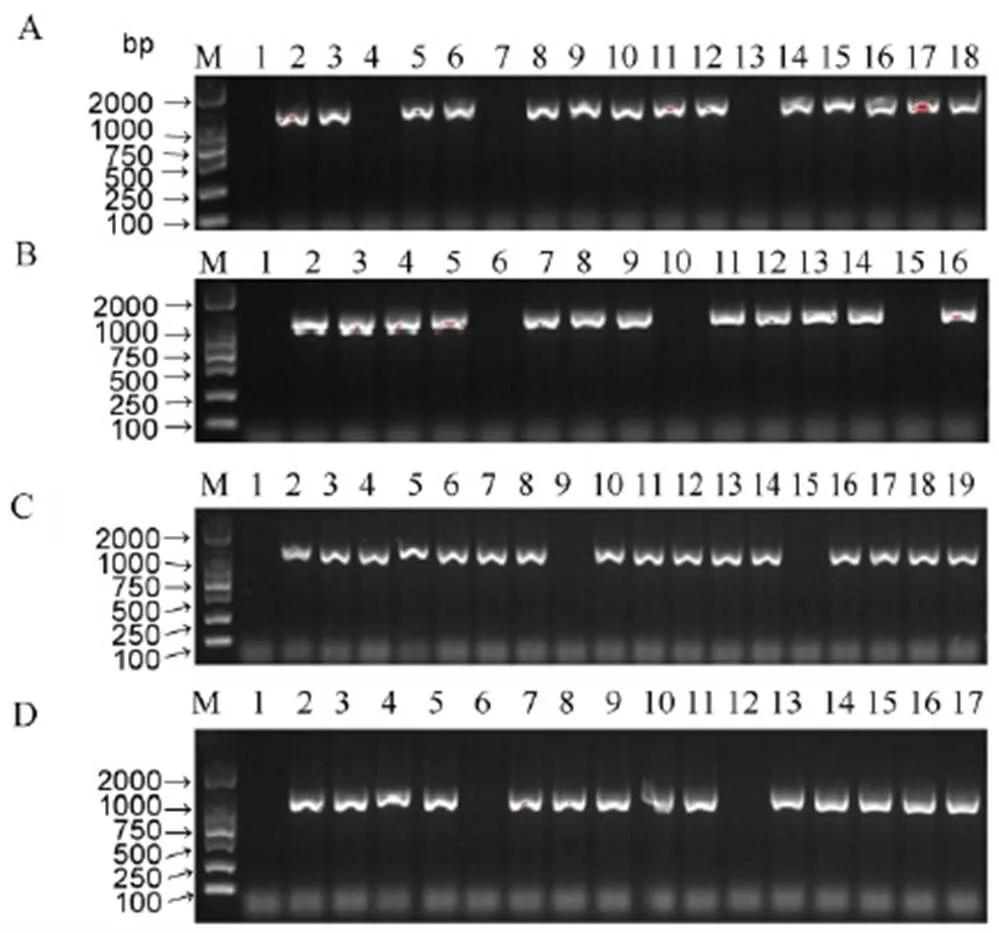
A-鉴定Os04g0275100基因编辑植株; B-鉴定Os07g0541900基因编辑植株; C-鉴定Os09g0353200基因编辑植株; D-鉴定Os01g0852100基因编辑植株。

A−Os04g0275100基因缺失突变体的突变类型;B−Os09g0353200基因缺失突变体的突变类型;C−Os07g0541900基因缺失突变体的突变类型;D−Os01g0852100基因缺失突变体的突变类型。

A−突变体和野生型的耐盐表型分析;B−突变体和野生型的存活率比较。
类受体蛋白激酶家族在调控植物的生长发育、抗逆性以及激素信号转导方面起着重要作用[23-24]。在水稻中,SIT1、SIK1、STRK1等类受体蛋白激酶已经被报道参与植物对盐胁迫的响应过程[10,11,23]。为了进一步鉴定响应盐胁迫的成员,本研究对4个转录水平受盐胁迫调控的基因进行功能分析。苗期盐胁迫处理实验结果表明,基因受到NaCl诱导,而其他3个基因受NaCl抑制,说明它们可能参与植物对盐胁迫的响应过程。此外,这4个基因的表达还受非生物胁迫相关激素ABA以及渗透胁迫物质甘露醇的影响。我们对这4个基因在不同组织器官中的表达进行检测,qPCR结果表明和基因在根中表达较多,基因主要在叶片中表达,基因在不同组织器官中有表达,说明4个基因的表达具有组织特异性。
为进一步分析这4个的功能,我们利用基因编辑技术分别对其进行敲除,得到了相应的功能缺失突变体。耐盐性表型结果表明,4个突变体在盐处理条件下的生长状态以及存活率均与野生型南粳9108没有明显差异。类受体蛋白激酶家族是植物最大的膜受体家族,成员众多,在水稻中数目超过1100个,单独缺失其中一个可能对植物表型并不产生显著影响。本研究鉴定的4个并未表现出NaCl超敏或者脱敏表型,可能是由于同源基因引起的功能冗余现象导致的。通过创制双突变或者多突变对进一步研究的功能至关重要。此外,与参考基因组相比,南粳9108中4个基因序列或者启动子区可能存在SNP或者其他变异,导致RLK功能发生变化。通过高表达以及比对耐盐程度不同的粳稻品种中这4个的基因型或表达水平可以进一步揭示其在响应植物盐胁迫中的功能,进而为耐盐粳稻育种提供候选基因资源。
[1] Gong Z, Xiong L M, Shi H Z, Yang S H, Herrera-Estrella L R, Xu G H, Chao D Y, Li J R, Wang P Y, Qin F, Li J, Ding Y L, Shi Y T, Wang Y, Yang Y Q, Guo Y, Zhu J K. Plant abiotic stress response and nutrient use efficiency.2020, 63(5): 635-674.
[2] Zhu J K. Plant salt tolerance., 2001, 6(2): 66-71.
[3] Flowers T J. Improving crop salt tolerance.,2004, 55(396): 307-319.
[4] Sahi C, Singh A, Kumar K, Blumwald E, Grover A. Salt stress response in rice: Genetics, molecular biology, and comparative genomics., 2006, 6(4): 263-284.
[5] Zelm E V, Zhang Y X, Testerink C. Salt tolerance mechanisms of plants., 2020, 71(1): 403-433.
[6] Hanin M, Ebel C, Ngom M, Laplaze L, Masmoudi K. New insights on plant salt tolerance mechanisms and their potential use for breeding., 2016, 7(11): 1787.
[7] Sivakumar P, Sharmila P, Saradhi P P. Proline suppresses rubisco activity in higher plants., 1998, 252(2): 428-432.
[8] Lemmon M A, Schlessinger J. Cell signaling by receptor tyrosine kinases., 2010, 141(7): 1117-1134.
[9] Morillo S A, Tax F E. Functional analysis of receptor-like kinases in monocots and dicots., 2006, 9(5): 460-469.
[10] Ouyang S Q, Liu Y F, Liu P, Lei G, He S J, Ma B, Zhang W K, Zhang J S, Chen S Y. Receptor-like kinase OsSIK1 improves drought and salt stress tolerance in rice () plants., 2010, 62(2): 316-329.
[11] Li C H, Wang G, Zhao J L, Zhang L Q, Ai L F, Han Y F, Sun D Y, Zhang S W, Sun Y. The receptor-like kinase SIT1 mediates salt sensitivity by activating MAPK3/6 and regulating ethylene homeostasis in rice., 2014, 26(6): 2538-2553.
[12] Shi C C, Feng C C, Yang M M, Li J L, Li X X, Zhao B C, Huang Z J, Ge R C. Overexpression of the receptor-like protein kinase genesandreduces the salt tolerance of., 2014, 217-218: 63-70.
[13] Osakabe Y, Yamaguchi-Shinozaki K, Shinozaki K, Tran L S. Sensing the environment: Key roles of membrane-localized kinases in plant perception and response to abiotic stress., 2013, 64(2): 445-458.
[14] Shiu S H, Bleecker A B. Receptor-like kinases fromform a monophyletic gene family related to animal receptor kinases., 2001, 98(19): 10763-10768.
[15] Tanaka H, Osakabe Y, Katsura S, Mizuno S, Maruyama K, Kusakabe K, Mizoi J, Shinozaki K, Yamaguchi-Shinozaki K. Abiotic stress-inducible receptor-like kinases negatively control ABA signaling in., 2012, 70(4): 599-613.
[16] Wang X F, Kota U, He K, Blackburn K, Li J, Goshe M B, Huber S C, Clouse S D. Sequential transphosphorylation of the BRI1/BAK1 receptor kinase complex impacts early events in brassinosteroid signaling., 2008, 15(2): 220-235.
[17] Shiu S H, Karlowski W M, Pan R, Tzeng Y H, Mayer K F, Li W H. Comparative analysis of the receptor-like kinase family inand rice., 2004, 16(5): 1220-1234.
[18] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants., 2015, 8(8): 1274-1284.
[19] 王才林, 张亚东, 赵凌, 路凯, 朱镇, 陈涛, 赵庆勇, 姚姝, 周丽慧, 赵春芳, 梁文化, 孙明法, 严国红. 耐盐碱水稻研究现状、问题与建议. 中国稻米, 2019, 25(1): 1-6.
Wang C L, Zhang Y D, Zhao L, Lu K, Zhu Z, Chen T, Zhao Q Y, Yao S, Zhou L H, Zhao C F, Liang W H, Shun M F, Yan G H. Research status, questions and suggestions on salt tolerance rice varieties., 2019, 25(1): 1-6. (in Chinese)
[20] 黄忠明, 周延彪, 唐晓丹, 赵新辉, 周在为, 符星学, 王凯, 史江伟, 李艳锋, 符辰建, 杨远柱. 基于CRISPR/Cas9 技术的水稻温敏不育基因突变体的构建. 作物学报, 2018, 44(6): 844-851.
Huang Z M, Zhou Y B, Tang X D, Zhao X H, Zhou Z W, Fu X X, Wang K, Shi J W, Li Y F, Fu C J, Yang Y Z. Construction ofmutants in rice based on CRISPR/Cas9 technology., 2018, 44(6): 844-851. (in Chinese with English abstract)
[21] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H, Liu Q Q.acts as a transcriptional activator to regulate rice grain shape and appearance quality., 2018, 9(1): 1240.
[22] 王才林, 张亚东, 朱镇, 姚姝, 赵庆勇, 陈涛, 周丽慧, 赵凌. 优良食味粳稻新品种南粳9108的选育与利用. 江苏农业科学, 2013, 41(9): 86-88.
Wang C L, Zhang Y D, Zhu Z, Yao S, Zhao Q Y, Chen T, Zhou L H, Zhao L. Breeding of the japonica rice varieties with good eating quality., 2013, 41(9): 86-88. (in Chinese)
[23] Zhou Y, Liu C, Tang D, Yan L, Wang D, Yang Y Z, Liu X M. The receptor-like cytoplasmic kinase STRK1 phosphorylates and activates CatC, thereby regulating H2O2homeostasis and improving salt tolerance in rice., 2018, 30(5): 1100-1118.
[24] Gao L L, Xue H W. Global analysis of expression profiles of rice receptor-like kinase genes., 2012, 5(1): 143-153.
Functional Analysis on Four Receptor-like Protein Kinases Under Salt Stress in Rice
LU Kai, CHEN Tao, YAO Shu, LIANG Wenhua, WEI Xiaodong, ZHANG Yadong*, WANG Cailin*
(Institute of Food Crops, Jiangsu Academy of Agricultural Sciences/Jiangsu High Quality Rice R&D Center/Nanjing Branch of China National Center for Rice Improvement, Nanjing 210014, China;*Corresponding author, E-mail: zhangyd@jaas.ac.cn; clwang@jaas.ac.cn)
Salinity stress is one of the main harsh environmental factor that greatly hinders rice growth and production, and it is meaningful to study salt-responsive genes for understanding the mechanisms behind plant response to salt stress and breeding salt tolerance variety. The receptor-like protein kinases are involved in regulation of plant cell signaling and responding to stress. In this study, we analyzed the expression pattern and function of fourunder salt stress.Quantitative real-time PCR was employed to detect the expression level of the fourunder NaCl treatment and its tissue specificity.The transcription ofwas induced by NaCl, which was expressed mainly in roots. The transcription ofwas inhibited by NaCl, which was expressed mainly in roots. The transcription ofwas inhibited by NaCl, which was expressed mainly in leaves. The transcription ofwas inhibited by NaCl, which was expressed in roots, shoots, leaves and sheath. Loss-of-function mutants of the fourwere obtained by sequencing, which exhibited the identical response to NaCl as compared with the wild-type.The expression of the fouridentified in this study were regulated by NaCl and were tissue-specified. Mutation of one of the fourhas no effects on rice salt tolerance. This study lays a basis for revealing the biological function and mechanisms of RLKs under salt stress.
receptor-like protein kinase; salt stress; CRISPR/Cas9;rice
10.16819/j.1001-7216.2021.0905
2020-09-05;
2020-09-18。
国家自然科学基金青年基金资助项目(31700229); 江苏省自然科学基金青年基金资助项目(BK20170594)。
