盐胁迫下蚕豆不同组织实时荧光定量PCR内参基因的筛选
2021-03-11樊有存张红岩杨旭升武学霞刘玉皎
樊有存,张红岩,韩 芊,杨旭升,武学霞,刘玉皎
(1.青海大学,青海 西宁 810016; 2.青海大学农林科学院,青海 西宁 810016;3.省部共建三江源生态与高原农牧业国家重点实验室,青海大学,青海 西宁 810016;4.青海大学生态环境工程学院,青海 西宁 810016)
实时荧光定量PCR (real-time quantitative PCR,RT-qPCR)是当前研究基因表达水平的主要技术手段[1]。为了准确检测目的基因的表达情况,一般会选用一个或多个表达相对稳定的内参基因进行校正[2-3]。目前,植物种常用的内参基因有α/β微管蛋白基因(TUA,TUB)、参与胞质核糖体小亚基及翻译因子(18SrRNA)、肌动蛋白基因(ACT)、蛋白合成中的翻译延伸因子(GAPA)、转录延伸因子基因(EF1a)及蛋白质加工代谢相关的亲环蛋白(CYP)等[4-7]。但实际上,在不同的细胞、组织、生理阶段,目的基因的表达量可能存在差异[8],亦会因所选用的内参基因的不同而影响实验结果。近年来,关于豆科植物内参基因的报道有很多。王骁等[9]筛选出CYP2和GAPDH为大豆籽粒发育过程中表达最为稳定的基因,UKN2和TUB4为胚轴中表达稳定的基因,而ACT2和CYP2为子叶中表达稳定的基因,逆境胁迫下UKN2、TUB4、EF1A较稳定。付媛媛等[10]发现,紫花苜蓿在各种胁迫(激素、盐胁迫、干旱和暗条件)下不同组织中表达量相对比较稳定的内参基因为MSC27、18SrRNA、EF-La和Actin2。
土壤盐渍化已成为影响植物生长发育和作物产量的主要因素之一,中国约20%耕地面积受到土壤盐渍化的影响[11]。蚕豆(ViciafabaL.)作为中国重要的食用豆类作物之一,具有粮、饲、菜、肥、药兼用和深加工增值等特点,且蚕豆能适应冷凉气候,并有较强的抗盐碱能力,同时还有“生物固氮之王”的誉称,是种植业结构调整中重要的养地作物和间套作物[12-14]。为了充分发挥蚕豆的多元化功能及生态价值,其种植区域不断扩大,而土壤盐渍化将成为制约蚕豆生产的主要因素之一。
筛选蚕豆在盐胁迫下的合适内参基因是研究耐盐基因表达特性的必要前提,目前,有关蚕豆盐胁迫方面的研究主要集中在盐胁迫对蚕豆生长、生理响应及提高蚕豆耐盐性的理化方法等[15-16]方面。Gutierrez 等[17]以不同的组织、基因型和接种几种常见病原体的蚕豆为试验材料,筛选出ACT1、CYP2和ELF1A可作为相对合适的内参基因,而蚕豆在盐胁迫下不同组织中适宜的内参基因筛选方面的研究尚未报道。本研究以不同氯化钠浓度胁迫下的蚕豆叶、茎和根为材料,筛选6个植物常用候选内参基因(ELF1A、ACT2、GAPA、18SrRNA、TUA和CYP2),利用Ct值分析及内参基因分析软件(GeNorm、NormFider和BestKeeper)分析评估候选内参基因的稳定性,由此筛选出蚕豆盐胁迫下不同组织中能稳定表达的内参基因,为以后研究蚕豆耐盐功能基因提供可靠的理论依据。
1 材料与方法
1.1 试验材料
(1)材料培养。本试验以筛选获得的耐盐蚕豆种质2014-04为材料(青海大学农林科学院蚕豆课题组提供)。用5%NaClO消毒处理种子,利用植物光照培养箱黑暗培养10 d后选择长势一致的幼苗移至装有等量石英砂的塑料盆中并放置培养箱 (25 ℃/18 ℃)(白天/夜晚),相对湿度(70±5)%,光照周期16 h/8 h(白天/夜晚),光照强度250 μmol/m2/s[18],待完全长出真叶后,分别以0、50、100、150 mmol/L氯化钠溶液于每天固定时间对蚕豆幼苗进行胁迫处理,培养7 d后对各浓度处理下蚕豆幼苗的根、茎和叶进行取样并置于-80 ℃冰箱保存备用。
(2)试剂及仪器。试剂:植物总RNA提取试剂盒(天根生化科技有限公司)、琼脂糖凝胶DNA回收试剂盒(天根生化科技有限公司);Reverse Transcriptase M-MLV反转录酶(TaKaRa)、TB Green Premix Ex TaqTMⅡ (Tli RNaseH Plus)酶(TaKaRa)。仪器设备:梯度PCR仪(Veriti SimpliAmp ProFlex 96,ABI)、低温高速离心机(Centrifuge 5430 R,Eppendorf )、超微量核酸蛋白测定仪(Implen NP80 Mobile,Implen)、实时荧光定量PCR仪(LightCycler®480,Roche)等。
1.2 试验方法
(1)RNA提取及cDNA合成。取50~100 mg蚕豆组织,利用天根植物总RNA提取试剂提取总RNA(RNA用DNase处理去除基因组DNA污染);利用超微量核酸蛋白测定仪测定总RNA浓度、纯度,并用1%的琼脂糖凝胶电泳检测RNA的完整性。用质量合格的总RNA反转录合成cDNA,在-20 ℃条件下保存备用。
(2)合成引物及RT-qPCR分析。试验选用的ACT2、18SrRNA、TUA、GAPA、CYP2和ELF1A六个候选内参基因[17,19]来源于已发表的蚕豆候选内参基因及其近缘植物中不同组织、不同非生物胁迫下筛选出的相对表达稳定的内参基因(表 1)。引物由天津金唯智生物科技有限公司合成。利用实时荧光定量PCR仪LightCycler®480,采用TB Green Ⅱ试剂盒进行RT-qPCR分析。程序设定为95 ℃预变性30 s,1个循环;95 ℃变性5 s,60 ℃退火30 s,40个循环;95 ℃,5 s,60 ℃,1 min,进行溶解曲线分析;50 ℃降温30 s,实验设定3个重复。

表1候选内参基因RT-qPCR引物序列Tab.1 RT-qPCR primer sequences of candidate reference genes
(3)数据处理与分析。分别利用GeNorm、NormFider和Bestkeeper软件分析候选内参基因的表达稳定性,筛选出在盐胁迫下蚕豆不同组织中适宜的内参基因。GeNorm和NormFider软件的计算原理相似,都需将原始Ct值转换为相对表达量(Q=2-ΔΔCt)后计算出稳定系数M值,并通过M值来评价内参基因的稳定性。一般默认阈值M=1.5,当M>1.5时,基因不稳定;当M<1.5时,可以考虑作为内参基因。且M值越小,基因的稳定性越高,反之越低。GeNorm软件亦可通过变异系数V值来确定适宜内参基因的数目,当Vn/Vn+1<0.15时,表明有n个内参基因即可满足实验要求。BestKeeper软件则直接对原始Ct值进行计算,通过计算的标准偏差(SD)、相关系数(r)及变异系数(CV)来选择表达稳定性较好的内参基因。其中r值越大,CV和SD就越小,内参基因的稳定性越好,反之,稳定性就越差;而当SD>1时,表示该内参基因表达不稳定[20]。
2 结果与分析
2.1蚕豆样品总RNA的提取及检测利用植物总RNA提取试剂提取在不同浓度NaCl处理过的蚕豆幼苗的叶、茎和根的总RNA。总RNA浓度检测结果显示:A260/A280为2.138~2.275 (表2),且叶片总RNA浓度在1 000 ng/μL左右,根与茎的总RNA浓度为303.12~650.84 ng/μL,表明总RNA的质量较好;1%琼脂糖凝胶电泳检测蚕豆幼苗叶、茎和根分别在0 mmoL/L、50 mmoL/L、100 mmoL/L和150 mmoL/L NaCl胁迫下的总RNA结果显示 (图1),28SrRNA、18SrRNA的条带清晰明亮, 5SrRNA条带较暗,这说明所提取的RNA未降解,完整性较好,可用于后续试验。

表2不同盐浓度下蚕豆不同组织的总RNA质量和纯度检测Tab.2 Total mass of RNA and purity testing of faba bean tissues under the different concentrations of NaCl

图1 蚕豆不同组织的总RNA电泳检测结果Fig.1 Electrophoresis results of total RNA in the different tissues of faba bean
2.2内参基因的特异性分析图2为1%琼脂糖溶液凝胶电泳检测候选内参基因普通PCR扩增产物的结果,图中1、2、3、4、5、6 分别为TUA、 18SrRNA、CYP2、ELF1A、GAPA和ACT2 六个候选内参基因PCR产物的电泳条带,均为单一条带,无引物二聚体和非特异性条带,且产物大小均为100~250 bp。在此基础上,利用RT-qPCR技术验证6对引物,结果显示候选内参基因的溶解曲线都表现为单一信号峰(图3)。以上分析结果表明,各候选内参基因的引物特异性较好,可用于后期分析。

图2 6对引物在盐胁迫下蚕豆不同组织PCR电泳检测Fig.2 PCR analysis with 6 pairs of primers in the different tissues of faba bean under the salt stress

图3 盐胁迫下蚕豆不同组织中6个内参基因 RT-qPCR 溶解曲线Fig.3 RT-qPCR dissolution curves of 6 reference genes in the different tissues of faba bean under the salt stress
2.3 候选内参基因表达稳定性分析

图4 六个候选内参基因在盐胁迫下蚕豆不同组织中的表达丰度Fig.4 Abundance of expressions of 6 candidate reference genes in the different tissues of faba bean under the salt stress
(1)候选内参基因表达丰度分析。内参基因的Ct值是衡量某一基因表达量的主要指标,Ct值越大,基因表达量越低;反之越高[20]。本试验所筛选的六个候选内参基因在蚕豆样品中表达丰度较高,Ct值在10~35(图4)。其中ELF1A基因Ct值范围为10.85~12.89,在根、茎、叶中表达量最高;而TUA基因Ct值范围为29.47~35.73,在根、茎、叶中的表达量最低。ELF1A、ACT2及18SrRNA三个基因在根茎叶中的表达量较稳定,而GAPA基因在根中表达量较低,在茎和叶片中的表达量较高。
(2)GeNorm软件分析结果。GeNorm软件分析结果显示(图5),盐胁迫下蚕豆不同组织中六个候选内参基因平均表达稳定系数M值均小于1.5。叶中候选内参基因的平均表达稳定性为ACT2>18SrRNA>GAPA>ELF1A>CYP2>TUA;茎中候选内参基因的平均表达稳定性为CYP2>ACT2>GAPA>ELF1A>18SrRNA>TUA;根中候选内参基因的平均表达稳定性为CYP2>ELF1A>18SrRNA>ACT2>TUA>GAPA。本次研究结果表明,在盐胁迫下蚕豆叶中稳定表达的内参基因为ACT2和18SrRNA,茎中相对稳定表达的内参基因是CYP2、ACT2,根中相对稳定表达的内参基因是CYP2和ELF1A。另外,从配对变异系数(V)可以看出Vn/Vn+1值均小于0.15(图6),表明在盐胁迫下的蚕豆不同组织中选取两个内参基因就可以得到准确结果。

图5 GeNorm软件分析内参基因表达稳定性Fig.5 Expression stability of reference genes analyzed via GeNorm
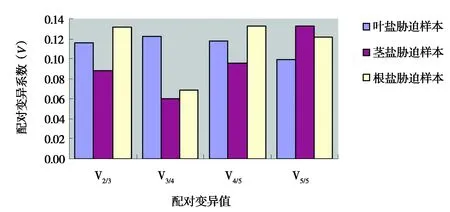
图6 GeNorm 分析候选内参基因的配对变异值Fig.6 Paired variation of reference genes analyzed via GeNorm
(3)NormFinder 软件分析。从NormFinder软件分析结果得出(表3),在盐胁迫下蚕豆叶中表达最稳定的内参基因是ACT2和18SrRNA,在茎中表达最稳定的内参基因是CYP2,在根中表达最稳定的内参基因是CYP2和ELF1A。

表3 NormFinder 软件分析结果Tab.3 Analysis results of NormFinder
(4)BestKeeper 软件分析结果。BestKeeper软件分析结果(表4)显示,除TUA以外的候选基因的标准差(SD)均小于1.5,说明所选候选内参基因的表达稳定较好。盐胁迫下蚕豆叶中ACT2基因表达最稳定,茎和根中CYP2基因表达最稳定。该结果与GeNorm和NormFinder软件分析的结果大致相同,部分差异可能由于软件间的计算原理不同所导致。

表4 BestKeeper 软件分析结果Tab.4 Analysis results of BestKeeper
3 讨论与结论
实时荧光定量PCR是研究基因表达变化的主要手段,为确保实验结果的准确性和可靠性,RT-qPCR需要选用表达较为稳定的持家基因来控制样品间的非特异性变异[21]。然而,近年来研究结果表明,并未有表达绝对稳定的内参基因,即使在同一物种或组织中稳定表达的内参基因,其稳定性会因实验条件的影响而表现出差异性[22-23]。因此,筛选出在不同物种、不同组织中适宜的内参基因就显得十分重要。Gutierrez等[17]以不同组织、基因型和接种几种重要的病原体的蚕豆为研究对象,进行内参基因的筛选,结果显示ACT1基因、CYP2基因和ELF1A基因可作为理想的内参基因。本研究以不同盐浓度胁迫下的蚕豆不同组织为材料,对六个植物常见的候选内参基因进行筛选,结果表明,在盐胁迫下的蚕豆叶片中,ACT2和18SrRNA基因表达相对稳定,在茎中CYP2基因表达相对稳定,而根中CYP2和ELF1A基因表达相对稳定。基于GeNorm、NormFinder和BestKeeper 软件分析候选内参基因的结果存在一些差异,可能由于软件间的计算原理不同所导致(GeNorm软件和NormFinder软件利用相对表达量Q来计算内参基因的表达稳定值,而BestKeeper软件直接利用原始Ct值进行计算)。由于在试验过程中,内参基因表达量也会受到样品质量的影响。因此,在试验过程中确保样本RNA、cDNA等的质量、纯度及操作细节的准确性是确保得到稳定内参基因的基础。本研究筛选的盐胁迫下蚕豆不同组织中稳定的内参基因,为后期研究蚕豆耐盐基因表达分析方面奠定了理论基础。
