新冠病毒刺突蛋白B细胞线性表位发掘
2021-02-28简春利汪佳琪张露瑶
简春利,汪佳琪,张露瑶,余 瑛,廖 飞
(重庆理工大学 药学与生物工程学院,重庆 400054)
新冠病毒(SARS CoV 2)引发的新冠肺炎疫情对人类生活和经济活动造成巨大冲击。病毒感染涉及病毒从外部入侵人体靶细胞、病毒在胞内复制、子代病毒感染更多细胞、病毒抗原诱发炎症反应等过程。新冠病毒感染患者潜伏期长且潜伏期患者仍有传染性,这迫切需要早期诊断感染的试剂及阻断病毒入侵靶细胞、抑制病毒体内复制、阻滞体内子代病毒感染等环节的药物。针对病毒抗原的高亲和力抗体,是用于新冠病毒感染预防、诊断及阻断感染的关键蛋白药物。所以,对冠状病毒入侵靶细胞所需关键蛋白进行生物信息学分析,发掘适合用于封闭入侵靶细胞必需位点以阻断感染的线性表位,及免疫检测病毒抗原所需特异表位,是研制预防、治疗及检测所需特异抗体的关键。
新冠病毒入侵靶细胞依赖其刺突蛋白(spike protein)与膜受体结合及刺突蛋白敏感位点水解[1];发掘新冠病毒刺突蛋白的重要线性表位,就成为研制所需抗体的关键。病毒蛋白的关键线性表位通常是溶液可及的连续肽段。抗体是蛋白质,识别/结合线性表位时对线性表位周围空间位阻很敏感。蛋白线性表位的空间位阻主要来自蛋白中折叠的肽链,及蛋白翻译后修饰基团。新冠病毒在人细胞内复制,其刺突蛋白在人体细胞内合成;人体细胞内合成的蛋白通常存在糖基化修饰[2]。病毒蛋白抗原的糖基化修饰遮蔽线性表位则抑制宿主体液免疫应答,使病毒逃避宿主免疫系统的识别和攻击[3]。诊断及治疗抗体所识别的刺突蛋白连续肽链线性表位如有糖基化修饰,则会因来自糖链的巨大空间位阻而使这类抗体失效[4-6]。因此,识别刺突蛋白的线性表位需排除该蛋白表面的糖基化修饰位点。
本文中预测新冠病毒刺突蛋白候选B细胞线性表位,同源建模刺突蛋白三维构象筛选暴露的候选线性表位,Emini、Karplus Schulz法定量比较候选线性表位空间可及性和构象柔韧性,搜索NCBI数据库判断候选线性表位特异性,预测刺突蛋白糖基化修饰位点并结合最新实验数据排除紧邻修饰位点的B细胞线性表位。综合比较,在新冠病毒刺突蛋白S区,发掘出以QLPP和RARS为代表的线性表位,为研制识别其刺突蛋白的抗体奠定基础。
1 材料与方法
1.1 序列来源
从美国生物信息中心NCBI数据库(https://www.ncbi.nlm.nih.gov/)中搜索并下载新冠病毒刺突蛋白氨基酸全序列(Gene ID:43740568)。
1.2 同源建模
由于本研究完成时S蛋白冷冻电镜晶体结构才报 道 (https://doi.org/10.1101/2020.02.17.951848),但同源建模的结构和晶体结构无显著差异(RMSD≈0.889?),故仍用建模构象分析候选线性表位的可及性。用Swiss Model在线同源建模;在molding任务栏下将新冠病毒刺突蛋白完整氨基酸序列以纯文本格式导入到目标序列框,点击“Start Modeling”开始自动进行S蛋白三维结构建模[7]。运行中,系统用BLASTP和HHblits自动搜索数据库中与待建模目标序列相似且有晶体结构的蛋白为模板,通过SIM程序比较模板与目标序列间相似性(最后选相似性近75%的模板建模),再对所得模型结构进行优化给出预测的目标蛋白质三维结构模型。建模结果通过DS(discov ery studio client v4.5.0.15071)软件获取拉氏图(Ramachandran Plot)[8],检查所得蛋白质结构模型中骨架及氨基酸残基分布的合理性。用PyMOL(PyMOLmolecular graphics system 2.2.0)展示蛋白质的三维模型并分析构象。
1.3 在线预测候选线性表位及判断其特异性
新冠病毒刺突蛋白氨基酸全序列,通过Kolas kar和Tongaonka方法[9],利用氨基酸残基的物理化学性质(亲水性、可及性、柔韧性)以及在已知线性表位中出现频率,计算每段候选肽段的抗原倾向值(Ap)。具体用在线服务器(http://imed.med.ucm.es/Tools/index.html),以纯文本格式导入目标蛋白的序列后提交至服务器,自动预测其肽段中候选的线性表位,再据三维空间结构定性筛选位于刺突蛋白同三聚体表面的肽段,用BLASTp搜索NCBI数据库判断候选线性表位的特异性。
1.4 可及性与刚性分析
在德泰生物(http://www.detaibio.com/tools/epitope prediction vr.html)在线服务器,输入纯文本格式新冠病毒刺突蛋白全氨基酸序列,开始自动预测线性表位的性质。用Emini方法据蛋白抗原中氨基酸残基被溶剂分子接触的可能性评价候选线性表位可及性[10];用Karplus Schulz方法,基于已知结构蛋白质Cx的温度效应值测量碳链的柔韧性,从而预测蛋白质骨架区的柔韧性(ht tps://doi.org/10.1007/BF01195768)。蛋白表面的高柔韧性连续肽段,易于与抗体经诱导契合紧密结合[11]。
1.5 糖基化修饰位点分析
在线网站预测新冠病毒刺突蛋白N型和O型糖基化位点 (http://www.cbs.dtu.dk/services/NetNGlyc),该服务器使用人工神经网络预测蛋白中糖基化位点,并自动检查Asn Xaa Ser/Thr易于糖基化的特征序列。具体是,在NetNGlyc和NetOGlyc任务栏下导入新冠病毒刺突蛋白氨基酸全序列的FASTA格式文件,提交后输出预测结果。最后,结合最新文献报道的糖基化信息排除大位阻候选表位。
2 结果
2.1 同源建模及质量评价
新冠病毒刺突蛋白与非典肺炎病毒刺突蛋白(PDB ID:6ACC)序列一致性达到76.47%,后者是前者同源建模的合适模板[12]。新冠病毒刺突蛋白三维模型中,各亚基仅获得与模板匹配的第15-1137位残基的原子座标(图1;紫色、绿色、蓝色分别代表3条肽链)。与非典病毒刺突蛋白类似,新冠病毒刺突蛋白的3个S1/S2异二聚体聚集成一个三聚体。新冠病毒刺突蛋白中,与ACE2相结合的3个C端结合域1位于伞形构象同侧且相距在3~7 nm。3个S2缠绕成束支撑伞形结构。模型拉氏图中绿色代表理想构象区域。据氨基酸分布,绝大部分残基都位于可接受区域,表明所建三维模型较合理(图2)。
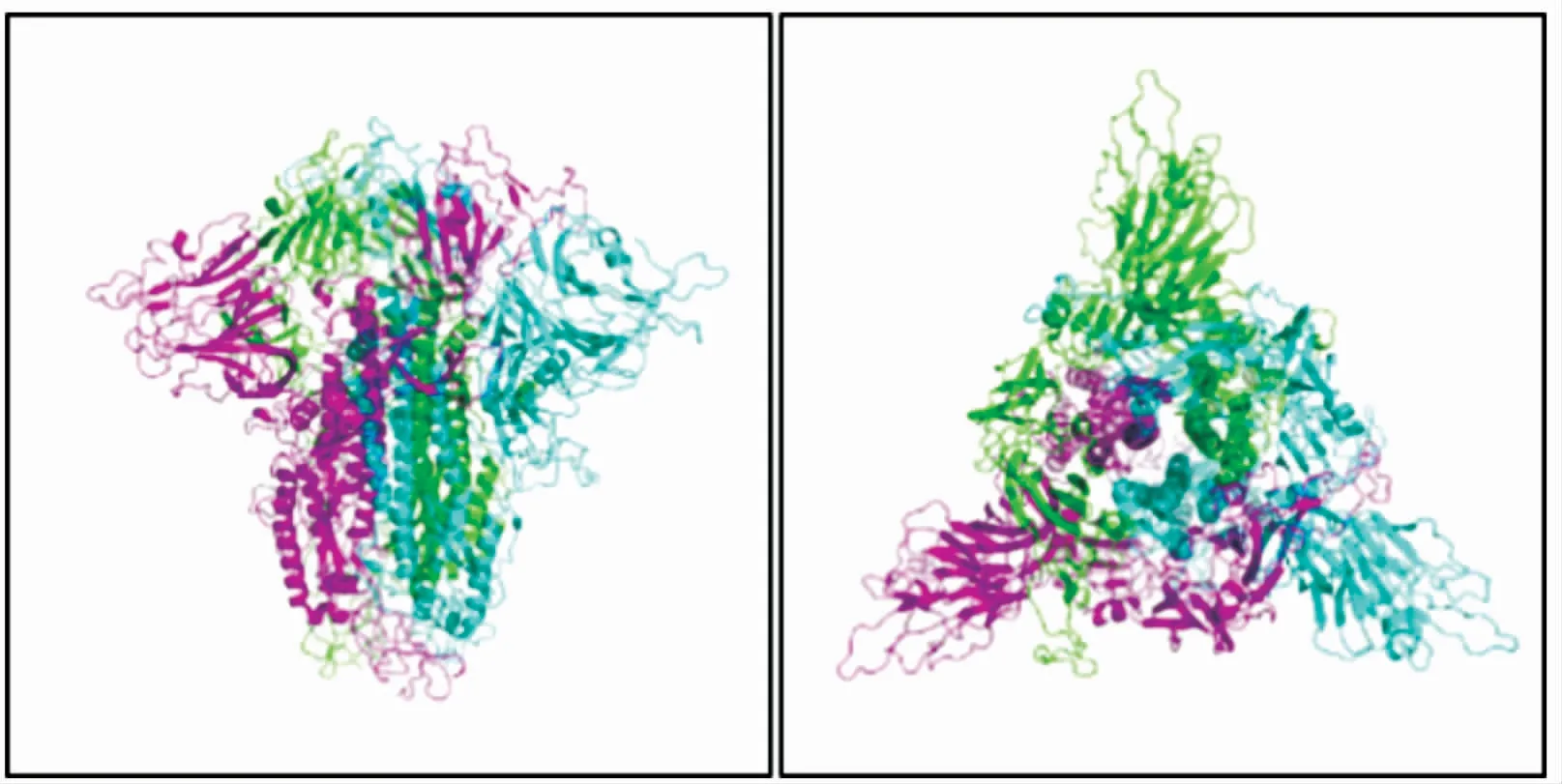
图1 新冠病毒刺突蛋白三维模型侧面视图、俯视图
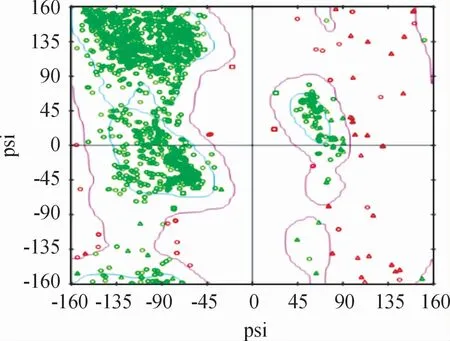
图2 新冠病毒刺突蛋白模型拉氏图
2.2 候选线性表位预测
新冠病毒刺突蛋白全长1 273个氨基酸残基,编码区位于基因组21549-25730区,较蝙蝠类SARS冠 状 病 毒、SARS CoV 和 MERS CoV 都更长[13]。
在线预测发现63个候选线性表位。检测及阻断病毒感染所用抗体识别的线性表位,理论上应位于病毒蛋白抗原三维构象的表面。据此定性要求,选出14段位于刺突蛋白三聚体构象表面的候选线性表位(表1)。其余候选表位多位于刺突蛋白三聚体的亚基接触面,显然不适合被本质为蛋白质的各种类型抗体识别。后续分析,主要考虑表1所列满足基本要求的候选表位。
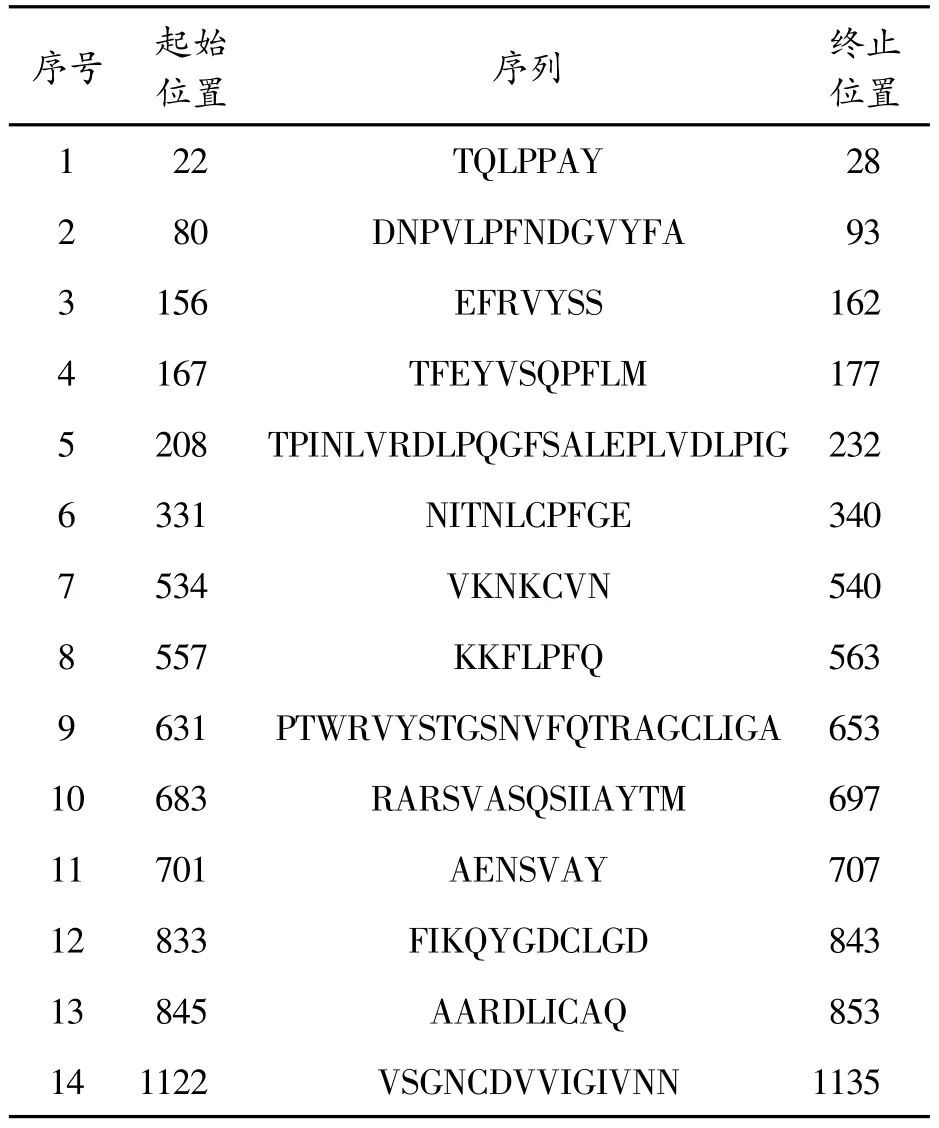
表1 新冠病毒刺突蛋白候选线性表位序列
2.3 候选线性表位空间可及性及柔韧性
位于蛋白抗原三维空间表面的连续肽段周围也有位阻。单链抗体及天然单抗识别的线性表位长度相近,为5~11个残基;纳米抗体识别的线性表位较短,为4~7个残基。据Emini法定量预测,可及性指数>1且含4个及以上残基的连续表位共20个,但位于新冠病毒刺突蛋白三聚体三维构象表面的线性表位则只有4个(表2)。
根据Karplus Schulz方法预测蛋白骨架柔韧性。含4个及以上残基且柔韧性指数>1的线性表位共有28个,而位于新冠病毒刺突蛋白三聚体表面的线性表位则只有8个(表2)。

表2 位于三维结构表面、可及性及柔韧性满足要求的连续4个以上残基新冠病毒刺突蛋白候选表位
2.4 糖基化位点预测
蛋白质常见糖基化类型含糖链连接在天冬酰胺与谷氨酰胺的酰胺残基的N 糖基化,以及连接在丝氨酸与苏氨酸的羟基残基的O 糖基化。
Asn Xaa Ser/Thr是易于发生糖基化的代表性位点(X是除pro氨酸以外残基)[14]。在线预测显示新冠病毒的刺突蛋白共有22个潜在N 糖基化修饰的候选天冬酰胺残基(见表3)。

表3 SARS CoV 2刺突蛋白糖基化位点
本研究修改过程中,新冠病毒刺突蛋白中S蛋白的N 糖基化位点已实验测定[6],在线预测N 糖基化位点与实测一致。但实验发现,随着所得刺突蛋白的表达系统(昆虫和HEK293)及表达批次不同,新冠病毒刺突蛋白糖基化水平和糖链种类有差别,且其刺突蛋白与ACE2的结合活性也存在差异[15]。可见,重组新冠病毒刺突蛋白的糖基化修饰存在多样性和异质性。
在线预测发现O 糖基化候选位点主要是S673(评分0.589),T678(评分0.631)和S6869(评分0 577),与早期文献预测结果一致[16],但实验证实O 糖基化主要发生在T323和S325[17]。可见,新冠病毒刺突蛋白糖基化修饰主要是N 糖基化。
2.5 综合多种标准的筛选结果
针对病毒蛋白线性表位的特异抗体是免疫检测病毒抗原的先决条件。针对多种病毒相同线性表位且能阻断感染的高亲和力抗体对多种病原体通用,对抗病毒治疗更有意义。将候选表位逐个通过BLASTp进行同源比对搜索相同及类似肽段,判断候选线性表位的特异性,并避开糖基化位点。
据以上要求,筛选出刺突蛋白表面QLPP和RARS为代表的候选连续线性表位;这2个肽段都属于Loop二级结构。QLPP位于刺突蛋白第23-26位氨基酸,在三维结构中位于S1区的N端结构域区;RARS位于S蛋白氨基酸序列第683-686位,在三维结构中位于S1和S2交界处(图3)。在线预测RARS的S686可能发生O 型糖基化,但实验未检测到哺乳动物细胞合成的刺突蛋白在此位点发生糖基化修饰,故RARS仍为候选连续表位。
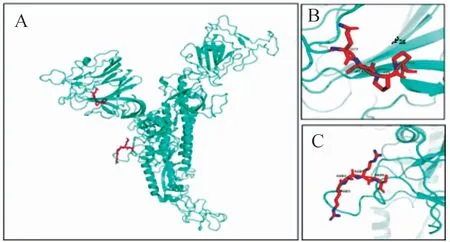
图3 QLPP和RARS在新冠病毒刺突蛋白单体中的空间位置(A)及其局部精细构象(B、C)
3 讨论
至今未报道适合阻断新冠病毒入侵靶细胞的特异抗体,也无高灵敏度检测新冠病毒蛋白抗原所需特异抗体。特异抗体仅识别蛋白抗原的特定表位[18]。实践中,蛋白的有效线性表位需同时有如下特征:
1)为表面凸出结构,以降低肽链折叠造成的结合位阻而保障抗体亲和力[19]。
2)避开三维结构内部二级结构区,以免可及性太低。蛋白表面二级结构可及性强而更适合成为识别位点[11]。
3)连续肽段长度适中,以适应抗体有限的结合空腔。
4)无糖基化等蛋白质修饰。
综合考虑上述要求,获得以QLPP和RARS代表的候选B细胞线性表位。可见,新冠病毒刺突蛋白表面可用的B细胞线性表位较少,这对其刺突蛋白的免疫检测带来挑战,也对重组刺突蛋白用作蛋白疫苗的预防效果带来压力。
获得针对所选线性表位的特异抗体,经典技术是筛选分泌单抗的杂交瘤。但是单抗生产成本高、筛选单克隆成本高且耗时、难保障单抗亲和力。筛选纳米抗体、scFv突变体库易获得高亲和力抗体,且所得抗体生产成本低。纳米抗体分子小,能耐受较大空间位阻[20-21]。抗体结合冠状病毒刺突蛋白家族中高度保守的融合肽阻断其作用是阻断感染的一种直接策略,此融合肽是广谱抗冠状病毒抗体的候选线性表位[22]。但是,此融合肽仅有小段凸出暴露且周围位阻大,天然单抗亲和力必然低。更重要的是,针对新冠病毒刺突蛋白的完整人源抗体可能面临抗体增强效应[23],会促进病毒感染多种细胞而加重病情。已经证实,高亲和力纳米抗体能有效阻断冠状病毒入侵人体及动物的靶细胞[24-25];纳米抗体有望通过雾化吸入,递送到人体肺部。纳米抗体结合抗原的构象互补区残基进行饱和突变可获得大容量突变体集中库[26]。基于磁分离和迭代竞争结合,本实验室建立指数富集高亲和力抗体展示载体的纳米抗体库高通量筛选新策略,两周内就获得抗FLAG标签纳米抗体(待发表)。目前,正用这种展示库筛选新策略,筛选所发掘新冠病毒刺突蛋白B细胞线性表位的高亲和力纳米抗体,以期用于应急阻止感染而降低重症死亡率。
