基于高通量测序分析紫贻贝冻藏过程中菌群组成变化
2021-01-25蒋慧丽水珊珊吴琼静陈晓楠苏来金
蒋慧丽 水珊珊 吴琼静 陈晓楠 张 宾 苏来金
(1.浙江海洋大学食品与药学学院,浙江 舟山 316022;2.温州市农业科学研究院温州市特色食品资源工程技术研究中心,浙江 温州 325006)
紫贻贝(Mytilusedulis)肉质肥美、营养丰富,在中国大部分产自渤海湾、黄海和东海沿岸地区,其中资源最为丰富的地区为浙江嵊泗列岛附近海域,嵊泗枸杞岛素有“贻贝之乡”的美称[1]。近年来,海鲜食品已成为人们餐桌上获得蛋白质的日常选择,其中紫贻贝深受广大消费者的喜爱。但紫贻贝由于产量大、采收期集中,加上捕后贮藏不当、微生物侵袭等因素,极易损害贝肉品质,造成营养和食用价值降低。其中,微生物侵袭及污染是影响紫贻贝及其制品品质的重要原因之一。另外生长水质、捕后贮运方式及加工环境等,也会影响紫贻贝制品中菌群组成及其丰度变化[2][3]17。据广东省动物性水产品微生物污染调查研究[4]发现,2017—2019年广东省部分地区虾、蟹、贻贝类中致病菌阳性检出水平超过半数。赵金丽等[5]研究发现,不同紫贻贝制品中的微生物组成情况不同;新鲜和自制紫贻贝干的微生物中,占比较大的是不动杆菌属和嗜冷杆菌属,而市售紫贻贝干产品中优势菌为肠球菌属和葡萄球菌属。不同状态下的紫贻贝样品的菌群组成均存在较大差异,关于紫贻贝肉冻藏过程中优势菌组成及其丰度变化情况还尚未见报道。
以往研究人员通常采用传统分离培养的方法来获取食品表面菌群信息,选取合适的样品稀释液涂布于各类平板上进行分离、纯化及鉴定,但该方法耗时长,人工成本高,且获取的菌群信息尚不准确。此外,采用传统分离方式培养得到的菌群信息较少,同时存在较大偏差。伴随科技的进步,现代基因测序技术已能够满足通量强、成本低、深度高和检测快速的需求,其中高通量测序技术作为一种分子生物学方法,可以实现一次检测大量样本,每次运行可产出几十千兆字节,甚至几百千兆字节的数据分析量,目前已被广泛应用于食品、养殖、生物医药等各个领域[6-7]。该技术将待测样品的全部基因进行提取分析,能够全面地反映菌群特征,兼具安全性高、误差小、分析全面等优势[8]。目前,已有多篇高通量测序技术应用于水产品微生物组成分析的报道,检测对象包括牡蛎[9]、南美白对虾[10-11]、蟹糊[12]、小龙虾[13]等,而利用该技术方法剖析冻藏过程中紫贻贝的细菌菌群结构变化情况的相关研究还鲜见报道。基于此,研究拟应用高通量测序技术,从基因水平层面全面揭示不同冻藏时间的紫贻贝肉中菌群的多样性和群落结构的动态变化情况,结合近期研究成果剖析其优势菌群的形成原因和发展趋势,为解决紫贻贝及其制品的贮藏品质问题提供基础理论支持。
1 材料与方法
1.1 材料
活体紫贻贝样品:壳(10.0±0.5) cm,壳宽(2.5±0.5) cm,壳高(5.1±0.5) cm,质量(50.0±3.0) g,运至实验室后,立即进行试验,浙江省舟山市东河市场。
1.2 试剂与仪器
TransStart FastPfu DNA Polymerase等测序试剂:化学纯,美国Illumina公司;
AxyPrepDNA 凝胶回收试剂盒:美国Axygen公司;
PCR仪:ABI GeneAmp©9700型,美国ABI公司;
蓝色荧光定量系统:QuantiFluorTM-ST 型,美国Promega公司。
1.3 样品处理与分组
鲜活的紫贻贝蒸汽处理2 min,开壳后解剖取出完整的紫贻贝肉样品,无菌纱布轻拭去表面水分,装入无菌袋中,参考吕丹丹[3]17-25的研究,根据冻藏期间理化指标变化节点,将紫贻贝肉分为:新鲜紫贻贝肉组(FS)、冻藏1周紫贻贝肉组(FS-1)、冻藏3周紫贻贝肉组(FS-3)和冻藏9周紫贻贝肉组(FS-9)。将以上各组样品置于-18 ℃冰箱中冻藏,分别在解剖即时和冻藏1,3,9周时取样测定。
1.4 贝肉表面微生物提取
向各组紫贻贝肉加入无菌生理盐水,配置成m紫贻贝∶V生理盐水=1∶9 (g/mL)的浊液,每组设置3个平行组,置于360 r/min下提取1 h,将表面微生物提取到生理盐水中,过滤,无菌水淋洗;然后将滤液10 000 r/min离心25 min,对获得的下层沉淀物进行微生物组成分析[14]。
1.5 微生物群落分析
根据赵金丽等[5]的方法进行高通量测序,从而进行生物学信息分析。
1.6 贝肉微生物信息分析
将测序得到的PE reads进行拼接,并对所得序列进行质控和过滤质量[15],再进行操作分类单元(Operational taxonomic units,OTU)聚类分析和物种分类学分析。最后进行多样性分析和主成分分析。基于物种分类学信息利用软件绘制出各试验组的群落柱形图和丰度聚类热图[5,16]。
1.7 数据处理
选择MiSeq测序平台进行数据分析,用FLASH对各试验组样品的reads进行拼接。基于相似性为97%的范围使用Uparse软件(v7.0.1001)进行OTU聚类分析。测序获得的基因序列与Silva数据库进行比对。
2 结果与分析
2.1 测序数据统计
基于97%相似水平下,对各样品序列信息进行聚类分析并统计得知,样品的有效序列区间为31 896~42 812条,平均长度区间为441.83~453.62 bp(表1)。统计各试验组OTU生物信息发现,OTU区间为289~681,新鲜紫贻贝肉OTU数量最高为681,其余3组紫贻贝肉的OTU数量依次为623,504,289。
2.2 Rank-Abundance曲线
Rank-Abundance曲线能够反映微生物的丰富度和均匀度。曲线越宽样品的物种丰度越高,曲线越平滑样品的均匀度越大[17]。分析结果可知,新鲜紫贻贝肉和冻藏1周紫贻贝肉的曲线较宽,而冻藏3周紫贻贝肉曲线宽度最小(图1),说明了前两组的紫贻贝肉中微生物的丰度很高。由于低温环境会抑制不耐冷微生物的生长繁殖,因此冻藏后期丰度降低。与新鲜样本和冻藏1周样本的曲线相比,冻藏3周和冻藏9周样本的曲线相对陡峭,说明冻藏3周和冻藏9周紫贻贝肉中的微生物分布不均匀,原因可能是冻藏后期贝肉表面聚集了对低温耐受性较高的微生物,致使贝肉中微生物的分布不均匀[18]。结果可知,由于长时间的低温胁迫,紫贻贝肉微生物丰度和分布均匀性降低。

表1 样品信息统计表Table 1 Sample information statistics
2.3 稀释曲线和Shannon-Wiener曲线
稀释曲线能够反映测序数据是否合理。由图2可知,当测序深度>25 000时,所有样品曲线上点的斜率均已趋于零,表示再扩大数据量也不会测出更多新微生物物种[19],说明该样本测序深度科学,样本中大多数的微生物物种能够被反映出来[20]。Shannon-Wiener曲线中各点的值愈大,群落所含的信息量愈大,能够反映测量序列的多样性。由图3可知,前两组样本菌群更多样。所有样本的曲线趋于平缓,说明测序数量充足,即能够反映样本绝大部分的菌群信息。稀释曲线和Shannon-Wiener曲线均表明试验中各贝肉样品测序数据合理,获得的测序结果真实有效。

图1 Rank-Abundance曲线图Figure 1 Rank-Abundance Graph
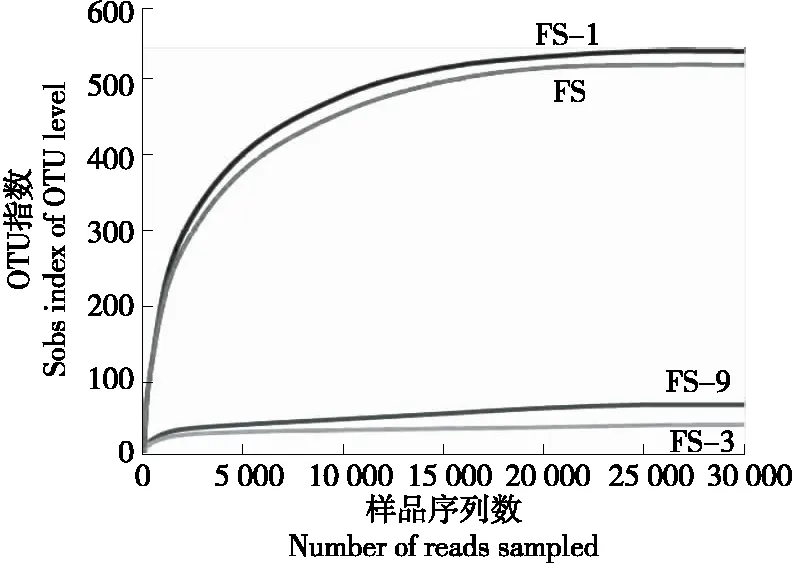
图2 稀释曲线Figure 2 Rarefaction curve

图3 Shannon-Wiener曲线Figure 3 Shannon-Wiener curve
2.4 Alpha多样性
由表2可知,各样本的Coverage值都高于0.998,说明检测序列深度足够,细菌多样性分析结果科学可信[21]。新鲜和冻藏1周样品的Ace、Chao1和Shannon指数均高于冻藏3周和9周的样品,说明新鲜和冻藏1周紫贻贝肉的群落丰富度高于冻藏3周和9周紫贻贝肉。这是由于随着冻藏时间的延长,一些对温度耐受性低的菌种(如嗜温菌)失去生理活性,致使贝肉中优势菌菌群结构发生了显著性变化,翁佩芳等[22]的研究也表明冻藏环境可抑制鲐鱼鳃体系中细菌的生长,与试验结果相似。此外,冻藏1周样品的丰富度和多样性均略高于新鲜样品,可能是因为短时间的冻藏只减缓了细菌的生长繁殖速度,并未完全抑制细菌生长活动。相较于冻藏3周,最后一组紫贻贝肉样品的Ace、Chao和Shannon指数值均增大,而且Simpson指数值减小,说明其菌群的丰度和多样性均升高。多半是因为此时贝肉已被很多腐败菌污染,同时其表面残留的部分嗜冷微生物,由亚致死状态逐渐恢复活力,从而导致紫贻贝肉表面菌群多样性升高。
2.5 微生物组成
在某一分类学水平上,群落柱形图能反映各样本中微生物的种类和序列数,即各微生物的相对丰度。在属水平上,各样本的优势菌群中均包含弧菌属(Vibrio),随着冻藏时间的延长,葡萄球菌属(Staphylococcus)的占比逐渐降低。冻藏1周时优势菌群增加了嗜冷菌属(Psychrobacter)。冻藏3周新增芽胞杆菌属(Bacillus)和肠杆菌属(Enterobacter),冻藏9周时新增希瓦菌属(Shewanella)且相对丰度很大,另外仅新鲜紫贻贝和冻藏9周样本的优势菌群中含有不动杆菌属(Acinetobacter)。
新鲜紫贻贝肉中不动杆菌为优势菌群,伴随长时间的低温胁迫,不动杆菌属的相对丰度逐渐降低,是由于其耐低温能力较弱导致的。由于各种腐败菌的生存能力是不同的,冻藏9周紫贻贝肉中不动杆菌亦为优势菌群,叶萍萍[23]在微冻鳙鱼保鲜研究中发现,微冻后期鳙鱼的优势菌中也含有不动杆菌属,与试验中冻藏9周的结果相似。与前两组相比,后两组的微生物组成存在显著的不同,菌群多样性减小,丰度升高。由于长时间的冻藏,葡萄球菌属的占比逐渐减小,尤其在第9周时葡萄球菌的占比减小至零。长时间的低温胁迫使葡萄球菌从鲜活到亚致死状态直至死亡。希瓦氏菌属也是水产品的腐败菌之一,耐低温[24],冻藏9周时其丰度占比非常高,表示此时紫贻贝肉已被很多腐败菌污染,会对其品质产生很大影响。虽然冻藏环境可以降低贝肉中菌群的多样性,但是依然会存在以上几种常见的嗜冷菌,所以对冻藏贝类产品,需要对这些腐败菌进行重点调控。另外,Illumina MiSeq测序目前无法鉴定到种,所以无法确定样品中是否还存在其他致病性菌株,但也应引起重视。
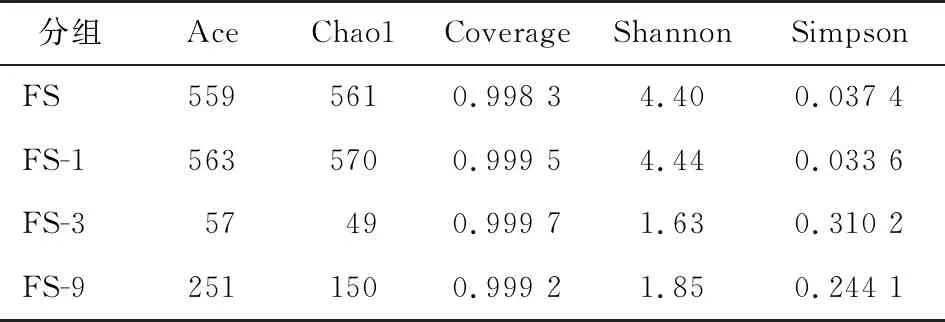
表2 多样性指数表Table 2 Diversity index
2.6 菌群组成(门水平)
Heatmap图是用定义的色彩深浅及相似水平来反映各样品在某一分类水平上微生物组成的相似程度和差异程度[25]。对各样本进行OTU聚类,绘制出各试验组的微生物丰度聚类热图,如图5所示。总的来说,由于长时间的低温胁迫,在冻藏后期紫贻贝肉样品中的微生物组成动态变化剧烈,菌群种类不断减少。由于冻藏时间较短,新鲜的和冻藏1周的紫贻贝肉菌群结构相似。分析丰度聚类热图可知,在门水平上,各试验组中变形菌门(Proteobacteria)均是占比最大的微生物。伴随长时间的低温胁迫,拟杆菌门(Bacteroidetes)、硬壁菌门(Firmicutes)、热脱硫杆菌门(Thermodesulfobacteria)和硝化菌(Nitrospinae)等微生物的占比均逐渐减小。可能是这些菌种生长繁殖需要较高水分活度,而冻藏后贝肉水分活度降低导致其占比降低。这与高乾坤等[26]关于冷藏带鱼的研究中菌群变化情况相同。对比各试验组,腐败菌、致病菌的相对丰度、菌群结构和组成菌发生了剧烈变化。
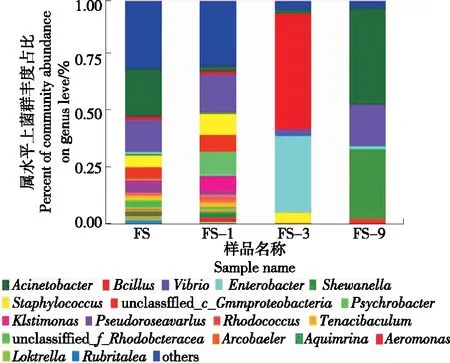
图4 不同冻藏时间贝肉微生物物种组成(属水平)Figure 4 Bacterial community composition at genus level in different frozen times mussel meat
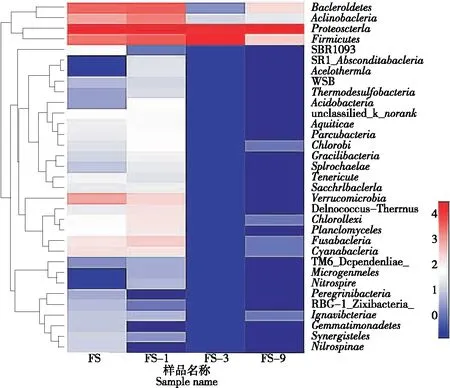
图5 不同冻藏时间样品微生物丰度聚类热图(门水平)Figure 5 Microbial community heatmap analysis at door level in different frozen times samples
所以,控制好紫贻贝肉的冻藏时间,对避免紫贻贝肉腐败至关重要。
2.7 β多样性
在主成分分析图中不同样本之间愈近,表示其菌群组成和结构愈类似。如图6所示,新鲜紫贻贝肉和冻藏1周的紫贻贝肉样本距离较近,而冻藏3周和冻藏9周的紫贻贝肉样本均相距较远,说明新鲜紫贻贝肉和冻藏1周的紫贻贝肉的菌群组成和结构相似,而在冻藏过程中各样本的微生物结构动态变化剧烈。这与丰度聚类热图的分析结果相同。
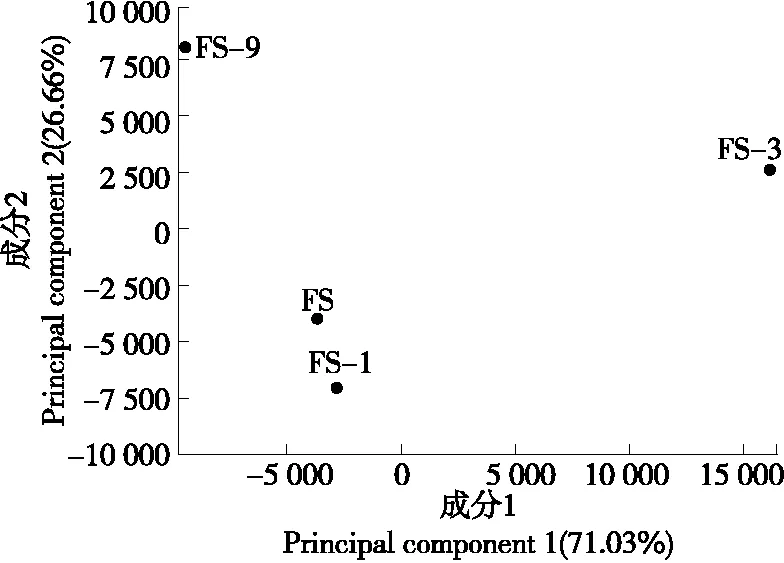
图6 不同冻藏时间样品主成分分析Figure 6 Principal component analysis of different frozen times samples
3 结论
采用高通量测序技术得到不同冻藏时间的紫贻贝肉中微生物基因序列,聚类分析了其微生物组成及结构变化情况。结果表明,各样品中OTU最少的是冻藏9周样本,最多的是新鲜紫贻贝肉。多样性指数分析得知,菌群丰富度和多样性最低的是冻藏3周紫贻贝肉。这可能是由于冻藏时间的延长,多数微生物在长期低温胁迫环境下,生命活动减弱直至死亡,仅有部分嗜冷菌存活。冻藏后期多样性、丰富度降低后又升高,说明冻藏9周时已污染较多腐败微生物。主成分分析得知,前2组的紫贻贝肉中的菌群组成和结构相似。聚类分析发现,在门水平上,各试验组中变形菌门均是占比最大的微生物,由于长时间的低温胁迫,拟杆菌门、硬壁菌门等占比均减少,多半是这些菌种生长繁殖需要高水分活度。在属水平上,各样本的优势菌群中均包含弧菌属,其可能是导致贝肉腐败的微生物之一。随着冻藏时间的延长,葡萄球菌属的占比逐渐降低。冻藏9周时希瓦菌属的相对丰度很大。低温贮藏环境虽可减少贝肉中微生物种类,但仍存在以上几种典型的嗜冷菌,而且希瓦氏菌属、不动杆菌属和弧菌属均是腐败菌和致病菌。在冻藏紫贻贝肉时,保证其品质和质量安全需要针对以上几种腐败菌重点调控。
