UBQLN1和GOLM1基因单核苷酸多态性与女性盆腔器官脱垂的相关性研究*
2021-01-21陶成斌毛宝宏刘希云
陶成斌 ,王 剑 ,毛宝宏 ,刘希云 ,魏 颖 ,刘 青 △
(1甘肃中医药大学临床医学院,甘肃 兰州 730030,2,*甘肃省妇幼保健院,甘肃 兰州 730050)
盆腔器官脱垂 (Pelvic Organ Prolapse,POP)是盆底支持结构缺陷、损伤及功能障碍造成的疾患,表现为盆腔器官位置下降及由此而引起的相应器官的功能改变,多因盆底支持组织薄弱引起[1]。既往研究证实阴道分娩、产次、高体质指数、高龄及慢性腹压增高等与POP发病相关[2-3],但临床中可见无慢性腹压增加的、年轻、未生育者患POP,而部分多产妇并未发病,提示POP发生可能与遗传因素相关[4]。本实验选取 UBQLN1 (rs3814507、rs7866234)、GOLM1(rs2225237、rs3750390、rs3750389、rs2297002)中SNP位点为待测位点,将241名POP患者及268名健康人群外周血DNA中待测位点基因多态性进行研究,旨在探讨单核苷酸基因多态性与盆腔脏器脱垂的相关性。
1 资料与方法
1.1 一般资料
本研究以241名女性POP患者为实验组,纳入标准:(1)甘肃省辖区内年龄≥20岁且在本地居住≥5年的女性;(2)符合国际尿控协会相关诊断标准及POP-Q分期标准[5]。排除标准:(1)合并妇科恶性肿瘤者;(2)中风致压力性尿失禁者;(3)既往有骨盆骨折史或相关损伤者。选取同期性别、年龄、产次、绝经状态相匹配的健康体检人群268名为对照。两组体型、分娩史、分娩方式、胎儿最大体重等资料差异无统计学意义(P>0.05)。见表1。留取外周血标本,对全血DNA进行提取,应用Sequenom技术对盆底功能障碍性疾病相关易感基因:UBQLN1、GOLM1进行多态性检测,分析与POP发生发展相关联的等位基因、基因型分布频率。本研究共纳入509份样本进行SNP位点检测。
1.2 检测方法
抽取两组静脉血5mL置EDTA抗凝管中,充分混匀以防凝血,并与流行病学资料使用统一编号,-80℃超低温冰箱保存备用。提取基因组DNA,采用Sequenom技术进行位点检测,通过引物延伸或切割反应与灵敏可靠的MALDITOF质谱技术相结合,实现基因位点检测。共选择2个基因,6个SNP位点。rs3814507反应失败及数据不准确未纳入数据分析,因此最终纳入5个SNP位点行数据分析,见表2。
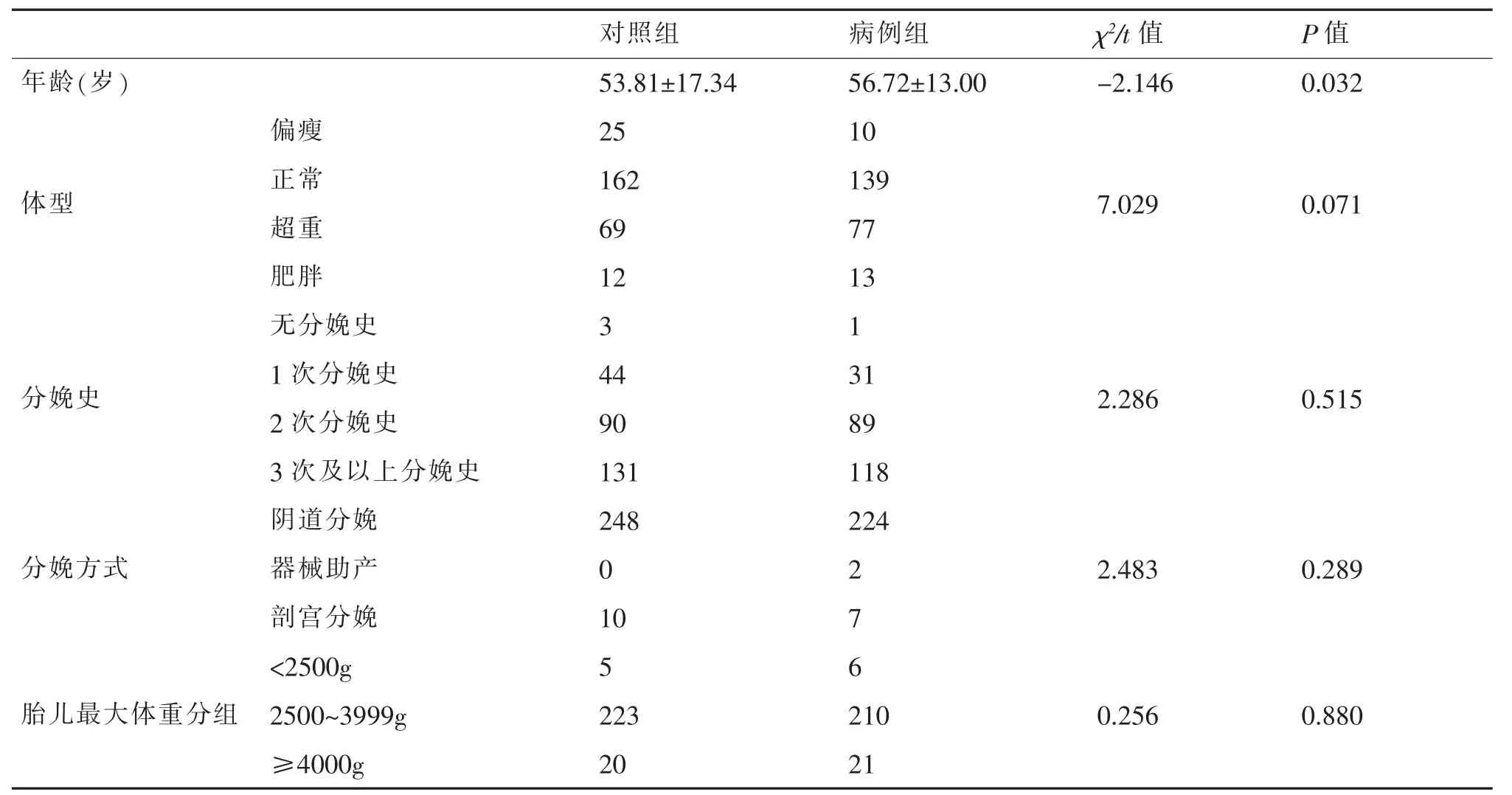
表1 两组人口学特征比较
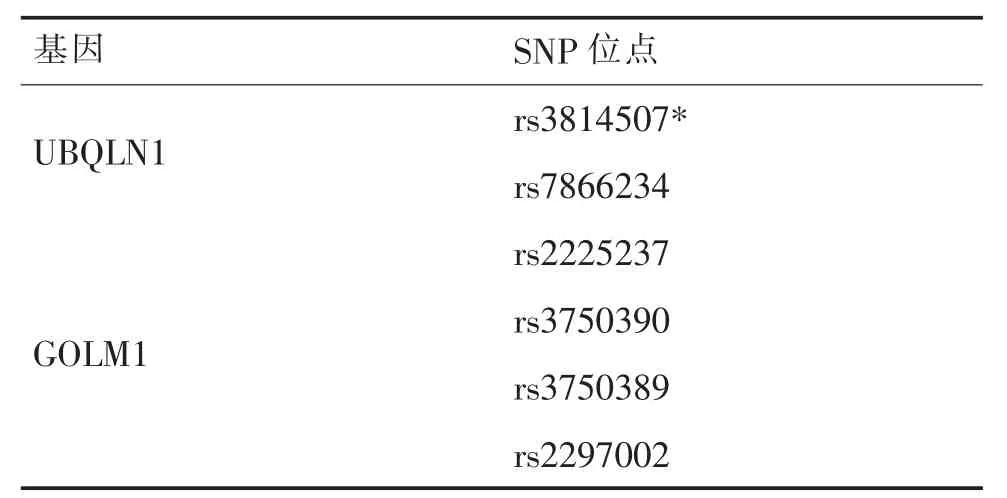
表2 SNP筛选结果
1.3 最小等位基因频率
最小等位基因频率 (Minor Allele Frequency,MAF)是指两等位基因中,频率较低的等位基因频率。若MAF<0.05,容易因为遗传漂变等原因造成选取实验群体与真实的大范围群体相比出现偏移,此外突变频率较低的位点在病例-对照组间往往差异较小,难以得出有效结论。本研究中选取SNP位点均大于0.05,即本研究选取的SNP位点有意义。详见表3。
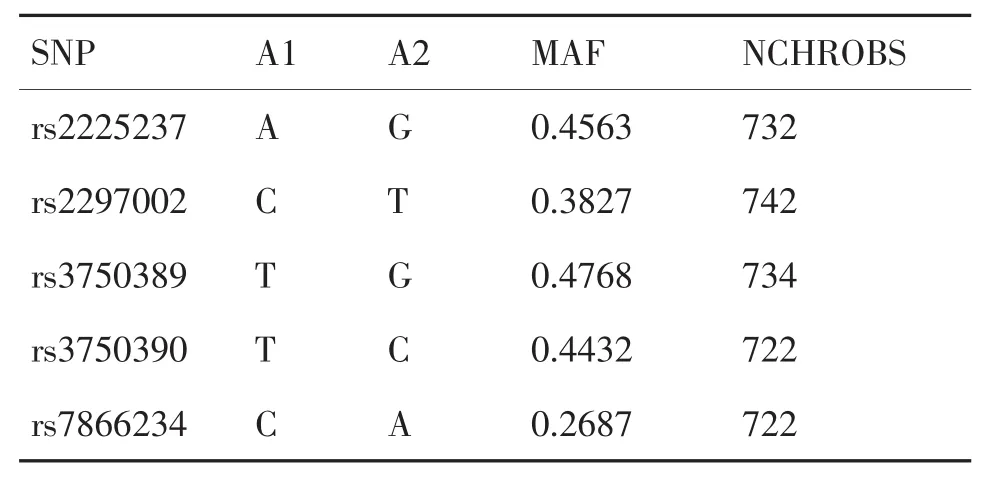
表3 SNP位点的MAF值
1.4 哈温平衡检测
分别统计所有样本,病例组,对照组是否符合哈温平衡定律,如果不符合哈温平衡 (结果中P值<0.05),说明选取的样本不是随机群体,需要进一步核实是否是家系来源样本,还是选取的样本来源有问题(即不能代表真实的大范围群体)。本研究包括的SNP位点仅rs2297002在病例组人群中分布不符合哈温平衡,其余所有位点在总体人群、病例组及对照组中分布均符合哈温平衡,即可以代表真实的大范围群体。详见表格4。
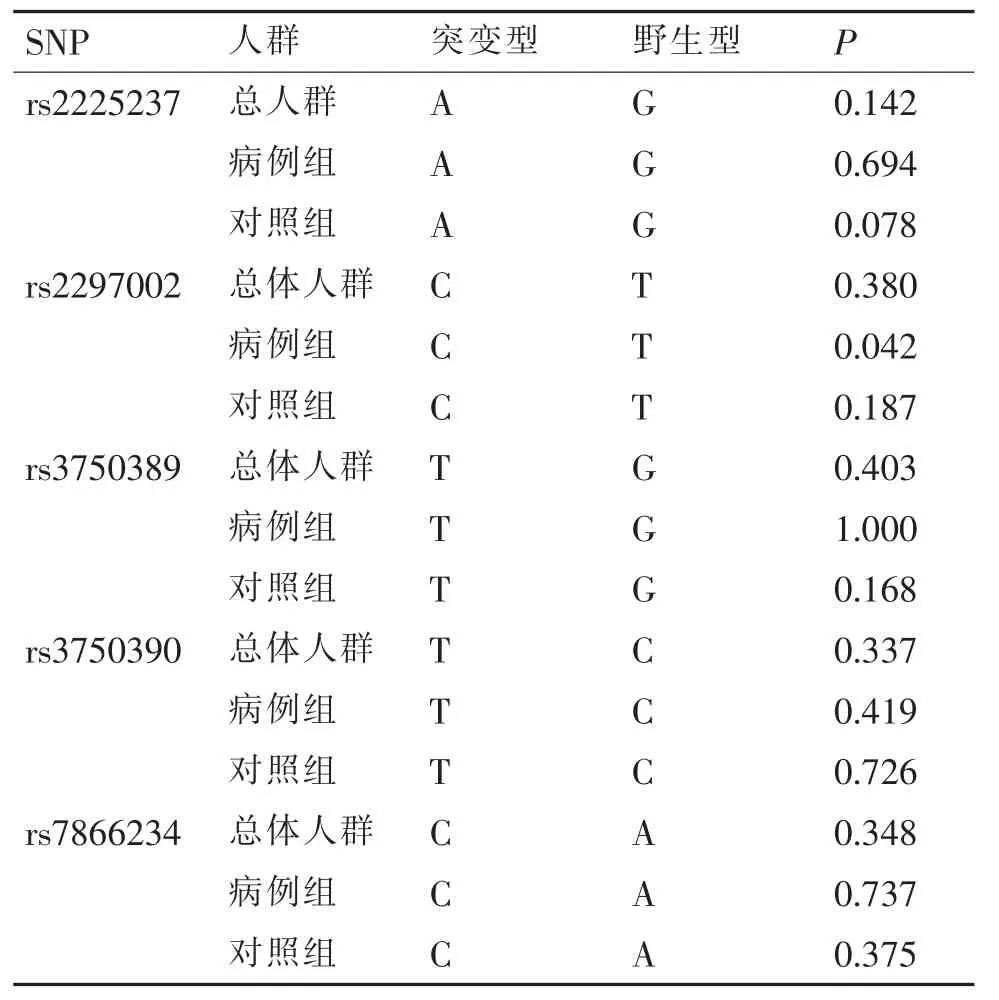
表4 SNP位点的哈温平衡检测
1.5 统计学方法
采用SPSS24.0软件进行数据分析,计量资料符合正态分布时以(±s)表示,予以 t检验,组间基因型、等位基因频率等计数资料予以χ2检验,将不同基因型在病例组及对照组间分布差异有统计学意义者纳入单因素Logistic回归分析,P<0.05为差异有统计学意义。
2 结果
2.1 等位基因频率
本研究共纳入5个SNP位点,发现rs2297002、rs3750389、rs7866234位点的等位基因频率在病例组及对照组之间差异有统计学意义 (P<0.05),余SNP位点等位基因频率组间无统计学差异 (P>0.05)。 见表 5。
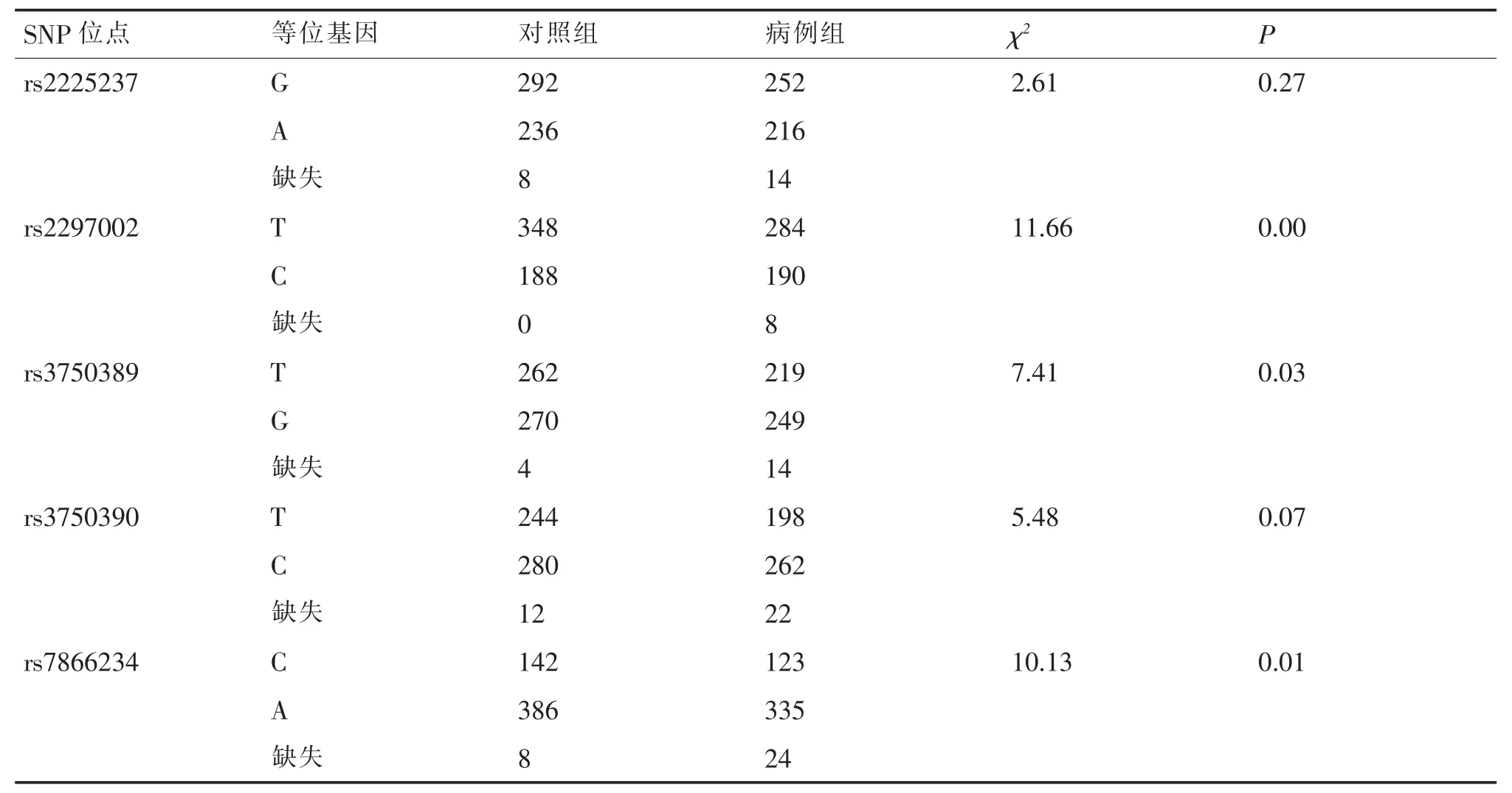
表5 等位基因频率及比较
2.2 基因型统计
本研究共纳入5个SNP位点,rs2297002 SNP位点基因型分布频率存在差异(P<0.05),详见表6。
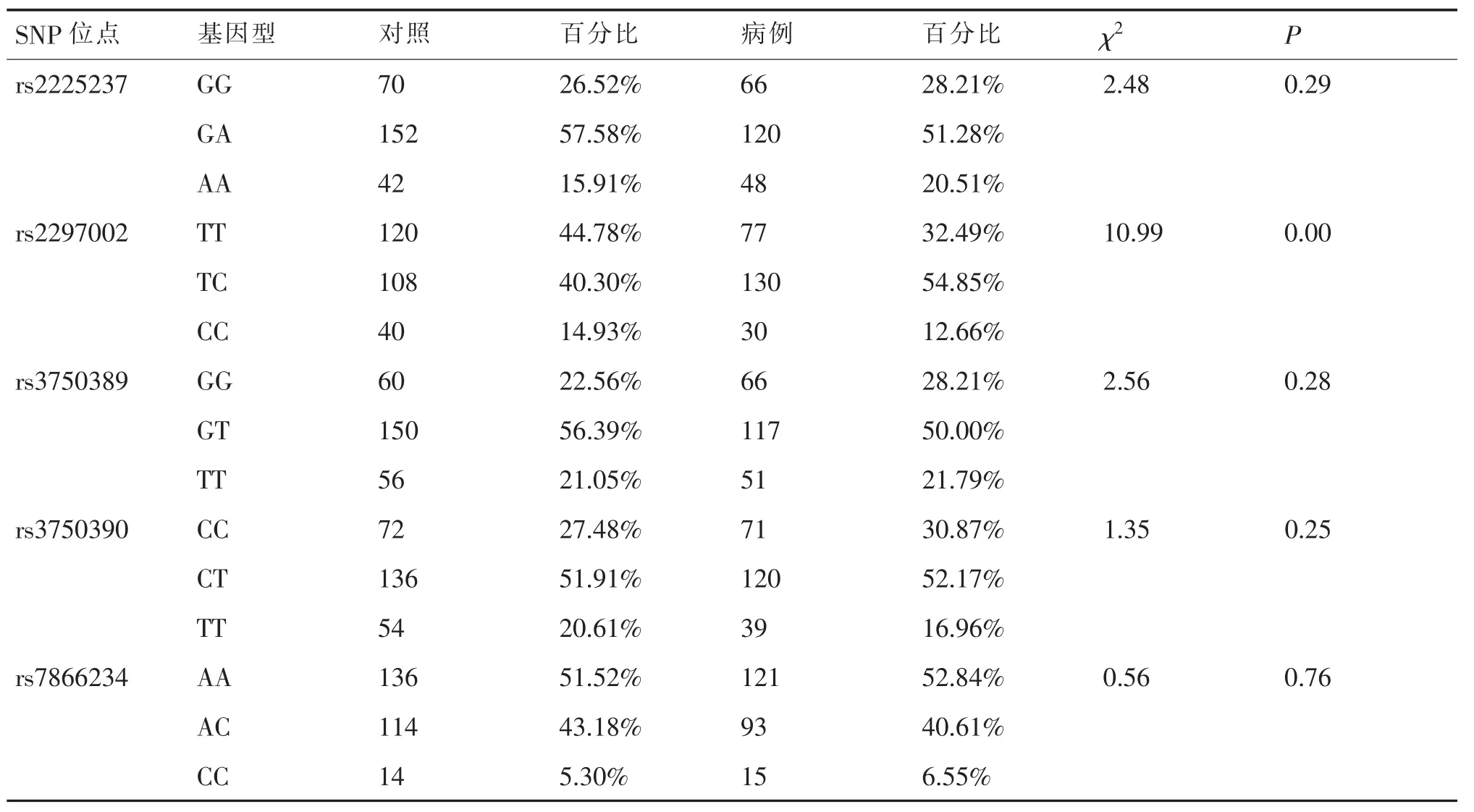
表6 基因型频率及比较
2.3 单因素logistic回归分析
rs2297002不同基因型在病例组及对照组间分布差异有统计学意义(P<0.05)。将上述基因型带入Logistic单因素回归模型分析SNP位点与POP的发病风险相关性,以野生型为对照,发现rs2297002位点TC型个体与TT型相比,可使POP发病风险增加至1.876倍。详见表7、表8。

表7 显性模型分析
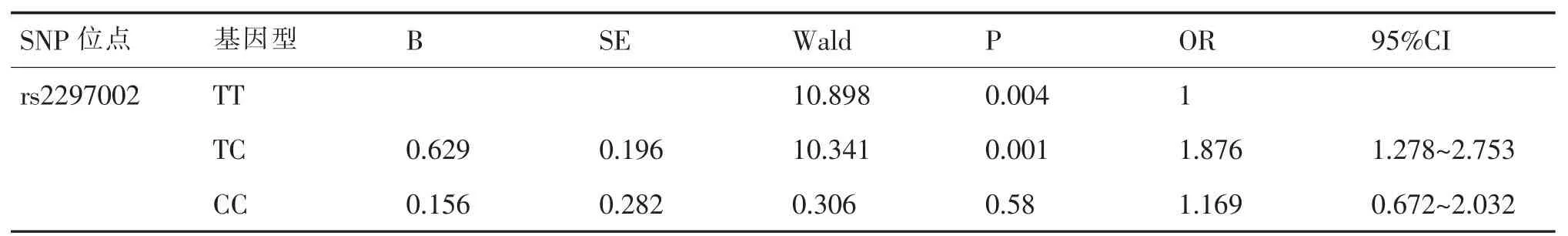
表8 rs2297002基因型单因素Logistic回归分析
3 讨论
目前POP的明确病因尚不明确,其发生可能因胶原蛋白、弹性蛋白、细胞外基质等原本保持的动态平衡被破坏所致[6]。目前国内外的多项研究都提示盆腔器官脱垂的发生、发展与遗传因素也有一定的关系。Altman,D.等[7]提出约40%的POP或SUI的发生与遗传因素相关。Allen-Brady,K.[8]等首次提出POP的发生可能与9号染色体长臂21区有关,既往研究表明多个位于该区的基因与肌肉蛋白功能相关或在肌肉组织中高表达,包括TLE4、TLE1、UBQLN1、MAK10、GOLM1 及 ADRB3 基因。
单核苷酸多态性(single nucleotide polymorphisms,SNPs)目前被广泛用于疾病相关基因定位及诊断、群体遗传学、药物开发及应用等相关研究中[9],是指基因组水平上因单个核苷酸变异致DNA序列多态性,各SNP位点通常有两个等位基因,在一定程度上表现出了每个个体之间存在着遗传差异,检测时无需分析片段长度,可通过简单的+/-分析进行基因型分型[10]。SNP位点在目前已经知道的多态性中占有相当大的比重,所占比例在90%以上。每500~1000个碱基对中就可存在1个SNPs位点。SNPs中所包含的各种等位基因,其等位基因频率均需大于1%。大部分SNPs对基因功能不存在影响,但当其位于编码区则可能影响基因功能。
本次研究以人群为基础,针对POP人群开展基因位点多态性与POP的关联分析,对照亦来自于同期人群,也是UBQLN1、GOLM1基因被较早的在POP领域研究。本实验检测了rs297002、rs3750389及rs7866234位点等5个位点SNPs与盆腔脏器脱垂的关系。研究实验结果显示,rs2225237、rs3750389、rs3750390、rs7866234 位点单核苷酸多态性与盆腔脏器脱垂无明显相关性;rs297002位点,等位基因C是盆腔脏器脱垂的危险等位基因,CC基因型是盆腔脏器脱垂的危险基因型;提示GOLM1基因的rs297002位点的单核苷酸多态性与中国汉族人群的盆腔脏器脱垂相关。rs297002位点位于GOLM1基因,但其生物学意义尚不完全明确。关于GOLM1基因SNP变异能否显著影响POP的发生尚无定论,由于本次实验样本量较小,故实验结果尚需大样本的重复性实验进一步证实,而GOLM1、基因单核苷酸多态性与盆腔脏器脱垂发病相关的具体作用机制,仍需在今后的研究中做进一步探究。
综上所述,GOLM1-rs297002C/T基因多态性与POP遗传易感性有相关性,TC型个体与TT型相比,可使POP发病风险增加至1.876倍。
