Shewanella marisflavi EP1高盐度下呼吸2,4-二硝基甲苯的全基因组分析
2021-01-20黄杰勋王汉林黄卫东刘先利
黄杰勋,王汉林,黄卫东,刘先利*
(湖北理工学院 a.环境科学与工程学院,b.矿区环境污染控制与修复湖北省重点实验室,c.化学与化工学院,湖北 黄石 435003)
2,4-二硝基甲苯(2,4-DNT)作为硝基芳香化合物的典型代表物之一,被广泛应用于制造推进剂、聚氨酯、炸药、杀虫剂、染料等化学品[1]。由于其具有较强的致癌性,在生产和使用过程中的不当处置会产生严重的土壤或水体污染问题[2]。
硝基具有较强的吸电子能力,这导致2,4-DNT难以通过好氧降解[3]。研究发现,二硝基甲苯的硝基可以先在厌氧环境下被微生物还原转化为氨基,以消去硝基的吸电子能力,从而使得还原产物二氨基甲苯在好氧条件下较容易被降解[4-5]。此外,海洋沉积物被认为是人类活动产生的大量污染物的汇,包括2,4-DNT污染[6]。研究表明,海洋沉积物中的希瓦氏菌属(Shewanellaspp.)在2,4-DNT污染的原位修复中起关键作用[7]。根据其16S rRNA基因序列,希瓦氏菌种可分为两大类。第1类希瓦氏菌是从海洋环境中分离得到的,为专性海洋菌,与分离自淡水环境中的第2类希瓦氏菌相比,其更耐盐分[8]。因此,研究专性海洋希瓦氏菌在较高盐度下还原转化2,4-DNT,有助于高盐度污水中的硝基芳香化合物的修复。
S.marisflaviEP1菌株为专性海洋菌,分离自厦门近海沉积物[7],具有厌氧还原转化2,4-DNT的能力[8]。本文将在高盐度条件下采用EP1对2,4-DNT的呼吸还原能力进行测定,并进一步对EP1的基因组进行测序和分析,以获得其呼吸还原2,4-DNT和耐盐性的相关遗传基础。
1 材料与方法
1.1 化学试剂
实验用化学试剂有2,4-二硝基甲苯(2,4-DNT,纯度≥ 99%,购自Sigma-Aldrich公司)、2,4-二氨基甲苯(2,4-DAT,纯度≥ 98%,购自Aladdin公司)。用于高效液相色谱分析的试剂均为色谱纯,购自美国Tedia公司;其他试剂均为分析纯或更高纯度,购自生工生物工程(上海)股份有限公司。
1.2 培养基和生长条件
EP1菌株分离自海洋沉积物,存于中国典型菌物保藏中心(编号:CCTCC M 209016)。采用文献[8]中的基础培养基(BM)进行厌氧培养,用1 mol/L HCl或NaOH调整至pH=7.0,用高纯氮气(99.999%)吹扫10 min,以除去微量的溶解氧,封装于50 mL血清瓶中,于121 ℃高温灭菌20 min。实验过程中乳酸钠始终作为电子供体。在含有20 mmol/L乳酸钠和40 mmol/L富马酸钠的厌氧培养基中,接种体预培养EP1 12 h后,取菌液8 000 ×g离心10 min,收集细胞,重悬于无菌BM中作为反应接种体。
1.3 高盐度下呼吸还原2,4-DNT
实验过程中使用严格的厌氧操作[9-10]。将EP1的预培养细胞接种到含有50 mL无氧BM的血清瓶中,分别加入10 mmol/L乳酸钠和100 μmol/L 2,4-DNT作为电子供体和电子受体。细胞的初始浓度约为2.0×105CFU/mL。通过添加浓度为0~10%的NaCl来控制培养基的盐度。所有反应体系均置于黑暗中30 ℃下静态培养。为测定反应,使用无菌注射器从血清瓶中取出1 mL试样,离心(10 000×g,2 min)后,取上清液分析。
1.4 全基因组测序
采用MO BIO公司的UltraCleanTM土壤DNA提取试剂盒提取并纯化EP1基因组DNA。基因组测序工作由上海美吉生物医药公司完成,测序文库采用SMRTbellTM模板制备试剂盒(Pacific Biosciences of California, USA)构建。使用PacBio RS II单分子实时(SMRT)测序平台联合Illumina测序平台对EP1基因组进行测序,且Illumina数据用于评估基因组的复杂性。EP1基因组序列已上传至NCBI的GenBank数据库,登录号为CP022272。
1.5 生物信息学分析
高通量测序读长采用PacBio SMRT portal版本2.3.0中的Hierarchical Genome Assembly Process 3(HGAP3)进行组装。用Quiver算法对装配进行校正,以获得高精度的基因组装配[11]。
采用Circos (v0.64)处理已图形化的环状基因组图谱[11]。一般基因组注释使用NCBI Prokaryotic Genome Annotation Pipeline(PGAP)2.10版本[12]。快速功能注释采用Rapid Annotation using Subsystem Technology(RAST)3.0版本以及Clusters of Orthologus Groups (COGs)[13]。
1.6 分析方法
使用配备C18色谱柱(Sun Fire,5 μm,φ4.6 mm×250 mm)的高效液相色谱(Waters e2695)测定2,4-DNT,流动相由水-甲醇组成。其程序如下:0 min时50%甲醇(V/V),11 min时70%甲醇(V/V),11~13 min时50%甲醇(V/V)。测定2,4-DAT,使用乙腈-磷酸盐缓冲液(20 mmol/L,pH=7.0,30/70,V/V)为洗脱液,流速1.0 mL/min。所有分析均在室温下进行,进样量为10 μL。使用菌落形成单位(CFU)测定细胞密度,将培养物在磷酸盐缓冲液(pH=7)中稀释,然后1式3份将100 μL等份试样涂布在Luria-Bertani(LB)琼脂平板上,于30 ℃培养24 h后,选取菌落个数为30~300的平板计数。
2 结果与讨论
2.1 高盐度下2,4-DNT的厌氧呼吸
不同盐度下菌株EP1对2,4-DNT的还原转化如图1所示。图1中,10 mmol/L乳酸钠和100 μmol/L 2,4-DNT分别作为电子供体和电子受体,百分数表示NaCl浓度,误差棒为3次平行的标准偏差。

图1 不同盐度下菌株EP1对2,4-DNT的还原转化
由图1可知,在非盐条件下观察不到2,4-DNT的转化。这与EP1属于专性海洋希瓦氏菌的事实相符,该类菌需要Na+才能生长[6]。转化还原速率最大值出现在2%~4% NaCl浓度下,2,4-DNT在12 h内几乎完全还原。在8%的盐度下,菌株EP1在60 h内还原转化约70%的2,4-DNT;在10%的盐度下,菌株EP1在60 h内还原转化约20%的2,4-DNT。
采用2,4-DNT上的硝基与细胞生长的相关性来考察EP1在8% NaCl高盐度下呼吸还原2,4-DNT的情况。8%盐度下硝基呼吸还原和细胞生长耦合情况如图2所示。图2中,硝基浓度由1 mol 2,4-DNT含2 mol 硝基计算。由图2可知,伴随硝基的还原,细胞密度增加了几个数量级,最大达到4.1×107CFU/mL。在无2,4-DNT作为电子受体的空白对照中没有观察到细胞生长。此外,细胞生长与硝基还原同时发生,并随着硝基耗尽而停止。
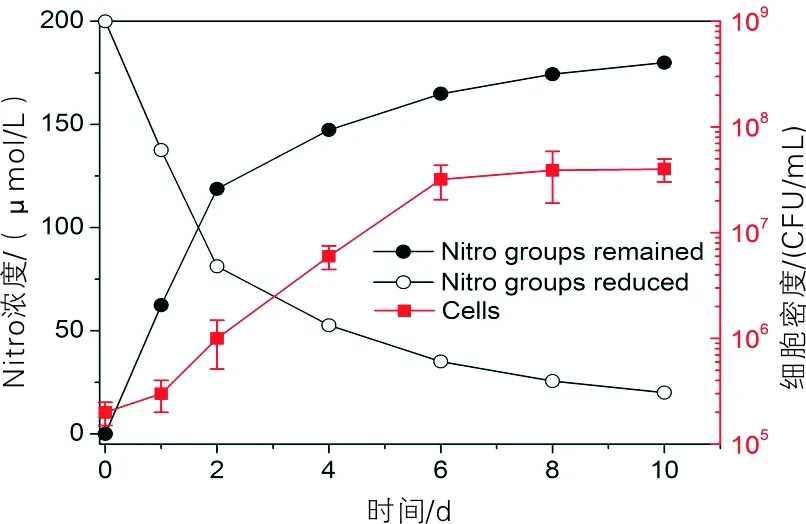
图2 8%盐度下硝基呼吸还原和细胞生长耦合情况
EP1菌株在含盐环境中显示出较强的DNT转化能力,特别是与2种非专性海洋希瓦氏菌相比,S.putrefaciensCN32和S.oneidensisMR-1的最适生长仅发生在盐度为0%时[5]。盐度偏好表明,EP1为专性海洋菌,且更耐高盐度。研究还发现,EP1可在高达20%的高NaCl浓度下对染料Xylidine Ponceau 2R还原脱色[13]。 为深入了解厌氧呼吸遗传基础并探索盐分耐受性的潜在应用,对EP1全基因组序列进行测定,并开展生物信息学分析。
2.2 全基因组测序
目前,希瓦氏菌S.marisflavi种内还没有获得任何菌株的基因组序列。因此,对菌株S.marisflaviEP1的全基因组进行测序,以期获得对遗传性状的更多了解。高通量测序共产生了52 966个高质量读长,平均读长7 518 bp。使用分层基因组组装技术从头开始重新组装读长,以生成仅含1个大重叠群(contig)的完整基因组。
S.marisflaviEP1基因组的一般特征见表1,S.marisflaviEP1的环状基因组图如图3所示。图3中,从外到内:环(1)显示了以Mbps为单位的刻度线;环(2)和环(3)显示了+和-链上的预测编码基因,并通过不同的COG功能分类进行着色;环(4)代表tRNA(红色)和rRNA(蓝色);环(5)和环(6)分别表示G+C含量和G+C偏移。由表1和图3可知,EP1的基因组为4 321 452 bp的环状染色体,G-C含量为50.0%。通过PGAP分析注释,染色体上总共预测了3 661个蛋白质编码基因,25个rRNA和98个tRNA(见表1)。此外,根据COGs数据库的基因组功能注释和分类,共有3 364个基因被划分为22个不同的功能类别,包括能量产生和保存、氨基酸转运和代谢、辅酶转运和代谢、碳水化合物转运和代谢、无机离子转运和代谢、次级代谢产物合成、转运和代谢等(见图3(b))。

表1 S. marisflavi EP1基因组的一般特征
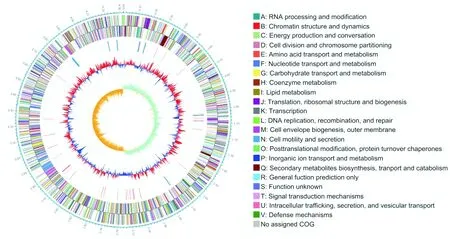
(a) S. marisflavi EP1的环状基因组图
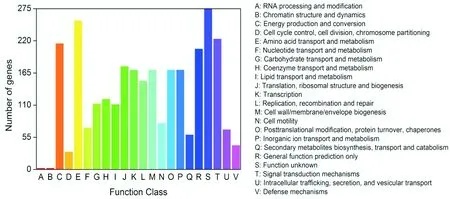
(b) EP1基因组在COGs数据库的功能注释
2.3 与2,4-DNT呼吸和耐盐性相关的基因
S.marisflaviEP1对2,4-DNT的还原转换能力可能是细胞内多种电子传递系统蛋白共同作用的结果,尤其是c型细胞色素蛋白可能在2,4-DNT呼吸还原中起到重要作用[14]。基因组分析表明,EP1共有多达41种推定的c型细胞色素,包括10种10-血红素细胞色素c和1种定位表面的非10-血红素细胞色素c脂蛋白。与其他希瓦氏菌类似,EP1具有1个编码c型细胞色素的基因座。这些色素构成Mtr电子的传递途径[15]。希瓦氏菌中编码胞外电子传递系统基因座对比如图4所示。由图4可知,EP1基因座中的基因排列与S.pealeana和S.halifaxensis最为相似。
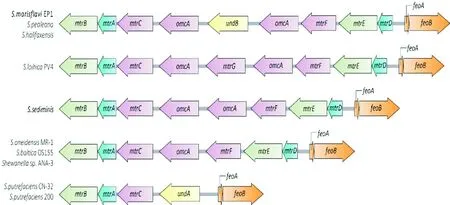
图4 希瓦氏菌中编码胞外电子传递系统基因座对比
对于希瓦氏菌属,Mtr呼吸途径被认为是硝基化合物厌氧呼吸中必不可少的胞外电子转移途径[16-17]。4种血红素c型细胞色素(包括MtrA,MtrB,MtrC和OmcA)是Mtr呼吸途径的主要成分,均参与DNT呼吸还原。然而,敲除Mtr途径并没有导致DNT生物还原能力完全丧失。这表明除Mtr途径之外的其他c型细胞色素可能也参与了DNT的还原[17]。这些结果与EP1的基因组分析结论一致,即除了Mtr途径外,大约还有30种推定的c型细胞色素由EP1基因组编码。c型细胞色素的多样性表明希瓦氏菌中电子传递至DNT的机理比预期的更复杂。
S.marisflaviEP1基因组中耐盐性相关基因见表2。由表2可知,EP1基因组包含多个盐度耐受相关基因。编码阳离子/H+反向转运蛋白nha的基因使得细菌具有对高Na+浓度和高碱性的耐受性[18-19]。yggT基因编码蛋白赋予细菌细胞对渗透休克的耐受性[19]。kef基因编码的阳离子特异性通道蛋白参与渗透压适应性[20-21]。trk基因也被发现存在于染色体上,其有助于Na+的胞外输出,并提高细胞抗盐能力[22]。
3 结论
EP1具有出色的2,4-DNT还原性能和耐盐性,在含8%~10%NaCl的高盐度下能有效地进行2,4-DNT的生物转化。菌株EP1的基因组为环状染色体,总长为4 321 452 bp,G-C含量为50%,包含3 661个编码基因。基因组注释发现包括Mtr呼吸途径在内的共41种c型细胞色素负责电子转移和硝基呼吸还原,多个耐盐性相关的基因也在EP1基因组中被识别。高盐度下EP1呼吸还原2,4-DNT的性能及其遗传特征会加深对环境中硝基芳香族化合物转化和归趋的理解,同时还可为硝基芳香族化合物环境污染修复研究积累基础。
