影响开花植物物种多样化速率变化的因素探讨
2021-01-12郭文钰黄建勋
郭文钰 黄建勋

摘 要:開花植物(被子植物)是目前最为繁荣兴盛的陆地植物,是地球上种类最多、分布最广、适应性最强的优势植物类群。然而,物种多样性在开花植物的谱系中极端不均匀,同时其物种并非匀速增加,而是集中在某些时期快速扩张。这种现象暗示了开花植物的多样性受到某些因素的调控,而揭示影响开花植物多样化速率的机制便是这数十年来植物学家孜孜不倦努力的目标。该文介绍了当前的研究进展。研究显示,那些与类群分类相关的关键性状大多与多样化无关,而早期被视为决定因素的花性状,目前被认为影响较低或是需要多性状联合才产生实际影响。多倍化是一个受到高度关注的候选因素,被认为能提供物种扩张所需的遗传物质,在适当的情况或其他因素的参与下促成物种多样化。然而,许多研究也呈现各类的争议,这些悖论或许代表了多倍化不是决定因素,而是扮演着间接的角色。另外,环境的变异(温度、湿度、空间)也能提高多样性的发生。整体而言,影响开花植物多样化速率的原因,很可能是一种综合因素共同促成的结果,而每个植物类群多样化速率提升的原因亦可能不尽相同。因此,先分别对植物类群进行探讨,再进行整合,或许更能提供一个确实且普遍的模型。
关键词:物种多样化速率,开花植物,多倍化,性状,地理环境
中图分类号:Q949
文献标识码:A
文章编号:1000-3142(2021)10-1707-12
Abstract:Flowering plants (angiosperms) constitute the most prosperous group of land plants. They are the dominant plant group with the highest diversity and the widest distribution. However,species richness across lineages of flowering plants is extremely uneven,and species expansion is not smooth,but concentrated in certain lineages during particular periods. This implies that the distribution of biodiversity of flowering plants is influenced by some factors,and revealing the underlying mechanism is the goal of botanists’ unremitting efforts for decades. In this review,we try to review the progress of current researches related to this scientific question. Previous studies showed that most of the synapomorphies used by systematists to define the major clades of flowering plants do not drive higher rates of diversification. Flower traits,which were considered as determinants in the first place,were currently considered to have low impact to diversification,while a combination of multiple traits seemed more influential than a single character. As a popular candidate,polyploidy has been widely studied with debates. These paradoxes imply that instead of a determinant factor,polyploidy may act indirectly by providing the genetic materials for and thus promoting species diversification under appropriate circumstances with the participation of other factors. Environmental variation (such as temperature,humidity,and space) seems to associated with improved biodiversity,too. Overall,what affects the biodiversity of flowering plants is likely the result of a combination of factors and may not be the same for different lineages. Therefore,exploring potential factors in different plant groups and integrating the information may provide a solid conclusion than considering one particular factor across the whole flowering plants.
Key words:diversification rate,flowering plant,polyploidization,trait,geographical factor
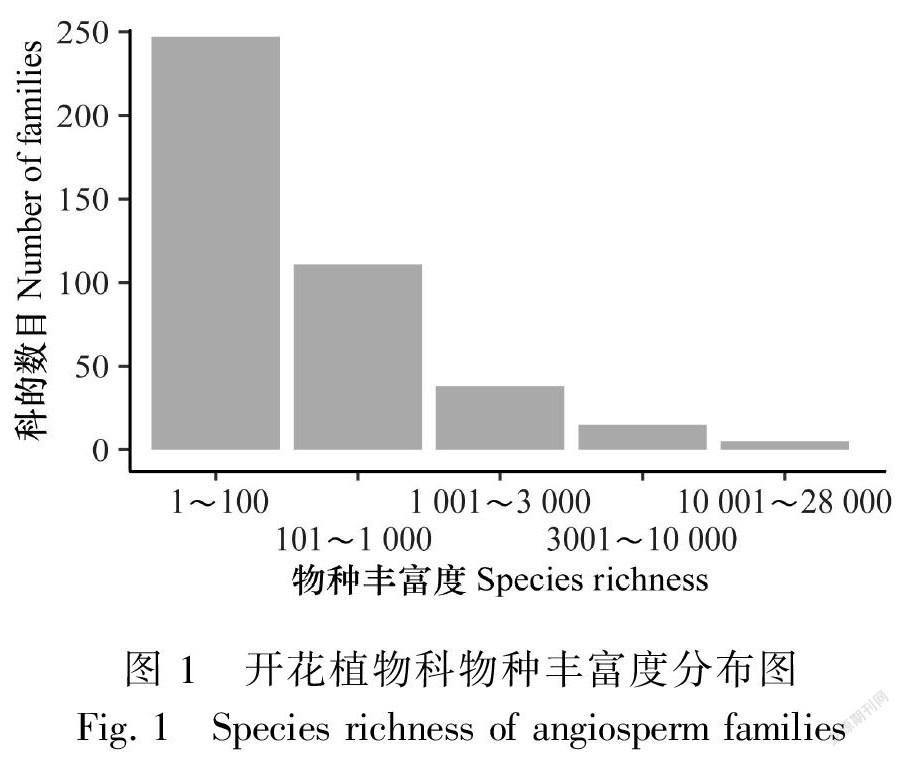
开花植物(又称为被子植物,angiosperm)是陆地植物王冠上最为璀璨的明珠。开花植物与人类生活密切相关,涵盖了大部分主要的粮食作物(包括水稻、小麦、玉米、大豆等)和经济作物(大部分的蔬菜、水果、观赏植物),而开花植物突然的起源和爆炸性的物种丰富度,更被达尔文称为“恼人之谜”,也是科学家们一直以来关注的焦点。开花植物起源于两亿五千万至一亿四千万年前(Foster et al.,2017; Magallón et al.,2015; Silvestro et al.,2015),具有约304 000个已命名物种,可能还有多达156 000个未命名物种(Pimm & Raven,2017);相较之下,与其关系最密切的裸子植物只有约1 000种,这两个姊妹类群的物种数目呈现了350倍的差异(35万 vs. 1 000)。这种极端差异暗示着开花植物在物种分化上的显著优势。然而,开花植物各类群之间的物种数目亦具有极大的差异(图1),大的科包含了超过两万个物种(兰科和菊科),小的科却仅有一个物种(29个科),大小科之间的物种数目差距达到了两万以上,而超过60%的科(274个科)物种数目则在10~3 000之间(Christenhusz & Byng,2016)。另外,Tank et al. (2015)研究发现,开花植物在时间维度上并不是均速地发生物种分化(图2),而是集中在某些时期快速增加(图2中尖峰的时期)。要注意的是,由于Tank et al. (2015)的分析是以科为单位进行物种多样化速率的分析,故此数据仅显示科以上物种多样化速率变化的情况。这两种现象暗示开花植物可能借由某些有利的因素,在特定时期的某些类群快速分化。因此,数十年来许多科学家对开花植物的进化进行了各种分析,以期揭示影响开花植物物种扩张的潜在因素。
促进物种多样化速率的过程及可能的影响因素相当的复杂。为此,科学家们数十年来经由各种不同层面的观察结果,找出了众多可能因素作为演化的推动力,并逐一进行关联性的分析。要强调的是,在对于小类群的研究中虽然能发现若干证据支持,但是往往受限于数据量太少、缺乏有意义的统计支持而流于主观的认定。相对地,在研究跨越长时间的大类群时,却往往很难有明确的证据支持,而流于一种笼统的印象。当前对开花植物的物种多样化速率变化成因的探讨,倾向于关注较大类群,并锁定单个或少数几个因素去进行关联性分析的方式,合并有意义的统计分析,以避免笼统性与模糊性。本文将介绍现有对开花植物的物种多样化速率变化有影响的可能因素,整合目前科学家对这个复杂问题的理解,大致包括了性状、多倍化、地理环境等方面(Vamosi et al.,2018)。 必须强调的是,这些结果受限于方法的假设、模型的设计、过于简化的划分或归类、不完整或有偏见的抽样等,得到的结论可能仍旧存在争议。再者,也不能排除其他未被研究的影响因素,或者物种多样性分布是由数个因素的联合作用而产生实际的影响,而非单个因素所造成的结果。整体而言,这个未解之谜仍需要科学家们的后续努力,协力揭开其神秘面纱。目前这些研究大致可以归类为三个角度:(1)开花植物发生多样化速率提升的时段,是否伴随着某些性状、地质气候、地理等方面的转换?(2)多倍化对于物种分化的影响究竟是促进还是抑制?(3)若是单一因素不足以促进物种多样化速率的变化,具体哪几个因素的协同作用能更明显地提升物种多样性?下面将探讨目前对这些问题的研究成果。
1 如何关注物种多样化速率的变化
影响演化的因素从达尔文开始即有非常多的研究,基因、性状、天择、地理隔离等都有所相关,然而这些研究限于研究方法往往在一些小类群之间才能呈现明显的证据,而在跨越大范围时间的尺度(数百万年以上)、广泛的类群上(数十万物种以上),从系统发育的角度来探讨的研究则较为稀少。在被子植物中,大的类群未必一定发生过快速增加的物种多样化速率(diversification rate)的变化,它也可能是随着时间逐步生成的。因此,欲研究影响物种多样化速率变化的原因,需要先确认物种多样化速率的估算,估算的原理与方法简介如下。
被子植物物种多样性分布不均的现象很早便已经在古生物学研究中被注意到(Magallon et al.,1999),然而并未引起广泛的关注。这是因为古生物学主要依赖化石证据去做推断,化石质量的差异容易引起物种丰富度的估算偏差(Xing et al.,2016),所以多数科学家对此现象仍存有疑虑。自Slowinski & Guyer (1993)开发出姊妹群测试(sister-group tests)后,合并系统发育数值对物种多样化速率进行估算的分析方法快速地发展(Rabosky & Lovette,2008; Ricklefs,2007; Sánchez-Reyes et al.,2017),相關研究也快速激增。具体方法是基于带有年份信息的系统发育树,合并现存的物种数目,借由估算物种形成率以及物种灭绝率来获得物种多样化速率)。这种基于系统发育树进行估算的方法目前已被普遍使用,其中有较为著名的两种方法。一种方法为 MEDUSA(modeling evolutionary diversification using stepwise akaike information criterion)(Alfaro et al.,2009)。此方法使用最大似然法(maximum likelihood)将可变速率之出生死亡模型(variable-rate birth-death models)套用到系统发育树上,并且使用AIC数值(akaike information criterion)判断挑选最贴近真实数据的计算模型,鉴定树的枝条之间物种多样化速率的显著增加或减少。此方法尽管经过广泛的使用,但是模拟研究表明此方法存在一些缺陷,主要因其估算的物种多样化速率的数值在一定的区块(regime)内是保持恒定的(比如在一条树枝内),当速率随时间变化且具有较高的错误发现率(false discovery rate)时,它会低估树中的多样化区块(diversification regimes)的数量,在此情况下实际速率与估计出的速率结果之间会出现较大差异(Rabosky et al.,2014;May & Moore,2016)。尽管如此,但是它仍然是目前很受欢迎的方法之一。另一种方法是BAMM(bayesian analysis of macroevolutionary mixtures)(Rabosky,2014; Rabosky et al.,2014)。此方法相似于MEDUSA,但基于贝叶斯方法进行估算,同时引进了在同一区块内其物种生成率与灭绝率可以随时间变化的模型(即一条树枝内的速率可以是不同的)。此方法也存在一些顾虑,比如没有考虑到沿着灭绝分支的多样化速率变化的可能性,也没有考虑到多样化速率变化之后的后验分布的准确性(Moore et al.,2016;Rabosky et al.,2017)。
整体而言,对于物种多样化速率的估算,现存的两种模型虽然仍存在一些算法上的疑虑,但是大体上还算是受到科学家所认可的,而其瑕疵也持续在改进中,或许随着时间的变化,这方面的问题能够更为减弱或被解决,能获取更为精准的估算结果。本文所介绍的文章都是经过物种多样化速率计算的被子植物类群,在此基础上,介绍与物种多样化速率变化相关的影响因子。
2 关键性状的创新与开花植物物种多样化速率的联系
科学家们最初的假设是关键形态的转变影响了物种多样化。此假设认为,当进化中出现一个关键形态时,可能带给这个类群极大的竞争优势,因此导致多样化速率提升,进一步造成辐射分化和种群扩张。早期科学家猜测所谓的关键形态是指在分类上具有独特性的、足以用来区分不同类群的显著特征,并由此进一步推论,多样化速率提升的时机应该和大类群(比如科、目)的分界点接近,甚至重合。Sanderson & Donoghue (1994)最早进行多样化速率与性状转变的相关性研究,他们利用纯出生模型(yule model)进行计算,发现开花植物进化早期一直维持着较低的多样化速率,后期才出现一些提高了多样化速率的独立类群。此结果显示,开花植物共有的独特性状(包括花器官、双受精、3N胚乳、保护种子的果实构造等),不足以解释开花植物的扩张。类似的结论也在后续使用出生-死亡模型(birth-death model)对不同类群进行的研究中得到了验证(Davies et al.,2004a; Magallón & Castillo,2009; Magallon & Sanderson,2001)。其中,Smith et al. (2011)使用超过55 000个物种的系统树,分析其多样化速率,提出了约2 700个速率的变化节点,发现这些变化点发生的位置更倾向于分支之内,而非分支的起源[比如科或目的冠点(crown node)]。这个结论同样显示,分类上用来划分与定义类群的那些性状,并非提升物种多样化速率的关键。
2.1 花朵两侧对称对多样化速率的影响
花朵是开花植物特有的生殖器官,被认为对于植物多样性有关键性的影响,因此科学家们对于花性状与多样性的关联也有着长久且专注的热忱。其中,花的两侧对称(zygomorphy)在发育、遗传、形态性状和进化等领域上受到大量关注与研究(Citerne et al.,2010; Cubas,2004; Endress,2001a; Hileman,2014; Reyes et al.,2016)。此性状借由特殊花的结构和角度去限制传粉者以特定方式接触花粉,从而提高了传粉的效率和专一性,而更易形成生殖隔离。由于传粉专一性和生殖隔离从而促进物种分化,此性状被视为是提高物种多样化速率的可能原因 (Citerne et al.,2010),然而从长远来看,专一化的传粉者也存在提高灭绝速率的风险,即专一传粉者灭绝时,也容易导致自己灭绝。为探索两侧对称和开花植物多样化速率是否相关,Sargent (2004)通过对姊妹枝的比较,发现两侧对称与开花植物中物种较丰富的那枝相关,Vamosi JC & Vamosi SM(2011)也通过系统进化回归分析(phylogenetic regression)得到类似的结论。尽管这两份研究都显示了正向的相关性,但是他们在方法上使用了较大范围的类群进行分析,对于性状的定义难免笼统简化。如某科有较为多数的物种为两侧对称,就简化为此科为两侧对称;又如向日葵,其花盘由舌状花与管状花组成,但舌状花为两侧对称,管状花为辐射对称,这类的花性状就应该要依据实际发生功能的花种类进行定义较为正确。加上还有着许多性状的反转(至少69个)(Reyes et al.,2016),真实的情况比他们当初假设的要复杂得多。若是考量性状的复杂情况,对数据进行更细致的分类,并且精确定义选择姊妹枝 (sister clade),后续研究中只得到了模棱两可的相关性(Kay et al.,2006)。而除了对姊妹枝的比较之外,BiSSE分析(binary state speciation and extinction)可针对二元性状测试其与物种多样化速率之间的相关性(FitzJohn et al.,2009; Maddison & FitzJohn,2015; Maddison et al.,2007)。然而,Reyes et al. (2016)对山龙眼科研究结果显示,并未发现花对称性状对于多样化速率有显著性的影响。因此,目前花对称性对于物种多样化速率的影响尚需要后续更多对不同植物类群的研究。
2.2 合生心皮和合生花瓣与多样性速率变化的关联
其他花性状中,心皮与花瓣的合生(syncarpy和sympetaly)也被视为是开花植物的关键创新(Armbruster et al.,2002; Endress,1982,2011a,b)。合生心皮(syncarpy)是大部分单子叶植物与真双子叶植物的特征,在开花植物中至少独立演化出现2~17次(Armbruster et al.,2002; Endress,2011),相较于离生心皮,能更好地提供花粉管生长,有利于花粉竞争,因此对于核果(drupes)与坚果(nuts)这些借由动物选择而传播的进化方式更有竞争力(Armbruster et al.,2002; Endress,1982,2006,2011)。合生花瓣(sympetaly)则是大部分菊类植物(asterids)的特征,不过开花植物的其他类群也时常可见(Endress,2001b,2011; Sauquet et al.,2017)。合生花瓣由于使花结构更加稳定,因此容许在花的大小与花管的长度上有较大的变异,而这样的变异已被證明有利于增加生殖隔离以及传粉者的多样性(Endress,2001b)。引申来说,这些借由调整花器官各种部位(比如雄蕊、花瓣)的变化,都有可能因为授粉者的专一化和高效化,而对物种多样化速率产生改变(Endress,2001a,b,2006,2010,2011)。然而,目前尚缺乏在取样上能高度涵盖整个开花植物的研究来对这些假设进行正式分析,因此这几种性状的影响仍有待验证。
3 多倍化简介与促进/抑制物种多样化速率的相悖观点
在众多被认为可能影响多样化速率的候选因素之中,多倍化(polyploidy)受到了广泛的关注与研究。多倍化事件可分为由两个物种杂交所形成的异源多倍化(allopolyploidy),或是一个物种自己发生了全基因组重复而造成的同源多倍化(autopolyploidy)(Ramsey & Schemske,1998)。这里常见的假说:多倍化将导致基因拷贝大量增加,这些新的拷贝便可能借由剂量效应(dosage effect)(多拷贝导致表达的蛋白增多,所以某方面的功能强化)或是因为减低的选择压力而增加突变率,故有较高的概率发生新功能化(产生新功能)(neofunctionalization)、亚功能化(原先功能分开到两个基因上去各自执行)(subfunctionalization)等的基因分化,这些都可能导致物种出现生理、结构或形态的变化,进而产生适应不同环境的竞争优势,提高物种分化的概率,因此被视为是促进物种多样性的可能原因(Chen et al.,2007; Soltis et al.,2009)。然而,刚形成不久的多倍体由于突增的遗传物质,干扰了减数分裂、细胞分裂等机制,存活不易。同时,倍化的配子要找到同样倍化的植株才能繁殖,在初期族群中多倍体数量稀少时是十分困难的(Levin,1975; Ramsey & Schemske,2002),使得这些多倍体具有繁殖力低、遗传变异性低的特质。目前,已知开花植物有30%~40%的物种在属内出现了多倍化(Stebbins,1938; Wood et al.,2009),尽管多倍体如此广泛分布,但仍有观点认为多倍化是进化上的噪音,他们虽然频繁地出现,但是经常走向进化的死胡同(Grant.,1981; Ohno,1970; Schultz,1970; Stebbins,1950; Wagner,1970)。
3.1 植物类群普遍经历过多倍化不能作为支持有利物种多样化速率的证据
值得一提的是,由于现存的类群中存在很多的多倍体,并且在大多数植物基因组的祖先中都检测到多次古代多倍化事件,部分学者认为这代表了多倍化在长期来看对植物进化是有利的,然而这些现象可以单纯来自于高频率发生的多倍化事件,并不能直接作为支持多倍化有利于物种多样性的证据。Meyers & Levin (2006)研究发现开花植物中多倍体形成的频率很高,而后逆转回二倍体状态(指的是染色体经历丢失、融合重组等过程而发生的去多倍化的现象)的概率却很低,因此出现众多的多倍体物种仅仅是一个不可避免的现象,而不涉及多倍化对于进化是有利还是有害。后续Scarpino et al. (2014)利用60个开花植物数据,辅以估算模型的优化与扩展进行分析,提出相较于二倍体,多倍体对多样化反而较为不利。他们进一步推论,多倍体的普遍性是由于它们频繁形成(Otto & Whitton,2000; Vamosi & Dickinson,2006),而不是有任何短期或长期的优势。然而上述两篇研究由于使用属水平的数据并且忽略属内关系后进行估算,因此无法区分其数据是由一个多倍化事件后产生了一个高度多样的进化枝,还是由数个多倍化分别产生的多个低度多样性的进化枝。Mayrose et al. (2011)则利用63个植物类群(大多为属)的数据,先使用chromEvol估算了倍性水平(Mayrose et al.,2010),然后使用BiSSE模型推断了倍性与多样化的相关性,结果发现多倍体分支的物种多样化速率显著较低。然而,当进一步将物种形成事件是否涉及了多倍化进行区分后,结果显示,二倍体具有较高的物种多样化速率这一结果,最适合的解释是由于经过多倍化后的二倍体(相较于新生多倍体)更容易发生物种分化。即:逆转回二倍体(随时间将倍化的染色体进行丢失融合等重组而恢复原本染色体倍数)的物种,由于发生多倍化时增加了基因拷贝,具有基因新/亚功能化的潜力,又因为恢复为二倍体而没有多倍体在细胞/减数分裂和匹配配子時的困难,因此更有可能促进物种分化。虽然Mayrose et al. (2011)和Scarpino et al. (2014)的分析是基于不同的原理和模型,以及不重复的数据集,但是分析涉及的开花植物比例还是太少,因此这些结论仍有待进一步的验证。另外,Barker et al. (2016)提出,理论上同源多倍体的形成率应该超过异源多倍体,但两者实际上出现的频率却差不多。这暗示了异源多倍体可能更有进化上的优势,可能是两种不同的基因组结合,涵盖了两方亲本的生态位以及更大的遗传变异,因此提高了异源多倍体在生存与维持上的竞争力。至于这样的现象是否会在进化上对物种多样化速率造成长期的影响,尚待进一步的验证。
3.2 多倍化对物种多样化的影响可能需要一段滞后时期
尽管从目前的结果来看,多倍化在短期而言对物种多样化是不利的,但是其长期的影响还不能肯定。科学家们假设多倍化或许对物种而言存在着长久而深远的影响,比如经过漫长时期的进化,植物已经克服了基因组不稳定,克服了细胞/减数分裂和繁殖上的困难,而借由重复的基因增添了大量的遗传变异,得以成功地从祖先的表型、栖地上分别开来而成为新种。这样的理论,指出多倍化之后,可能要间隔一段时间,才能消化掉多倍化的害处而获取它的优势。Schranz et al. (2012)提出多倍体对多样化的积极影响需要经过一段滞后时期 (lag period),在通过二倍体化、新的关键性状产生和遗传多样性建立之后,才能显现出来。不过这个假说目前的支持证据仅来自于较近期发生的物种类群,比如车前科的婆婆纳属(Veronica,Plantaginaceae)(Meudt et al.,2015)和十字花科(Brassicaceae)(Hohmann et al.,2015),依然需要其他更广泛的研究进一步验证。Tank et al. (2015)评估过开花植物中的多倍化与多样化速率上升之间的关联性,他们使用定年后的科水平物种树,以MEDUSA测算物种进化速率的变化,结果显示有速率增加的节点与多倍化发生的位置很少重合,但有六个多倍化事件滞后的三个节点内发生了多样化速率大幅提升的现象。此研究可视为对滞后理论(Schranz et al.,2012)的一种支持,但其包含的多倍化事件大多位于容易取得基因组的大类群中,为了确认这一关联性是否受到此种潜在偏差的影响,后续仍需增加多倍化事件的数据进一步验证。Landis et al. (2018)则进一步综合了大范围的开花植物类群及其中可能的多倍化节点,针对多样化速率和多倍化之间的相关性进行分析,其中姊妹枝比较的结果支持多倍化伴随着较高的丰富度,而当直接比较多倍化和多样性变化节点时,虽然有一定比例的数据符合滞后理论,但是这种分布和随机分布的数据组相比并没有显著差异。另外,此分析中采用的多倍化结果,其部分事件在不同分析方法中并未得到一致的结果(其采用有多种判断多倍化的方法,很多多倍化发生的位点却往往只有一种方法侦测到,其他的方法却不支持),取信上存在疑虑。因此这部分结果还须未来更深入地分析并进一步验证。
3.3 其他导致基因重复的方式是否有利于物种速率变化
由于目前的分析方式除了使用基因组的共线性分析(synteny)能够较为确定多倍化是为全基因组重复(即同源多倍化),至于同义替换速率分布(Ks distribution)和系统基因组学(phylogenomics)等方法目前尚无法准确地分辨同源或异源多倍化。另外,某些由串联重复、转座子所导致的关键基因家族的进化,是否也起到重要的作用,这些都是将来应加强关注的方向,不过这些信息在很大程度上需要依赖于基因组测序后获得的基因所在位置的信息来加以分辨,在目前大量材料为转录组的前提下有些难以实现。不过在大量转录组为材料中,只要某多倍化发生的祖先物种其后代覆盖了至少一个基因组测序的物种,即可达成继续深入追踪多倍化事件的来源基础。
4 气候与空间因素对开花植物物种多样化速率的影响
温度和湿度等气候条件皆会影响物种丰富度,而纬度与海拔的变化皆会造成不同的气候条件。和生物界其他的类群一样,植物的物种丰富度随着纬度呈现梯度分布,其多样性在热带地区是最高的,随后往温带与极地逐渐下降(Gentry,1988)。一些研究显示热带地区的多样化速率更高(Davies et al.,2004b; Jablonski et al.,2006; Jansson & Davies,2008),另外的研究则支持热带保守假说(tropical conservatism hypothesis),推论温带物种是由属于热带的祖先物种在气候变化时适应了温带化的栖地而产生,并提出证据显示分布在温带的大部分类群起源于全球寒冷化(始新世-渐新世界线)之后,并且嵌套在热带物种谱系之中(Kerkhoff et al.,2014)。这种现象也同样出现在鸟类以及哺乳类(Rolland et al.,2014; Weir & Schluter,2007)中,皆符合此假说的预测。整体而言,随着纬度而变化的生物多样性,很可能是因为热带地区稳定而四季恒常的气候,再加上古地质历史上的气候更接近热带,因此保留更多植物多样性的结果(Crane & Lidgard,1989; Crisp et al.,2009)。
4.1 热带高山拥有较高的多样化速率
其他研究则发现热带高山地区的植物类群具有特别高的多样化速率(Hughes & Eastwood,2006; Lagomarsino et al.,2016; Merckx et al.,2015; Pennington et al.,2010; Uribe-Convers & Tank,2015)。这可能是由于在高山地区中,因海拔变化造成了较大的温度梯度变化和物种群落差异所造成的现象。在过去对植物多样化速率变化的研究中,通常没有同时考量纬度或海拔的影响,也就是大多将纬度与海拔分成两个方向去考量,但低纬度(热带)地区的高度生物多样性是否是因为低纬度地区的高山其丰富的物种多样性所造成的?目前的分析方式是否存在将同一现象(热带地区高物种丰富度)赋予两种解释的疑虑(低纬度、高山物种多样化速率较快)?建议未来的研究可拆分这部分数据去进行验证。
多倍化后的植物通常会因为染色体倍数的差异导致多倍体后代谱系与其祖先之间的生殖隔离,再加上多拷贝基因因为降低的选择压力而增进突变的可能,容易造成多倍化物种与原种之间产生形态和生态上的差异,起到驱动新物种形成的作用(Zhan et al.,2016)。目前一些研究在高纬度或高山地区中观察到较高的物种形成率,也被认为可能与较多的多倍化相关(Brochmann et al.,2004; Vamosi & McEwen,2012)。
4.2 群体连通性影响物种多樣化速率
除了温度和湿度之外的地理因素也会造成物种丰富度的差异,特别是影响群体连通性(population connectivity)的情况下,例如生活在海岛(Baldwin & Sanderson,1998)或山峰(Comes et al.,2008; Merckx et al.,2015)等空间隔离的环境中的某些开花植物类群,便发生了适应性辐射的现象。其他研究也发现,在地中海区域的一些干燥栖地有高度的物种形成率(Buerki et al.,2012; Goldberg et al.,2011; Reyes et al.,2015),这很可能是由于这些栖地的干燥度不均匀,其中水的可利用性和土壤养分的异质性形成了群体之间的空间隔离,而导致较高的新物种的形成率(Savolainen et al.,2006)。
5 复合原因的协同影响
除了上述已被研究的可能因素之外,是否还有其他目前未知的、从未考虑的因素,实际上才是推动一系列性状变化而导致多样化速率提升的真正原因(Maddison & FitzJohn,2015; Rabosky & Goldberg,2015)?这是对于多样化速率影响因素相关研究的一大挑战。尽管我们不可能列入所有可能的因素,但是将多重因素同时列入考虑则或许更贴近事实。比如,O′Meara et al.(2016)使用500个开花植物的数据,分析六个花性状的复合效应对于多样化的影响,结果发现有花冠、两侧对称、低雄蕊数目三个性状的联合作用,比单独一个性状与多样化速率变化之间的关联都更显著。此发现同时符合了长久以来植物学家认为诸多花性状之间并非独立进化的观念 (Jabbour et al.,2008; Sauquet et al.,2017)。虽然这两篇研究在方法上仍有值得改进之处,比如对性状归属的简化,取样范围过小等,但是可说是多性状复合作用研究方向的一篇重要代表作。
5.1 复合效应的关联性
广义而言,复合效应可以包含形态、地理、基因遗传的多元因素。首先,研究性状在系统发育过程中进化的模型已经被开发出来,用来明确地测试性状是独立进化还是以互相具有关联性的方式进化,现有的计算模型亦可以适用于离散型(Huelsenbeck et al.,2003; Lewis,2001; Pagel,1994,1999; Pagel & Meade,2006; Schluter et al.,1997)或是连续型(Pagel,1997; Schluter et al.,1997)的多个性状的研究。目前已有一些研究使用这些模型估算多种特征之间的关联性,比如花瓣的微结构与授粉者的种类(Ojeda et al.,2016),特定基因与病原体感染树木的致病能力(Nowell et al.,2016)等。
虽然这些方法并不能显示性状间的因果关系,但是离散数据的模型也可以用来推论一个性状对其他性状转变的影响。比如,在分析环境与C3/C4植物进化的关联性时,C4植物与干燥栖地的关联性是很显著的,至于具体的原因,Osborne & Freckleton (2009)研究发现,这并非是由于C4植物较容易出现在干燥的栖地,而是C4植物一旦演化出现后,就更容易适应而转移到干燥的环境。又比如在对大戟属与枫属植物的研究中(Armbruster,2002),不但发现花色与秋天的叶色两者之间具有关联性,而且显示叶色的改变是在花色变化之前发生的,代表植物中叶的色素(如花青素)在需要时会先被选择,随后才被合并进入花中。
5.2 未经研究的性状或性状-环境的组合或许才是真正的主因
目前大部分的研究结果普遍显示,代表关键创新的许多形态可能不是影响开花植物多样化速率变化的唯一原因,甚至不是主要原因。相反地,未经研究的性状或性状-环境的组合或许更可能才是刺激物种形成的主因(Bouchenak-Khelladi et al.,2015)。我们同时也需要考虑以下的可能性:即便观察到了某个性状与多样化速率的相关性,此一性状仍可能是通过其他性状的转变产生了间接影响,而非导致多样化速率变化的直接因素(Maddison & FitzJohn,2015)。這种情况会大幅影响对分析结果的判读。比如,具有纤细导管的植物,由于在寒冷的气候下结冰的问题较小,在低温环境中生活的适应成本较低,因此更容易在寒冷的环境中定居(Zanne et al.,2014)。这样的性状可以视为是在进化过程中产生的一种基底(前置)性状,具有更容易适应某种环境的前提而更容易导致生态位扩张。
5.3 多倍化-环境气候-多样性之间的关联性结论
另外,也有一部分的文章分析并讨论了多倍化-环境气候-多样性之间的关联。Vanneste et al. (2014)首先提出,在现存植物基因组中所侦测到的古多倍化事件,大多聚集在白垩纪-古近纪的交界处(K-Pg boundary,65 Mya)(Fawcett et al.,2009; Vanneste et al.,2014),而非是均匀分布在开花植物中的。由于这段时期的环境剧变,导致地表众多生物灭绝(包括称霸了地球1.6亿年的恐龙),这一发现暗示了重复的基因可能提供了植物适应上的优势,赋予多倍体更大的表型可塑性以应对环境剧烈变化的冲击。然而,对此现象,也有其他的说法。如Vanneste et al. (2014)也提出一个中性的解释,认为这种现象可能只是由于在剧烈变化的环境下,形成多倍体的概率较高,并且在种群减少的情况下减轻了多倍体面临的少数细胞型缺陷,不一定代表多倍体更有优势。此外,Freeling (2017)也提出,白垩纪-古近纪的交界处发现很多的古多倍体事件也可能是一种搭便车效应(hitchhiking effect),是由于无性繁殖或许更容易在剧变环境中生存,使得多倍体在这段灭绝期间得以活下来。更有研究认为多倍化并没有在气候剧烈变化的时期更加密集(Ren et al.,2018)。然而,确实也有一些研究结果显示正向的结论,比如在菊科、葫芦科、豆科中,其科内物种快速扩张时期,通常与多倍化事件,剧烈的气候变化甚至是某些性状的转变协同发生(Guo et al.,2020; Huang et al.,2016; Zhang et al.,2021; Zhao et al.,2021)。对于这种多因素之间的关联性,还需要未来更多研究,对更多类群或更广泛的类群,加入各层面的因素考量的分析结果去进行验证。
6 要点总结
(1)影响物种多样化速率提升的性状大多不是分类上用来定义分支的性状。比如开花植物共有的独特性状,包括花器官、双受精、3N胚乳、保护种子的果实构造等,皆不足以解释开花植物的扩张(Magallón & Castillo,2009; Magallon & Sanderson,2001; Sanderson & Donoghue,1994; Smith et al.,2011)。
(2)花的性状对物种多样化有其重要性,花的两侧对称,合生心皮与合生花瓣在植物学家眼中很早便被视为与物种扩张有关,然而这些性状与物种多样化速率变化之间的关联性,在目前多个研究的结论中有所冲突,尚无法获得肯定结论(Citerne et al.,2010; Cubas,2004; Endress,2001a; Fitzjohn et al.,2009; Hileman,2014; Maddison & FitzJohn,2015; Maddison et al.,2007; Reyes et al.,2016)。
(3)多倍化对多样化的影响尚未有定论(Chen et al.,2007; Landis et al.,2018; Mayrose et al.,2011; Meyers & Levin,2006; Otto & Whitton,2000; Scarpino et al.,2014; Soltis et al.,2009; Vamosi & Dickinson,2006)。短期而言,多倍化对物种形成了存活和繁殖上的阻碍,对多样化是不利的。然而从长期来说,多倍化可通过增加的拷贝促进改变而获得适应上的优势,存在促进物种多样化的可能。现存类群中有很多的多倍体,并且多数植物基因组中都能检测到多次古代的多倍化事件,因此部分学者认为多倍化对植物的长期进化是有利的,然而亦有学者认为这可能单纯来自于高频率发生的多倍化事件,而并不涉及其对进化有利还是有害。
(4)当环境中有大幅度的温度、湿度变化,或空间上的隔离产生的不连续栖地,通常是物种多样化的热点,导致较高的物种形成率(Baldwin & Sanderson,1998; Buerki et al.,2012; Comes et al.,2008; Goldberg et al.,2011; Merckx et al.,2015; Reyes et al.,2015; Savolainen et al.,2006)。
(5)近期研究开始分析复合因素协同影响多样化速率的可能性。比如花冠、两侧对称、低雄蕊数目三个性状的联合作用与多样化速率变化之间的关联,较任一单独性状的关联都更显著(O′Meara et al.,2016),而某些非关键创新性状-环境的组合亦被认为能够刺激物种形成(Maddison & FitzJohn,2015; Rabosky & Goldberg,2015)。另外亦有多篇研究基于多个古多倍化事件被定年在白垩纪-古近纪的交界处(K-Pg boundary,此时期发生了生物大灭绝事件),而多倍化-环境气候-多样性之间的关联推论(Fawcett et al.,2009; Vanneste et al.,2014)认为多倍化可能提供了植物适应上的优势,赋予多倍体更大的表型可塑性以应对环境剧烈变化的冲击。然而,此观察亦可中性地解读为剧变的环境下减轻了多倍体的少数细胞型缺陷或者提高了其无性繁殖的优势,因而提高其生存率。不过,有部分研究结果确实显示了多倍化可能对多样化具有正向的影响,如在菊科、葫芦科、豆科中(Guo et al.,2020; Huang et al.,2016; Zhao et al.,2021),其科内物种快速扩张时期,通常与多倍化事件,剧烈的气候变化甚至是某些性状的转变协同发生。关于多个因素的协同作用和影響,还有待更多研究验证。
(6)同时不可忽略存在目前未知的、从未考虑的因素去推动物种多样化速率的可能,这或许是先前研究常得到模棱两可或甚至冲突结论的原因(Bouchenak-Khelladi et al.,2015)。举例来说,或许多倍化对物种多样化速率的影响是间接的,而直接影响的因素是目前未知的,因为尚未纳入研究分析,所以多倍化对多样化速率影响的研究结果仍无法肯定。
7 未来展望
综观物种多样化分布相关研究,最关键的困难点在于研究方向的模糊性与研究方法的提升。大体说到演化时,多数人都可以提出诸多影响的原因,如基因、性状、环境等的变化,然而在跨越大时间尺度的演化过程中,这些因素的影响将逐渐流于广泛、模糊不清,甚至互相交杂,如果不能分辨这些影响因素之间的前后、主从关系,将很难取得明确可靠的证据来说明其相关性。从科学研究的角度出发,牵涉多因素的复杂问题,应当抓住其中的主控因素,化繁为简,方有可能找出本质的规律。这就是为何本文中列举的文章,往往只关注某几个点对物种多样化速率的影响,这正是因为他们试图从复杂的原因中提出某因素确实影响物种多样化速率的证据。然而,当前对不同植物类群研究的结果似乎暗指开花植物中多次的多样化速率提升很可能不是基于同一种共有的原因,而是各类群特有的性状结合其生态环境共同造就而成。若此为真,专注于个别植物科的多样化速率改变过程,再加以整合,或许是探索问题答案的重要方法。要注意的是,现存的各种假说看起来都对其实际观察到的现象做出了合理的解释,而后续研究则必须注意是否先基于某假说立场而引导了数据分析和解读方向,否则,若假说并不真实,可能会将研究方向推离真相,并且越推越远。本文尝试将研究结果进行客观表述,让更多的科研学者能同时接收到相关研究的正反论点,而能以更加公正的态度去解读自己的研究发现,并发散思维,做出突破。
7.1 此类研究面临的困难之处
此外,目前各种事件之间(物种多样化速率提升、多倍化、性状转变,或气候、地理环境因素剧变等)具有关联的证据,主要都基于其发生时间位于系统发育树上相同或者邻近的节点上,并且具有统计显著性。然而,由于一些涉及系统发育学最基础的数据和方法上的限制,结果往往存在是否可靠的疑虑,这也是许多相关的研究结果较难获得广泛认同的原因。这些问题基本如下。
(1)系统发育树取样密度。基于对取样、测序、运算等成本的考量,一般研究中往往只收集代表类群进行分析,加上某些关键类群可能难以取得,很容易有取样不全的问题。即便真的将现存物种完整取样,也无法排除某些物种已灭绝的事实。因此,大多数的系统发育树,难免在某些节点上有所疏漏,尤其是大时间尺度的分析下,每个节点之间实际时间跨度平均在5百万~10百万年之间,对于进一步比较影响因素的关键程度,分辨其之间的主从关系造成了阻碍。同时,事件发生的时间段也可能因为节点跨度太大而显得笼统。
(2)代表类群对性状的归类。举例来说,进行性状重建分析之前,在归类一个代表类群的性状时,往往先会进行简化。一种方式是在量上进行简化,比如此类群有90%物种是A性状,有10%物种是B性状,一般就会将代表类群简化为A性状,反之,如果此类群A、B性状的物种比例是60%、40%,很可能就会将代表类群简化为A与B两种性状。这种简化难免忽略了一些重要信息,比如B性状数目虽然很少,但是出现在此类群的基部物种,可能表示此性状实际的转变是在类群之内,然而将整个类群简化为A性状,将忽视了类群之内的变化。另外可能的影响是当性状转换发生在代表类群出现之前,简化方式对分析结果的影响可能相差数个节点,对跨越长时间尺度的分析就可能产生很大的误差,与其他事件的关联性也就存在更高的不确定性。另一种简化方式是质上的简化,这涉及了某些性状在归类或者定义存在偏差,比如要对向日葵花朵的对称情况进行归类时,由于其花盘由舌状花与管状花组成,舌状花为两侧对称,管状花为辐射对称,就会产生了如何简化的问题。因此,性状归类时容易遇到这种两难的情况;不简化就不容易看出规律,简化了却又存在如何简化的争议。
(3)有些事件与物种多样化速率的变化之间可能存在时间差。以多倍化事件为例,滞后时期 (lag period)(Schranz et al.,2012)的论点认为多倍化发生后需要一段时间才会导致物种快速分化。从理论上来讲,多倍化之后,多出的重复基因由于选择压力降低可能导致更高的变异度,为物种适应环境提供了转变的基础,只是需要多长的时间才会发生实际的变化呢。假设因基因变化确实产生了新的功能和性状,然而环境没有显著差异而无法呈现优势,无法与原种竞争,这种变异或许会消失在种群之中,也可能会保存在种群内,等到适合的新环境出现后,使其与原种分开,进一步成为新种。这个假设意味着多倍化与物种多样化速率的变化很可能存在时间差。然而,由于取样上的限制,系统树上的一个节点差距可能代表了一段非常长的时间段,实际上这样的事件究竟会在多久以后才发生影响却又不好界定。假设这个时间差非常远,我们要如何判断这两者确实存在关联,而非是一厢情愿地坚持其相关性?
7.2 从演化与模型分析观点上对研究方式的改进建议
在研究方向上,我们则可以考虑以下改进方向:
(1)由于不同类群的栖地环境等差异,是否真的能有一个普遍适用于所有被子植物、跨越时间与各种环境空间皆普遍适用的影响因素?对于不同类群而言,促进其多样化速率的因素,是否应在细节上保留一定的弹性?
(2)尽量挑选能够更完整进行采样的类群为研究材料,或是至少代表类群要能更加周全地覆盖目标性状变化,同时也最好选择多倍化证据较为明显的类群。越古远的多倍化,一方面丢失较多的基因,另一方面还可能有其他近期多倍化事件重叠(Leebens-Mack et al.,2019),古多倍化痕迹将更不清晰。因此,最好选多倍化事件发生时间较近的类群,同时最好包含有基因组测序的物种,以便依照基因组的共线性等更多信息判断多倍化的过程。根据这些考量,若直接以整个被子植物为背景进行研究,很容易发生取样不足导致的结论偏差,因此可以考量从较小的类群进行分析,之后再整合各个小类群的研究结果来获取整个被子植物多样化速率变化的原因。
除此之外,还有一些研究方法与模型的问题,这些问题的改进应该能大幅地提升分析结果的精准度与结论的可靠度。这些改进有赖世界上众多科学家的共同努力,本文只能抛砖引玉,提出一些问题与建议:
(1)一般的研究很难在种水平进行完整取样,尤其是当取样范围愈广泛时,通常只会有数个代表性物种。然而,即便在属水平,其性状大多不是单态。当前的SSE(state speciation & extinction)分析(FitzJohn et al.,2009)仅适用于单态编码的数据,未来可考量朝向多态编码发展,以适应目前广泛化的研究走向。以多态编码估算祖先性状同样也是一个趋势。虽然目前有使用多态编码的估算模型,但是对于单一数据的多态单纯假设平均分布,实际情况要复杂得多。未来若能在多态编码中加上其分布比例的相关信息,更能提高估算的准确度。
(2)需要有更好的方法来检测性状转变与多样化速率变化之间的关联性。尤其是对那些出现次数较少,或仅出现一次的性状,由于样本稀少,较难有统计上的显著性,其重要性容易被忽略。
(3)目前分析所使用的系統发育树基本上是二叉化的(Guo et al.,2020; Landis et al.,2018; Leebens-Mack et al.,2019; Ren et al.,2018; Tank et al.,2015; Vanneste et al.,2014; Zhang et al.,2021),因而简化了杂交、渐渗等过程的影响,这很可能导致后续分析结果的偏差。未来的模型发展可加入这方面的考量。
(4)多倍化事件基本上分为同源多倍化和异源多倍化(Ramsey & Schemske,1998),然而目前的分析方法并不能很好地进行区分,甚至像串联重复、转座子引起的大规模重复等,是否也有促进多样化的机制,也是值得探讨的方向,仍有待将来加入研究方法中进行深入判断。
参考文献:
ALFARO ME,SANTINI F,BROCK C,et al.,2009. Nine exceptional radiations plus high turnover explain species diversity in jawed vertebrates [J]. Proc Natl Acad Sci USA,106(32):13410-13414.
ARMBRUSTER WS,2002. Can indirect selection and genetic context contribute to trait diversification? A transition-probability study of blossom-colour evolution in two genera [J]. J Evol Biol,15(3):468-486.
ARMBRUSTER WS,DEBEVEC EM,WILLSON MF,2002. Evolution of syncarpy in angiosperms:theoretical and phylogenetic analyses of the effects of carpel fusion on offspring quantity and quality [J]. J Evol Biol,15(4):657-672.
BALDWIN BG,SANDERSON MJ,1998. Age and rate of diversification of the Hawaiian silversword alliance (Compositae) [J]. Proc Natl Acad Sci USA,95(16):9402-9406.
BARKER MS,ARRIGO N,BANIAGA AE,et al.,2016. On the relative abundance of autopolyploids and allopolyploids [J]. New Phytol,210(2):391-398.
BOUCHENAK-KHELLADI Y,ONSTEIN RE,XING Y,et al.,2015. On the complexity of triggering evolutionary radiations [J]. New Phytol,207(2):313-326.
BROCHMANN C,BRYSTING AK,ALSOS IG,et al.,2004. Polyploidy in arctic plants [J]. Biol J Linn Soc,82:521-536.
BUERKI S,JOSE S,YADAV SR,et al.,2012. Contrasting biogeographic and diversification patterns in two Mediterranean-type ecosystems [J]. PLoS ONE,7(6):e39377-e39387.
CHEN ZJ,HA M,SOLTIS D,2007. Polyploidy:genome obesity and its consequences [J]. New Phytol,174(4):717-720.
CHRISTENHUSZ MJM,BYNG JW,2016. The number of known plants species in the world and itsannual increase [J]. Phytotaxa,261(3):201-217.
CITERNE H,JABBOUR F,NADOT S,et al.,2010. The evolution of floral symmetry [J]. Adv Bot Res,54:85-137.
COMES HP,TRIBSCH A,BITTKAU C,2008. Plant speciation in continental island floras as exemplified by Nigella in the Aegean Archipelago [J]. Philos Trans R Soc B:Biol Sci,363:3083-3096.
CRANE PR,LIDGARD S,1989. Angiosperm diversification and paleolatitudinal gradients in cretaceous floristic diversity [J]. Science,246:675-678.
CRISP MD,ARROYO MTK,COOK LG,et al.,2009. Phylogenetic biome conservatism on a global scale [J]. Nature,458(7239):754-756.
CUBAS P,2004. Floral zygomorphy,the recurring evolution of a successful trait [J]. Bioessays,26(11):1175-1184.
DAVIES TJ,BARRACLOUGH TG,CHASE MW,et al.,2004a. Darwin’s abominable mystery:insights from a supertree of the angiosperms [J]. Proc Natl Acad Sci USA,101(7):1904-1909.
DAVIES TJ,BARRACLOUGH TG,SAVOLAINEN V,et al.,2004b. Environmental causes for plant biodiversity gradients [J]. Philos Trans R Soc B:Biol Sci,359(1450):1645-1656.
ENDRESS PK,1982. Syncarpy and alternative modes of escaping disadvantages of apocarpy in primitive angiosperms [J]. Taxon,31(1):48-52.
ENDRESS PK,2001a. Evolution of floral symmetry [J]. Curr Opin Plant Biol,4(1):86-91.
ENDRESS PK,2001b. Origins of flower morphology [J]. J Exp Zool,291:105-115.
ENDRESS PK,2006. Angiosperm floral evolution:morphological developmental framework [J]. Adv Bot Res,44:1-61.
ENDRESS PK,2010. Flower structure and trends of evolution in eudicots and their major subclades [J]. Ann Mo Bot Gard,97:541-583.
ENDRESS PK,2011. Evolutionary diversification of the flowers in angiosperms [J]. Am J Bot,98(3):370-396.
FAWCETT JA,MAERE S,VAN DE PEER Y,2009. Plants with double genomes might have had a better chance to survive the Cretaceous-Tertiary extinction event [J]. Proc Natl Acad Sci USA,106(14):5737-5742.
FITZJOHN RG,MADDISON WP,OTTO SP,2009. Estimating trait-dependent speciation and extinction rates from incompletely resolved phylogenies [J]. Syst Biol,58(6):595-611.
FOSTER CSP,SAUQUET H,VAN DER MERWE M,et al.,2017. Evaluating the impact of genomic data and priors on bayesian estimates of the angiosperm evolutionary timescale [J]. Syst Biol,66(3):338-351.
FREELING M,2017. Picking up the ball at the K/Pg boundary:the distribution of ancient polyploidies in the plant phylogenetic tree as a spandrel of asexuality with occasional sex [J]. Plant Cell,29(2):202-206.
GENTRY AH,1988. Changes in plant community diversity and floristic composition on environmental and geographical gradients [J]. Ann Mo Bot Gard,75(1):1-34.
GOLDBERG EE,LANCASTER LT,REE RH,2011. Phylogenetic inference of reciprocal effects between geographic range evolution and diversification [J]. Syst Biol,60(4):451-465.
GRANT V,1981. Plant speciation [M]. Columbia:Columbia University Press.
GUO J,XU W,HU Y,et al.,2020. Phylotranscriptomics in Cucurbitaceae reveal multiple whole-genome duplications and key morphological and molecular innovations [J]. Mol Plant,13(8):1117-1133.
HILEMAN LC,2014. Trends in flower symmetry evolution revealed through phylogenetic and developmental genetic advances [J]. Philos Trans R Soc B:Biol Sci,369(1648):20130348. http://doi.org/10.1098/rstb.2013.0348.
HOHMANN N,WOLF EM,LYSAK MA,et al.,2015. A time-calibrated road map of Brassicaceae species radiation and evolutionary history [J]. Plant Cell,27:2770-2784.
HUANG CH,ZHANG C,LIU M,et al.,2016. Multiple polyploidization events across Asteraceae with two nested events in the early history revealed by nuclear phylogenomics [J]. Mol Biol Evol,33(11):2820-2835.
HUELSENBECK JP,NIELSEN R,BOLLBACK JP,2003. Stochastic mapping of morphological characters [J]. Syst Biol,52(2):131-158.
HUGHES C,EASTWOOD R,2006. Island radiation on a continental scale:exceptional rates of plant diversification after uplift of the Andes [J]. Proc Natl Acad Sci USA,103(27):10334-10339.
JABBOUR F,DAMERVAL C,NADOT S,2008. Evolutionary trends in the flowers of Asteridae:is polyandry an alternative to zygomorphy? [J]. Ann Bot,102(2):153-165.
JABLONSKI D,ROY K,VALENTINE JW,2006. Out of the tropics:evolutionary dynamics of the latitudinal diversity gradient [J]. Science,314:102-106.
JANSSON R,DAVIES TJ,2008. Global variation in diversification rates of flowering plants:energy vs. climate change [J]. Ecol Lett,11(2):173-183.
KAY KM,VOELCKEL C,YANG JY,et al.,2006. Floral characters and species diversification [M]//HARDER LD,BARRETT SCH. Ecology and evolution of flowers [M]. Oxford:Oxford University Press:311-325.
KERKHOFF AJ,MORIARTY PE,WEISER MD,2014. The latitudinal species richness gradient in New World woody angiosperms is consistent with the tropical conservatism hypothesis [J]. Proc Natl Acad Sci USA,111(22):8125-8130.
LAGOMARSINO LP,CONDAMINE FL,ANTONELLI A,et al.,2016. The abiotic and biotic drivers of rapid diversification in Andean bellflowers (Campanulaceae) [J]. New Phytol,210(4):1430-1442.
LANDIS JB,SOLTIS DE,LI Z,et al.,2018. Impact of whole-genome duplication events on diversification rates in angiosperms [J]. Am J Bot,105(3):348-363.
LEEBENS-MACK JH,BARKER MS,CARPENTER EJ,et al.,2019. One thousand plant transcriptomes and the phylogenomics of green plants [J]. Nature,574:679-685.
LEVIN DA,1975. Minority cytotype exclusion in local plant populations [J]. Taxon,24(1):35-43.
LEWIS PO,2001. A likelihood approach to estimating phylogeny from discrete morphological character data [J]. Syst Biol,50:913-925.
MADDISON WP,FITZJOHN RG,2015. The unsolved challenge to phylogenetic correlation tests for categorical characters [J]. Syst Biol,64(1):127-136.
MADDISON WP,MIDFORD PE,OTTO SP,2007. Estimating a binary character’s effect on speciation and extinction [J]. Syst Biol,56(5):701-710.
MAGALLN S,CASTILLO A,2009. Angiosperm diversification through time [J]. Am J Bot,96:349-365.
MAGALLON S,CRANE PR,HERENDEEN PS,1999. Phylogenetic pattern,diversity,and diversification of Eudicots [J]. Ann Mo Bot Gard,86(2):297-372.
MAGALLN S,GMEZ-ACEVEDO S,SNCHEZ-REYES LL,et al.,2015. A metacalibrated time-tree documents the early rise of flowering plant phylogenetic diversity [J]. New Phytol,207:437-453.
MAGALLON S,SANDERSON MJ,2001. Absolute diversification rates in angiosperm clades [J]. Evolution,55(9):1762-1780.
MAY MR,MOORE BR,2016. How well can we detect lineage-specific diversification-rate shifts? A simulation study of sequential AIC methods [J]. Syst Biol,65(6):1076-1084.
MAYROSE I,BARKER MS,OTTO SP,2010. Probabilistic models of chromosome number evolution and the inference of polyploidy [J]. Syst Biol,59(2):132-144.
MAYROSE I,ZHAN SH,ROTHFELS CJ,et al.,2011. Recently formed polyploid plants diversify at lower rates [J]. Science,333(6047):1257.
MERCKX VSFT,HENDRIKS KP,BEENTJES KK,et al.,2015. Evolution of endemism on a young tropical mountain [J]. Nature,524:347-350.
MEUDT HM,ROJAS-ANDRS BM,PREBBLE JM,et al.,2015. Is genome downsizing associated with diversification in polyploid lineages of Veronica? [J]. Bot J Linn Soc,178(2):243-266.
MEYERS LA,LEVIN DA,2006. On the abundance of polyploids in flowering plants [J]. Evolution,60(6):1198-1206.
MOORE BR,HHNA S,MAY MR,et al.,2016. Critically evaluating the theory and performance of Bayesian analysis of macroevolutionary mixtures [J]. Proc Natl Acad Sci USA,113(34):9569-9574.
NOWELL RW,LAUE BE,SHARP PM,et al.,2016. Comparative genomics reveals genes significantly associated with woody hosts in the plant pathogen Pseudomonas syringae [J]. Mol Plant Pathol,17(9):1409-1424.
OHNO S,1970. Evolution by gene duplication [M]. Berlin:Springer-Verlag.
OJEDA DI,VALIDO A,FERNNDEZ DE C,et al.,2016. Pollinator shifts drive petal epidermal evolution on the Macaronesian Islands bird-flowered species [J]. Biol Lett,12(4):20160022.
O′MEARA BC,SMITH SD,ARMBRUSTER WS,et al.,2016. Non-equilibrium dynamics and floral trait interactions shape extant angiosperm diversity [J]. Proc R Soc Lond,Ser B:Biol Sci,283(1830):20152304.
OSBORNE CP,FRECKLETON RP,2009. Ecological selection pressures for C4 photosynthesis in the grasses [J]. Proc R Soc Lond,Ser B:Biol Sci,276(1663):1753-1760.
OTTO SP,WHITTON J,2000. Polyploid incidence and evolution [J]. Annu Rev Genet,34(1):401-437.
PAGEL M,1994. Detecting correlated evolution on phylogenies:a general method for the comparative analysis of discrete characters [J]. Proc R Soc Lond,Ser B:Biol Sci,255(1342):37-45.
PAGEL M,1997. Inferring evolutionary processes from phylogenies [J]. Zool Scr,26(4):331-348.
PAGEL M,1999. The maximum likelihood approach to reconstructing ancestral character states of discrete characters on phylogenies [J]. Syst Biol,48(3):612-622.
PAGEL M,MEADE A,2006. Bayesian analysis of correlated evolution of discrete characters by reversible-jump Markov chain Monte Carlo [J]. Am Nat,167(6):808-825.
PENNINGTON RT,LAVIN M,SRKINEN T,et al.,2010. Contrasting plant diversification histories within the Andean biodiversity hotspot [J]. Proc Natl Acad Sci USA,107(31):13783-13787.
PIMM SL,RAVEN PH,2017. The fate of the world’s plants [J]. Trends Ecol Evol,32:317-320.
RABOSKY DL,2014. Automatic detection of key innovations,rate shifts,and diversity-dependence on phylogenetic trees [J]. PLoS ONE,9(2):e89543-e89557.
RABOSKY DL,DONNELLAN SC,GRUNDLER M,et al.,2014. Analysis and visualization of complex macroevolutionary dynamics:an example from Australian scincid lizards [J]. Syst Biol,63(4):610-627.
RABOSKY DL,GOLDBERG EE,2015. Model inadequacy and mistaken inferences of trait-dependentspeciation [J]. Syst Biol,64(2):340-355.
RABOSKY DL,LOVETTE IJ,2008. Explosive evolutionary radiations:decreasing speciation or increasing extinction through time? [J]. Evolution,62(8):1866-1875.
RABOSKY DL,MITCHELL JS,CHANG J,2017. Is BAMM flawed? Theoretical and practical concerns in the analysis of multi-rate diversification models [J]. Syst Biol,66(4):477-498.
RAMSEY J,SCHEMSKE DW,1998. Pathways,mechanisms,and rates of polyploid formation in flowering plants [J]. Ann Rev Ecol Syst,29:467-501.
RAMSEY J,SCHEMSKE DW,2002. Neopolyploidy in flowering plants [J]. Annu Rev Ecol Syst,33:589-639.
REN R,WANG H,GUO C,et al.,2018. Widespread whole genome duplications contribute to genome complexity and species diversity in angiosperms [J]. Mol Plant,11:414-428.
REYES E,MORLON H,SAUQUET H,2015. Presence in Mediterranean hotspots and floral symmetry affect speciation and extinction rates in Proteaceae [J]. New Phytol,207:401-410.
REYES E,SAUQUET H,NADOT S,2016. Perianth symmetry changed at least 199 times in angiosperm evolution [J]. Taxon,65(5):945-964.
RICKLEFS RE,2007. Estimating diversification rates from phylogenetic information [J]. Trends Ecol Evol,22(11):601-610.
ROLLAND J,CONDAMINE FL,JIGUET F,et al.,2014. Faster speciation and reduced extinction in the tropics contribute to the mammalian latitudinal diversity gradient [J]. PLoS Biol,12(1):e1001775. DOI:10.1371/journal.pbto.1001775.
SNCHEZ-RYES LL,MORLON H,MAGALLN S,2017. Uncovering higher-taxon diversification dynamics from clade age and species-richness data [J]. Syst Biol,66(3):367-378.
SANDERSON MJ,DONOGHUE MJ,1994. Shifts in diversification rate with the origin of angiosperms [J]. Science,264(5165):1590-1593.
SARGENT RD,2004. Floral symmetry affects speciation rates in angiosperms [J]. Proc R Soc Lond,Ser B:Biol Sci,271(1539):603-608.
SAUQUET H,VON BALTHAZAR M,MAGALLN S,et al.,2017. The ancestral flower of angiosperms and its early diversification [J]. Nat Commun,8:16047. DOI:10.1038/ncomms16047.
SAVOLAINEN V,ANSTETT MC,LEXER C,et al.,2006. Sympatric speciation in palms on an oceanic island [J]. Nature,441:210-213.
SCARPINO SV,LEVIN DA,MEYERS LA,2014. Polyploidformation shapes flowering plant diversity [J]. Am Nat,184:456-465.
SCHLUTER D,PRICE T,MOOERS A,et al.,1997. Likelihood of ancestor states in adaptive radiation [J]. Evolution,51(6):1699-1711.
SCHRANZ EM,MOHAMMADIN S,EDGER PP,2012. Ancient whole genome duplications,novelty and diversification:the WGD radiation lag-time model [J]. Curr Opin Plant Biol,15(2):147-153.
SCHULTZ RJ,1980. Role of polyploidy in the evolution of fishes[M]//LEWIS WH. Polyploidy:Biological Relevance. Boston,MA:Springer US:313-340.
SILVESTRO D,CASCALES-MIANA B,BACON CD,et al.,2015. Revisiting the origin and diversification of vascular plants through a comprehensive Bayesian analysis of the fossil record [J]. New Phytol,207(2):425-436.
SLOWINSKI JB,GUYER C,1993. Testing whether certain traits have caused amplified diversification:an improved method based on a model of random speciation and extinction [J]. Am Nat,142(6):1019-1024.
SMITH SA,BEAULIEU JM,STAMATAKIS A,et al.,2011. Understanding angiosperm diversification using small and large phylogenetic trees [J]. Am J Bot,98(3):404-414.
SOLTIS DE,ALBERT VA,LEEBENS-MACK J,et al.,2009. Polyploidy and angiosperm diversification [J]. Am J Bot,96(1):336-348.
STEBBINS CL JR,1950. Variation and evolution in plants [M]. London:Oxford University Press (Geoffrey Cumberlege).
STEBBINS JR GL,1938. Cytological characteristics associated with the different growth habits in the dicotyledons [J]. Am J Bot,25(3):189-198.
TANK DC,EASTMAN JM,PENNELL MW,et al.,2015. Nested radiations and the pulse of angiosperm diversification:increased diversification rates often follow whole genome duplications [J]. New Phytol,207(2):454-467.
URIBE-CONVERS S,TANK DC,2015. Shifts in diversification rates linked to biogeographic movement into new areas:an example of a recent radiation in the Andes [J]. Am J Bot,102(11):1854-1869.
VAMOSI JC,DICKINSON TA,2006. Polyploidy and diversification:a phylogenetic investigation in Rosaceae [J]. Int J Plant Sci,167(2):349-358.
VAMOSI JC,MAGALLN S,MAYROSE I,et al.,2018. Macroevolutionary patterns of flowering plant speciation and extinction [J]. Annu Rev Plant Biol,69:685-706.
VAMOSI JC,MCEWEN JR,2012. Origin,elevation,and evolutionary success of hybrids and polyploids in British Columbia,Canada [J]. Botany,91(3):182-188.
VAMOSI JC,VAMOSI SM,2011. Factors influencing diversification in angiosperms:at the crossroads of intrinsic and extrinsic traits [J]. Am J Bot,98(3):460-471.
VANNESTE K,MAERE S,VAN DE PEER Y,2014. Tangled up in two:a burst of genome duplications at the end of the Cretaceous and the consequences for plant evolution [J]. Philos Trans R Soc B:Biol Sci,369(1648):20130353.
WAGNER JR WH,1970. Biosystematics and evolutionary noise [J]. Taxon,19(2):146-151.
WEIR JT,SCHLUTER D,2007. The latitudinal gradient in recent speciation and extinction rates of birds and mammals [J]. Science,315(5818):1574-1576.
WOOD TE,TAKEBAYASHI N,BARKER MS,et al.,2009. The frequency of polyploid speciation in vascular plants [J]. Proc Natl Acad Sci USA,106(33):13875-13879.
XING Y,GANDOLFO MA,ONSTEIN RE,et al.,2016. Testing the biases in the rich Cenozoic angiosperm macrofossil record [J]. Int J Plant Sci,177(4):371-388.
ZANNE AE,TANK DC,CORNWELL WK,et al.,2014. Three keys to the radiation of angiosperms into freezing environments [J]. Nature,506:89-92.
ZHAN SH,DRORI M,GOLDBERG EE,et al.,2016. Phylogenetic evidence for cladogenetic polyploidization in land plants [J]. Am J Bot,103(7):1252-1258.
ZHANG C,HUANG CH,LIU M,et al.,2021. Phylotranscriptomic insight into Asteraceae diversity,polyploidy,and morphological innovation [J]. J Integr Plant Biol,63(7):1273-1293.
ZHAO YY,ZHANG R,JIANG KW,et al.,2021. Nuclear phylotranscriptomics/phylogenomics support numerous polyploidization events and hypotheses for the evolution of rhizobial nitrogen-fixing symbiosis in Fabaceae [J]. Mol Plant,14(5):748-773.
(責任编辑 何永艳)
