基于生物信息学对胃癌预后基因的筛选
2021-01-12李高勤牛帆苏欢李俊杰宋忠阳祁亚峰雍文兴张志明
李高勤,牛帆,苏欢,李俊杰,宋忠阳,祁亚峰,雍文兴,张志明*
(1.甘肃中医药大学,甘肃 兰州;2.甘肃中医药大学附属医院,甘肃 兰州)
0 引言
胃癌作为最常见的恶性肿瘤之一,据世界卫生组织( WHO)最新报道,在全球范围内胃癌发病率位列第5 位,死亡率位列第3 位[1]。早发现、早诊断、早治疗是提高胃癌患者生存时间的关键。随着临床诊疗方法和预后标志物的完善,胃癌患者的早期诊出率得到明显提高,但对胃癌患者预后标志物的研究仍存在不足。因此,应寻找更可靠的预后标志物,作为提高治疗效果和延长患者生存时间的靶点。基因芯片作为一种可靠的技术,经过多年的应用,能够快速检测出差异表达的基因[2]。
本研究从GEO 数据库中筛选出GSE19826、GSE54129 和GSE79973 三个同时含有肿瘤样本与正常样本的数据集。利用GEO2R 在线工具和Venn 作图软件,获得上述三个数据集中差异表达基因(DEGs)。然后利用DAVID 数据库对这些DEGs 进行分析,包括分子功能(MF)、细胞成分(CC)、生物过程(BP)与KEGG分析。接着通过STRING 在线工具建立了蛋白质相互作用(PPI)网络,然后应用MCODE(分子复合物检测)对DEGs 进行分析以确定其核心基因。然后,将这些核心DEGs 导入Kaplan-Meier Plotter 在线生存分析数据库,以获得显著的预后信息(P<0.05)。采用基因表达谱交互分析(Gene Expression profiling interactive analysis GEPIA)对胃癌组织与正常胃组织间的DEGs 表达再次进行检测(P<0.05)。最后,产生四个DEGs(COL1A2,BGN,THBS2,COL1A1)。总之,本研究的生物信息学研究为胃癌患者提供了一些有用的生物预后标记物,可以作为胃癌患者的有效靶点。
1 资料与方法
1.1 资料来源
NCBI-GEO 是一个免费的基因芯片/ 基因图谱公共数据库,我们获得了GSE19826、GSE54129 和GSE79973 在胃癌和正常胃组织中的基因表达谱。GSE19826、GSE54129 和GSE79973 的基因芯片数据均基于GPL570 平台([HG-U133_Plus_2]Affymetrix Human Genome U133 Plus 2.0 Array),分别包括12 个正常组织和12 个胃癌组织、21 个正常组织和111个胃癌组织、10 个正常组织和10 个胃癌组织。
1.2 DEGs 的数据处理
用GEO2R 在线工具[3]按|logFC|>2 与P值<0.05 鉴定胃癌标本与正常标本之间的DEGs。然后,用VENN 软件在线分析原始数据,找出三个数据集中的共同基因。logFC<0 的DEGS 为下调基因,logFC>0 的DEGS 为上调基因。
1.3 GO 功能分析与KEGG 通路富集分析
基因本体分析(GO)是定义基因及其RNA 或蛋白质产物以识别高通量转录组或基因组数据的独特生物属性的常用方法[4]。KEGG 是处理基因组、疾病、生物途径、药物和化学材料的在线数据库[5]。David 是一个旨在识别大量基因或蛋白质的功能的在线生物信息学工具[6]。我们可以使用David 可视化显示BP、MF、CC 和通路的DEGS 富集(P<0.0 5)。
1.4 PPI 网络与模块分析
本研究通过在线工具STRING(用于检索基因相互作用的搜索工具)[7]来绘制PPI 网络。然后,应用Cytoscape[8]中的STRING APP 来检索这些DEG 之间的潜在相关性(最大交互作用数=0 和置信度分数≥为0.4)。此外,Cytoscape 中的MCODE 应用程序用于构建PPI 网络的模块(degree cutoff=2,max. Depth=100, k-core=2, and node score cutoff=0.2)。
1.5 核心基因的生存分析
Kaplan Meier-Plotter[9]是一个基于EGA、TCGA 和GEO数据库来评估基因对生存的影响的网站工具。为了验证这些设计,本研究应用GEPIA 网站对Kaplan Meier-Plotte 筛选的基因作二次验证。
2 结果
2.1 胃癌中DEGs 的鉴别
本研究共有133 例胃癌组织和43 例正常组织。通过GEO2R在线工具,我们分别从GSE19826、GSE54129 和GSE79973 中提取80、768 和415DEGs。然后,利用Venn 图软件对三个数据集中的DEG 取交集。结果表明,共检测到22 个DEGs,包括8 个下调基因(logFC<0;)和14 个上调基因(logFC>0;),下调基因有RDH12、AKR7A3、MFSD4A、DPCR1、VSIG1、MUC5AC、PSAPL1、RASSF6;上调基因有SULF1、FAP、INHBA、PDLIM7、SPP1、COL1A1、COL10A1、SFRP4、THBS2、BGN、COL1A2、MFAP2、ADAMTS2、COL8A1。

图1 三组数据中有14个DEGs上调(logFC>0),8 个DEGs下调(logFC<0)
2.2 DEGs 基因在胃癌中的GO 与KEGG 通路分析
DAVID 软件对22 个DEGs 进行了分析,GO 分析结果表明:对于生物过程(BP),上调的DEGs 在胶原纤维组织、皮肤形态发生、蛋白质异三聚、细胞粘附、内皮细胞分化等方面都有显著的富集作用;细胞组分(CC)主要富集在蛋白质的细胞外基质、胶原三聚体、细胞外间隙、I 型胶原三聚体、细胞表面等方面;分子功能(MF)主要集中在细胞外基质结构组成方面,而下调基因无显著性富集(表1)。
KEGG 分析结果如表2 所示,结果显示,上调的DEGs 在ECM 受体作用、局灶性粘连、PI3K-Akt 信号通路、蛋白质消化吸收等方面尤为丰富,差异有统计学意义(P<0.05),而下调的DEGs 在信号通路中无明显富集。
2.3 蛋白质相互作用网络(PPI)构建
共有16 个DEGs 被导入DEGs-PPI 网络复合体,其中包括16个节点和29 条边,包括3 个下调基因和13 个上调基因(图2a)。然后我们应用MCODE 插件进一步分析(degree cutoff = 2, node score cutoff = 0.2, k-core = 2, and max. Depth = 100),结果显示在16 个节点中鉴定出5 个中心节点,这些节点都是上调基因(图2b)。
2.4 Kaplan-Meier Plotter 和GEPIA 分析核心基因预后
利用Kaplan-Meier Plotter(http://kmplot.com/analysis)鉴定5个核心基因存活数据。结果发现,4 个基因的存活率明显下降,而1 个基因的存活率则无显著性差异(P>0.05,图3)。然后,用GEPIA 方法检测癌细胞与正常人之间4 个基因的表达水平。结果显示,与正常胃粘膜样本相比,COL1A2、BGN、THBS2、COL1A1 等4 个基因在胃癌样本高表达(P<0.05,图4)。
3 讨论
本 研 究 以GSE19826、GSE54129 和GSE79973 三 个 数 据 为基础,采用生物信息学方法,对胃癌的预后进行了研究。本研究共收集了133 例胃癌标本和43 例正常胃标本。通过GEO2R和Venn 软件,我们发现共有22 个差异表达的DEG(|LogFC|>2,并调整P 值<0.05),包括14 个上调基因(logFC>0)和8 个下调基因(logFC<0)。然后,利用DAVID 方法对基因本体和途径富集分析表明:对于生物过程(BP),上调的DEGs 在胶原纤维组织、皮肤形态发生、蛋白质异三聚、细胞粘附、内皮细胞分化等方面都有显著的富集作用;细胞组分(CC)主要富集在蛋白质的细胞外基质、胶原三聚体、细胞外间隙、I 型胶原三聚体、细胞表面等方面;分子功能(MF)主要集中在细胞外基质结构组成方面,而下调基因无显著性富集;在通路分析中,ECM 受体作用、局灶性粘连、PI3K-Akt 信号通路、蛋白质消化吸收上调的DEGs 尤其丰富,而下调的DEGs 则没有显著富集的信号通路(P>0.05)。其次,利用STRING 在线数据库和Cytoscape 软件构建了16 个节点、29 条边的DEGs-PPI 网络复合体。然后,通过MCODE 分析从PPI 网络复合体中筛选出5 个核心的上调基因。此外,通过Kaplan-Meier Plotter 分析,我们发现5个基因中有4 个存活率明显下降。而通过GEPIA 在线分析发现这4 个基因在正常人与胃癌患者中差异表达(P<0.05)。最后得出结论,这4 个基因可作为改善胃癌患者预后的新的有效靶点。

表1 胃癌差异表达基因的GO 分析

表2 胃癌差异表达基因的KEGG 通路分析

图3 Kaplan-Meier Plotter 在线分析显示COL1A1、BGN、COL1A2、THBS2 的生存率明显下降(P<0.05)。

图4 GEPIA 在线分析显示上述4 个基因在胃癌患者高度表达(*表示P<0.05)。红色表示肿瘤组织,灰色表示正常组织。
细胞外基质(ECM)是一个复杂的非细胞3D 网络,由胶原、蛋白多糖/糖胺聚糖、弹性蛋白、纤维连接蛋白、层粘连蛋白和其他几种糖蛋白组成[10]。细胞外基质的主要成分是胶原[11],I 型胶原存在于大多数结缔组织和胚胎组织中[12]。通常,I 型胶原由I 型胶原α1 链(COL1A1)和一条I 型胶原α2 链(COL1A2)组成[13-14]。I 型胶原蛋白α1 链(collagen type I alpha 1chain,COL1A1),由COL1A1 基因编码,它可以构成胶原纤维,并且参与细胞增殖、浸润、转移和血管生成,与多种类型肿瘤有关[15-17]。有研究表明I 型胶原蛋白形成的交叉网状结构能够支持卵巢癌细胞的生长[18],I 型胶原蛋白基因缺乏可促进乳腺癌细胞转移[19],而在脑肿瘤中I 型胶原蛋白是肿瘤微环境的重要组成部分[20]。有报道称,COL1A2与胰腺癌[21]、颅内动脉瘤关系密切[22],COL1A2 基因的突变与成骨不全的发生具有相关性[23]。
血小板反应蛋白 2 (thrombospondin-2,THBS2) 属于凝血酶敏感蛋白(THBS/TSP)家族,由5 种钙结合的基质细胞糖蛋白THBS1-THBS5 组成。根据寡聚状态和结构域结构,它们可分为三聚体蛋白和五聚体蛋白两个亚类。THBS1 和THBS2 是三聚体蛋白,而其他的是五聚体蛋白[24]。THBS2 与各种细胞表面受体、生长因子、细胞因子和蛋白酶相互作用,调节细胞-基质粘附、运动、趋化、伤口愈合、血管抑制等[25]。它主要通过抑制血管生成和负调控MMP-2 和MMP-9 参与肿瘤的发生[26]。在前列腺癌组织和细胞系中观察到THBS2基因下调[27]。在Chijiwa 等人的研究中,肺腺癌的THBS2 转录水平反而显著高于正常肺组织(P<0.0001)[28]。
双链蛋白聚糖(biglycan,BGN)是一种细胞外基质(extracellular matrix ECM)蛋白,属于富含亮氨酸的小蛋白聚糖家族[29]。BGN在人体几乎每个器官中都有发现,但在每个器官中分布并不均匀。BGN 在细胞表面表达,有时在一系列特殊细胞类型的细胞外基质中表达[30]。最近的研究表明,与邻近的正常组织相比,BGN 在肿瘤组织中的表达显著增高,包括子宫内膜癌、胰腺癌、结肠癌和肿瘤血管以及食管鳞状细胞癌[31-35]。BGN 在肿瘤组织中的异常表达提示BGN 在肿瘤的发生、发展中具有重要意义。
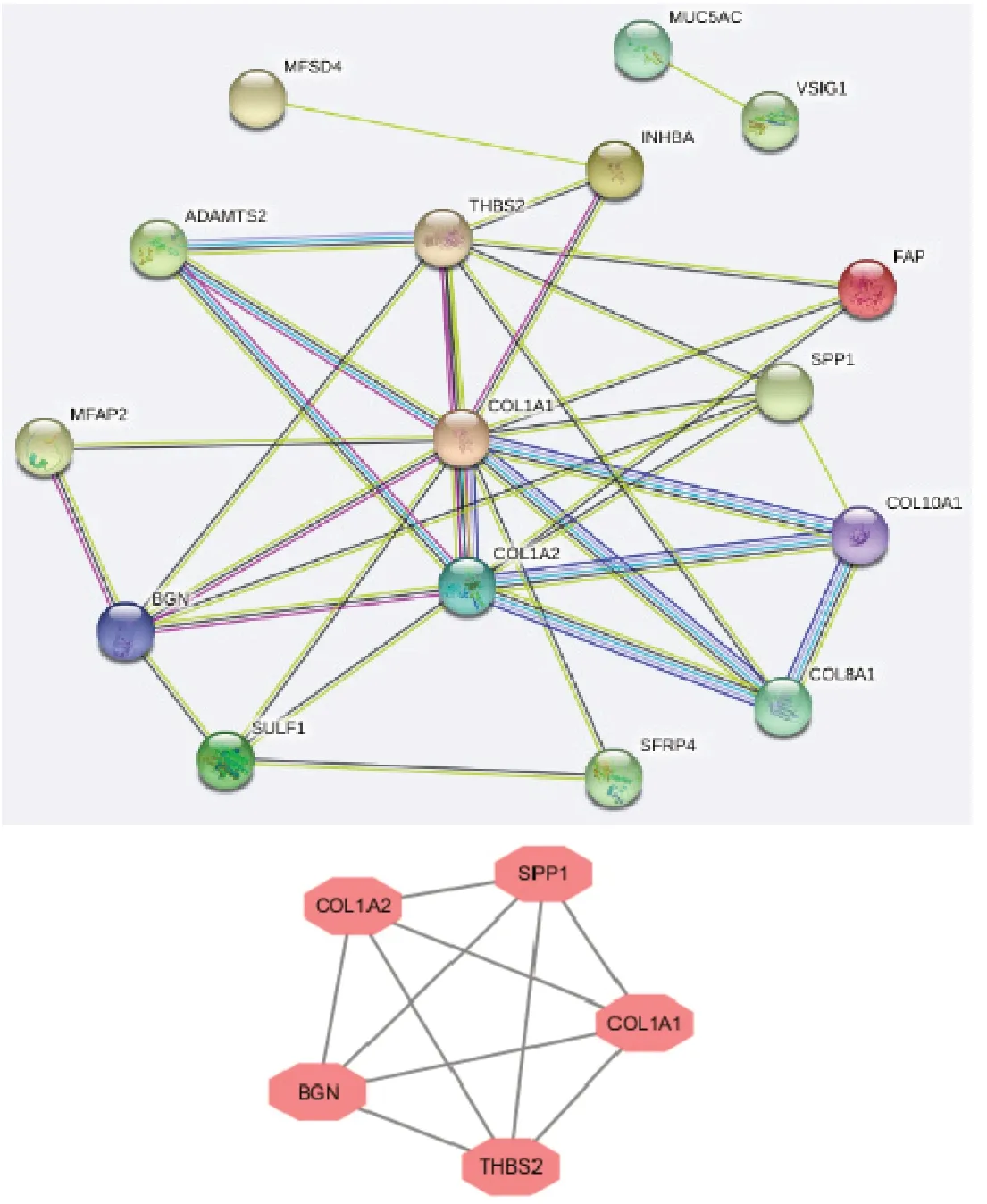
图2 a.PPI 网络中共有16 个DEGs;b.MCODE 插件获得5 个核心基因
4 结论
综上所述,本研究通过生物信息学分析研究基于三个微阵列数据集,在胃癌组织和正常胃组织之间鉴定出四个DEGs(COL1A2,BGN,THBS2,COL1A1)。这些差异基因在胃癌组织高度表达,且与胃癌患者不良预后具有密切关系,因此表明,这4 个基因可能在胃癌的发生发展中起关键作用。这些数据可能会为研究胃癌的潜在生物标志物和生物学机制提供一些有用的信息和方向。
