多组学数据分析中关联网络融合ANF算法及应用研究*
2021-01-09哈尔滨医科大学卫生统计学教研室150081徐臻旖
哈尔滨医科大学卫生统计学教研室(150081) 徐臻旖 王 策 侯 艳 李 康
【提 要】 目的 引入关联网络融合(affinity network fusion,ANF)方法对多组学数据进行整合分析,并应用于肿瘤分子分型研究。方法 模拟产生两组学数据,改变总体差异大小等情况比较多种多组学整合方法的效果。实例分析中目标人群选择TCGA数据库中对铂类药物敏感并拥有mRNA和甲基化两个组学的卵巢癌患者,目标基因是TCGA数据库和ImmPort数据库中共有基因,目标甲基化位点是目标基因对应的所有甲基化位点。使用ANF、SNF、K-means、系统聚类和iCluster五种方法比较聚类效果。结果 模拟实验提示存在总体差异的两亚型间差异较小时ANF方法的效果明显优于其他方法。实例分析结果表明,通过ANF方法进行多组学数据整合得到的分子分型较单组学得到的分子分型有更好的生物学意义且多组学聚类效果优于其他方法。结论 ANF方法可以应用于多组学数据整合分析,具有实际应用意义。
多组学指基因组、转录组、蛋白质组及代谢组等,利用多组学进行整合分析能提供更多的信息。传统的分析方法是对各组学分别进行分析,然后再根据生物学知识对其进行解释。这种分析方法的不足之处在于不能充分利用数据提供的信息,从根本上建立各组学之间的关系。根据不同目的可将多组学整合方法分为不同类型,如基于奇异值分解发现组学间调控关系的JIVE(joint and individual variation explained)方法[1],基于联合潜在变量模型的iCluster方法以及基于样本间网络结构得到分子亚型的相似网络融合(similarity network fusion,SNF)方法等[2-3]。本文引进关联网络融合法(affinity network fusion,ANF),与SNF方法相比计算过程更简洁,可利用先验信息对不同组学进行加权。ANF将不同组学的数据矩阵进行转化再整合分析,为多组学研究提供有价值的信息[4]。
原理和方法
ANF是一种利用多组学数据构建矩阵的方法,即通过随机游走的方式将分别构建的组学样本间关联矩阵进一步整合[4],对最终得到的包含多组学信息的关联矩阵进行谱聚类分析。
具体步骤如下:
1.构建关联矩阵
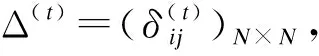
(1)
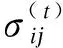

(2)
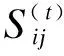
(3)
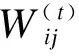
2.不同组学关联矩阵融合
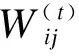
(4)
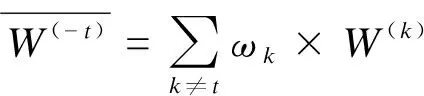
(5)
对得到的关联矩阵W进行多分类谱聚类分析[5]。多分类谱聚类由Stella等人提出,与标准谱聚类不同在于该方法使用奇异值分解进行计算,聚类数目通过启发式特征间隙法(eigengap heuristic)进行确定,假设求得m个特征值λ1,…,λm,存在i∈[1,m],使得λi与λi+1的差值较大,则认为聚类类别数为i类。
3.聚类效果评价
聚类效果的衡量指标通常分为两大类:第一类为有监督方法,已知真实标签,用一定的度量评判聚类结果与真实标签的符合程度,例如标准化互信息(NMI)[0,1],NMI的值越接近1表明得到聚类结果与真实情况越吻合;第二类为无监督方法,根据是否满足类内聚集程度高与类间离散程度大对聚类效果进行评价,例如轮廓系数(silhouette coefficient),其值越接近1表明聚类越合理,越接近-1表示可能存在错分情况;还可通过比较生存分析的预后情况评价聚类效果。
模拟实验
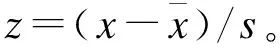
可以看到,随着差异变量比例和不同亚组差异变量均值差异增加,NMI逐渐增加,ANF在不同亚型总体差异较小时效果优于其他四种方法,如均数差值为4,变量数为200,差异变量为15%时,ANF的NMI值为0.750,明显大于其他聚类方法的NMI值。当不同亚型总体差异逐渐增加达到一定程度时,除系统聚类外以上方法的聚类效果均较好。
实例分析
本研究对使用铂类药物敏感的卵巢癌患者进行免疫亚型分析。选择TCGA数据库中同时拥有mRNA、甲基化数据以及患者临床信息的卵巢癌样本,使用ImmPort数据库确定与免疫相关的mRNA。化疗敏感患者定义为使用大于等于六个周期的铂类药物且化疗后无进展生存期为六个月以上的患者;将化疗耐药患者定义为第一次使用铂类药物化疗耐药以及在治疗周期完成后六个月内复发的患者[7]。
对数据进行预处理,将样本ID匹配,要求样本同时具有mRNA数据和甲基化数据,选择TCGA与ImmPort数据库中共有的mRNA,同时确定mRNA所对应的甲基化位点。删除重复变量,将mRNA中超过70%为0的变量剔除,将甲基化位点中超过70%缺失的变量剔除,并用最近邻KNN算法填补剩余缺失值。最终铂类药物化疗敏感人群136人,1211个mRNA以及2140个甲基化位点纳入本次研究。对mRNA数据的表达值进行log2(count+1)转化,由于不同组学存在异质性问题,将转化后的mRNA数据以及甲基化数据进行Z-score标准化处理。
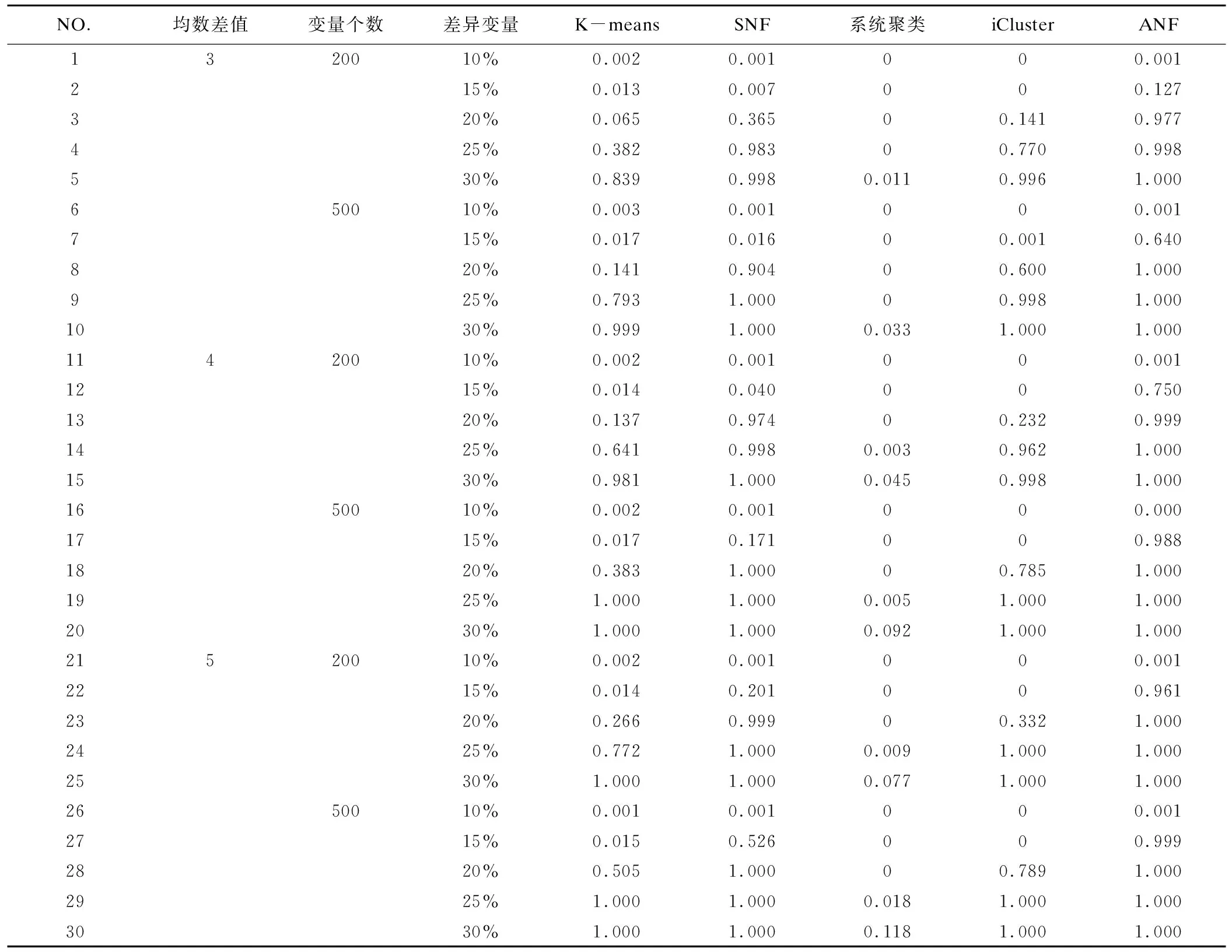
表1 基于NMI评价指标对五种无监督聚类方法结果比较
对预处理得到的数据分别使用ANF、SNF、K-means、iCluster以及系统聚类方法分析,提示聚类分为两类,因此将卵巢癌铂类药物敏感患者分为两个免疫亚型即免疫I型和免疫II型,通过log-rank检验比较亚型间的生存差异。为增加方法间的可比性,对于SNF和ANF两种方法,分别选择log-rank检验得到P值最小时的参数k,其中ANF中k分别选择30和15。结果参见表2。

表2 无监督聚类得到免疫分子分型的log-rank生存分析结果比较
结果显示对ANF得到的无监督聚类标签进行log-rank检验P<0.001(HR=2.900,95%CI(1.809,4.648)),P值小于其他四个方法的P值,其中通过系统聚类进行生存分析模型不收敛,同时多组学整合无监督聚类结果优于单组学无监督聚类结果。图1表示利用以上方法进行多组学整合得到的Kaplan-Meier生存曲线。
图1-A显示利用ANF方法得到的免疫分型I型和II型的生存曲线,发现I型预后好于II型预后。为了进一步证明多组学聚类效果优于单组学聚类效果,使用ANF方法得到的mRNA、甲基化以及mRNA和甲基化多组学整合的聚类结果计算轮廓系数,参见图2,A~C图中纵轴表示每个样本及其所在的亚组,横轴表示每个样本的轮廓系数值,最终取平均得到该组学的轮廓系数值,值越接近1表示聚类效果越合理。结果显示,通过多组学聚类的轮廓系数0.42高于单组学聚类轮廓系数(mRNA:0.32;甲基化:0.3),进一步说明多组学在无监督聚类亚型分析中的价值。

图1 多组学整合的化疗敏感患者免疫分型生存曲线
为了控制其他因素对生存的影响,将年龄(≥60岁和<60岁),化疗缓解情况(完全缓解和非完全缓解),肿瘤分化程度(高分化G1,中分化G2,低分化G3,未分化G4),肿瘤临床分期(I期,II期,III期,IV期)列为协变量,Cox比例风险回归模型结果见表3。
结果表明,校正上述协变量后,I型预后仍好于II型预后。可以看出ANF得到的免疫亚型是卵巢癌化疗敏感人群的独立预后因素。
为了进一步确定I型和II型的差异变量,本研究使用LASSO算法进行组间差异变量筛选,得到43个差异mRNA以及39个差异甲基化位点,将得到的差异变量在不同样本间表达量尺度归一化,使用R包pheatmap画出热图,表示如图3。
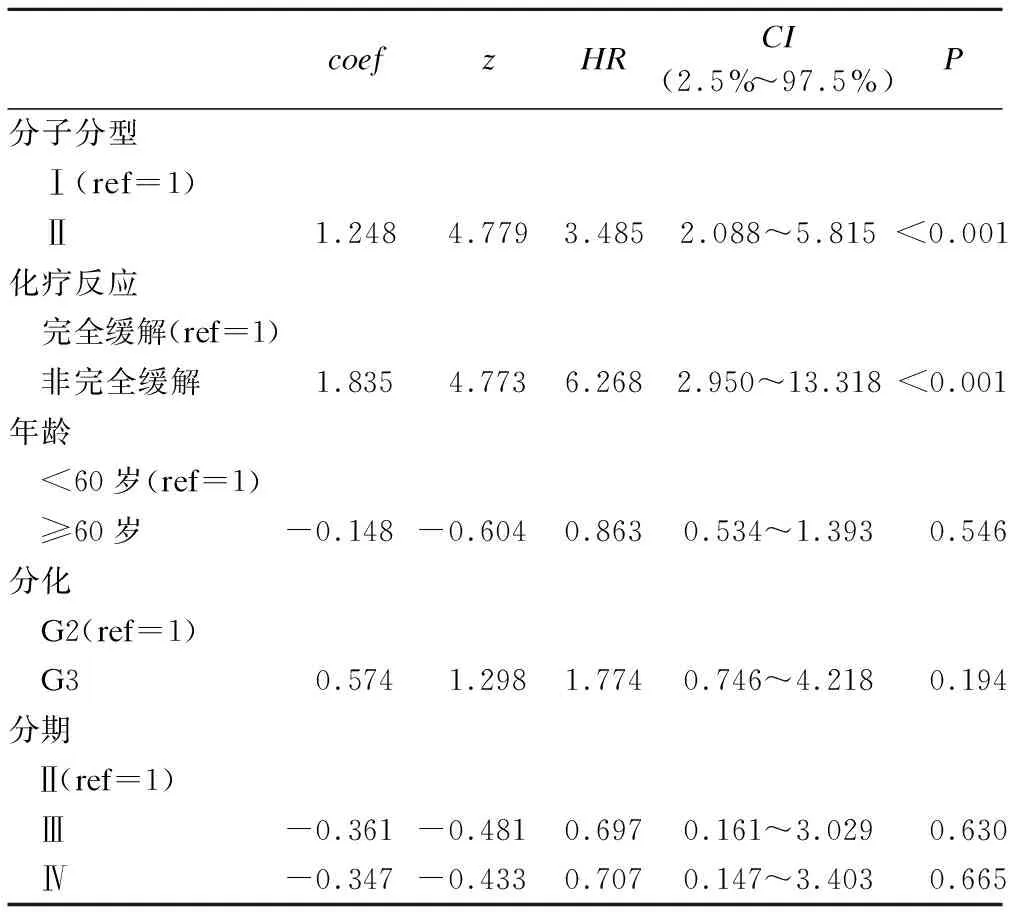
表3 调整协变量后的Cox比例风险回归模型结果
根据热图可以看出不同亚型的组间差别。据文献报道,BMPR1B在乳腺癌中高表达与较差的生存预后相关[8]。PIK3CD在其他癌症如结肠癌组织和CRC细胞系中过表达,是结肠癌患者总体生存的独立预测因子,并且PIK3CD与CILA4和原发性免疫缺陷有关,有理由认为BMPR1B和PIK3CD的高表达可能与卵巢癌化疗敏感的患者生存预后较差相关[9-10]。甲基化位点中cg16097079,cg17475918分别对应HLA-C、HLA-E,属于主要组织相容性复合体中(MHC)的I类分子,其表达的丧失提供了关键的免疫逃避策略[11]。巨噬细胞迁移抑制因子(MIF)是一种炎性介质,可促进炎症细胞因子的产生,并具有趋化特性,可招募巨噬细胞和T细胞,目前有研究探讨可将MIF作为靶标对卵巢癌以及泌尿生殖系统癌症患者进行治疗,本研究中在甲基化位点cg20377673所对应的基因以及mRNA中均发现MIF表达在得到的免疫亚型间存在差异[12-14]。因此,针对铂类药物敏感的卵巢癌患者的不同免疫亚型,使用特定的免疫靶点治疗可能增加患者的预后生存。

图3 不同分子亚型中差异基因表达的热图
讨 论
与传统的使用单组学数据聚类发现疾病亚型相比,包含多种组学信息可以弥补单组学的局限性,生物学解释更合理,有效提高患者预后效果。利用样本多组学数据进行网络构建的ANF算法在原始SNF算法的基础上简化了迭代过程,使计算上更为简便且效果良好,提高运算效率。通过矩阵变换使其在解释上更为合理,同时可对不同组学信息进行加权,更符合生物学意义。
模拟实验结果提示通过改变差异变量的均数差值以及差异变量百分比,当亚型总体差异较小时,ANF方法聚类效果明显优于其他方法。均数差异越大,差异变量百分比越高时,NMI越高,聚类效果越好,当不同亚型总体差异足够大时,除系统聚类外其他几种方法效果趋于一致。
由于铂类药物敏感的卵巢癌患者总生存较低,有必要对这类患者进行进一步治疗提高患者生存。本次研究发现对铂类药物敏感的卵巢癌患者的新的免疫亚型,且该免疫亚型中通过LASSO筛选得到的差异mRNA和差异甲基化位点中,有目前报道的免疫靶点MIF等,因此针对不同免疫亚型,选择免疫靶点进行治疗可能是铂类药物化疗后的又一治疗策略。
由于收集多组学数据较为困难,目前大多数利用多组学得到的分子亚型在验证上难度较大。对于无监督聚类,生存分析的结局指标常用来对获得亚型进行评价分析。方法中参数的选择可能会对假设检验中的P值产生较大影响,因此这并不能作为唯一的评价指标,在未知组学权重的情况下,权重的设置通常较难选择,实际中可以根据已有先验知识进行设置。
