水稻OsFRK1与OsFRK2基因CRISPR敲除突变体的构建
2021-01-04张宗飞刘媛媛欧阳解秀李绍波王鑫
张宗飞,刘媛媛,欧阳解秀,李绍波,王鑫
水稻与基因CRISPR敲除突变体的构建
张宗飞,刘媛媛,欧阳解秀,李绍波,王鑫*
(南昌大学生命科学学院,江西 南昌 330031)
为揭示水稻果糖激酶(OsFRK)家族基因的生物学功能,采用CRISPR/Cas9技术,成功创建了2个已鉴定了的水稻OsFRK家族基因的敲除突变体;获得28株的T0代转基因植株,其转基因阳性率、突变率与纯合突变率分别为100%、46.43%和10.71%;获得14株的T0代转基因植株,其转基因阳性率、突变率与纯合突变率分别为92.86%、92.86%和21.43%;T0代敲除纯合突变体中目的蛋白质的功能结构域均发生不同程度的破坏。
水稻;OsFRK家族基因;CRISPR/Cas9技术;载体构建;敲除突变体
果糖激酶在植物的生长发育过程以及逆境适应中具有重要的功能,属于pfkB碳水化合物激酶家族中的一类亚家族[1-2]。该类酶具有高度保守的ATP结合结构域和果糖底物结合结构域[3-4]。在植物中,对番茄的果糖激酶基因的功能研究比较深入,KANAYAMA等[5]从番茄中克隆到了4个果糖激酶基因、、和,这4个同源基因位于不同的染色体上,在番茄生长发育过程中发挥着不同的作用。基因下调表达,突变体的花期延迟,说明基因与花期的调控有关[6];基因在花粉发育的后期和花粉萌发期间在花药中特异性表达,表明这种酶可能是参与花粉管发育过程中所必需的碳水化合物[7]。研究发现,拟南芥家族基因对种子油的积累与维管的发育非常重要[8]。基因在玉米响应短期盐胁迫过程中上调表达[9];在甜菜()的根中,检测到伤口应激反应中FRK活性增加[10]。说明FRK在维持植物正常的生长发育以及逆境适应过程中都具有重要的功能。
水稻中,目前已鉴定了2个家族基因,分别为和[11]。有研究结果表明,在无氧条件下,水稻幼苗中的基因表达上调,基因表达下调[12]。GUGLIELMINETTI等[13]研究发现,乙醇处理后水稻基因在幼苗的胚与胚芽鞘中的表达上调,基因下调表达,这些结果暗示了和很有可能参与了水稻响应氧胁迫的作用过程,但还需要进一步证实。
本研究拟利用成熟的CRISPR/Cas9基因编辑技术[14],构建水稻和的敲除表达载体,利用根癌农杆菌介导的水稻遗传转化法、靶点序列的PCR扩增与测序分析等方法创建和的CRISPR敲除表达突变体,为深入研究和的生物学功能提供基础。
1 材料与方法
1.1 材料与试剂
粳稻‘中花11’由江西省分子生物学与基因工程重点实验室提供。主要试剂有KOD-Plus (TOYOBO)、酶(TransGen)、限制性内切酶Ⅰ(NEB)、T4DNA ligase (NEB)、超薄DNA产物纯化试剂盒(TIANGEN)和质粒小提中量试剂盒(TIANGEN)等。
1.2 实验菌株与载体
大肠埃希菌DH5α和根癌农杆菌EHA105购于上海唯地生物技术有限公司;CRISPR/Cas9载体构建的pYLCRISPR-Cas9Pubi-H(MH)及sgRNA表达盒构建所需的启动子载体pYLsgRNA-OsU6a和pYLsgRNA-OsU6b由华南农业大学刘耀光惠赠。
1.3 CRISPR/Cas9靶点设计
在Phytozome网站(https://phytozome.jgi.doe. gov/pz/portal.html)分别下载水稻(LOC_ Os01g66940)和(LOC_Os08g02120)基因组序列、mRNA序列和氨基酸序列;随后在CRISPR-P网站(http://crispr.hzau.edu.cn/CRISPR2/)上,以NGG为PAM结构,分别在目的基因的第1个外显子上设计特异性的pYLCRISPR/Cas9靶点;并利用SMART在线网站(http://smart.embl-heidelberg.de/ smart/batch.pl?tdsourcetag=s_pctim_aiomsg)对这2个家族基因的蛋白保守结构域进行分析;进而在CRISPR-GE网站(http://skl.scau.edu.cn/)对已设计出的靶点进行脱靶预测分析[15],挑选出编辑效率较高、脱靶可能性较低且位于蛋白保守结构域序列上的靶点,并根据特异性靶点的特性设计靶点接头引物。
1.4 CRISPR/Cas9 敲除表达载体的构建
参考MA等[14]的方法构建CRISPR/Cas9敲除表达载体。先进行靶点接头的制备,再采用Ⅰ对sgRNA 启动子载体进行酶切,其中选择pYLsgRNA-OsU6b启动子载体,选择pYLsgRNA-OsU6a和pYLsgRNA-OsU6b 2个靶点,进行第1轮PCR。各取1 μL第1轮PCR产物,用ddH2O稀释10倍,作为第2轮PCR的模板。采用30 μL体系进行sgRNA表达盒的构建,加入0.5 μL第2轮PCR引物(最终浓度0.167 μmol/L),使用适量的KOD-Plus进行巢式PCR。程序设置为20个循环:95 ℃预变性3 min;95 ℃变性10 s,58 ℃退火15 s,68 ℃延伸20 s。将第1轮与第2轮PCR产物进行电泳鉴定,成功之后再把包含特异性靶点的sgRNA 表达盒的第2轮PCR产物进行纯化回收,成功构建sgRNA 表达盒。采用Gold Gate cloning方法[16],将其与pYLCRISPR/Cas9骨架载体同时进行酶切-酶连;取5 μL产物转化大肠埃希菌DH5α感受态细胞,在含卡那霉素的LB培养基上长出菌落后,进行菌落PCR和测序验证;最后再利用质粒小提中量试剂盒提取相应的CRISPR/Cas9质粒。
1.5 水稻组织培养与遗传转化
采用冻融法[17],将构建好的载体转化至EHA105感受态细胞中,挑取阳性菌加入含卡那霉素和利福平抗生素的液体LB培养基中进行培养。参考前人建立的水稻遗传转化体系[18],完成敲除载体的遗传转化。首先,利用成熟的粳稻‘中花11’种子经诱导培养基培养,获得结构致密、呈淡黄色的愈伤组织;挑选硬实的胚性愈伤组织于预培养基中,预培养3 d后进行农杆菌浸染转化;将浸染处理后的愈伤组织接种到筛选培养基(9 cm方皿)中,培养约1个月;将新长出的抗性愈伤组织移至分化培养基中,分化处理约1个月后长出幼苗;将幼苗去根后移至生根培养基中培养10 d左右,获得根系发达的转基因幼苗,经过练苗处理后,移至大田中正常生长。
1.6 水稻叶片基因组DNA的提取
采用CTAB法[19]提取水稻T0代材料的叶片基因组DNA。T0代幼苗移栽大田后15 d,每棵植株剪取0.2 g叶片于2 mL离心管中(加有直径0.4 cm的不锈钢珠),加入800 μL 的CTAB 提取液(含β-巯基乙醇),置于DNA-extractor (高通量组织球磨仪)上,打碎3 min,重复2 次;65 ℃水浴处理30 min;采用氯仿、异戊醇(氯仿与异戊醇的体积比为24∶1)溶液进行抽提,随后利用无水乙醇进行DNA沉淀;获得DNA沉淀后,再用ddH2O溶解,最后采用超微量分光光度计(Thermo Nanodrop 2000)对DNA溶液进行检测。
1.7 敲除突变体材料的鉴定
因CRISPR/Cas9 载体构建的骨架载体MH的T-DNA插入区包含有潮霉素抗性基因(HPT)序列,通过Addgene质粒数据库(http://www.addgene.org/)获取MH载体的序列,再将序列导入SnapGene软件中自动识别出载体的HPT序列,针对该序列设计PCR引物(表1)并验证,以确定敲除载体的T-DNA区是否插入到水稻基因组中。在靶点上、下游各200 bp区段附近设计特异性扩增引物,进行PCR扩增产物测序验证[20],其中只需设计1对引物就可将双靶点序列扩增在内,对扩增产物进行测序,测序结果采用Geneious软件进行比对分析,以此鉴定突变体的靶点序列突变情况。采用Geneious软件进行突变体的氨基酸序列分析,先将突变后的CDS序列导入Geneious软件中,进行翻译,即可看到氨基酸序列的变化情况,再与粳稻‘中花11’编码蛋白质保守结构域的序列进行对比,可知保守结构域是否遭到破坏。
2 结果与分析
2.1 OsFRK1和OsFRK2敲除靶点的设计与获取
在Phytozome数据库中下载和的基因序列,利用CRISPR-P和CRISPR-GE在线网站设计最佳的靶点(图1),在基因的第1个外显子上设计了1个敲除靶点,由OsU6b启动;在基因的第1个外显子上设计了2个敲除靶点,分别由OsU6a和OsU6b启动。最后针对以上特异性靶点设计相应的靶点接头引物(表1)。

图1 OsFRK1和OsFRK2的CRISPR/Cas9敲除靶点序列与位置

表1 本研究所用的引物

表1(续)
2.2 OsFRK1和OsFRK2敲除表达载体的构建
参考MA等[14]的方法,首先成功构建了sgRNA表达盒,其2轮巢式PCR产物电泳结果(图2-A、图2-B)显示,第2轮PCR产物OsFRK1-OsU6b- gRNA、OsFRK2-OsU6a-gRNA、OsFRK2-OsU6b- gRNA电泳条带大小与预期的基本相符,分别为485 bp、599 bp和485 bp;随后把纯化回收的靶标sgRNA表达盒与pYLCRISPR/Cass9骨架载体同时进行酶切-酶连反应;产物转化大肠埃希菌后,经菌落PCR鉴定,获得阳性单菌落(图2-C、图2-D),其中图2-C中的泳道2~4为敲除表达载体转化后的阳性单菌落,泳道1为假阳性;图2-D中除泳道10为假阳性单菌落外,其余的皆为敲除表达载体转化后的阳性单菌落。分别挑取3个不同的阳性单菌落进行测序和NCBI的Blastn比对分析,发现预期的特异性靶点序列已成功装载,由此确定成功构建了和的CRISPR敲除表达载体。

M DNA Marker;H2O 空白对照;MH pYLCRISPR/Cas9-MH质粒。A 泳道1、2为OsFRK1-OsU6b第1轮扩增产物,泳道3为OsFRK1-OsU6b第2轮扩增产物;B 泳道4、5为OsFRK2-OsU6a第1轮扩增产物,泳道6、7为OsFRK2-OsU6b第1轮扩增产物,泳道8、9为OsFRK2-OsU6b第2轮扩增产物;C 泳道1~5为OsFRK1 基因敲除单菌落;D 泳道6~13为OsFRK2基因敲除单菌落。
2.3 水稻遗传转化及转基因植株的获得
成功获得CRISPR/Cas9敲除表达载体后,利用冻融法将其转化至根癌农杆菌(EHA105)感受态细胞中,经农杆菌介导法转化水稻愈伤组织,获得T0代转基因水稻植株(图3)。最终获得28株基因的T0代转基因植株和14株基因的T0代转基因植株。这些转基因植株移至大田种植后的存活率均为100%。

A 水稻愈伤组织的诱导;B 农杆菌浸染前愈伤组织的预培养;C 浸染后的共培养;D 愈伤组织的筛选培养;E 愈伤组织的分化培养;F T0代组培苗的生根培养。
2.4 T0代转基因植株的鉴定
在转基因幼苗移栽至大田15 d后,提取其叶片基因组DNA,对其转基因植株的阳性率和敲除突变情况进行鉴定。对潮霉素抗性基因()进行PCR扩增条带检测(图4),其中扩增片段大小为733 bp;若无条带,则表明无T-DNA插入。
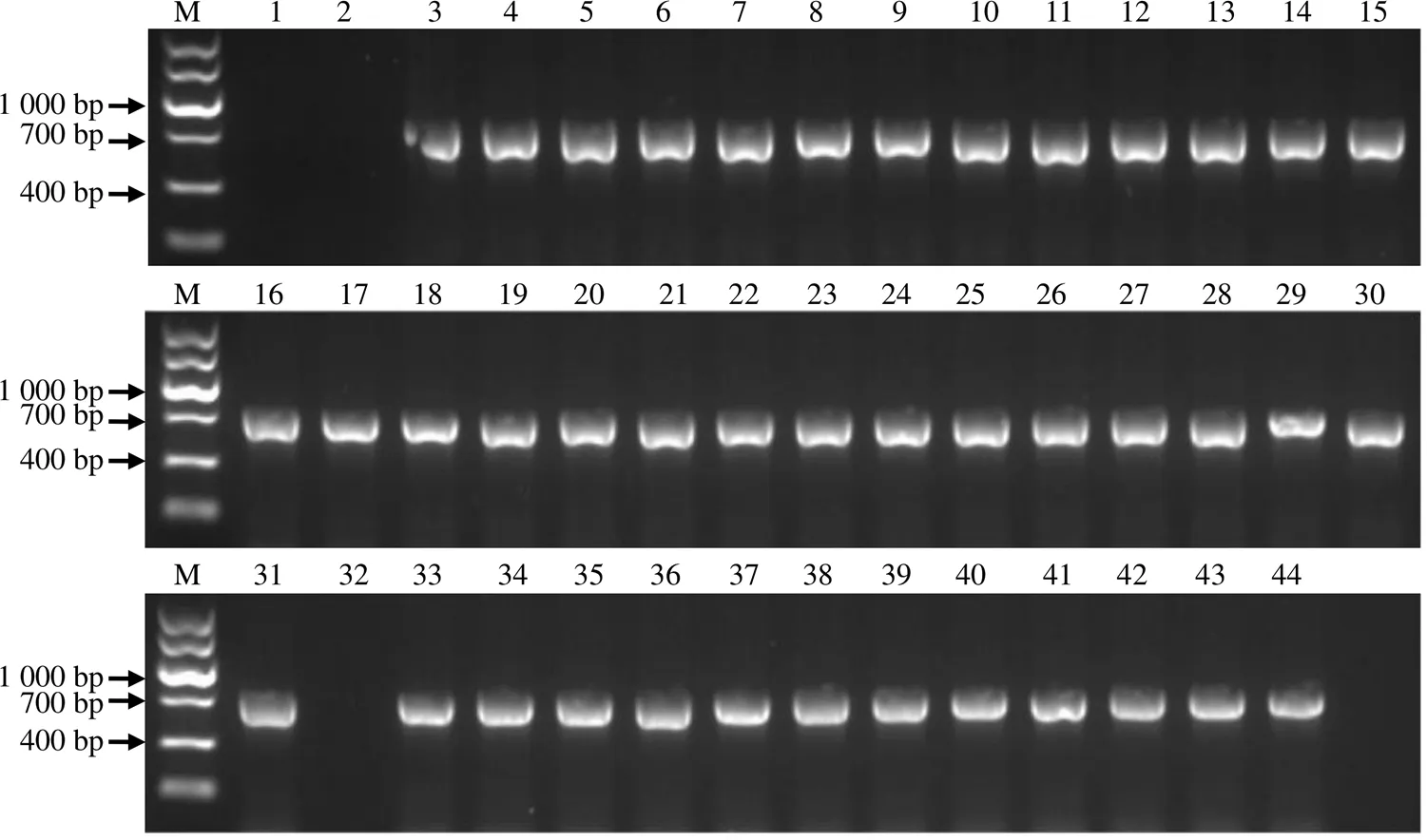
M DNA Marker;泳道1 H2O 空白对照;泳道2 野生型‘中花11’;泳道3~30 OsFRK1转基因潮霉素抗性苗;泳道31~44 OsFRK2转基因幼苗。
对T0代转基因植株进行分析,结果(表2)显示,28株为T0代转基因阳性植株,转基因阳性率为100%;13株为T0代转基因阳性植株,其阳性率为92.86%。对转基因植株靶位点序列进行PCR扩增和扩增产物的测序分析,结果(表2)发现,转基因植株突变率为46.43%,纯合突变率为10.71%,同时获得2种纯合突变体材料,分别是osfrk1-2/17(碱基C缺失)和osfrk1-14(碱基CGC缺失且由C突变为A);转基因植株突变率为92.86%,纯合突变率为21.43%,也获得2种纯合突变体材料,分别是osfrk2-5/7(靶点1处插入碱基T;靶点2处缺失碱基C)和osfrk2-9(靶点2处插入碱基A);纯合突变体的测序结果没有杂峰(图5)。
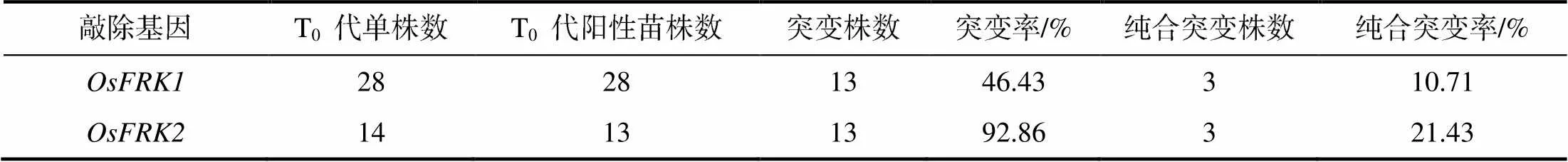
表2 OsFRK1和OsFRK2的T0代转基因植株的敲除效率

A、B、C 分别为OsFRK1b、OsFRK2a 和OsFRK2b 靶点序列测序结果。
进一步对纯合突变体进行氨基酸序列预测,结果(图6)发现:所获得的T0代纯合突变体材料的蛋白功能结构域都遭到不同程度的破坏,osfrk1-2/17的靶位点序列缺失C碱基,导致之后的氨基酸序列发生移码突变,翻译提前终止;osfrk2-5/7在靶点a处增添T碱基,导致天冬氨酸(D)直接突变为终止密码子,严重破坏了目的蛋白的功能结构域。

灰色部分序列为目的蛋白的功能结构域;红色序列代表突变体发生突变后的序列。
3 结论与讨论
有研究[21]表明,优化Cas9基因表达的启动子,可以提高基因组编辑的效率。在拟南芥中,由PcUbi4-2驱动Cas9基因时,T1代转基因植株突变效率低至26%[22],而改为YAO启动子时,拟南芥基因组的突变率高达88.5~90.5%[23]。MA等[14]研究发现,在水稻中用PZmUbi启动Cas9基因,表达效果最佳,T0代转基因植株的突变率高达85.4%。本研究中,采用PZmUbi启动的CRISPR/Cas9技术成功创建了与的T0代转基因植株,它们的突变率分别为46.43%和92.86%,其纯合突变率分别为10.71%和21.43%,说明了PZmUbi启动的CRISPR/Cas9系统对水稻基因组的编辑效率较高。
前人研究结果表明,在拟南芥和番茄中,FRK家族基因的表达变化会影响植株的生长发育[5-8];在玉米、向日葵和甜菜等植物中,FRK家族基因表现出对多种非生物胁迫具有显著的响应[9-10]。说明FRK家族基因在植物的生长发育以及逆境适应中具有重要的生物学功能。本研究中,采用CRISPR/Cas9技术,构建了与的敲除突变体,获得了13株敲除突变体T0代植株,并且在2个基因的T0代敲除突变体中均获得了2种不同突变类型的纯合突变体,为进一步探究和的生物学功能提供了遗传材料。
[1] PETERSEN L A,DOWNS D M.Identification and characterization of an operon ininvolved in thiamine biosynthesis[J].Journal of Bacteriology,1997,179(15):4894-4900.
[2] SIGRELL J A,CAMERON A D,JONES T A,et al. Structure ofribokinase in complex with ribose and dinucleotide determined to 1.8 å resolution:insights into a new family of kinase structures[J]. Structure,1998,6(2):183-193.
[3] FENNINGTON G J,HUGHES T A.The fructokinase frombiovarbelongs to group I fructokinase enzymes and is encoded separately from other carbohydrate metabolism enzymes[J]. Microbiology,1996,142(2):321-330.
[4] 陈毅鸿,张积森.植物果糖激酶基因家族演化及其功能[J].分子植物育种,2016,14(2):359-369. CHEN Y H,ZHANG J S.A review for gene evolution and functional study in plant fructokinase[J].Molecular Plant Breeding,2016,14(2):359-369.
[5] KANAYAMA Y,DAI N,GRANOT D,et al.Divergent fructokinase genes are differentially expressed in tomato[J].Plant Physiology,1997,113(4):1379-1384.
[6] ODANAKA S,BENNETT A B,KANAYAMA Y. Distinct physiological roles of fructokinase isozymes revealed by gene-specific suppression ofandexpression in tomato[J].Plant Physiology,2002,129(3):1119-1126.
[7] GERMAN M A,DAI N,CHMELNITSKY I,et al.,a novel tomato (Mill.) fructokinase specifically expressed in stamens[J].Plant Science,2002,163(3):607-613.
[8] STEIN O,AVIN-WITTENBERG T,KRAHNERT I,et al. Corrigendum:fructokinases are important for seed oil accumulation and vascular development[J]. Frontiers in Plant Science,2017,8:2047.
[9] ZÖRB C,SCHMITT S,MÜHLING K H.Proteomic changes in maize roots after short-term adjustment to saline growth conditions[J].Proteomics,2010,10(24):4441-4449.
[10] KLOTZ K L,FINGER F L,ANDERSON M D. Wounding increases glycolytic but not soluble sucrolytic activities in stored sugarbeet root[J].Postharvest Biology and Technology,2006,41(1):48-55.
[11] JIANG H W,DIAN W M,LIU F Y,et al.Isolation and characterization of two fructokinase cDNA clones from rice[J].Phytochemistry,2003,62(1):47-52.
[12] GUGLIELMINETTI L,MORITA A,YAMAGUCHI J,et al.Differential expression of two fructokinases inseedlings grown under aerobic and anaerobic conditions[J].Journal of Plant Research,2006,119(4):351-356.
[13] GUGLIELMINETTI L,VOLTERRANI M.Effect of ethanol on the expression of two fructokinases in rice seedlings[J].Plant Production Science,2014,17(4):305-310.
[14] MA X L,ZHANG Q Y,ZHU Q L,et al.A robust CRISPR/Cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants[J].Molecular Plant,2015,8(8):1274-1284.
[15] XIE X R,MA X L,ZHU Q L,et al.CRISPR-GE:a convenient software toolkit for CRISPR-based genome editing[J].Molecular Plant,2017,10(9):1246-1249.
[16] ENGLER C,KANDZIA R,MARILLONNET S.A one pot,one step,precision cloning method with high throughput capability[J].PLoS One,2008,3(11):e3647.
[17] HOLSTERS M,WAELE D D,DEPICKER A,et al. Transfection and transformation of[J].Molecular and General Genetics,1978,163(2):181-187.
[18] WANG X,ZHOU W,LU Z H,et al.A lipid transfer protein,OsLTPL36,is essential for seed development and seed quality in rice[J].Plant Science,2015,239:200- 208.
[19] 张焕,仇忠凯,阎新,等.水稻种子特异表达基因的克隆与表达[J].湖南农业大学学报(自然科学版),2018,44(3):278-283. ZHANG H,QIU Z K,YAN X,et al.Cloning and expression analysis of a seed-specific genein rice[J].Journal of Hunan Agricultural University (Natural Sciences),2018,44(3):278-283.
[20] 李东昊,姜玲,刘春林,等. 甘蓝型油菜基因CRISPR/Cas9敲除载体的构建及功能探究[J]. 湖南农业大学学报(自然科学版),2018,44(4):346-352. LI D H,JIANG L,LIU C L,et al.Construction of CRISPR/Cas9 knockout vectorand its genetic transformation in[J]. Journal of Hunan Agricultural University(Natural Sciences),2018,44(4):346-352.
[21] LI J F,NORVILLE J E,AACH J,et al.Multiplex and homologous recombination-mediated genome editing inandusing guide RNA and Cas9[J].Nature Biotechnology,2013,31(8):688-691.
[22] FAUSER F,SCHIML S,PUCHTA H.Both CRISPR/Cas- based nucleases and nickases can be used efficiently for genome engineering in[J].The Plant Journal,2014,79(2):348-359.
[23] YAN L H,WEI S W,WU Y R,et al.High-efficiency genome editing inusing YAO promoter- driven CRISPR/Cas9 system[J].Molecular Plant,2015,8(12):1820-1823.
Generation of CRISPR knock-out mutantsof theandin rice
ZHANG Zongfei, LIU Yuanyuan, OUYANG Jiexiu, LI Shaobo, WANG Xin*
(College of Life Sciences, Nanchang University, Nanchang, Jiangxi 330031, China)
To reveal the biological function of fructokinase (OsFRK) family genes in rice, two knock-out mutants for identified OsFRK family genes were constructed using CRISPR/Cas9 technology. The results showed that 28and 14T0transgenic plants were successfully constructed. The transgenic positive rate, mutation rate and homozygous mutation rate ofis 100%, 46.43% and 10.71%, whileis 92.86%, 92.86% and 21.43%. In addition, the functional domains of targeted proteins of the T0knock-out homozygous mutants were damaged in the mutatnts.
; OsFRK family genes; CRISPR/Cas9; vector construction; knock-out mutants
S511;Q789
A
1007-1032(2020)06-0679-07
张宗飞,刘媛媛,欧阳解秀,李绍波,王鑫.水稻与基因CRISPR敲除突变体的构建[J].湖南农业大学学报(自然科学版),2020,46(6):679-685.
ZHANG Z F, LIU Y Y, OUYANG J X, LI S B, WANG X. Generation of CRISPR knock-out mutants of theandin rice[J]. Journal of Hunan Agricultural University (Natural Sciences), 2020, 46(6): 679-685.
http://xb.hunau.edu.cn

2019-12-09
2020-03-01
国家自然科学基金项目(31760080、31960422)
张宗飞(1993—),男,广东茂名人,硕士研究生,主要从事水稻遗传学研究,zzf520cn@163.com;*通信作者,王鑫,博士,讲师,主要从事植物分子遗传学研究,wangxin@ncu.edu.cn
10.13331/j.cnki.jhau.2020.06.007
责任编辑:毛友纯
英文编辑:柳正
