35株非洲猪瘟病毒全基因组序列的系统生物学分析
2020-12-21于婷婷陈江华丰志华张扬帆林章
于婷婷 陈江华 丰志华 张扬帆 林章


摘 要:非洲猪瘟(African Swine Fever,ASF)源于非洲猪瘟病毒(African Swine Fever Virus,ASFV)感染所引起的烈性传染病,主要是以蜱和野猪为天然宿主。ASFV感染能够导致家猪及野猪直接死亡,死亡率近乎100%。目前在非洲、欧洲、亚洲等地区均出现大面积爆发,却尚未出现对该病毒进行预防的商业化疫苗。因此,需要对病毒基因组信息有更深入的了解。通过对不同地区发生的ASFV全基因组进行多序列比对、系统进化树构建、保守区域检测等,获得与ASFV毒力、进化等相关的基因片段,并研究这些基因片段在不同区域的病毒株中的关系。结果表明: Ⅶ与Ⅱ基因型相比较,后者出现很多大片段基因序列的增加,这些增加的片段主要出现在V110、360、505/530多基因家族中。其中,对多基因家族中MGF 505/5307R、MGF 3601L基因研究分析表明,不同基因型之间由于这些基因的差异影响巨噬细胞的存活能力、病毒复制强弱与病毒变异和进化有关,结果为研究病毒进化及未来病毒疫苗研发提供新的方向。
关键词:非洲猪瘟病毒;全基因组序列;系统生物学;病毒进化
中图分类号:S852.651 文献标志码:A 文章编号:0253-2301(2020)05-0012-08
Abstract: African Swine Fever (ASF) is a severe infectious disease caused by the African Swine Fever Virus (ASFV). The ticks and wild boars are the main natural hosts. The infection of ASFV can cause the direct death of domestic pigs and wild boars, with the death rate of nearly 100%. At present, the widespread outbreaks have occurred in Africa, Europe and Asia, but there was no commercial vaccine to prevent the virus. Therefore, a deeper understanding of viral genomic information was required. Through the multi-sequence alignment, the construction of phylogenetic tree, the detection of conserved region of ASFV whole genomes occurring in different regions, the gene fragments related to the virulence and evolution of ASFV were obtained, and then the relationship between these gene fragments in virus strains from different regions was also studied. The results showed that compared with the genotype Ⅶ, the genotype Ⅱ showed many increases in large fragment of gene sequence, most of which were found in the multigene family of V110, 360 and 505/530. Among them, the analysis of MGF 505/5307R and MGF 3601L genes in the multigene family showed that the differences of these genes among different genotypes would affect the survival ability of macrophages and the strength of virus replication was related to the variation and evolution of viruses. The results provided a new direction for the study of the evolution of viruses and the development of virus vaccines in the future.
Key words: African Swine Fever Virus; Whole genome sequence; Systematic biology; Viral evolution
非洲豬瘟病毒(African Swine Fever virus,ASFV)是一种大型多囊膜结构的DNA病毒,主要感染猪单核细胞和巨噬细胞,并在细胞核周围离散的病毒工厂内进行复制[1]。该病毒基因组为170~190 kbp的线性双链DNA分子,编码150多种多肽[2-3]。结构上内部由脂质膜将基因组核心包裹,外部由二十面体蛋白衣壳和外部脂质膜组成[1]。其中内部脂质膜由内质网的膜碎片组成,外部脂质膜是由病毒胞吐期间在质膜上发芽形成[4]。ASFV是目前唯一已知的DNA基因组的虫媒病毒[5],是导致野猪、家猪高度致死,出血性疾病的病原体,并且在细胞内外的ASFV均具有传染性[4]。在近期研究中尚未有应对非洲猪瘟的疫苗,因此急需对ASFV病毒基因组进行全方面分析,以探究病毒在不同地区传染过程中基因组发生的变化。
目前,对ASFV基因研究主要是病毒检测方法、传播途径、入侵宿主的分子过程、病毒变异等,其中包括开发最新PCR检测法用于检测ASFV[6];ASFV进入宿主和体内途径的机制[7];病毒基因
MGF360、530对宿主干扰素影响[5];在Georgia株种敲除9GL和UK基因为疫苗提供参考等[8]。尽管关于ASFV研究已有很多,但是大多数研究只是对单一区域ASFV进行的研究,缺乏从不同区域、不同时间上对ASFV全基因组进行纵向比较。
本研究主要从不同区域的ASFV全基因组层面上,使用生物信息学进行系统研究,相比对于单一病毒株的研究更具有意义。其次选择全基因组数据分析,对比只关注于单个基因更具全面性。方法上选用MAFFT、MEGA软件进行多序列比对、构建系统进化树,MAFFT多重序列比对软件与其他类型软件相比速度和精度相对较高[9-10]。MEGA构建系统进化树更加简洁、方便。经过对比得到的差异片段使用CDD数据库进行保守区域鉴定[11]。最后由Blast系列工具鉴定基因片段重复出现频率及相似度,完成从基因组上对ASFV的研究。
1 数据来源与分析方法
由美国国家生物技术信息中心(NCBI)获得全部的非洲猪瘟全基因组测序数据,并收集相关的病毒类型、病毒发生地和时间等。
为描述所收集到非洲猪瘟的样本数据之间的关系,通过MAFFT、MEGA进行全基因组序列比对和系统进化树构建。与使用部分片段进行比对,能够直接获得更多基因片段的取代、插入、缺失等信息,并且依靠全基因组所构建进化树也更加可靠。构建进化树使用最大似然法(ML)從全基因组层面进行计算,与依据P72基因构建进化树进行病毒基因分型使用的方法一致[12-13]。
MAFFT软件使用mafftautothread 32 in.file>out.file命令进行全基因组序列比对,其中in.file为输入文件out.file为比对结果输出文件,结果文件中包含序列详细的比对信息。根据结果文件相互比较结果中存在取代、插入、缺失信息的片段进行检验:(1)对于缺失较大片段通过CDD(CDD保守域数据库能够对蛋白质特定结构域、保守区域、注释信息进行筛查,CDD保守区域检索可以使用NCBI提供的在线数据库,也可以通过本地构建的保守序列数据库进行检索)进行验证,确定是否是大片段蛋白插入形成新的基因类型;(2)缺失较小片段由Blast工具进行检索,验证小片段基因的来源。先由Makeblastdb命令构建35株病毒库,其次使用Blastn命令对自建数据库进行检索;(3)其他差异片段从NT/NR数据库寻找来源信息。
2 结果与分析
2.1 系统进化树
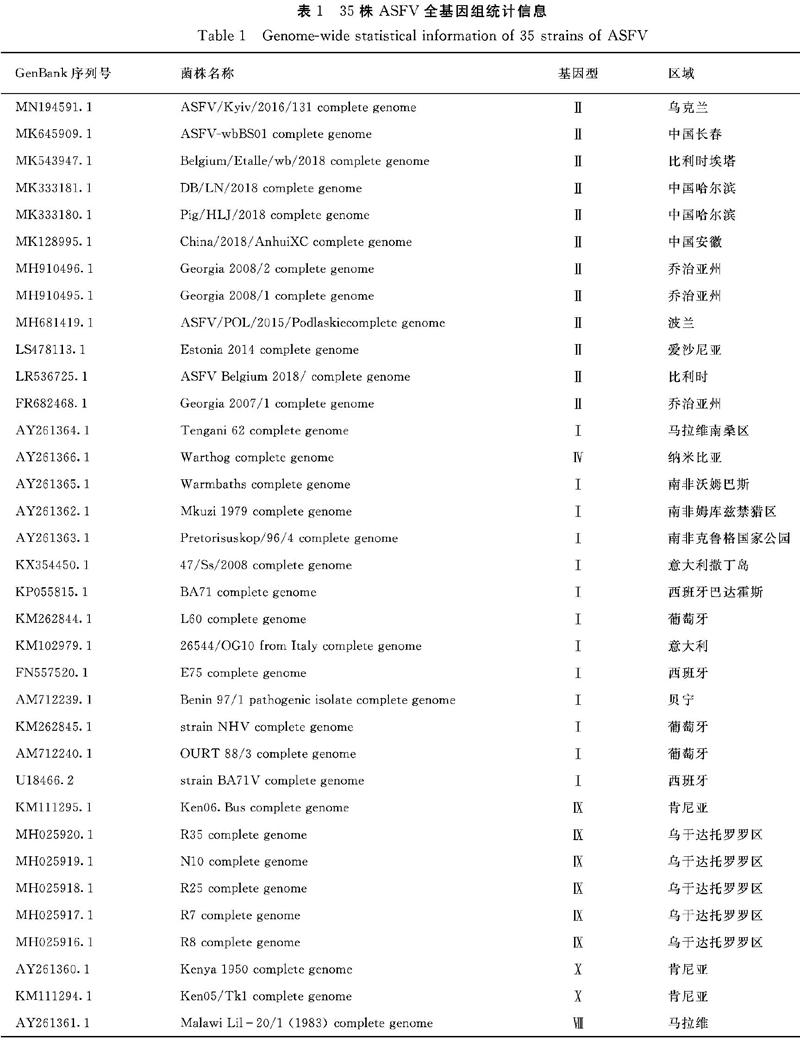
通过NCBI获得40多种非洲猪瘟的全基因组数据,对这些数据中未验证的数据进行剔除,最终得到35种基因组数据、基因型信息(表1)[12-15]。使用MAFFT、MEGA软件比对、构建系统进化树,描述35种不同基因组数据之间的关系。并结合对ASFV基因型研究,绘制进化树和基因分型关系图(图1),发现亚洲地区与欧洲地区病毒基因型相同,并且与使用全基因构建进化树分类一致。在进化树中出现最早的病毒可能是马拉维Ⅷ基因型,同时Ⅰ与Ⅳ基因型在全基因组层面上具有一定的相似性。
2.2 CDD保守区域检索
根据进化树将同一分支、不同分支之间进行相互比较,发现在同一分支内基因组相差异小,但对于不同分支之间差异大。其中Ⅱ与Ⅷ基因型比较结果差异较大,两者相比存在很多缺失、差异片段。因此对这两株中较大缺失片段进行CDD检索,大多数差异片段出现在V110、360、505/530多基因家族中。由于MGF 505/5307R、MGF 3601L基因影响宿主干扰素、促进巨噬细胞存活等[16-17],因此选择MGF 505/5307R、MGF 3601L基因为主要对象,并通过CDD检索比较,绘制出该基因MGF 505/5307R蛋白保守区域图(图2)。
2.3 MGF 505/5307R与MGF 3601L基因重复片段检测
为研究Ⅷ基因型中缺失片段在35株中存在的总体情况,使用Makeblastdb工具对35株ASFV全基因组构建核酸数据库,由Blastn命令对所有株型中进行检索得到统计信息(表2),结果发现在ASFV中可能存在2~4个重复片段,与Ⅱ基因型各个片段基本没有差异,在Ⅰ、Ⅹ、Ⅸ、Ⅳ基因型中存在15%左右的差异,和基因型Ⅷ相比差异最大。
同时也发现 MGF 3601L 基因在比较中也存在类似差异(表3)。与Ⅱ基因型各个片段基本没有差异,在部分Ⅹ基因型、部分Ⅰ基因型和Ⅳ基因型中没有重复片段,Ⅸ基因型有大概15%的差异。
3 讨论与结论
ASFV最早是1920年在肯尼亚报道[18],20世纪中叶蔓延到欧洲再到南美、加勒比海,1990年在欧洲对所有野猪、家猪进行捕杀,以达到将ASFV规模根除的目的[1]。但2007年该病毒再次从非洲传播到高加索地区、波兰等地区[19],并在2018年传入中国,并带来巨大经济损失[20]。因此目前急需解决的是在防治方面得到突破。本研究从生物信息分析角度出发,利用现有的软件、数据库,发现在病毒基因组中存在的潜在规律,为未来ASFV研究提供一定的参考信息。采用MAFFT进行多序列比对、MEGA构建系统进化树、Blast比对重复序列等,得到基因与病毒发生时间、进化方式之间的关系。
研究中构建35株ASFV全基因组构建的进化树,与前期研究中根据P72基因所构建进化树大体一致[12]。但在全基因组构建进化树中,Ⅷ型为最早的原始株,同时推断出由Ⅷ型突变衍生出其他基因型。进化树中I型与Ⅳ型基因型被划分在同一分支中,表明二者在全基因组上相近。为此猜想Ⅳ型可能是由Ⅰ型突变或重组得到。进一步结合进化树和分布区域的信息,发现Ⅱ与部分Ⅰ、Ⅸ型基因型同一分支,可能是部分Ⅰ型与Ⅸ型重组得到,同时也符合区域分布、传染信息等。
全基因組进化树中,将分支最远的两个基因型Ⅱ与Ⅷ全基因组进行比较。发现Ⅷ基因组中存在很多缺失、差异片段。经过CDD检索大多数缺失片段为V110、360、505/530多基因家族。进一步说明这些缺失片段可能与病毒存活能力、复制能力有关。综合ASFV前期研究,多基因家族中MGF 505/5307R、MGF 3601L基因,对宿主干扰素产生、促进巨噬细胞存活等具有一定的影响[17]。对35株中这两种基因片段相似度、重复情况进行检索统计。发现Ⅱ型基因型具有全部基因型出现的重复片段,并且相似度较高。在进化树中随着距离Ⅱ型分支越远逐渐出现部分片段缺失、相似度降低。综合35株不同区域、不同基因型的图谱信息及爆发时间,发现随着病毒不断传播,在新地区出现病毒复制能力强弱程度,以及其他文献研究报道则表明505/530、360多基因家族的功能。说明ASFV中505/530、360多基因家族重复片段数量,与病毒存活能力、复制息息相关。以及在Ⅱ型基因型中MGF 505/5307R、MGF 3601L出现的重复片段,可能是早期基因型出现重组的结果。
在ASFV防治方面,是否通过对505/530、360多基因家族产生影响,获得弱毒病毒株以加快疫苗的研制。同时在病毒进化方面,提供了基因变化与病毒变异、进化关系,为进一步研究病毒进化和预防等提供更多的证据,也为未来ASFV疫苗研发提供新方向。
参考文献:
[1]GALINDO I,ALONSO C.African Swine Fever Virus:A Review [J].Viruses,2017,9(5):103.
[2]YANEZ R J,RODRIGUEZ J M,NOGAL M L,et al.Analysis of the Complete Nucleotide Sequence of African Swine Fever Virus[J].Virology,1995,208(1):249-278.
[3]HERNAEZ B,ESCRIBANO J M,ALONSO C.Visualization of the African swine fever virus infection in living cells by incorporation into the virus particle of green fluorescent proteinp54 membrane protein chimera[J].Virology,2006,350(1):1-14.
[4]SALAS M L,GERMAN A.African swine fever virus morphogenesis[J].Virus Research,2013,173(1):29-41.
[5]AFONSO C L,PICCONE MEZAFFUTO K M, NEILAN J,et al.African swine fever virus multigene family 360 and 530 genes affect host interferon response[J].Journal of Virology,2004,78(4):1858.
[6]LUO Y,ATIM S A,SHAO L,et al.Development of an updated PCR assay for detection of African swine fever virus[J].Archives of Virology,2016,162(1):191-199.
[7]SNCHEZ E G,PREZ-NEZ D,REVILLA Y.Mechanisms of Entry and Endosomal Pathway of African Swine Fever Virus[J].Vaccines,2017,5(4):42.
[8]O′DONNELL V,RISATTI G R,HOLINKA L G,et al.Simultaneous deletion of the 9GL and UK genes from the African swine fever virus Georgia 2007 isolate offers increased safety and protection against homologous challenge[J].Journal of Virology,2017,91(1):e01760-16.
[9]WONG K M,SUCHARD M A,HUELSENBECK J P.Alignment uncertainty and genomic analysis[J].Science,2008,319(5862):473-476.
[10]KUMAR S,STECHER G,TAMURA K.MEGA7:Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets[J].Molecular Biology & Evolution,2016,33(7):1870-1874.
[11]ARON M B,DERBYSHIRE M K,GONZALES N R,et al.CDD:NCBI′s conserved domain database[J].Nucleic Acids Research,2015,43(Database issue):D222-D226.
[12]VILLIERS E P,DE,CARMINA G,MARISA A,et al.Phylogenomic analysis of 11 complete African swine fever virus genome sequences[J].Virology,2010,400(1):128-136.
[13]BASTOS A D S,PENRITH M L,CRUCIRE C,et al.Genotyping field strains of African swine fever virus by partial p72 gene characterisation[J].Archives of Virology,2003,148(4):693-706.
[14]BISHOP R P,FLEISCHAUER C,VILLIERS E P D,et al.Comparative analysis of the complete genome sequences of Kenyan African swine fever virus isolates within p72 genotypes IX and X[J].Virus Genes,2015,50(2):303-309.
[15]BOSHOFF C I,BASTOS A D,GERBER L J,et al.Genetic characterisation of African swine fever viruses from outbreaks in southern Africa(1973-1999)[J].Veterinary Microbiology,2007,121(1):45-55.
[16]CHRISTOPHER N,ISABELLE R,THOMAS W.The subcellular distribution of multigene family 110 proteins of African swine fever virus is determined by differences in C-terminal KDEL endoplasmic reticulum retention motifs[J].Journal of Virology,2004,78(7):3710-3721.
[17]AFONSO C L,PICCONE M E,ZAFFUTO K M,et al.African Swine Fever Virus Multigene Family 360 and 530 Genes Affect Host Interferon Response[J].Journal of Virology,2004,78(4):1858-1864.
[18]MONTGOMERY R E.On a form of swine fever occurring in British East Africa(Kenya Colony) [J].Journal of comparative pathology & therapeutics,1921,34:159-191.
[19]WOZNIAKOWSKI G,KOZAK E,KOWALCZYK A,et al. Current status of African swine fever virus in a population of wild boar in eastern Poland(2014-2015)[J].Archives of Virology,2016,161(1):189-195.
[20]GE S,LI J,FAN X,et al.Molecular Characterization of African Swine Fever Virus, China, 2018[J].Emerging infectious diseases,2018,24(11):2131-2133.
(責任编辑:柯文辉)
