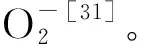蒲公英抗氧化酶基因的克隆与盐胁迫表达分析
2020-12-04张海洋梁晓艳邢延富宋延静李茹霞李俊林郭洪恩王向誉
付 娆,张海洋,乔 鹏,梁晓艳,邢延富,宋延静,李茹霞,李俊林,郭洪恩,王向誉
(1.山东省蚕业研究所,山东烟台 264002;2.烟台市福山区农业农村局,山东烟台 265500)
随着淡水资源危机以及土壤盐渍化问题的加剧,探索微咸水或海水资源在农业生产中的应用、充分利用盐渍化土壤是未来农业发展的新方向。中国的盐碱地总面积达3630万hm2,其中,近100万hm2滨海盐碱地是直接或间接受海水或海潮影响的地区,土壤含盐量一般为0.6%~1.0%,高者达3%以上,盐分组成以氯化物为主[1]。近十几年来,盐碱地改良利用技术不断创新发展,主要集中于利用淡水洗盐、暗管排盐和修筑台田等工程措施将土壤的含盐量降低到作物能适应的程度,或直接种植盐生植物、耐盐经济作物等[2]。加强耐盐植物种质资源的筛选与培育,对推动盐碱地的绿色高效利用有重要意义。要实现兼具经济和生态价值的耐盐植物新品种(品系)的培育,阐明植物对盐碱地生境的生理生态响应及分子调控机制是基础亦是关键。
陈华等[20]将蒲公英作为耐海水蔬菜开发的新成员,其集食用、药用、保健和生态价值于一身。笔者前期的试验结果也证实在NaCl和海水胁迫下,蒲公英叶片中的3种抗氧化酶SOD、POD和CAT活性迅速增强,本研究拟在此基础上,以蒲公英叶片cDNA为模板,利用同源克隆的方法得到蒲公英TmoCu/ZnSOD、TmoAPX和TmoCAT基因,对基因编码的氨基酸序列进行生物信息学分析,并测定NaCl单盐和海水复合盐胁迫下基因的表达情况,以期了解盐胁迫下蒲公英抗氧化酶系统变化机理,为蒲公英耐盐分子机制的研究提供基础数据。
1 材料与方法
1.1 幼苗培养及盐胁迫处理
试验材料为烟台本地采集的野生蒲公英种子,单盐为200 mmol/L的NaCl,等Na+含量的海水(盐质量浓度16 g/L)为天然海水和蒸馏水配制,其中天然海水取自烟台近海,盐质量浓度为33.33 g/L,蒲公英(TaraxacummongolicumHand.-Mazz.)播种于花盆中,待幼苗长至8~10片真叶时进行盐处理。对照组浇灌蒸馏水,单盐处理组浇灌200 mmol/L的NaCl,复合盐处理组浇灌盐质量浓度16 g/L的海水,分别设置3个生物学重复。
1.2 总RNA的提取和cDNA第一链的合成
对照组、NaCl和海水胁迫处理组,分别处理0 h、3 h、6 h和12 h,采集长势基本一致的相同部位的叶片,且每一处理选用3株不同植株的叶片,液氮速冻,-80 ℃冰箱中保存。利用Trizol试剂(InvitrogenTM)进行总RNA的提取,凝胶电泳检测提取的RNA的完整性,并用NanoDrop 2000C(Thermo Scientific)超微量分光光度计测定其 浓度。
1.3 抗氧化酶基因的克隆
由于蒲公英尚未有基因组数据,在植物基因组数据库Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)查找与其亲缘关系相近且已有基因组数据的向日葵和莴苣3种抗氧化酶基因的cDNA和氨基酸序列,下载后与拟南芥、棉花和水稻相关基因的核酸和氨基酸序列进行同源比对分析,查找与其他物种保守性较高的基因,最终选取向日葵Cu/ZnSOD基因(GenBank登录号XM_022153724)、CAT基因(GenBank登录号XM_022141874)和莴苣APX基因(GenBank登录号XM_023880576)的核酸序列进行引物设计。为尽可能多地获得蒲公英抗氧化酶基因的遗传信息,正向和反向引物的设计分别从向日葵和莴苣相关基因的起始和终止密码子开始(表 1)。以蒲公英叶片cDNA为模板进行 PCR扩增,反应体系为25 μL:cDNA 1 μL、前后引物(10 μmol/L)各0.75 μL、10×EasyPfu Buffer 2.5 μL、2.5 mmol/L dNTPs 2.5 μL、EasyPfu DNA Polymerase 0.5 μL和Nuclease-free water 14.5 μL。反应程序为94 ℃ 2 min;94 ℃ 20 s,50 ℃ 20 s,72 ℃ 30 s,35个循环;72 ℃ 5 min。产物经 10 g/L琼脂糖凝胶电泳检测、切胶回收,胶回收产物与pEASY-Blunt Simple Cloning Kit载体(TRANSGEN)连接,转化,挑取单克隆进行菌液PCR验证,阳性克隆送华大基因测序。
1.4 抗氧化酶基因氨基酸序列的生物信息学分析
蒲公英抗氧化酶基因的编码区序列,经测序后进行拼接,用Editseq预测基因氨基酸序列,通过在线软件Pfam(http://pfam.sanger.ac.uk/)和NCBI Conserved Domain分析氨基酸序列的保守结构域。利用ClustalX2软件将预测的氨基酸序列与NCBI下载的莴苣(LactucasativaLinn.)、向日葵(HelianthusannuusL.)、棉花(Gossypiumraimondii)、毛果杨(Populustrichocarpa)、葡萄(VitisviniferaL.)、番茄(Solanumlycopersicum)、大豆(GlycinemaxLinn. Merr.)、拟南芥(Arabidopsisthaliana)、甘蓝(BrassicaoleraceaL.)、水稻(OryzasativaL.)和玉米(ZeamaysL.)相关基因的氨基酸序列进行比对分析,并利用MEGA 7.0软件构建NJ(Neighbor-joining)进化树。
1.5 抗氧化酶基因的表达分析
以肌动蛋白基因Actin为内参基因[21],检测对照组和试验组中抗氧化酶基因的表达情况。根据克隆得到的抗氧化酶基因编码区序列,利用primer 5.0软件设计qRT-PCR引物(表 1),由北京六合华大基因科技有限公司合成。以蒲公英叶片cDNA为模板,利用TransStart Tip Green qPCR Super Mix试剂,进行RT-qPCR扩增,反应体系为20 μL:cDNA 0.5 μL、前后引物(10 μmol/L)各0.5 μL、2 ×TransStart Tip Green qPCR SuperMix 10 μL和Nuclease-free water 8.5 μL;反应条件为94 ℃预变性2 min;94 ℃ 20 s,60 ℃ 20 s,72 ℃ 30 s,40个循环。每个扩增反应设置3个重复,扩增后采用2-ΔCt法分析基因相对表达量[22],ΔCt=Ct目的片段-Ct内参基因。

表1 试验所用引物序列Table 1 Sequences of primers used in this study
2 结果与分析
2.1 蒲公英抗氧化酶基因克隆和生物信息学 分析
采用同源克隆的方法,从蒲公英叶片中克隆到3个抗氧化酶基因,分别命名为TmoCu/ZnSOD1、TmoAPX1和TmoCAT1(图1),其中TmoCu/ZnSOD1基因CDS序列全长474 bp,编码1个含157个氨基酸残基的铜锌超氧化物歧化酶和1个终止密码子(图2);TmoAPX1基因CDS序列全长852 bp,编码1个含283个氨基酸残基的L-抗坏血酸过氧化物酶和1个终止密码子(图3);TmoCAT1基因CDS序列全长1 479 bp,编码1个含492个氨基酸残基的过氧化氢酶和1个终止密码子(图4)。多序列比对结果表明TmoCu/ZnSOD1、TmoAPX1和TmoCAT1与来自其他植物的Cu/ZnSODs、APXs和CATs有相同的家族特征,包括高度保守结构域(图2、3和4)。
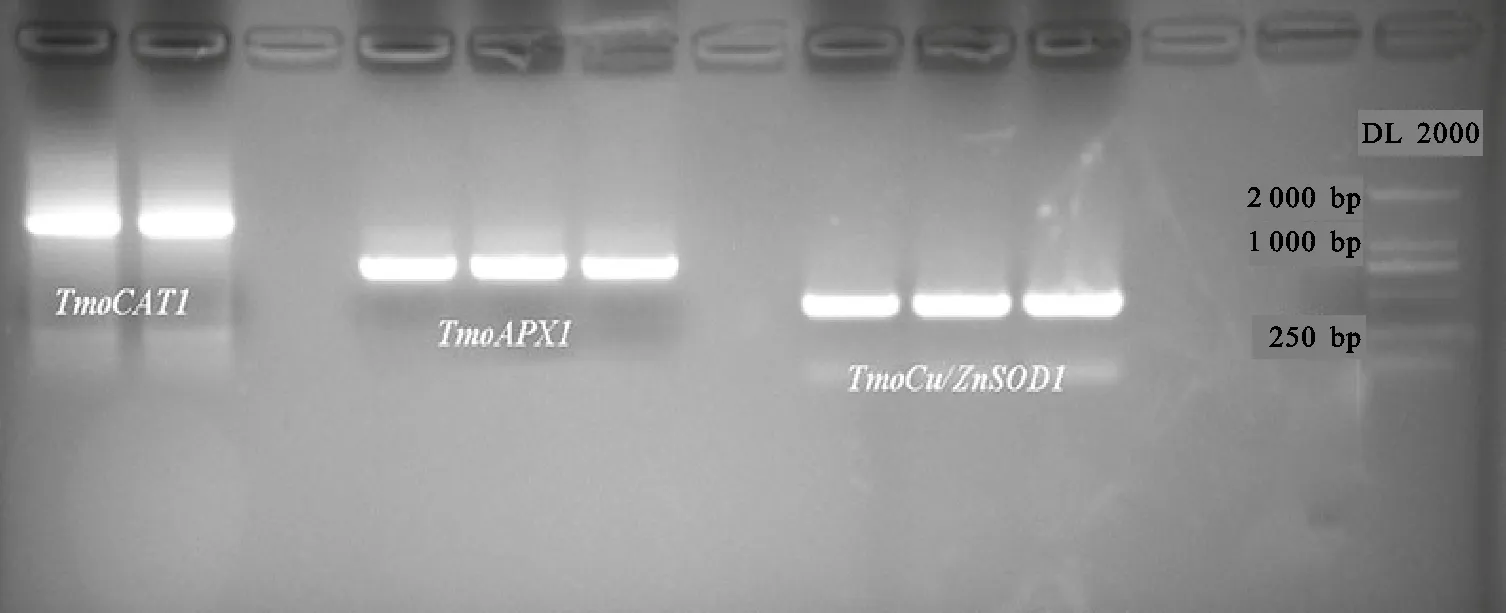
图1 TmoCu/ZnSOD1、 TmoAPX1和 TmoCAT1基因扩增产物Fig.1 Amplification products of TmoCu/ZnSOD1, TmoAPX1 and TmoCAT1

图2 TmoCu/ZnSOD1与其他植物Cu/ZnSODs氨基酸序列的比对Fig.2 Multiple sequence alignment of TmoCu/ZnSOD1 and amino acid sequence of Cu/ZnSODs from other plants

图3 TmoAPX1与其他植物APXs氨基酸序列的比对Fig.3 Multiple sequence alignment of TmoAPX1 and amino acid sequence of APXs from other plants
将克隆得到的蒲公英3个抗氧化酶基因与其他11个物种的抗氧化酶基因的氨基酸序列进行多序列比对,序列同源性见表2,结果表明,TmoCu/ZnSOD1与莴苣中的Cu/ZnSOD氨基酸同源性最高(93.63%),其次为向日葵(84.71%),然后是番茄(84.08%)、棉花(81.17%),与其他物种氨基酸同源性小于80%。TmoAPX1与莴苣和向日葵APX氨基酸同源性均大于90%,与棉花、毛果杨、葡萄和番茄同源性均大于80%,与其他物种氨基酸同源性小于80%。TmoCAT1与其他11个物种的CAT氨基酸同源性均大于80%,与莴苣、向日葵和毛果杨同源性较高。
基于抗氧化酶基因氨基酸序列的系统进化关系分析表明:TmoCu/ZnSOD1与同属菊科的莴苣进化关系最近,其次为菊科的向日葵、茄科的番茄(图5-A);TmoAPX1与同属菊科的莴苣和向日葵的进化关系最近,其次为单子叶植物水稻和玉米(图5-B);TmoCAT1与同属菊科的莴苣和向日葵的进化关系最近(图5-C)。

图4 TmoCAT1与其他植物CATs氨基酸序列的比对Fig.4 Multiple sequence alignment of TmoCAT1 and amino acid sequence of CATs from other plants
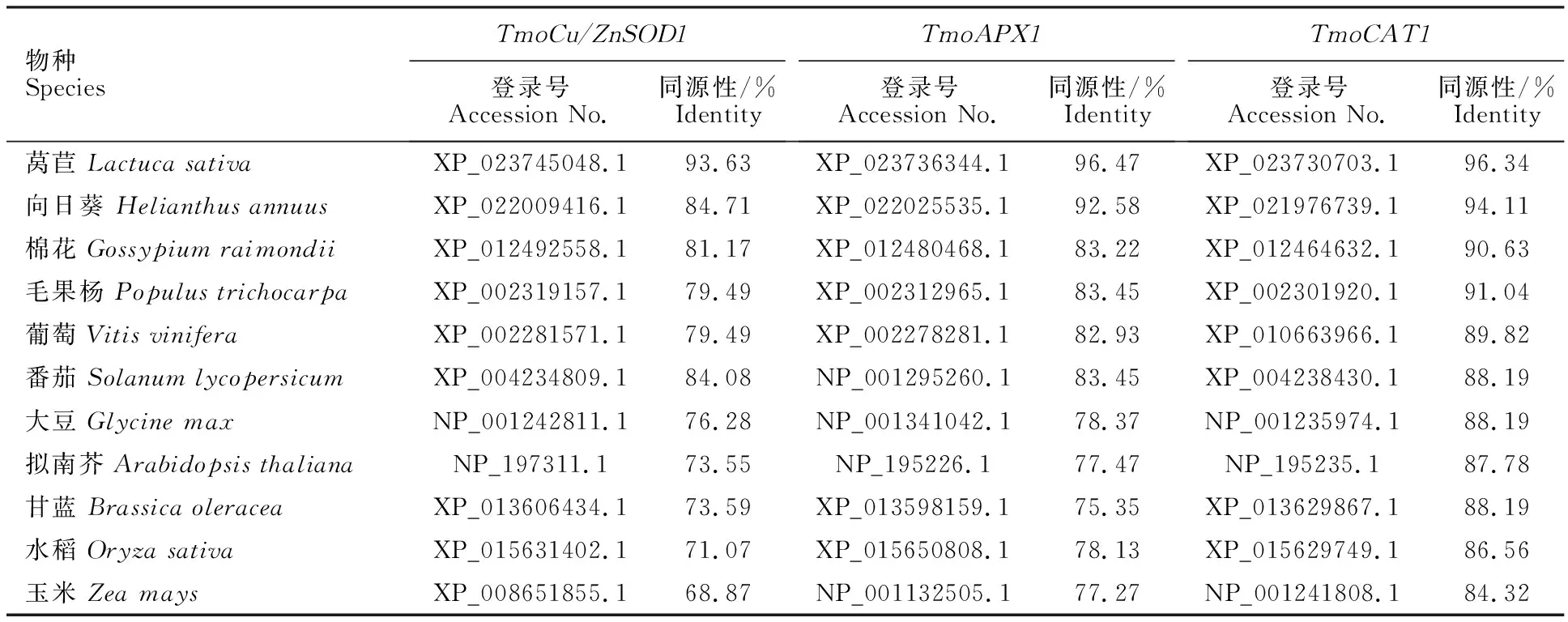
表2 蒲公英3种抗氧化酶与其他植物抗氧化酶基因氨基酸序列同源性比较Table 2 Comparison of amino acid sequence homology between three antioxidant enzymes of dandelion and other plants
2.2 盐胁迫对蒲公英叶片抗氧化酶基因表达的影响
蒲公英抗氧化酶基因在NaCl和海水胁迫后的表达分析(图6)显示,对照组TmoCu/ZnSOD1、TmoAPX1和TmoCAT1的表达量均无显著变化(P>0.05),用200 mmol/L NaCl和盐浓度16 g/L的海水处理后,TmoCu/ZnSOD1、TmoAPX1和TmoCAT1表达量均增加。NaCl处理3、6和12 h,TmoCu/ZnSOD1表达量相比0 h分别增加140.93%、263.41%和351.13%,且P值均小于0.05;海水处理3、6和12 h,TmoCu/ZnSOD1表达量相比0 h分别增加 570.97%、421.95%和220.96%,且P值均小于0.05(图6-A)。NaCl处理3、6和12 h,TmoAPX1表达量相比0 h分别增加168.35%、103.35%和127.93%,且P值均小于0.05;海水处理3、6和12 h,TmoAPX1表达量相比0 h分别增加209.84%、71.89%和112.64%,且P值均小于0.05(图6-B)。NaCl处理3、6和12 h,TmoCAT1表达量相比0 h分别增加 184.79%、123.66%和161.80%,且P值均小于0.05;海水处理3、6和12 h,TmoCAT1表达量相比0 h分别增加226.85%、157.27%和106.94%,且P值均小于0.05(图6-C)。
3 结论与讨论
盐碱化是一个世界性的问题,受盐碱化影响的地区由于降雨少、灌溉系统差、盐分入侵、水污染等环境因素而日益严重。植物盐分胁迫耐受机制是非常复杂的现象,需要各种生理生化过程的协同作用和基因水平的综合调控[23-24]。近年来,盐生或甜土植物抗氧化酶系统对盐胁迫的响应已取得重大进展,盐胁迫下抗氧化酶活性及相关基因表达的变化依植物种类、胁迫程度而异。植物对逆境胁迫耐受能力的增加,常伴随抗氧化酶基因表达量的上升。水稻中的抗氧化酶基因OsAPx8[25]、cCuZn-SOD2[26]和OsAPX7[27]在盐胁迫下表达上调,积极参与ROS的清除。盐胁迫下植物抗氧化酶系统的变化机理也已有相关研究,一些研究结果表明抗氧化酶基因转录产物的构成和转录表达与其活性相关,Hu等[28]研究发现,盐胁迫下,多年生黑麦草耐盐品种中的APX活性高于盐敏感品种,同时其抗氧化酶基因的转录表达高于盐敏感品种,APX基因表达水平与其酶活性正相关,进而推测黑麦草的耐盐性与抗氧化酶基因的组成型或诱导型表达相关。但也有研究表明,抗氧化酶基因的转录水平与其酶活性水平的变化可能并不一致,Lopez等[29]研究表明,盐胁迫下,萝卜中APX活性增加但其基因的转录表达并未增加;Hu等[30]在多年生黑麦草中的研究发现,盐胁迫下叶片POD和CAT酶活性降低,而POD和CAT基因的转录明显升高。本研究结果显示,盐处理后蒲公英TmoCu/ZnSOD、TmoAPX和TmoCAT基因的表达量迅速增加,同时前期的试验结果显示,蒲公英叶片中SOD、POD和CAT酶活性在盐胁迫处理条件下与对照组相比明显增加,证实抗氧化酶基因的表达水平与其酶活性正相关。但随着盐胁迫时间的继续延长,基因表达量有所回落,可能是由于长时间受到较高浓度的盐胁迫,过量的活性氧大量积累,对其细胞造成严重的损害,导致活性氧的产生与清除失去平衡。此外,在前期的研究中笔者比较分析了NaCl单盐和海水复合盐胁迫下蒲公英叶片SOD、POD和CAT酶活性的差异,相较于NaCl单盐胁迫,海水复合盐胁迫下酶活性有所增加。因此,本研究基于基因表达转录水平分析NaCl单盐胁迫和海水复合盐胁迫对蒲公英的影响,结果显示,TmoCu/ZnSOD1表达量在海水复合盐胁迫下表达量的增加速率显著高于NaCl单盐,TmoAPX和TmoCAT基因表达量的增加速率和量在NaCl单盐和海水复合盐胁迫中差异不显著,表明复合盐诱导的酶活性增加也可能是转录后调控。
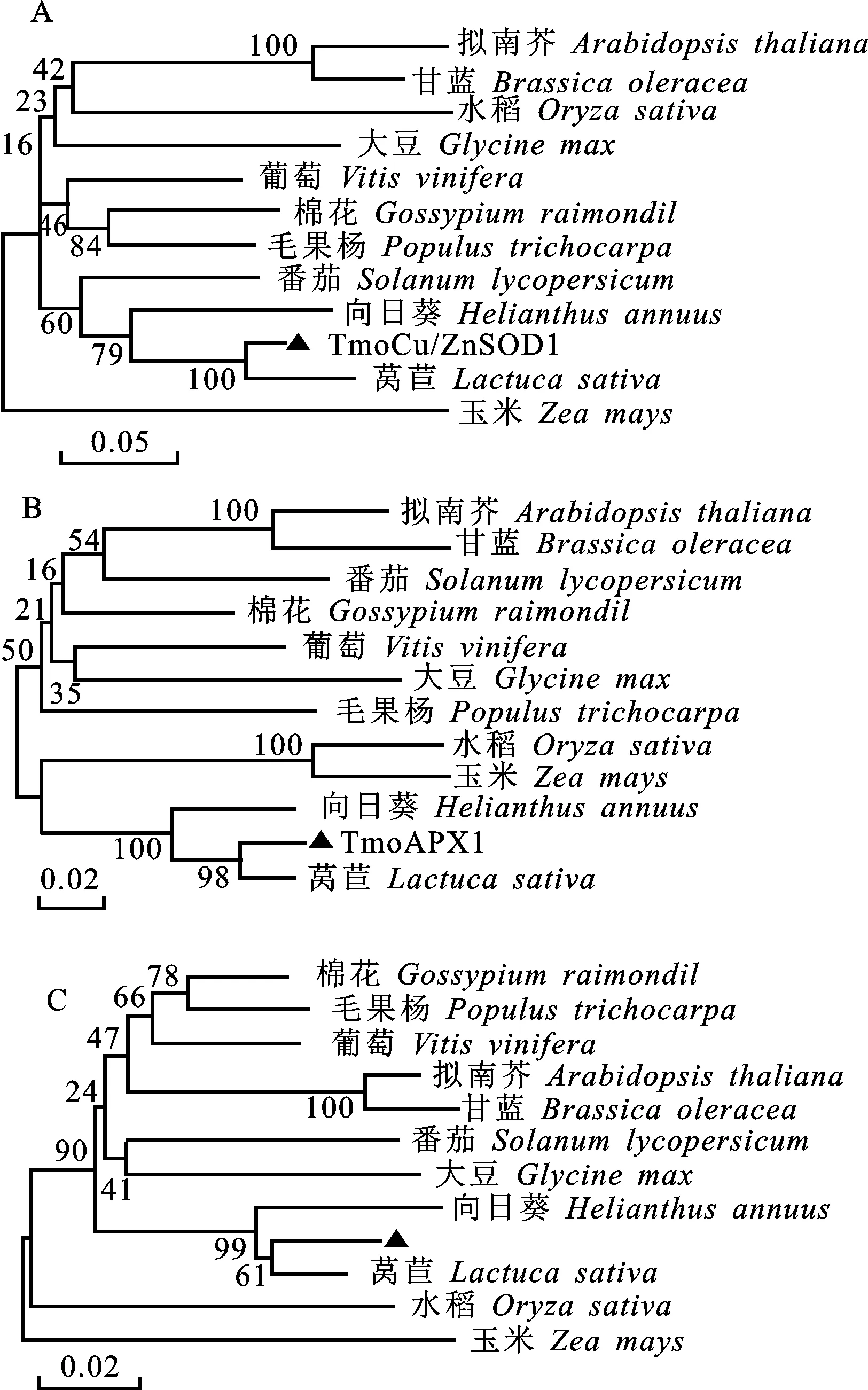
图5 蒲公英 TmoCu/ZnSOD1(A)、 TmoAPX1(B)和 TmoCAT1(C)与其他植物抗氧化酶的进化分析Fig.5 Evolutionary analysis of TmoCu/ZnSOD1 (A), TmoAPX1(B) and TmoCAT1(C) in dandelion and other plants
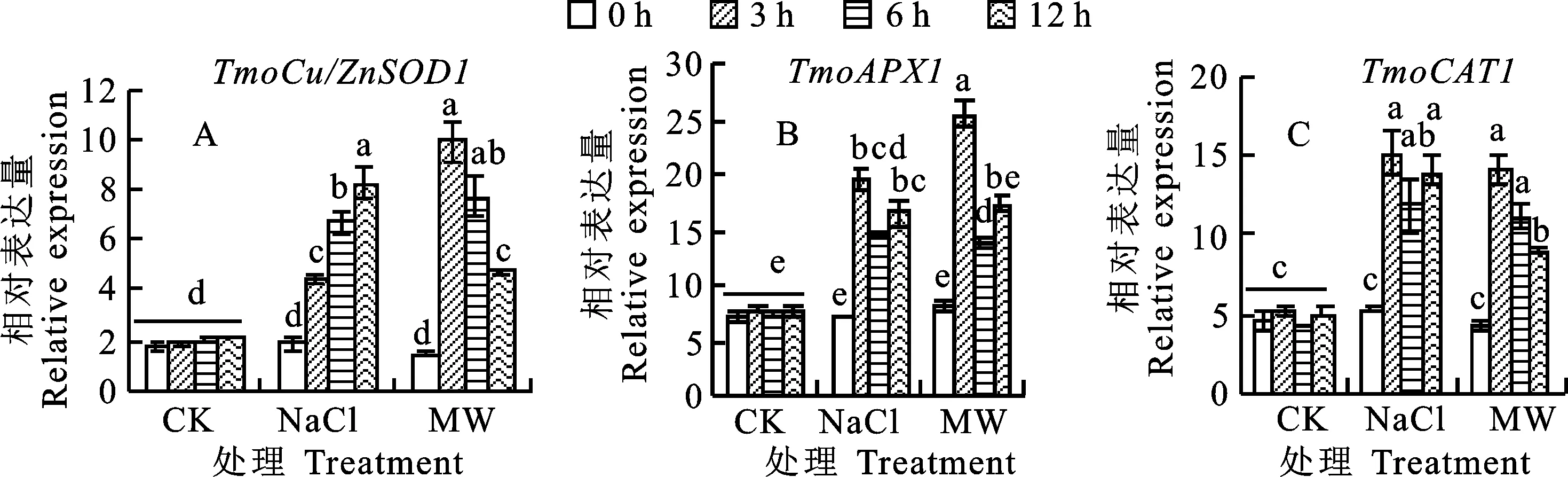
柱形上不同小写字母表示差异显著(P<0.05)