苋菜AmSPL基因家族转录组鉴定及表达分析
2020-11-26陈家兰赵春丽郑友峰赖钟雄刘生财
陈家兰 赵春丽 王 晓 郑友峰 赖钟雄 刘生财
(福建农林大学 园艺植物生物工程研究所,福州 350002)
转录因子(Transcription factors, TFs)是在转录水平上调控基因表达的DNA结合蛋白。SPL是植物特异转录因子的重要组成部分,对植物生长发育过程中起着重要调控作用[1]。这个转录因子家族的成员具有一个高度保守的DNA结合结构域,称为SBP结构域[2]。SBP结构域由约76个氨基酸残基组成,包含2个锌离子结合位点(Zn 1和Zn 2)。2个锌离子结合位点组成一个Cys3HisCys2HisCys或Cys6HisCys,其中第一个锌指是Cys3His或Cys4,第二个是Cys2HisCys[3]。在金鱼草中鉴定出第一个SPL基因,能够与SQUAMOSA基因启动子结合来控制花的早期发育[4]。目前,许多陆生植物中已经鉴定出SPL基因,例如水稻[5]、番茄[6]、拟南芥[7]、毛白杨[8]和葡萄[9]等。迄今为止,已知SPLS调节许多植物的生长发育过程,包括叶发育、相变[10]、芽发育[11]、孢子发生、GA信号[12]、铜稳态[13]和花青素的合成[14]等。
MicroRNAs (miRNAs) 对植物基因功能的调节中起着关键作用。miRNAs是20~24 bp的内源性单链非编码小RNA分子,在转录和转录后水平调节基因功能[15]。miRNA是转录后水平的调控因子,主要作用于靶基因的3′UTR。一般认为,当miRNA与靶位点完全互补,那么这些miRNA的结合往往引起靶基因mRNA的降解,这在植物中较为常见,而当miRNA与靶基因mRNA不完全互补,可导致miRNA在蛋白翻译水平上抑制靶基因表达(哺乳动物中较为常见),miRNAs已被发现在植物中调节生长、发育、代谢、非生物和生物胁迫应答反应[16-17]。
miR156家族成员通过负向调节SPLs,进而调控植物营养生长和生殖生长的转变、花的发育和浆果果皮颜色的形成。miR156的过表达上调了花青素合成关键基因表达,从而促进了花青素在细胞质中合成[18-19]。在拟南芥的SPL基因中有11个具有miR156的靶位点[20]。大多数拟南芥SPL基因miRNA反应元件(MREs)位于保守的SBP结构域的下游,是最后一个外显子的编码序列的一部分[21]。在水稻中,Os-miR156靶向11个OsSPL[22]。水稻与拟南芥一样,除OsSPL4和OsSPL13的靶位点位于3′UTR,大多数MREs都位于编码区[23]。同样,番茄中15个SPL基因中有10个被发现是miR156的靶点[24]。苋菜是石竹目苋科苋属的一年生保健蔬菜,品种繁多,营养价值高,生长速度快,抗逆性强。石竹目植物中红、黄甜菜色素取代花青素[25],苋菜茎和叶中富含甜菜红素,具有较高的营养价值和药用价值[26]。同时花青素和甜菜色素在许多方面都存在相似之处,包括发生机理、发展模式及色素合成的环境影响因子。虽然有研究报道miR156通过负调控SPL调节花青素合成[14],但关于SPL是否参与甜菜色素代谢的研究尚未见报道。本研究基于‘全红’苋菜的转录组数据库(SAR登录号:SRR11196359-SRR11196364),利用生物信息学手段对SPL家族进行筛选分析,同时分析苋菜SPL家族与miR156之间的调控模式,旨在明确苋菜SPL家族基因结构特征及响应蓝光条件的表达模式,以期为进一步研究和应用苋菜SPL转录因子的功能奠定基础。
1 材料与方法
1.1 材料与处理
‘全红’苋菜种子由福建农林大学园艺植物生物工程研究所提供。将‘全红’苋菜种子均匀地播种于铺有3层滤纸的塑料培养皿上,加入适量的去离子水,分别放于蓝光(25 ℃,24 h/d,光强115.96 μmol/(m2·s))和黑暗(25 ℃)的条件下,培养36~40 h后,取胚轴时期苋菜的茎段作为转录组测序的材料,每种处理取样3次,每次0.1 g,液氮速冻,由深圳华大基因股份有限公司进行转录组测序及分析。
1.2 方法
1.2.1数据来源及AmSPL家族基因的鉴定
本课题组测序获得‘全红’苋菜转录组数据库,根据测序数据在NT、NR、PFAM和Swiss-Prot 4个数据库进行关键词搜索,获得SPL家族相关基因,去除掉重复序列后,运用SMART(http:∥smart.embl-heidelberg.de/)在线软件对候选基因进行保守结构域分析,删除不含有SPL家族特征结构域的序列[27]。模式植物拟南芥中已知的16个SPL基因的核苷酸序列及其推导的氨基酸序列从TAIR数据库(https:∥www.arabidopsis.org/)中下载得到[28]。使用pfam(http:∥pfam.sanger.ac.uk)进行识别AtSPLs的SBP域[27]。
1.2.2AmSPL家族蛋白生物信息学分析
对筛选获得的SPL蛋白序列进行分析,分别利用在线软件ProtParam(http:∥web.expasy.org/protparam/)[29]对SOPMA (https:∥npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)[30]和Plant-mPLoc Server(http:∥www.csbio.sjtu.edu.cn/bioinf/plant-multi/#)[31]对氨基酸理化性质、蛋白的二级结构和亚细胞定位进行分析。‘全红’苋菜SPL家族蛋白的结构域使用SMART进行分析,蛋白结构图使用IBS软件作出。
利用在线软件 MEME Suite 5.0.5(http:∥meme-suite.org/tools/meme)分析‘全红’苋菜SPL家族蛋白序列的保守结构域[32]。利用软件MEGA-X构建‘全红’苋菜SPLs蛋白序列系统进化树,同时与拟南芥SPL家族蛋白共同构建系统进化树[33-34]。
利用在线分析软件psRNATarget(http:∥plantgrn.noble.org/psRNATarget/)对获得的17个AmSPL的cDNA序列进行miR156的靶向位点预测,期望值设置为5.0,整理后绘制靶位点图。苋菜miR156序列从small RNA数据库中获得。
1.2.3蓝光和黑暗处理下苋菜‘全红’SPL家族基因的差异表达分析
根据蓝光和黑暗处理下‘全红’苋菜胚轴的转录组测序数据中的FPKM(Fragments per kilobase per million, 每千个碱基的转录每百万映射读取的碎片)值,计算出均值后,通过GraphPad Prism 8.0.2软件对蓝光和黑暗处理下苋菜胚轴SPL家族基因的表达数据进行分析。
然后以转录组测序的蓝光和黑暗处理的‘全红’苋菜胚轴为试验材料,对本研究所筛选的SPL基因进行实时定量荧光PCR扩增,选用AmEF1a为内参,检测AmSPL基因的表达情况,根据SYBR®Premix Ex TaqTMII (Tli RNaseH Plus,Takara, Japan)试剂盒说明书进行qRT-PCR反应液的配制,在LightCycler480 Real-time PCR 仪器上进行试验,所有试样进行3次重复,采用2-ΔΔct的方法计算AmSPLs基因的相对表达量。根据AmSPL特异序列设计上下游定量引物,内参基因及其引物序列,见表1。

表1 qPCR 所用引物序列及退火温度Table 1 Primer sequences and their annealing temperature used in qPCR
2 结果与分析
2.1 AmSPL家族基因的筛选及蛋白特性
基于苋菜转录组数据库,通过关键词对4个蛋白数据库的注释进行搜索,初步获得52个SPL家族的相关基因,进一步通过SMART分析,最终得到 17个SPL家族基因,为使后续分析更加方便,根据转录组编号的顺序命名为AmSPL1(CL10801.Contig1_All) ~AmSPL17(Unigene9395_All)。由表2和表3可知,AmSPLs蛋白长度在186~777 个氨基酸,蛋白相对分子量在20.53~88.27 ku,其中有6个AmSPL蛋白属于酸性蛋白,其余11个为碱性蛋白;除了AmSPL16其余蛋白不稳定系数均>40,表明除AmSPL16,其余均为不稳定蛋白;平均亲水系数均为负值,表明17个AmSPL蛋白都是亲水蛋白。二级结构分析发现,无规则卷曲占据的比例最大,均>50%,亚细胞定位预测结果均定位在细胞核,并且推测AmSPL4,AmSPL11和AmSPL12同时定位在细胞质中。

表3 AmSPL家族蛋白二级结构和亚细胞定位分析Table 3 Two level structure and subcellular localization of AmSPL family proteins
由图1(a)可知,17个AmSPL蛋白中有3对基因自展值为100,这表明这3对AmSPL亲缘关系非常近,其可能存在功能冗余的现象。SMART分析的AmSPL家族结构(图1(b))表明AmSPL家族蛋白均含有SBP结构域。
2.2 AmSPL家族结构域分析
SPL家族之间所含保守基序种类和数目存在一定的差别,其中AmSPL1、AmSPL3、AmSPL4、AmSPL5、AmSPL6、AmSPL7、AmSPL8、AmSPL9、AmSPL11、AmSPL14和AmSPL17含有Motif 1,而AmSPL4、AmSPL14、AmSPL17含有Motif 4。出现频率最高的Motif 1是SPL家族结构域中非常重要的保守基序,SBP结构域,说明本研究筛选得到的17个AmSPL家族成员大都具有SBP结构域。通过Jalview软件对其序列进行比对和分析(图2),发现其在‘全红’苋菜中高度保守。
2.3 ‘全红’苋菜与拟南芥SPL家族蛋白系统进化树
由图3可知, 33个SPL成员共分为6个组,GroupⅠ含有9个AmSPL家族蛋白,分别为AmSP 4,AmSPL 12,AmSPL 14,AmSPL 17,AmSPL2,AmSPL1,AmSPL15,AmSPL3和AmSPL16,并含有4个AtSPL家族蛋白;GroupⅡ含有AmSPL10和AmSPL8这2个AmSPL家族蛋白,同时含有3个AtSPL家族蛋白;Group Ⅲ含有3个AmSPL家族蛋白,分别为AmSPL 7,AmSPL 5和AmSPL 9,同时含有6个AtSPL家族蛋白;GroupⅣ含有2个AmSPL家族蛋白,分别为AmSPL 6和AmSPL 13;GroupⅤ不含有AmSPL家族蛋白;Group Ⅵ含有1个为AmSPL11。

红色是SBP结构域,粉红色是DEXDc结构域,蓝色是低复杂区域,绿色是跨膜区。图中数字表示自展值。Red represents SBP domain. Pink represents DEXDc domain. Blue represents low complex region. Green represents the transmembrane region. Number represents bootstrap value.图1 AmSPL家族进化树(a)及蛋白结构分析(b)Fig.1 Phylogenetic tree (a) and structural analysis (b) of AmSPL family proteins

图2 AmSPL家族蛋白SBP保守结构域Fig.2 SBP conserved domains of AmSPL

不同的分支颜色代表分属于不同的组。图中数字表示自展值。Different branch colors represent belonging to different groups. The digital represents bootstrap value.图3 ‘全红’苋菜(Am)与拟南芥(At)SPL家族蛋白系统进化树Fig.3 Phylogenetic tree of SPL family proteins of Amaranthus ‘Quanhong’ and Arabidopsis thaliana
‘全红’苋菜和拟南芥分别在GroupⅡ和Group Ⅵ含有1对自展值为100(AmSPL10和AtSPL8,AmSPL11和AtSPL7)的直系同源基因对(图3)。此外,大部分含有相同基序的蛋白序列被划分为同一组,相近分枝的SPL蛋白分析,推测它们可能具有相同或相似的功能。
2.4 AmSPL家族基因中miR156作用位点的预测
由图4可知,17个AmSPL家族成员中有8个成员含有与miR156成熟体互补序列,分别为AmSPL1,AmSPL2,AmSPL4,AmSPL5,AmSPL6,AmSPL7,AmSPL8和AmSPL13,初步表明这8个AmSPLs可能是miR156的靶基因。

图4 Am-miR156a-5p、Am-miR156-1与8个AmSPLs互补序列的序列比对Fig.4 Sequence alignment of Am-miR156a-5p and Am-miR156-1 with their complementary sequence of 8 AmSPLs
2.5 AmSPL基因家族的表达分析
由图5可知,AmSPL基因表达丰度各不相同,黑暗条件下大部分的AmSPL基因表达量显著高于蓝光条件下,其中AmSPL1在黑暗处理下的表达量最强;其中AmSPL3的表达量最弱,但在蓝光条件下的表达量显著上升,与其相似度最高的AmSPL16表达量较高,且呈现相反趋势,说明虽然它们同属一个基因家族,但功能不同。在蓝光条件下,AmSPL11的表达量最高,其余AmSPL的表达量都相对较低。FPKM均值与实时荧光定量PCR结果相比较,除了AmSPL3,AmSPL11,AmSPL12,AmSPL13和AmSPL17,其余的AmSPLs在黑暗和蓝光处理下胚轴中的相对表达量趋势与转录组数据的FPKM值相符,并且实时荧光定量PCR结果表明,所有AmSPL基因全都表现为黑暗条件下表达量显著高于蓝光处理的表达量,虽然趋势率上存在差异,但这些相似的表达模式也表明这些AmSPLs可能存在功能上的冗余,并推测出AmSPLs的表达可能通过受到蓝光的负调控,其变化的调控机制还需要进一步研究。
3 讨 论
目前基于全基因组研究的深入,越来越多物种的SPL基因家族被鉴定出来,其中拟南芥中发现16个,水稻19个,小麦58个,番茄15个。然而对于没有全基因组的物种更多的是通过转录组进行分析鉴定。SPL是植物的特异性转录因子,含有高度保守的SBP结构域[35],它能够与花分生组织中的特性基因SQUAMOSA和其同源基因的启动子特异性结合,从而在植物的生长发育过程中起着重要的调控作用[36-38]。有研究发现,拟南芥miR156的过表达在较低的环境温度(16 ℃)下导致开花延迟[39]。在miR156的靶基因中,SPL3表达水平主要降低,这可能是因为在16 ℃时,miR156更易使SPL3降解。同时,miR156的过表达植株可上调花青素合成关键基因表达,从而促进花青素的合成。本研究在转录组范围内鉴定‘全红’苋菜的SPL基因,并进行生物信息学分析和表达情况分析。通过鉴定分析,共获得17个AmSPL家族成员,比拟南芥中的成员数量多1个。通过对AmSPL家族蛋白保守结构域预测,同时与拟南芥进行比较分析,发现许多保守的序列特征,例如,SPL都含有高度保守的SBP结构域,约有78个氨基酸残基。系统进化分析表明,‘全红’苋菜和拟南芥共33个SPL蛋白聚成6个类群,除类群Ⅴ,其余每个类群都至少含有1个AmSPL和1个 AtSPL,不同物种间的SPL基因功能具有相似性,也证明SPL家族之间具有一定的功能保守性。Wang等[40]研究表明AtSPL2,AtSPL10,AtSPL11可以调控茎生叶和花的形态特征,在系统发育树(图3)中发现,AmSPL2,AmSPL4,AmSPL12,AmSPL14和AmSPL17与AtSPL2,AtSPL10和AtSPL11处于同一个类群,也可能调控苋菜茎生叶和花的形态特征;AtSPL9和AtSPL15可以调控拟南芥幼年到成年生长期的转变和叶片生长的起始速率[41],AmSPL8与AtSPL9和AtSPL15属于同一类群且自展值为100,因此AmSPL8在苋菜中很有可能调控着幼年到成年的转变。转录组的FPKM值与qRT-PCR结果基本一致,蓝光条件下的AmSPL家族基因表达量显著低于黑暗条件下的表达量,同时蓝光条件下积累更多的甜菜色素,可推测AmSPL参与苋菜甜菜色素的合成。Yang等[17]研究发现,处于同一组的基因显示相同或相似的表达模式,其中一些会显现出差异模式,这也表明植物SPLs具有多样性,所以SPLs基因在苋菜中的功能还需要进行进一步的探究与鉴定。
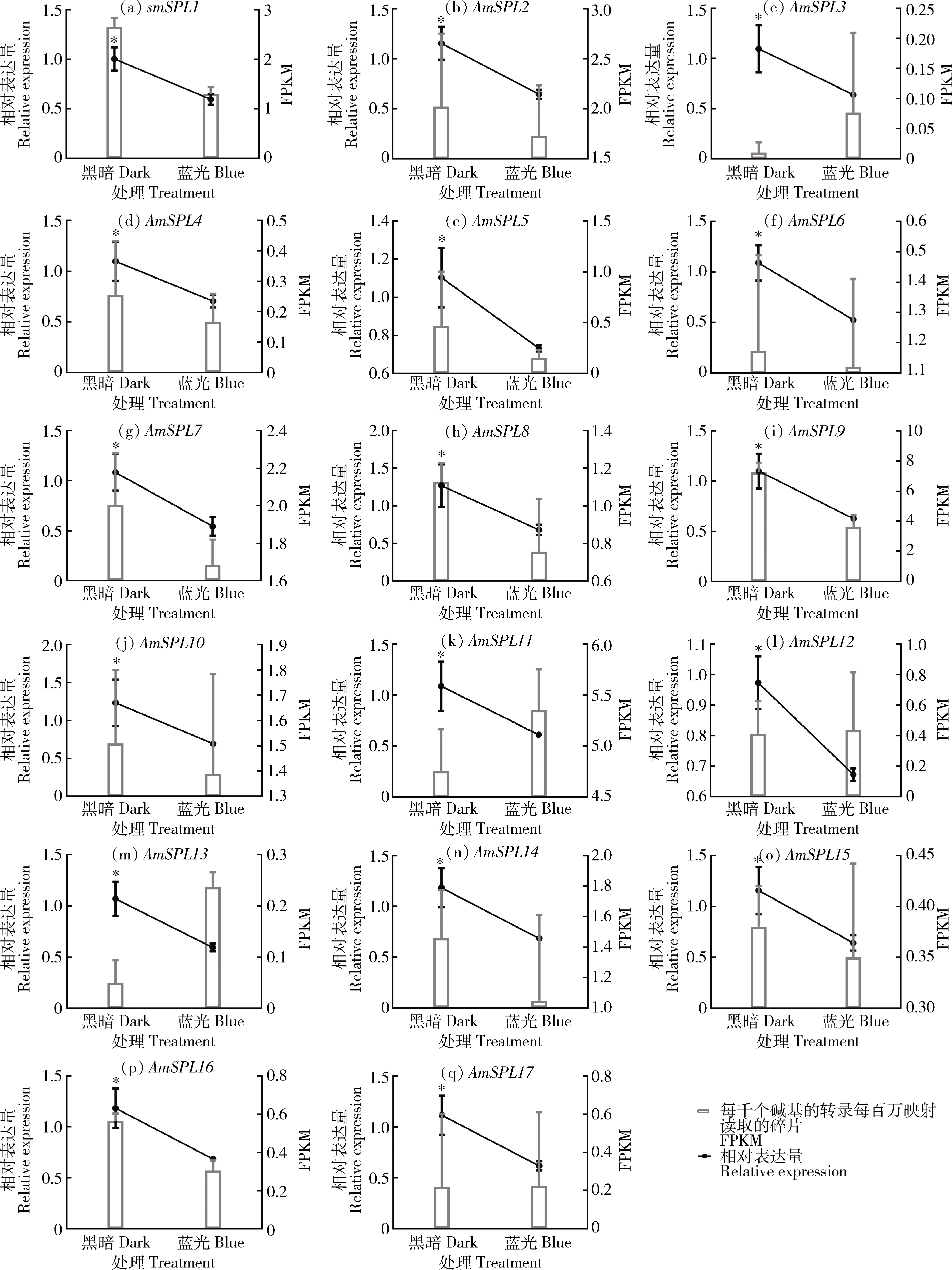
*表示不同处理间差异显著(P<0.05)。* indicate significant difference in different treatment, at 0.05 level.图5 苋菜AmSPLs的qRT-PCR定量结果与其对应的FPKM值Fig.5 qRT-PCR quantitative results of AmSPLs and the FPKM values
研究表明植物中的大部分SPL家族基因是 miR156的靶基因,而且miR156/SPL调控模式在调节植物生长发育中起重要作用[42]。拟南芥[10]、番茄[6]、大豆[43]和水稻[22]等植物中SPL家族的大部分成员被证实是miR156的靶基因,本研究也预测17个AmSPLs中有8个是miR156的靶基因,这表明miR156介导的转录后调控在植物中具有保守性。这些结果说明miR156对SPL家族调控起着重要的作用。虽然未被预测出其余9个AmSPLs是Am-miR156的靶基因,但也不能排除其存在的可能性。同时也有研究表明miR156靶向SPL转录因子对拟南芥花青素生物合成存在负调控[14], Kishima等[44]早已证明蓝光可以诱导甜菜色素的积累,而花青素和甜菜色素的合成在环境影响因子中存在相似之处,本研究中的qRT-PCR结果也显示蓝光条件下AmSPL的表达量均显著低于黑暗中,因此可以推测蓝光诱导miR156过表达从而抑制AmSPL的表达,促进甜菜色素的积累。
