广东省部分地区禽产品中印第安纳沙门菌的耐药性和耐药基因分子特征研究
2020-11-18刘海霞黄雪欢邬雨倩王存敏詹泽强高凤磊张建民
刘海霞,黄雪欢,邬雨倩,王存敏,詹泽强,高凤磊,廖 明,张建民
(1.广东农工商职业技术学院热带农林学院,广州510507;2.华南农业大学兽医学院 人兽共患病防控制剂国家地方联合工程实验室 农业农村部人畜共患病重点实验室 广东省动物源性人兽共患病预防与控制重点实验室,广州510642)
沙门菌(Salmonella)是一种受到全世界广泛关注的人兽共患病原菌,也是引起世界范围内细菌性食物中毒的重要致病菌,可以通过动物、食物以及一些其他途径传递,给人类健康及畜禽生产带来极大的危害。全世界每年约有9380万人感染沙门菌,造成约15.5万人死亡[1-2]。在我国细菌性食物中毒中,70%~80%是由沙门菌造成的[3]。它广泛分布于自然界,常污染猪肉、蛋类、鸡肉等动物性食品[4-5]。造成食品污染和人源感染的沙门菌优势血清型主要为鼠伤寒和肠炎沙门菌[6]。印第安纳沙门菌在过去比较少见,1955年首次被分离于美国印第安纳州一个临床病例中[7],随后,在北美和欧洲,有几起由印第安纳沙门菌引起的呕吐、腹泻、发烧等临床病例[7]。但近几年逐渐成为重要的多重耐药血清型之一。1984年,中国首次在一个外国游客体内分离得到印第安纳沙门菌,在随后的24年(1984-2007年)共分离到13株菌。自2008年以来,由印第安纳沙门菌感染引起的临床报告数量急剧增加[7]。值得注意的是,印第安纳沙门菌在动物、食物和环境中的流行程度也逐渐提高,从而使印第安纳沙门菌成为了中国沙门菌的主要血清型,取代了传统上占主导地位的血清型[7]。在印第安纳沙门菌流行的同时,还出现了多重耐药印第安纳沙门菌[7-8],其迅速增长的耐药性己严重削弱了传统抗生素药物的临床作用,对于畜禽动物性疾病防控以及人类的安全健康造成了巨大的威胁,因此亟需对印第安纳沙门菌进行深入的研究。
本研究检测了沙门菌特定血清型印第安纳沙门菌耐药性及携带耐药基因的情况,并基于PFGE技术进行分型,探究采集于广东省广州市和佛山市禽样品中的印第安纳沙门菌分离株的耐药性和耐药基因分子特征,为了解印第安纳沙门菌耐药情况和保障食品安全提供数据支持。
1 材料与方法
1.1 菌株来源36株印第安纳沙门菌(SalmonellaIndiana)于2018年分离自广东省广州市(n=15)和佛山市(n=21)禽样品;标准菌株SalmonellaBranderup H9812以及肠杆菌(Escherichia coli)ATCC 25922标准质控菌株由本实验室保藏。
1.2 主要试剂和培养基抗生素购自广州翔博生物科技有限公司;木糖赖氨酸脱氧胆盐(XLD)、四硫磺酸钠煌绿(TTB)增菌液、缓冲蛋白胨水(BPW)购自美国Becton公司;PFGE专用琼脂糖(seaKem gold agarose)购自美国Bio-rad公司;沙门菌属诊断血清购自S&A公司;沙门菌显色培养基、MH琼脂、LB琼脂培养基和麦康凯培养基购自广东环凯微生物科技有限公司;限制性内切酶XbaⅠ、细胞悬浮缓冲液(cell suspension buffer,CSB)、细胞裂解缓冲液(cell lysis buffer,CLB)购自宝生物(大连)有限公司。
1.3 血清型鉴定沙门菌诊断血清购自宁波天润生物药业有限公司。对沙门菌菌株做O多价血清凝集反应及H抗原鉴定等,按GB4789.4-2010操作,依据White- Kauffmann-Le Minor抗原表对血清学分型鉴定的结果进行血清型鉴定。
1.4 药敏试验参照临床实验室标准化协会(CLSI,2010)推荐的琼脂微量稀释法[9],对菌株进行14种抗菌药物的最小抑制浓度(the minimum inhibitory concentration,MIC)试验,根据CLSI提供的标准判读药敏结果(表1)。

表1 抗生素及稀释浓度范围Table 1 Antibiotics and dilution concentration range
1.5 印第安纳沙门菌耐药基因的PCR检测提取菌株的DNA模板进行沙门菌相关基因的检测及序列测定,鉴定引物参考文献[10],引物由广州艾基生物技术有限公司合成。PCR鉴定结果为阳性的产物送至生工生物工程(上海)有限公司进行测序。PCR反应程序:94℃预变性7 min;94℃变性1 min,55℃退火30 s,72℃延伸1 min,34个循环;72℃延伸5 min。PCR产物通过1%琼脂糖凝胶电泳进行检测。1.6 沙门菌的PFGE分型 方法参考美国CDC提供的标准方案进行[11],先用琼脂糖包埋印第安纳沙门菌和标准菌株沙门菌H9812后,加入50 UXbaⅠ酶,在37℃的水浴中消化1.5~2 h。使用Chef Mapper电泳系统(Bio-Rad),在14℃条件下电泳分离基因组18 h。使用BioNumerics 6.5版软件(Applied Maths)分析PFGE结果。
2 结果与讨论
2.1 药敏试验结果36株印第安纳沙门菌药敏结果显示:菌株对四环素的耐药率最高,达100%,其次为磺胺异恶唑(97.2%)、氧氟沙星(97.2%)、氟苯尼考(75%)、氯霉素(75%)、萘啶酸(72.2%)、庆大霉素(63.9%)、环丙沙星(55.6%)、阿米卡星(30.6%)、头孢噻肟(30.6%)、氨 西林(22.2%),对头孢吡肟、多粘菌素B和亚胺培南完全敏感(表2)。97.2%(35/36)印第安纳沙门菌呈现多重耐药性(耐3种及3种以上的抗生素),1株(2.8%)耐1种抗生素,23株(63.9%)可耐8~10种抗生素。通过对耐药分析可知,菌株对磺胺异恶唑、氧氟沙星、萘啶酸药物以及酰胺醇类药物耐药严重,这与国内和国外相关研究报道基本一致[7,12-14],可能由于在食品动物生产和临床环境中使用不同种类和剂量的抗菌剂,也可能与这些传统的抗菌药物在畜禽养殖中已被长时间广泛使用和滥用、给药剂量次数不规则等给药方法密切相关。特别值得注意的是,菌株对四环素的耐药率达100%,这可能是由于四环素类药物是高效的广谱性抗菌药,在临床上应用广泛,因不合理的应用,导致近年来细菌对四环素类药物出现高水平耐药[15-16]。喹诺酮类和头孢类药物是目前临床上治疗沙门菌感染并具有良好治疗作用的重要抗生素[15-16],但是本次研究结果发现,禽样品中分离到的印第安纳沙门菌对喹诺酮类和头孢类药物出现耐药,对氧氟沙星、萘啶酸、环丙沙星、头孢噻肟的耐药率达30%以上。值得注意的是,临床上治疗细菌感染性疾病的“最后一线”药物粘菌素类和碳青霉烯类抗生素均不耐药,如多粘菌素B和亚胺培南,这与之前的研究结果[8,17]一致。本研究中,高达97.2%的菌株表现出多重耐药性,耐药谱多样,表明广东省部分地区禽产品中印第安纳沙门菌的耐药性严重,警醒我们在畜禽生产中要科学应用、避免滥用抗菌药物,以减少耐药性的产生。
2.2 印第安纳沙门菌相关耐药基因检测结果β-内酰胺酶类耐药基因是介导头孢菌素耐药的主要机制之一[13,16],本研究对36株印第安纳沙门菌头孢类常见耐药基因(blaOXA、blaTEM、blaCTX-M、blaCMY和blashv)进行PCR检测及测序。结果显示,共有17株菌株携带有β-内酰胺酶基因。其中,有17株(47.2%)携带blaOXA-1基因,5株(13.9%)含有blaTEM(blaTEM-1和blaTEM-135)以及1株(2.8%)含有blaCTX-M-55基因。没有菌株检测到blaCMY和blashv基因,这与以前的报道类似[7,16],可以被用来解释本次研究中菌株对头孢噻肟耐药的可能原因。喹诺酮类耐药决定区基因parC和gyrA突变率分别为86.1%(31株)和61.1%(22株)。在gyrA基因中,最为常见的突变是位于83位点处的丝氨酸突变成苯丙氨酸,所占的比例为58.3%(21株)。除此之外,gyrA基因还存在87位点的天冬氨酸突变为甘氨酸或天冬酰胺,所占的比例分别为59.4%(19株)、8.3%(3株)。在parC基因中,83.3%(30株)菌株突变发生在62位点苏氨酸突变为丝氨酸(Thr62→Ser)、52.8%(19株)菌株在85位点丝氨酸突变为精氨酸(Ser85→Arg)、44.4%(16株)在140位点谷氨酸突变为天冬氨酸(Glu140→Asp);94.4%(34株)菌株携带至少1个parC或gyrA突变,66.7%(24株)菌株携带至少2个QRDRs突变(表3)。PMQR基因检测中检出aac(6')-Ib-cr最多(21株,58.3%),其次是qnrS(13株,36.1%),检出qnrB最少(4株,11.1%)。本研究并未检出qnrA、qnrC、qnrD和qepA。36株印第安纳沙门菌中共有33株携带至少1种喹诺酮类耐药基因(plasmid-mediated quinolone resistance,PMQR),阳性率为91.7%。共有5株携带至少2种PMQR基因,阳性率为13.9%(表4)。喹诺酮类耐药决定区(quinolone resistance determining region,QRDR)基因突变和质粒介导的PMQR基因是沙门菌对喹诺酮类药物产生耐药性的主要原因[16]。在本研究中,94.4%菌株至少发生1个parC或gyrA突变。有研究表明,QRDR的点突变可以降低对喹诺酮类药物的敏感性[18-19],QRDR突变结果与菌株对氧氟沙星和萘啶酸的高耐药率结果具有一致性。质粒介导的喹诺酮类耐药基因可通过外排泵等机制增强细菌对喹诺酮类药物的耐药性[20],研究结果显示,高达91.7%的菌株携带PMQR耐药基因,且检测出有PMQR基因的菌株对氧氟沙星都耐药。表明高携带率的PMQR基因和QRDR位点高突变率与印第安纳沙门菌对喹诺酮表现出的高耐药存在一定的关系。
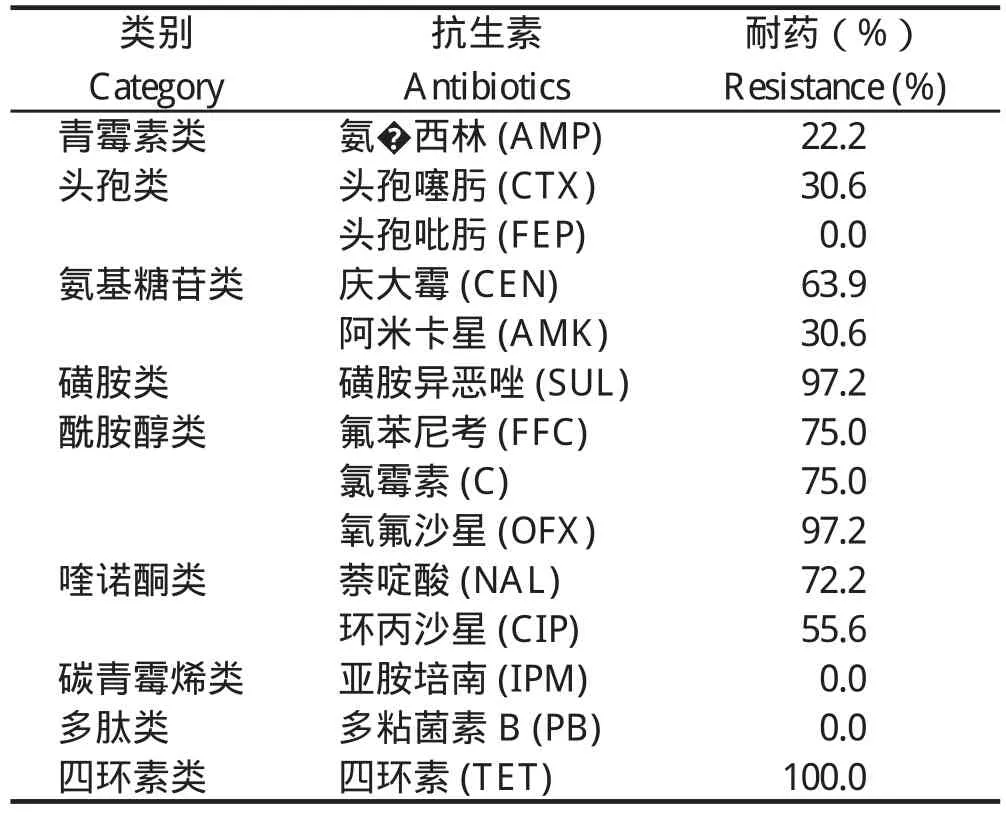
表2 36株印第安纳沙门菌药敏试验结果Table 2 Drug susceptibility test results of 36 strains of Salmonella Indiana

表3 36株印第安纳沙门菌QRDR突变的检出结果Table 3 Detection results of QRDR mutations in 36 strains of Salmonella Indiana
2.3 PFGE分型结果目前国内外研究者常用PFGE技术进行病原菌的分子分型及溯源研究[11],广东省广州市和佛山市禽样品中分离出的36株印第安纳沙门菌的PFGE分型结果显示,共有32种PFGE基因指纹图谱产生,根据菌株间相似度80%来划分PFGE簇,将36株印第安纳沙门菌分为8个PFGE型簇和2个单独条带,8个基因簇分别是A簇、B簇、C簇、D簇、E簇、F簇,G簇和H簇,包含的菌株数分别为4株、4株、7株、3株、2株、2株、2株和8株。所含的基因型数分别为:4个、4个、4个、3个、2个、2个、2个、7个。其中H簇最大,由22.2%(8/36)的菌株组成(图1)。结果表明:广州市和佛山市流行的印第安纳沙门菌基因型种类较复杂,且基因型差异性较大,呈现遗传多样性。H簇是一个优势簇,其中包含不同采样点,不同样品来源的菌株,有不同源菌株带型相似度达到了100%,这提示禽产品间可能存在交叉污染或者共同来源。
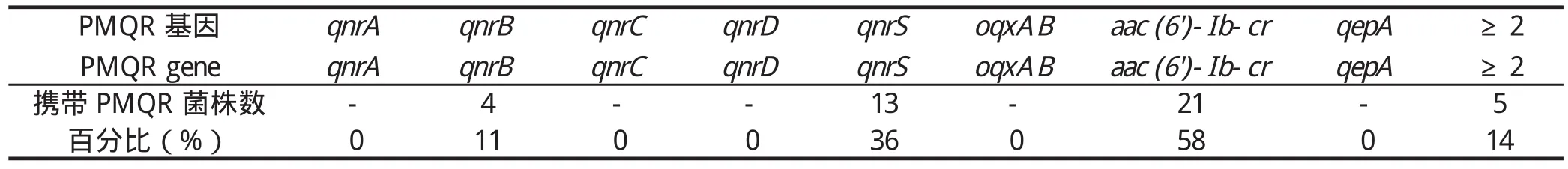
表4 印第安纳沙门菌PMQR的检出率Table 4 Detection rates of Salmonella Indiana PMQR
本研究结果显示,广东省部分地区禽源样品中印第安纳沙门菌多重耐药性严重,喹诺酮类耐药决定区gyrA和parC基因突变和质粒介导的喹诺酮类耐药基因(PMQR)在菌株中普遍存在,而PFGE分子分型显示其遗传是多样性的。同源菌株的存在表明存在交叉污染或共同来源。为减少印第安纳沙门菌引起的公共卫生健康威胁,应加强食品安全监督并规范抗菌药物的使用。

图1 印第安纳沙门菌PFGE图谱Fig.1 PFGE map of Salmonella Indiana
