大斑芫菁转录组编码序列密码子使用偏好性分析
2020-11-16刘春雷陈艾萌刘燕琴曹敏陈强
刘春雷,陈艾萌,刘燕琴,曹敏,陈强
论著·中药研究与开发
大斑芫菁转录组编码序列密码子使用偏好性分析
刘春雷1,陈艾萌2,刘燕琴1,曹敏1,陈强1
1.重庆市药物种植研究所,重庆 408435;2.内江市农业科学院,四川 内江 641000
探讨大斑芫菁转录组编码序列的密码子使用特点及其影响因素,为运用基因工程技术实现斑蝥素异源生物合成提供依据。利用Codon W和EMBOSS软件分析10 264条大斑芫菁转录组编码序列的密码子使用模式。大斑芫菁转录组编码序列的平均GC、GC3含量分别为0.387、0.321,GC12与GC3呈显著正相关(=0.369,<0.001);ENc-GC3及偏倚性分析结果显示,大部分基因分布于标准曲线和平面中心点周围,有少部分基因偏离较远。通过基因高表达优越密码子方法确定了6个大斑芫菁最优密码子,分别为GCC、CGC、GGC、AUC、UUC、ACC。密码子使用频率分析结果显示,大斑芫菁与果蝇整体密码子使用偏好性差异最大,与酵母菌的差异最小。大斑芫菁转录组编码序列的密码子使用偏好性以突变影响为主,多种因素共同作用的结果。酵母菌可作为大斑芫菁目的基因异源转化的理想受体系统。
大斑芫菁;转录组;密码子偏好性;最优密码子
大斑芫菁Pallas又名南方大斑蝥,为鞘翅目芫菁科斑芫菁属昆虫,是现行《中华人民共和国药典》收录的斑蝥来源昆虫之一,具有破血逐瘀、散结消癥、攻毒蚀疮等功效[1]。斑蝥素(cantharidin,C10H12O4)为斑蝥体内产生的一种防御物质,具有抗炎、抗肿瘤作用[2-3]。斑蝥素及其衍生物是许多抗癌制剂的原料药[4-5]。目前斑蝥主要来源于野生,随着过度搜捕,部分地区野生资源已经濒危。因此,开展天然斑蝥素生物合成机理研究,探索斑蝥素生物合成途径,是解决斑蝥素资源匮乏的思路之一。
遗传密码子是DNA与蛋白之间信息传递的基本单位。大多数氨基酸由多个同义密码子编码,然而生物体对同义密码子的选择具有偏好性。物种间[6]、基因间[7-8]、组织间[9-10]的密码子使用偏好性均存在差异。研究表明,生物体密码子偏好性与GC含量[11]、基因表达水平[12]、RNA结构[13]、蛋白结构[14]、基因长度[15]、tRNA丰度[16-17]、环境压力[18]等因素有关。随着测序技术及天然产物研究的发展,药用动植物的密码子偏好性研究积累了一定成果[19-23],但鲜见药用昆虫的相关报道。生物体对密码子的选择能够提高基因的翻译效率及表达量[24],因此,研究密码子偏好性有助于外源基因密码子的优化,筛选适宜宿主,并对转录、翻译的调控机制研究具有重要意义[23-25]。
笔者通过对大斑芫菁转录组数据进行密码子偏好性分析,揭示大斑芫菁基因的密码子使用规律,并与4种模式生物进行比较,以期为斑蝥目的基因选择适宜的异源表达宿主及密码子优化提供依据。
1 材料与方法
1.1 数据来源
大斑芫菁转录组数据来源于本课题组前期选取大斑芫菁蛹期、成虫期、成熟期样品,并基于Illumina HiSeq 2000平台进行转录组测序和组装获得的Unigene。采用TransDecoder软件预测Unigene的编码区序列及其对应氨基酸序列,编写perl程序筛选其中同时预测到起始密码子和终止密码子及长度>300 bp的完整编码序列,用于密码子偏好性分析。
4种模式生物大肠杆菌、酵母菌、果蝇、家蚕的密码子偏好性数据来自Codon Usage Database在线数据库(http://www.kazusa.or.jp/codon/)。
1.2 碱基含量及中性绘图分析
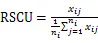
1.3 GC3关联及偏倚性分析
ENC-GC3关联分析用于探究密码子偏好性与碱基成分之间的关系。以ENC为纵坐标、GC3为横坐标绘制散点图,并进行非线性拟合回归分析。偏倚性分析以A3/(A3+U3)为纵坐标、G3/(G3+C3)为横坐标绘制散点图,计算基因平均分布位置,用于评价各基因密码子中嘌呤与嘧啶之间的关系。
1.4 最优密码子及密码子使用频率分析
采用EMBOSS软件的CUSP模块计算大斑芫菁编码序列密码子使用频率(frequency of optional codons,FOP),并与4种模式生物的密码子进行比较分析,当两两之间密码子使用频率比值>2.0或<0.5时,表明两者具有明显的密码子偏好差异。
参照Yang等[27]和李滢等[21]的方法,将大斑芫菁的编码序列按CAI值由高到低排序,提取前后各5%的序列分别代表高低表达的基因,然后计算各密码子平均RSCU值。当密码子在高低表达基因中RSCU值的差异>0.3,且在高表达基因中RSCU≥1、在低表达基因中RSCU≤1,即为最优密码子。
2 结果与分析
2.1 GC含量及中性绘图分析
从大斑芫菁转录组数据中共筛选获得10 264条完整的编码序列,密码子使用模式分析结果见图1。
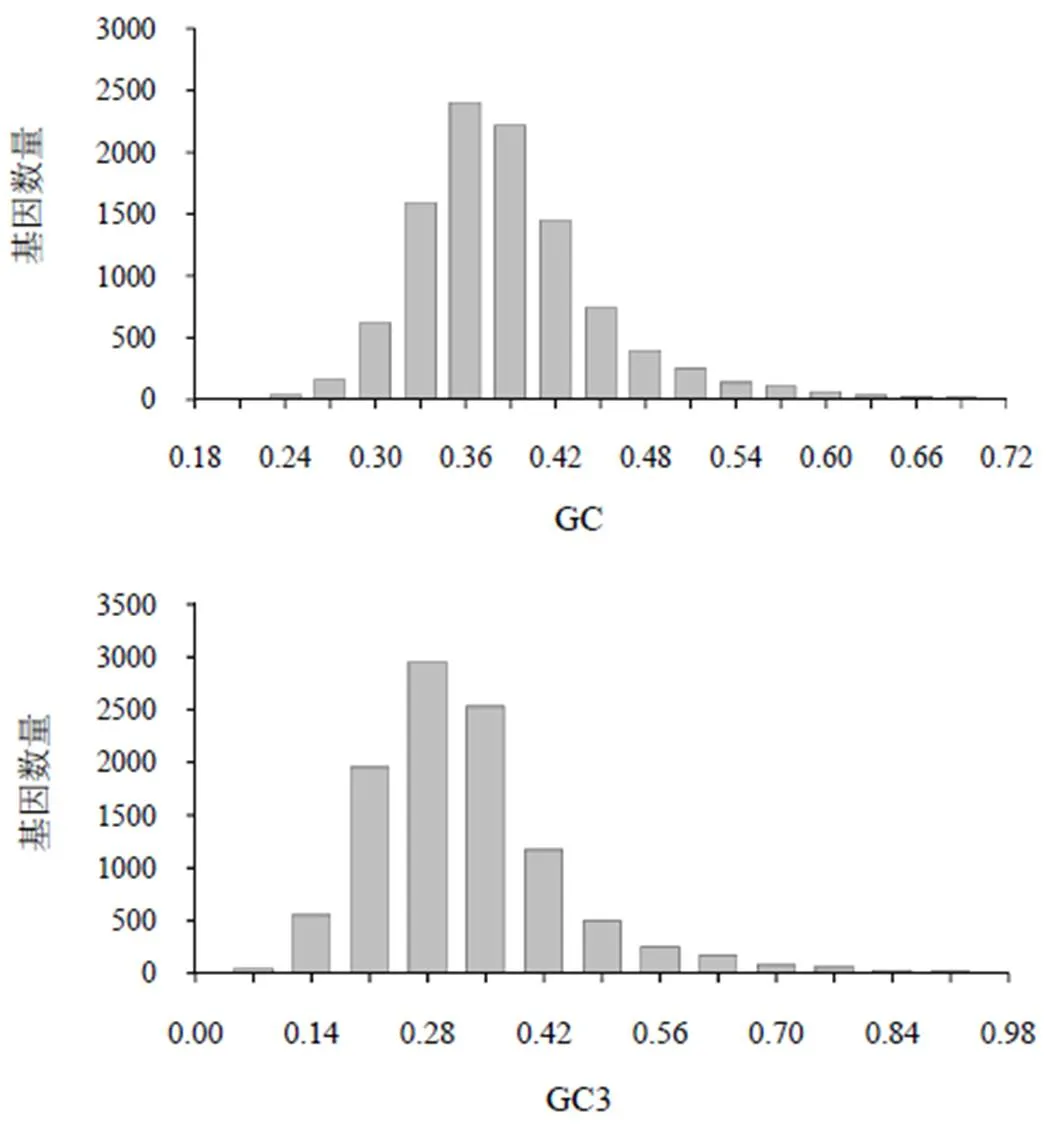
图1 大斑芫菁转录组编码序列GC及GC3含量分析
平均GC含量为0.387,变化范围为0.199~0.736。87.9%的基因GC含量为0.30~0.45。GC3含量为0.025~0.959,平均值仅为0.321,表明不同基因间密码子使用偏好性具有一定差异。但93.9%的基因GC3含量为0.14~0.49,表明大斑芫菁转录组密码子及末位碱基均偏好使用A或U。
GC12变化范围为0.238~0.721,与GC3含量呈显著正相关(=0.369,<0.001),回归方程为=0.180 7+0.362 3,2=0.136 2,见图2。表明大斑芫菁转录组密码子的使用主要受突变压力影响。但GC12与GC3含量的相关性较弱,表明选择压力可能同时影响着大斑芫菁转录组密码子的使用偏好性。
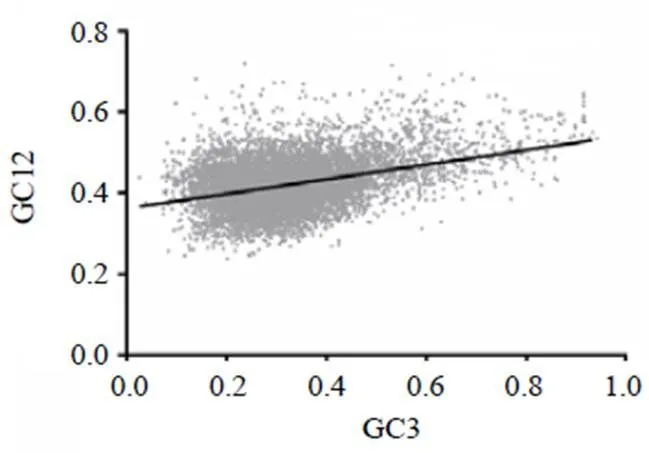
图2 大斑芫菁转录组编码序列中性绘图
2.2 有效密码子数及偏倚性分析
ENC变化范围为24.34~61,均值为46.86,其中ENC≤35的基因有312个(3.04%),ENC=61的基因有128个(1.25%)。CAI值介于0.093~0.635,均值为0.209,表明大斑芫菁转录组的编码序列除少数具有较强的偏好性外,其整体密码子的偏好性较弱。ENC与GC3关联性分析结果显示,大部分基因均匀分布在标准曲线周围,其回归方程为=-144.62+139.12+19.048,2=0.544 7,表明密码子的偏好性主要来自突变压力。少部分基因偏离曲线较远,表明部分基因受到选择压力的影响。见图3。
图3 大斑芫菁转录组编码序列ENC-GC3s相关性分析
偏倚性分析结果见图4。大斑芫菁基因平均分布位置为(0.505,0.527),大部分基因样本分布于中心点附近,其碱基中嘌呤与嘧啶出现的概率相对一致,少部分基因偏离中心点较远,进一步表明大斑芫菁密码子使用偏好性主要受到突变压力的影响。
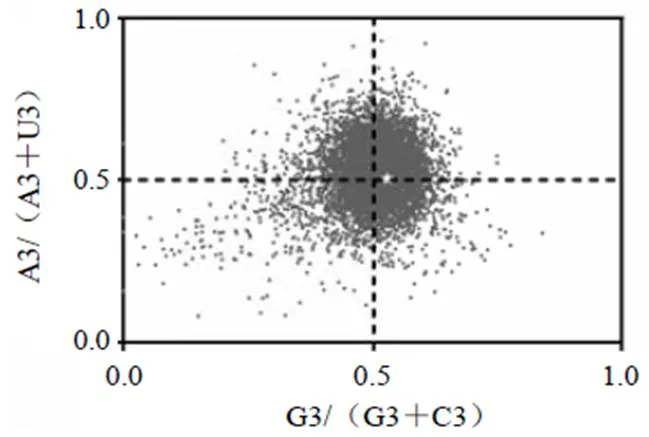
注:☆为基因平均分布位置
2.3 最优密码子分析
大斑芫菁转录组高、低表达序列RSCU分析结果见表1。筛选到6个密码子作为大斑芫菁转录组编码序列的最优密码子,分别为GCC、CGC、GGC、AUC、UUC、ACC。这些密码子均以C结尾,表明大斑芫菁转录组的最优密码子偏好以G或C结尾。
表1 大斑芫菁转录组高低表达基因的RSCU比较分析
氨基酸密码子RSCU 氨基酸密码子RSCU 高表达低表达 高表达低表达 AlaGCA0.851.29 LeuCUA0.590.82 GCC*1.140.83 CUC0.870.28 GCG0.600.56 CUG0.880.32 GCU1.411.29 CUU0.600.41 ArgAGA0.881.39 UUA1.542.71 AGG0.520.57 UUG1.501.46 CGA0.811.25 LysAAA1.231.57 CGC*1.230.50 AAG0.760.43 CGG0.550.61 PheUUC*1.210.80 CGU1.921.67 UUU0.751.19 AsnAAC0.900.39 ProCCA1.371.78 AAU1.081.61 CCC0.740.37 AspGAC0.720.36 CCG1.201.22 GAU1.281.62 CCU0.670.62 CysUGC0.770.41 SerAGC0.870.45 UGU1.001.43 AGU1.101.58 GlnCAA1.221.61 UCA1.041.61 CAG0.760.38 UCC0.920.45 GAA1.191.44 UCG1.191.23 GAG0.800.54 UCU0.880.68 GlyGGA0.650.96 StopUAA1.831.71 GGC*1.250.74 UAG0.500.55 GGG0.300.39 UGA0.670.74 GGU1.801.88 ThrACA1.101.91 IleAUA0.491.07 ACC*1.140.52 AUC*1.270.62 ACG0.700.73 AUU1.221.32 ACU1.050.83 ValGUA0.781.20 TrpUGG0.780.85 GUC0.980.57 TyrUAC0.970.54 GUG0.820.65 UAU0.951.44 GUU1.411.57 HisCAC0.900.53 MetAUG1.001.00 CAU1.001.36
注:*最优密码子
2.4 大斑芫菁与模式生物密码子使用频率比较
大斑芫菁与大肠杆菌、酵母菌、果蝇、家蚕密码子使用频率比较结果见图5。大斑芫菁与4种模式生物间的整体密码子使用偏好性差异较大,与果蝇存在偏好性差异的密码子数量最多,为29个,其中亮氨酸密码子UUA使用频率比值最高,达9.36;与大肠杆菌和家蚕存在偏好性差异的密码子均为14个;与酵母菌存在偏好性差异的密码子数量最少,为10个。因此,在开展大斑芫菁目标基因异源表达研究时,可选择酵母菌作为受体系统,但仍需进行密码子优化。
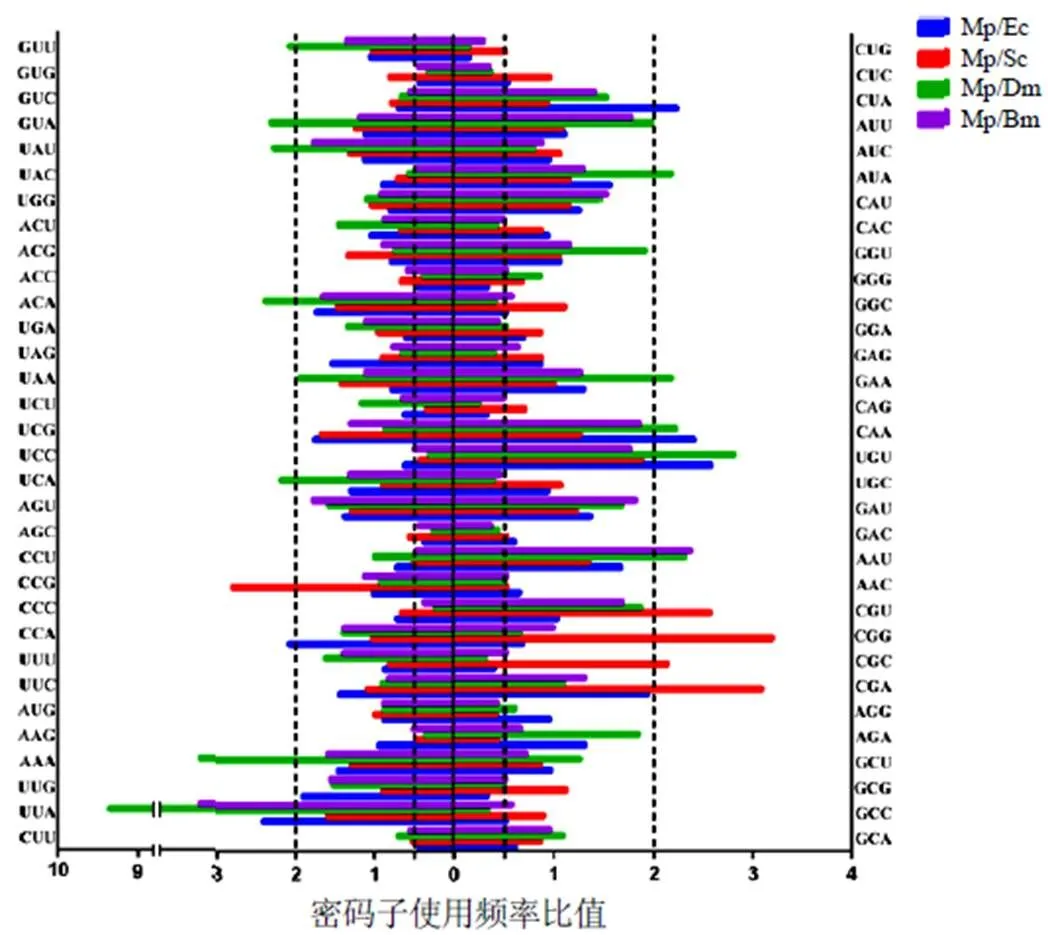
注:Mp.大斑芫菁;Ec.大肠杆菌;Sc.酵母菌;Dm.果蝇;Bm.家蚕
3 讨论
在复杂多样的进化过程中,各个物种均形成了一套特定的密码子使用模式。近年来,随着代谢工程及合成生物学研究的逐步深入及应用,解析天然药物成分的合成途径,进行异源表达,成为研究热点。因此,研究大斑芫菁转录组编码序列的密码子使用偏好性,可为揭示其密码子使用规律、异源表达宿主系统的选择及密码子优化提供理论基础。
本研究对大斑芫菁转录组10 264条完整编码序列的密码子使用模式分析结果显示,大斑芫菁转录组编码序列的平均GC、GC3s含量分别为0.387、0.321,略低于眼斑芫菁转录组平均GC含量0.41[28],但均小于50%,表明大斑芫菁转录组偏好使用富含A、U且以A/U结尾的密码子,这与斑翅草螽[29]、赤拟谷盗[30]、大头金蝇[31]密码子使用模式一致。研究表明,大多数昆虫组线粒体基因组具有较高AU含量[32-35],而植物中的小麦()[36]、水稻()[37]、玉米()[38]等则富含GC碱基,且偏好以G/C结尾,川贝母转录组GC含量与GC3s平均值几乎一致,均接近50%,整体上无明显的密码子使用偏性[21],表明碱基组成是影响密码子偏好性的重要因素之一。
此外,突变和选择也影响着密码子使用的偏好性。本研究结果显示,大斑芫菁转录组GC12s与GC3s含量具有显著相关性,97%基因的ENC≥35,且ENc-GC3s关联分析及偏倚性分析均发现,大部分基因分布于标准曲线及平面中心点周围,少部分基因偏离较远,表明大斑芫菁密码子偏好性是以突变压力为主导因素,多种作用方式共同影响的结果。
在构建异源表达体系中,不同基因密码子偏好性差异较大,因此,分析外源基因和宿主生物的密码子使用模式对于优化改造密码子及提高转录、翻译效率等具有重要意义[24]。本研究筛选到6个大斑芫菁基因偏好的密码子,分别为GCC、CGC、GGC、AUC、UUC、ACC,均以C结尾,表明大斑芫菁基因密码子的偏好性可能与第3位碱基的GC含量呈正相关。有研究表明,高表达基因中偏好强烈的密码子通常以G/C结尾[11],且对碱基C的偏好性强于碱基G,与本研究结果一致。密码子使用频率分析结果显示,大斑芫菁与果蝇整体密码子使用偏好性差异最大,与酵母菌差异最小,因此可选择酵母菌作为大斑芫菁目的基因异源表达的受体系统。
本研究对大斑芫菁转录组编码序列的密码子使用模式进行分析,探讨了影响其密码子使用偏好性的作用因素,并筛选出大斑芫菁基因的6个最优密码子及适宜异源表达受体酵母菌,可为大斑芫菁的基因工程和遗传机理研究提供依据。
[1] 国家药典委员会.中华人民共和国药典:一部[M].北京:中国医药科技出版社,2015:331-332.
[2] JAEMOO C, KYOUNG P M, HYEJIN K, et al. Bioassay-guided isolation of cantharidin from blister beetles and its anticancer activity through inhibition of epidermal growth factor receptor-mediated STAT3 and Akt pathways[J]. Journal of Natural Medicines,2018, 72(4):937-945.
[3] LIU Y P, LI L, XU L, et al. Cantharidin suppresses cell growth and migration, and activates autophagy in human non-small cell lung cancer cells[J]. Oncology Letters,2018,15(5):6527-6532.
[4] 陈中建,苟菲,张天锋.复方斑蝥注射液辅助结直肠癌根治术治疗结直肠癌的效果及安全性分析[J].中华中医药学刊,2019,37(7):1718-1720.
[5] 王平,王晓晶,杨淬,等.去甲斑蝥素酰胺类衍生物的合成及抗肝癌活性评价[J].中国药物化学杂志,2018,28(4):261-266.
[6] PALIDWOR G A, PERKINS T J, XIA X. A general model of codon bias due to GC mutational bias[J]. PLoS One,2010,5(10):101-105.
[7] 晁岳恩,吴政卿,杨会民,等.11种植物psbA基因的密码子偏好性及聚类分析[J].核农学报,2011,25(5):927-932.
[8] 李冠英,王政,张志燕,等.甘蓝型油菜WRKY基因家族碱基组成及密码子使用特性分析[J].生物学杂志,2013,30(4):42-45.
[9] LIU Q. Mutational bias and translational selection shaping the codon usage pattern of tissue-specific genes in rice[J]. PLoS One, 2012,7(10):e48295.
[10] SHOKRI E. Codon bias patterns in photosynthetic genes of halophytic grass[J]. Journal of Plant Molecular Breeding,2014,3(1):12-20.
[11] WANG L, ROOSSINCK M J. Comparative analysis of expressed sequences reveals a conserved pattern of optimal codon usage in plants[J]. Plant Molecular Biology,2006,61(4):699-710.
[12] PAUL P, MALAKAR A K, CHAKRABORTY S. Codon usage and amino acid usage influence genes expression level[J]. Genetica,2018,146(1):53-63.
[13] MARTÍNEZ-PÉREZ F, BENDENA W G, CHANG B S W, et al. Influence of codon usage bias on FGLamide-allatostatin mRNA secondary structure[J]. Peptides,2011,32(3):509-517.
[14] XU C, CAI X, CHEN Q, et al. Factors affecting synonymous codon usage bias in chloroplast genome of oncidium gower ramsey[J]. Evolutionary Bioinformatics,2011,2011(7):271-278.
[15] COMERON J M, KREITMAN M, AGUADE M. Natural selection on synonymous sites is correlated with gene length and recombination in drosophila[J]. Genetics,1999,151(1):239-249.
[16] NOVOA E M, DE POUPLANA L R. Speeding with control:codon usage, tRNAs, and ribosomes[J]. Trends in Genetics,2012,28(11):574-581.
[17] SHAH P, GILCHRIST M A, ALLAN D. Effect of correlated tRNA abundances on translation errors and evolution of codon usage bias[J]. PLoS Genetics,2010,6(9):e1001128.
[18] GOODARZI H, TORABI N, NAJAFABADI H S, et al. Amino acid and codon usage profiles:Adaptive changes in the frequency of amino acids and codons[J]. Gene,2008,407(1/2):30-41.
[19] 吴正常,王靖,赵乔辉,等.猪脂多糖结合蛋白基因(LBP)的密码子偏好性分析[J].农业生物技术学报,2013,21(10):1135-1144.
[20] 李滢,匡雪君,朱孝轩,等.长春花密码子使用偏好性分析[J].中国中药杂志,2016,41(22):4165-4168.
[21] 李滢,匡雪君,孙超,等.川贝母转录组密码子使用偏好性分析[J].中国中药杂志,2016,41(11):2055-2060.
[22] 梁娥,齐敏杰,丁延庆,等.竹节参转录组使用密码子偏好性分析[J].江苏农业科学,2019,47(2):67-71.
[23] 吴彦庆,赵大球,陶俊.芍药花色调控基因的密码子使用模式及其影响因素分析[J].中国农业科学,2016,49(12):2368-2378.
[24] ADI Y, SOPHIA K, RUTH H. The codon usage of lowly expressed genes is subject to natural selection[J]. Genome Biology and Evolution,2018,10(5):1237-1246.
[25] BEHURA S K, SEVERSON D W. Codon usage bias:causative factors, quantification methods and genome-wide patterns:with emphasis on insect genomes[J]. Biological Reviews,2013,88(1):49-61.
[26] 吴宪明,吴松锋,朱云平,等.密码子偏性的分析方法及相关研究进展[J].遗传,2007,29(4):420-426.
[27] YANG X, LUO X, CAI X. Analysis of codon usage pattern inbased on a transcriptome dataset[J]. Parasites & Vectors,2014,7(1):527.
[28] HUANG Y, WANG Z, ZHA S, et al. De novo transcriptome and expression profile analysis to reveal genes and pathways potentially involved in cantharidin biosynthesis in the blister beetle[J]. PLoS One,2016,11(1):e0146953.
[29] 周志军,尚娜,黄原,等.斑翅草螽线粒体基因组序列测定与分析[J].昆虫学报,2011,54(5):548-554.
[30] WILLIFORD A, DEMUTH J P. Gene expression levels are correlated with synonymous codon usage, amino acid composition, and gene architecture in the red flour beetle,[J]. Molecular Biology and Evolution,2012,29(12):3755-3766.
[31] 张玉波,周正湘,吴小玉,等.基于转录组的大头金蝇密码子的偏好性分析[J].江苏农业科学,2019,47(11):78-81.
[32] JIA W, HIGGS P G. Codon usage in mitochondrial genomes:Distinguishing context-dependent mutation from translational selection[J]. Molecular Biology & Evolution,2007,25(2):339-351.
[33] 郭仲龙,袁明龙.半翅目昆虫线粒体基因组学研究进展[J].中国科学:生命科学,2016,46(2):151-166.
[34] GUO C, ZHANG Q, HUANG Y. The complete mitochondrial genome of the Oedaleus infernalis sauss (Orthoptera:Oedipodidae)[J]. Mitochondrial DNA Part A DNA Mapping Sequencing & Analysis, 2017,28(1):1-2.
[35] RICHARDS S, GIBBS R A, WEINSTOCK G M, et al. The genome of the model beetle and pest[J]. Nature,2008, 452(7190):949-955.
[36] ZHANG W, ZHOU J, LI Z, et al. Comparative analysis of codon usage patterns among mitochondrion, chloroplast and nuclear genes inL[J]. Journal of Integrative Plant Biology, 2007,49(2):246-254.
[37] LIU Q, FENG Y, ZHAO X, et al. Synonymous codon usage bias in Oryza sativa[J]. Plant Science,2004,167(1):101-105.
[38] 刘汉梅,何瑞,张怀渝,等.玉米同义密码子偏爱性分析[J].农业生物技术学报,2010,18(3):456-461.
Analysis on Codon Usage Bias ofPallas Transcriptome Coding Sequence
LIU Chunlei1, CHEN Aimeng2, LIU Yanqin1, CAO Min1, CHEN Qiang1
To explore the characteristics and influencing factors of the codon usage of the coding sequence of the transcriptome ofPallas, so as to provide a basis for the realization of heterologous biosynthesis of cantharidin by using genetic engineering technology.The codon usage bias patterns of 10 264 transcriptome coding sequences ofPallas were analyzed by Codon W and EMBOSS software.The average contents of GC and GC3 of the coding sequence ofPallas were 0.387 and 0.321, respectively. There was a significant positive correlation between GC12 and GC3 (=0.369,<0.001). The results of ENC-GC3 and bias analysis showed that most genes were distributed around the standard curve and the center point of the plane, and a small number of genes deviated far away. Six optimal codons ofPallas were determined through high gene expression and superior codon method, namely GCC, CGC, GGC, AUC, UUC, ACC. The results of codon usage frequency analysis showed that the difference of codon usage bias betweenandPallas was the largest, whileandPallas was the smallest.The codon usage bias ofPallas transcriptome coding sequences is mainly due to the effect of mutation pressure, but there are other factors.can be used as an ideal system for heterologous expression.
Pallas; transcriptome; codon usage bias; optimal codons

R282.3
A
1005-5304(2020)10-0064-05
10.19879/j.cnki.1005-5304.202002103
重庆市卫生计生委中医药科技项目(ZY201802116)
陈强,E-mail:157557589@qq.com
(2020-02-08)
(2020-02-20;编辑:陈静)
