PRRSV GP5多表位融合基因的生物信息学分析及真核表达载体的构建
2020-11-06王小月蓝肇煜武静桥范真玮姚景丽何敬文金天明
李 丹,王小月,蓝肇煜,武静桥,范真玮,姚景丽,陈 婷,何敬文,赵 微,金天明
(天津农学院动物科学与动物医学学院,天津 300384)
猪繁殖与呼吸综合征(PRRS)是由猪繁殖与呼吸综合征病毒(PRRSV)引起的以母猪繁殖障碍和生长期猪的呼吸道症状为特征的高度接触性传染病,可感染不同年龄、品系的猪,给全世界的养猪业造成了巨大的经济损失[1]。PRRSV具有高度的变异性,能够引起免疫抑制和继发性感染,灭活疫苗和弱毒疫苗对PRRSV防控效果较差,且弱毒苗存在疫苗返毒的潜在风险[2-3]。GP5蛋白为PRRSV的主要结构蛋白,在病毒的致病性、复制、装配、变异和保护性反应等方面具有重要意义[4-5]。
抗原表位又称抗原决定簇,是抗原分子中决定其特异性的化学基团。抗原表位与相应的淋巴细胞表面抗原受体相结合,进而激活淋巴细胞,引起免疫应答反应[6]。在免疫应答过程中,被T细胞受体(TCR)识别的表位称作T细胞表位,被B细胞受体(BCR)识别的表位称作B细胞表位[7]。表位疫苗具有安全性好、保护力强、应答类型可以有效调控等优点,且重组质粒容易构建,使基因重组表达模式的表位疫苗具有较高的研究和应用价值[8]。研究显示,在抗原表位连接时,应注意各抗原表位之间的连接序列,如在抗原表位间插入柔性短肽“GPGPG”,可以避免抗原表位间形成新的表位[9-10]。柔性连接头还能够提高蛋白酶水解和抗原表位递呈的效率,增强疫苗的免疫效果。
本试验通过分析5株常见的美洲型PRRSV毒株的GP5蛋白序列,并选取预测的其保守区的T细胞和B细胞表位序列进行连接,组建新的GP5多表位融合基因(GP5 recombinant epitopes sequence,GRE),并对GRE序列进行生物信息学分析;然后分别构建GP5和GRE基因重组真核表达载体并分析其细胞毒性。
1 材料与方法
1.1 材料重组质粒pMD-GP5(AmpR)及pMD-GRE(AmpR)由上海捷瑞生物工程有限公司合成;pEGFP-N1(KanR)质粒及大肠杆菌感受态细胞XL-10Glod由天津农学院预防兽医学实验室保存。
1.2 GP5基因保守区筛选对5株美洲型PRRSV毒株JL580(AKZ66195.1)、HENAN(AHB29375.1)、VR2332(ABU87666.1)、NADC30(AFP43978.1)和JXA1(ABL60902.1)的GP5序列进行同源性分析,确定其保守区域。将获得的较长保守区域利用NCBI Protein Blast软件验证,验证所得保守序列是否为GP5蛋白的特异性序列。
1.3 GRE基因序列的构建利用生物信息学软件ABCPred、BepiPred 2.0对B 细胞表位进行预测,选择评分较高的2个作为B细胞表位预测结果;利用MHC Class Ⅰ Binding Peptide Prediction Server 预测细胞毒性T 细胞表位,选择HLA-A2、HLA-A*0201、HLA-A*0202、HLA-A*0203、HLA-A*0205五种预测类型,综合预测结果确定细胞毒性T淋巴细胞(CTL)最佳预测方案;利用ProPred.MHCⅡ Binding Peptide Prediction Server预测辅助性T细胞表位,选择DRB1-0101、DRB1-0102和DRB1-0301三种预测类型,综合预测结果选择最优方案作为Th细胞预测结果。
将上述获得的B细胞及T细胞表位序列串联,相同类型细胞间表位连接选择短肽GPGPG,促进其二、三级结构的形成;不同类型细胞间的表位连接选择刚性短肽GPLS,以此增强B细胞和T细胞表位各自的独立性,构建合成GRE基因序列。
1.4 GRE序列蛋白二级结构、跨膜结构域及亲水性分析利用生物信息学软件ExPASy tools对GRE序列进行二级结构分析,利用生物信息学软件TMpred对GRE序列跨膜区进行分析,利用生物信息学软件ProtScaleon Expasy对GRE序列亲水性进行分析。
1.5 引物设计根据NCBI中5株GP5基因保守序列,构建表位预测序列;使用Primer 6.0软件设计序列引物,选择最优的设计结果,添加保护性序列,引物均由上海捷瑞生物工程有限公司合成(表1)。
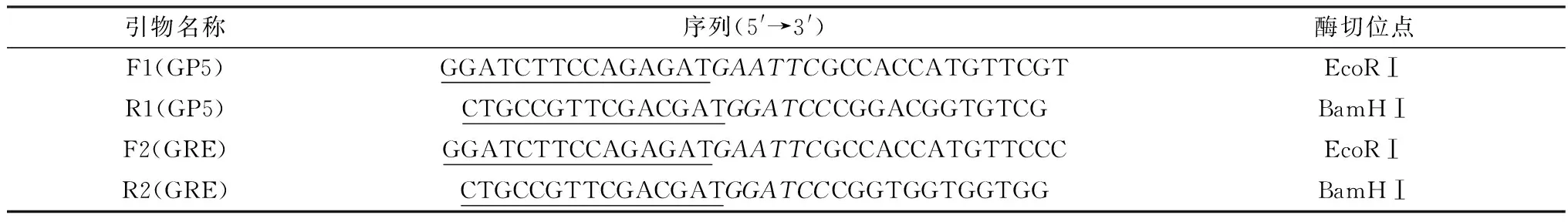
表1 引物信息
1.6 重组真核表达载体的构建及酶切鉴定利用限制性内切酶BamHⅠ和EcoRⅠ分别对质粒pMD-GP5、pMD-GRE和pEGFP-N1进行双酶切,T4DNA连接酶连接后分别获得pEGFP-GP5、pEGFP-GRE重组质粒。将上述2个质粒和pEGFP-N1分别转化XL-10Glod感受态细胞,通过卡那霉素抗性培养基筛选阳性菌株。利用引物F1/R1、F2/R2进行PCR鉴定,将阳性重组菌分别命名为XL-10Glod/pEGFP-GP5和XL-10Glod/pEGFP-GRE,-80℃保存备用。
1.7 重组质粒转染Marc145细胞按照每孔l×105个Marc145细胞加入6孔板,于10% FBS的MEM完全培养基中培养至细胞汇合度大于70%;将6孔板内更换新鲜培养基,约为2 mL,分别转染质粒pEGFP-GP5、pEGFP-GRE和pEGFP-N1,使用 LipoFiter 3.0TM转染试剂对质粒进行转染,分别采集12,24,36和48 h时的细胞荧光照片,观测重组质粒在Marc145细胞中不同时间的表达情况。
1.8 RT-PCR验证重组质粒在Marc145细胞中的表达情况将重组质粒转染Marc145细胞,方法同1.7。转染48 h后,经0.25%胰酶处理后收集细胞,加入350 μL裂解液RL裂解细胞,振荡,按照试剂盒操作步骤提取RNA,-70℃保存。
利用逆转录试剂盒获得cDNA后,以其为模板,分别以F1/R1、F2/R2为引物,PCR检测目的基因。反应程序:95℃ 5 min;95℃ 30 s,60℃ 30 s,73℃ 1 min,共35个循环;73℃ 15 min。
1.9 CCK-8检测重组质粒对Marc145细胞存活率的影响按照每孔100 μL 5 000个 Marc145细胞加入96孔板,于10% FBS的MEM完全培养基中培养至细胞汇合度大于50%;分别以不同质量梯度(0.40,0.20,0.10和0.05 μg)于每孔加入重组质粒pEGFP-GP5、pEGFP-GRE和pEGFP-N1,每个处理设置5次重复;分别将96孔板置于CO2培养箱中孵育至24,48,72和96 h时,于96孔板中每孔加入10 μL配置好的CCK-8溶液,在37℃孵育1.5 h,用酶标仪检测D450 nm值。
2 结果
2.1 5株PRRSV GP5蛋白保守区域的筛选及特异性验证通过对所得保守区域B细胞、CTL细胞、Th细胞进行表位预测(图1),根据结果选取67~91氨基酸作为GP5所筛选保守区域的目的片段。
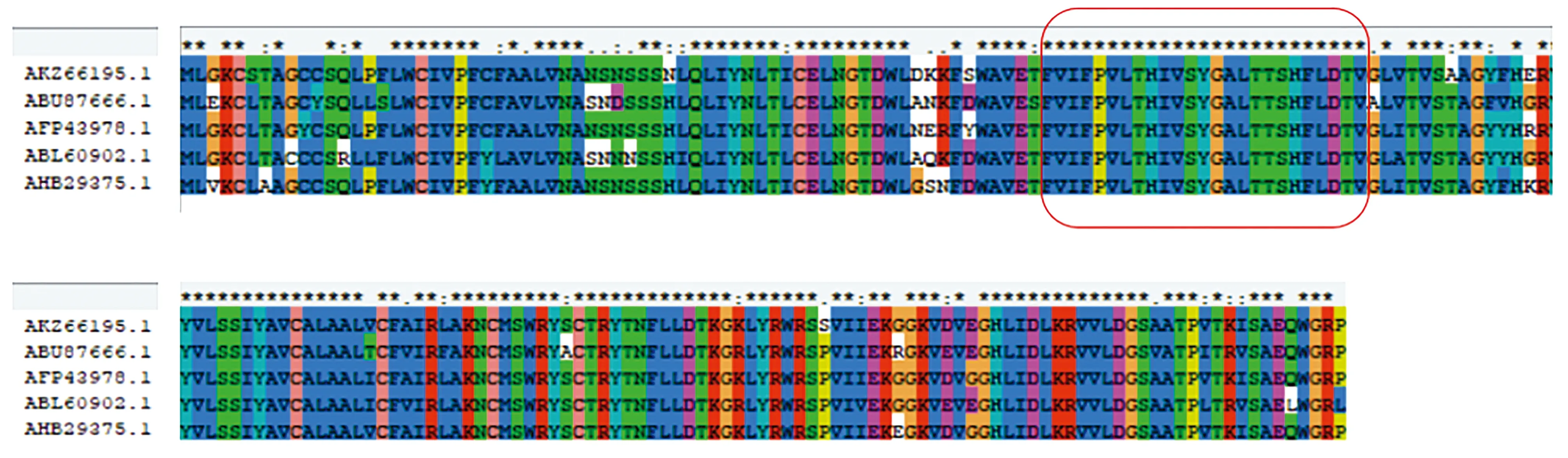
图1 5株PRRSV GP5蛋白同源性比较及保守区筛选
使用NCBI 中Protein Blast在线工具对筛选到的GP5目的片段进行特异性验证,结果显示,该目的片段为PRRSV GP5特异性蛋白(图2),可以作为表位预测目的片段。
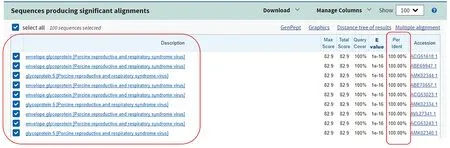
图2 GP5保守区域片段特异性验证
2.2 GP5保守区表位预测结果对目的片段的3种表位进行预测后的结果,按照评分标准高低进行选择和组合(表2),不同表位序列之间加入连接肽,序列末端添加His标签便于蛋白纯化表达,最终所得GRE预测序列:FPVLTHIVSYGALTTSHFLDTVGPLSVIFPVLTHIVSYGALTTSHFLDT-VGPGPGFVIFPVLTHIVSYGALTTHHHHHH。
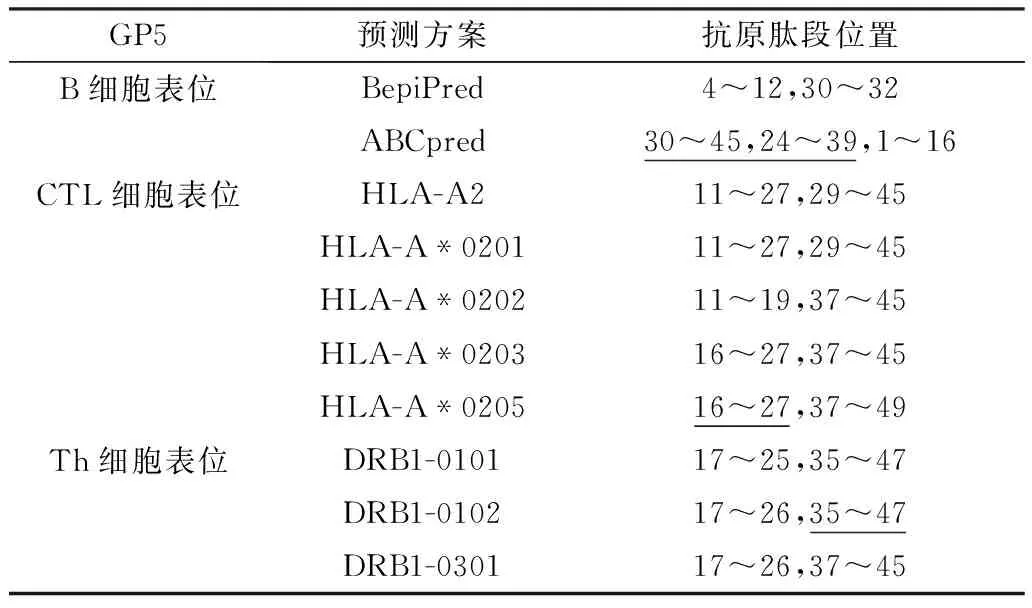
表2 GP5保守区B细胞及T细胞表位预测结果
2.3 GRE序列蛋白质二级结构分析使用ExPASy tools软件对GRE序列进行分析,结果显示,α螺旋为21.52%,延伸链为31.65%,β转角为11.39%,无规则卷曲为35.44%(图3)。
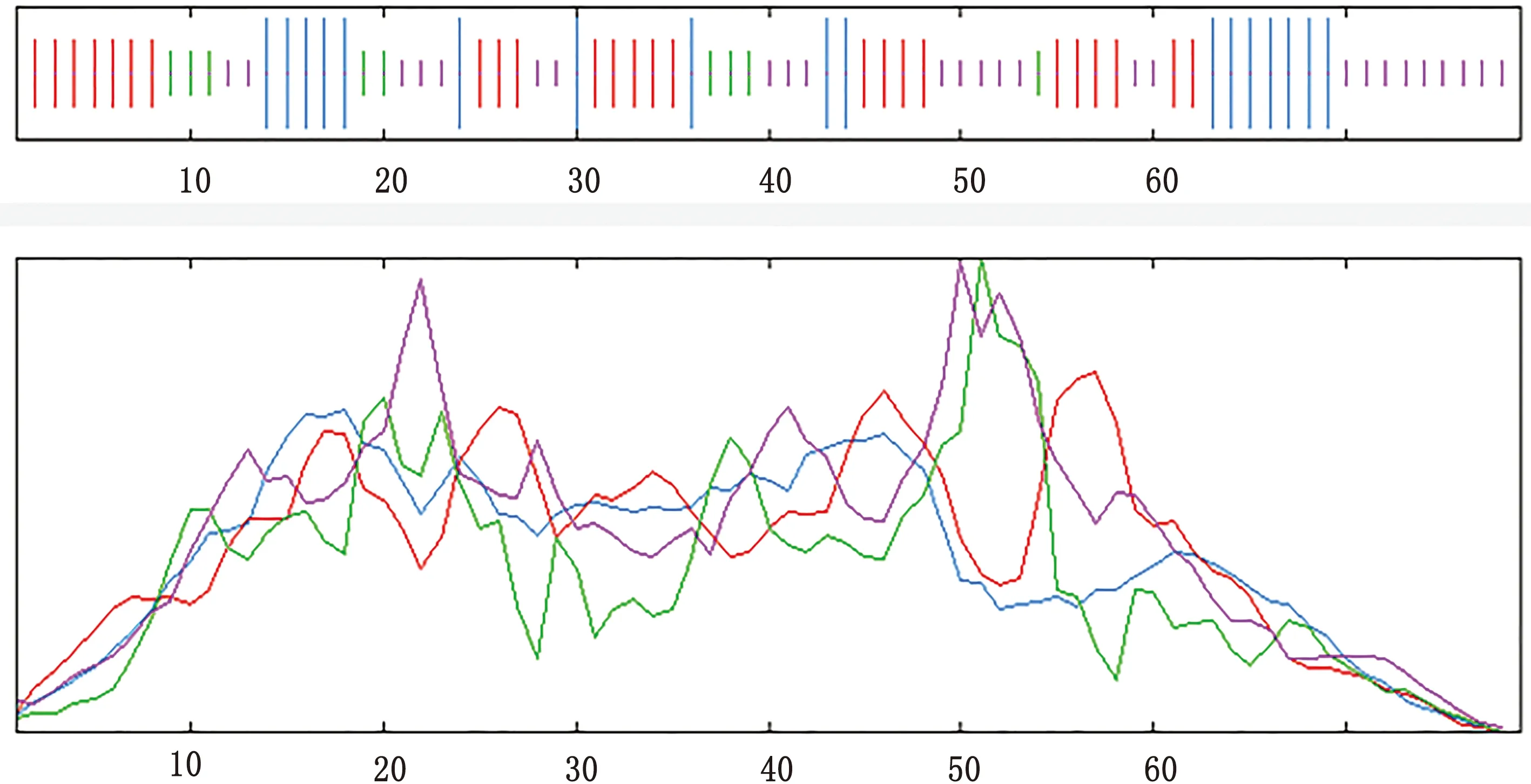
图3 GRE序列蛋白质二级结构分析
2.4 GRE序列跨膜结构域分析使用TMpred分析GRE序列跨膜结构域,结果显示,其在第7~44个氨基酸及第49~75个氨基酸处存在两处跨膜区域,预测其为跨膜蛋白(图4)。
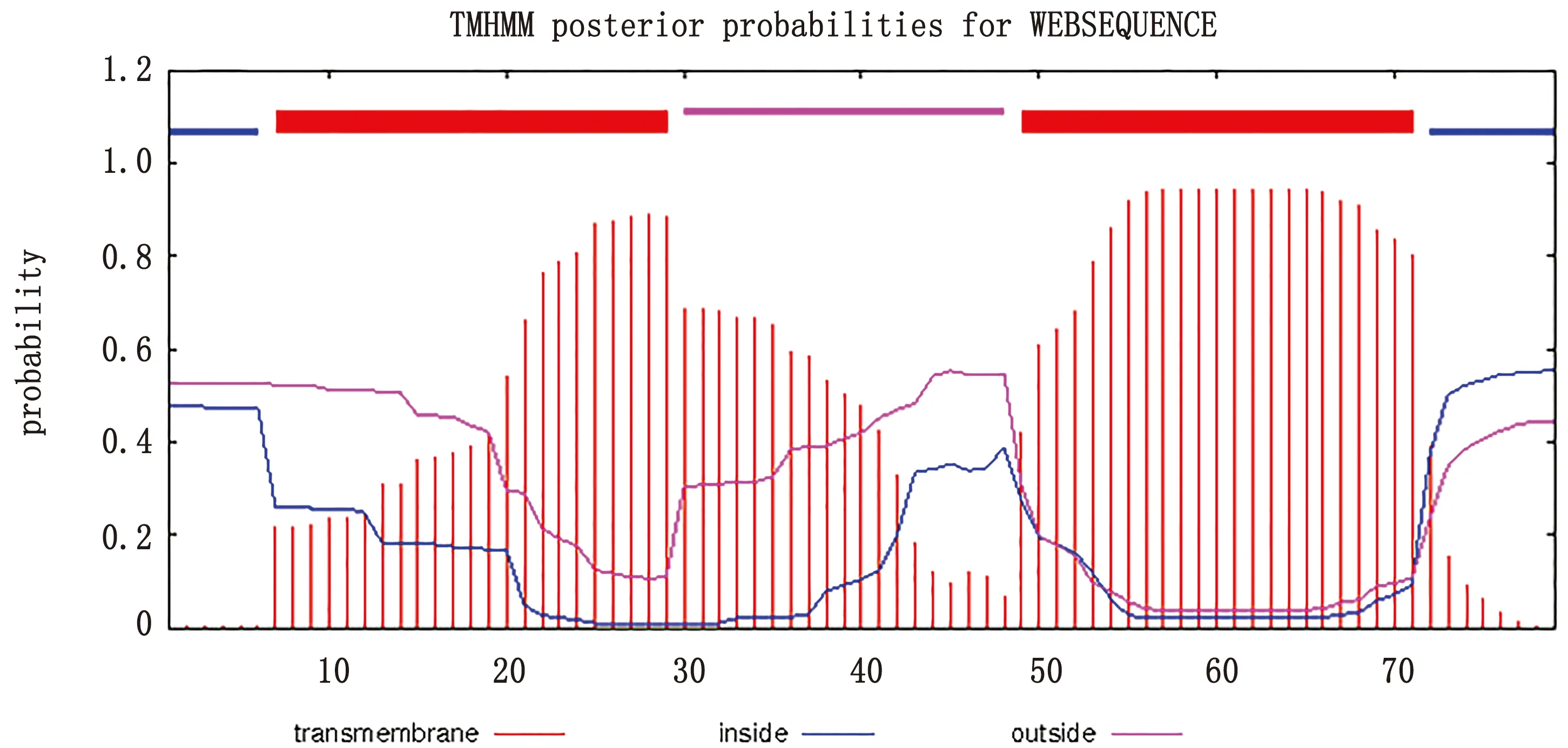
图4 GRE序列跨膜结构域分析
2.5 GRE序列的亲水性分析使用ProtScaleon Expasy软件对GRE序列亲水性进行分析,结果显示,其亲水性氨基酸评分较高,预测其为亲水性蛋白(图5)。
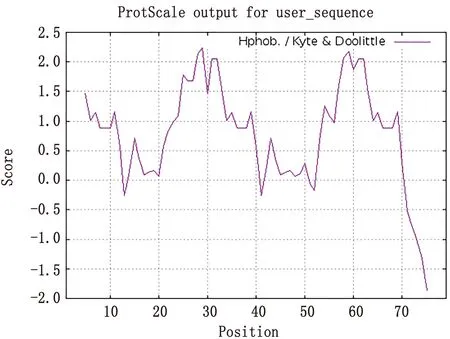
图5 GRE序列亲水性分析结果
2.6 重组质粒的构建及鉴定提取携带pEGFP-GP5和pEGFP-GRE的重组菌质粒,经PCR鉴定,酶切鉴定及测序,结果显示,均获得大小分别为98,260 bp 的目的片段,与预期结果相符(图6)。经测序和序列比对,结果发现无碱基缺失、增添和突变,表明重组质粒pEGFP-GP5和pEGFP-GRE构建成功。

图6 GP5和GRE基因片段的PCR扩增结果 M.DL2000 DNA Marker;1,2.GP5、GRE基因的扩增产物
2.7 重组质粒在Marc145细胞中的表达情况荧光显微镜下观察,重组质粒pEGFP-N1、pEGFP-GP5和pEGFP-GRE在转染细胞12 h后开始表达荧光,荧光表达强度随转染时间逐渐增强。结果表明,重组质粒pEGFP-GP5及pEGFP-GRE在Marc145细胞中成功表达,并且在48 h时表达效果最好(图7)。
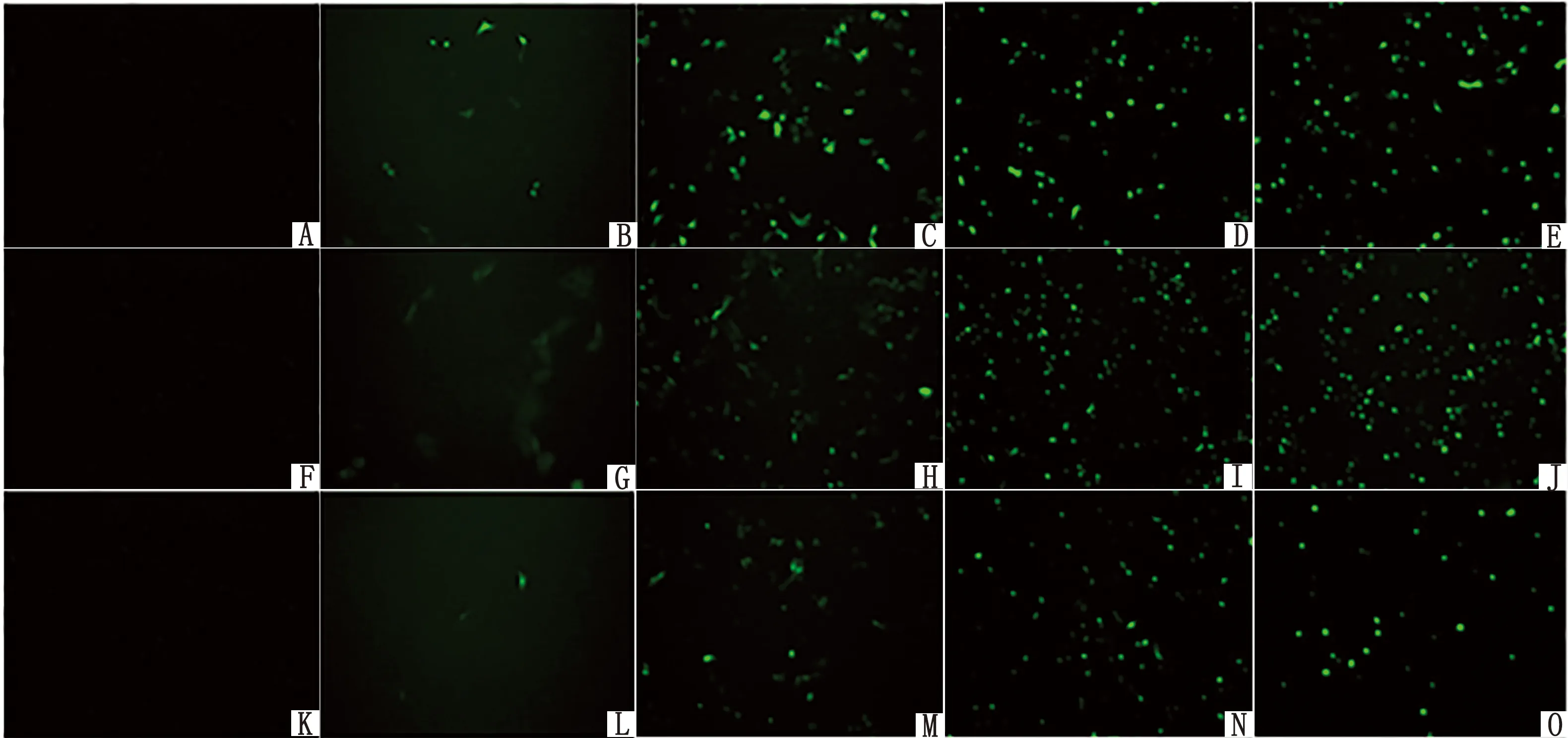
图7 重组质粒转染Marc145细胞在不同时间的表达结果 A,F,K.空白对照;B~E.空白质粒pEGFP-N1转染Marc145细胞在12,24,36,48 h的荧光表达;G~J.重组质粒 pEGFP-GP5转染Marc145细胞在12,24,36,48 h的荧光表达;L~O.pEGFP-GRE转染Marc145细胞在12,24,36 h的荧光表达
2.8 RT-PCR 验证重组质粒在Marc145细胞中的表达情况重组质粒转染Marc145细胞48 h后,提取RNA,反转录试剂盒进行反转,以F1/R1、F2/R2为引物检测重组质粒在细胞中的转录情况,结果显示,在98,260 bp处获得目的基因条带,经测序与目的基因序列一致(图8),表明重组质粒pEGFP-GP5及pEGFP-GRE成功在Marc145中转录表达。

图8 重组质粒目的基因检测结果 M.DL2000 DNA Marker;1,2.GP5、GRE基因的扩增产物
2.9 CCK-8检测重组质粒对Marc145存活率的影响通过CCK-8试验分别检测重组质粒pEGFP-GP5和pEGFP-GRE在转染Marc145细胞后24,48和72 h时的细胞存活率,结果显示,转染后24,48 h时重组质粒pEGFP-GP5和pEGFP-GRE组的细胞存活率极显著高于PBS组细胞存活率(P<0.01),且随着质量浓度增加,高质量浓度的重组质粒pEGFP-GRE对Marc145细胞不产生抑制作用,不同质量浓度组之间差异不显著(P>0.05)(图9)。

图9 不同质量浓度重组质粒转染Marc145后在24,48,72 h的细胞存活率 A~C.分别为24,48,72 h的细胞存活率。*.P<0.05;**.P<0.01;ns.P>0.05
3 讨论
细胞免疫是动物感染病原体后出现的一种保护性免疫应答。研究发现,猪群感染PRRSV后在产生体液免疫反应的同时,也出现了细胞免疫反应[11]。动物机体受到外来病原体入侵时,除了做出相应的体液免疫反应外,还会激发细胞免疫,细胞免疫是清除细胞内寄生微生物的最为有效的防御反应[12]。
近年来研究较多的是DNA疫苗和亚单位疫苗等基因工程疫苗,其中PRRSV的DNA疫苗是PRRSV结构基因的DNA疫苗,使用DNA疫苗免疫动物,从而使机体产生特异性抗体来保护机体。亚单位疫苗主要是针对M蛋白、GP5、GP4和GP3等结构蛋白[13]。
PRRSV GP5参与细胞免疫与体液免疫应答,尤其是刺激中和抗体的产生,因此,GP5被认为是研究DNA疫苗及活病毒载体疫苗等新型疫苗的最佳候选基因[14-15]。此外,GP5蛋白能刺激机体产生T淋巴细胞增殖反应,诱导机体产生针对PRRSV的特异性细胞免疫,其诱导细胞免疫的能力仅次于M蛋白,表明GP5在诱导机体细胞免疫中发挥重要作用[16-17]。
表位是宿主免疫系统识别病原体上的关键区域,优势中和表位的发现是诊断试剂和疫苗研发的基础[18]。有研究表明,重组真核质粒免疫小鼠能够诱导动物机体产生针对GP4、GP5的特异性抗体[19]。因此本试验选择PRRSV GP5蛋白进行表位预测,所构建的多表位融合基因经生物信息学软件分析后发现其亲水性较好,且存在两段跨膜结构域,转染细胞后重组质粒成功表达;且重组质粒对Marc145细胞的存活率在24,48 h时较对照组相比无抑制作用,在72 h时由于细胞的生存空间及培养基中营养物质的消耗,此时各组细胞存活率较24,48 h时相比显著降低,结合转染后细胞形态观察及CCK-8结果可评定重组质粒安全性较高。
PRRSV传统疫苗存在一定的弊端和局限性,随着PRRSV核酸全基因序列测定及其编码蛋白功能分析等病毒分子生物学方面的深入研究,基于蛋白质结构信息的分子对接技术为解决这一矛盾提供了新的研究思路和技术手段,拓展了现代病毒学在相关疾病的感染机制和预防控制的研究领域,特别是在病毒表位预测的研究上,可节省大量时间及资源,在现代病毒学的药物及疫苗开发中具有不可或缺的地位[20]。开发新型疫苗取代现有的传统疫苗己势在必行,生物学技术的迅速发展为新型疫苗的研究提供了理论基础。本试验构建的GRE重组真核表达载体及其细胞毒性分析为PRRS表位基因工程疫苗的后续动物试验提供依据。
