DNA条形码技术在白云山国家森林公园苔藓植物群落研究中的应用
2020-10-31胡永春杨子习靓靓李雨姗周紫羽符强邵毅贞叶永忠袁志良
胡永春,杨子,习靓靓,李雨姗,周紫羽,符强,邵毅贞,叶永忠,袁志良
(1.河南农业大学生命科学学院,河南 郑州 450002;2.河南农业大学林学院,河南 郑州 450002)
DNA条形码技术是利用短DNA片段对物种进行快速准确识别和鉴定的技术,在系统发育群落生态学研究中较为常见,多用于探索群落物种的生态和进化机制,以及群落内物种之间相互作用对进化和生态系统过程的影响[1-3]。目前,该技术主要应用于木本植物的研究中。KRESS等[4]使用matK+rbcL+psbA-trnH的DNA条形码片段组合对巴罗科拉多岛(Barro Colorado Island,BCI)样地中299个物种进行准确识别,识别率高达98%。裴男才等[5]利用DNA条形码技术构建了鼎湖山大样地木本植物系统发育关系,从森林群落系统发育结构来了解该区域生态状况。卢孟孟等[6]以被子植物系统发育研究组系统(angiosperm phylogeny group,APG)为框架,结合DNA条形码技术构建哀牢山森林群落系统发育进化树,为从谱系结构了解木本植物群落多样性维持机制提供了新的视角。
苔藓植物(Bryophyte)是生物多样性的重要组成部分,在水土保持、积蓄养分和林地更新等方面具有重要的生态价值[7]。苔藓植物体型微小、构造简单、对环境变化敏感,经常作为环境变化的指示物种,然而单从形态特征和生理结构上鉴定苔藓植物难度较大,从而限制了苔藓植物生态学的发展[8]。近年来,有关苔藓植物分子生物学研究主要集中在苔藓植物生长状况记录[9]、苔藓植物系统发育与系统学研究以及来自不同基因组标记进化和系统发育效用等方面[10-11]。郭磊等[12]在小秦岭自然保护区通过生态位来研究了苔藓植物物种多样性。陈云等[13-14]在小秦岭自然保护区用群落数量分类和排序的方法研究了苔藓植物物种多样性与样地环境因子的关系。目前,苔藓植物DNA条形码技术尚不成熟,同时苔藓植物整体系统发育框架的构建还未完成,无法像木本植物一样根据已有的分类系统与数据进行群落生态学分析。刘艳等[15]根据Gen-Bank中3 365条rps4基因序列,利用遗传距离法和分子系统学方法证明rps4基因作为苔藓植物候选条形码片段具有较好的可行性。张安世等[16]基于叶绿体trnL-F基因序列对10种苔藓植物亲缘关系进行分析,把不同科的苔藓植物分类到不同的谱系树的分支上。李丹丹等[17]用trnL、trnG、psbT和rps4基因序列进行系统发育分析并构建了15棵贝叶斯系统发育树,发现trnL-trnG-psbT组合适合用于蓑藓属植物的系统发育分析。赵丽嘉等[18]通过atpB和rbcl基因序列进行藓类物种系统重建时,发现rbcl基因序列不仅长度适合做PCR扩增,且有助于系统重建。并非所有基因片段都适合苔藓植物系统发育群落生态学研究。rps4基因属于保守基因,在进化水平上比较慢,容易扩增和成功测序;rbcl基因是部分苔藓植物的保守性片段,对于苔藓植物的识别效果较高;trnL-F基因存在于植物叶绿体的非编码区,其功能编码区进化速度小,受外界因素变动压力小,适合用于分类水平上系统关系的重建[15-17]。因此,rps4、rbcl和trnL-F基因常用来评估DNA条形码技术在苔藓植物系统发育群落生态学中可适性。但是,上述研究是基于NCBI数据库中理论上的苔藓植物序列以及苔藓植物标本提取的DNA序列进行的,缺乏对森林动态监测样地内的苔藓植物的实地考察与验证。有关苔藓植物DNA条形码技术在物种层面的鉴定以及苔藓植物群落系统发育树重建方面的研究需要进一步推进。
本研究基于白云山国家森林公园4个1 hm2森林动态监测样地,对样地内苔藓植物进行采集并提取物种DNA,利用单一DNA条形码和组合DNA条形码技术进行苔藓物种鉴定并构建样地苔藓植物物种系统发育进化关系树,通过3种DNA条形码片段鉴定样地内苔藓植物物种的成功率,构建快速有效鉴定苔藓植物体系,进而构建白云山国家森林公园动态监测样地苔藓植物系统发育进化关系树,了解苔藓植物群落中物种亲缘关系的远近以及物种组成,为苔藓植物生态学发展和苔藓植物物种多样性保护奠定基础。
1 样地概况
1.1 研究区概况
白云山国家森林公园位于河南省洛阳市嵩县南部伏牛山原始林区,经纬度坐标为111°48′~112°16′E,33°33′~33°56′N,总面积168 km2。区域内地势西高东低,山体南北平等延伸,地处南北地理气候分界线,属亚热带暖温带气候过渡区。该区域内年均气温13.1~13.9 ℃,最低气温-14.4 ℃,最高气温42.1 ℃;年降雨量达1 200 mm,多集中在7—9月。土壤类型为山地棕壤、山地黄棕壤和山地褐土,且阴暗潮湿;有机质含量高,区域内土壤质地多以轻壤为主,pH值5.5~6.5,呈酸性。
1.2 样地概况
2015年在对白云山国家森林公园全面踏查的基础上,参照BCI样地技术规范,选择具有代表性的森林植物群落建立了4个1 hm2(100 m×100 m)永久性森林动态监测样地。样地建设时用全站仪(徕卡TC2003,德国)打点定位,以西南角为原点,将1 hm2的样地细划分为25个20 m×20 m子样方,子样方4个角用水泥柱做标记[19]。
2 研究方法
2.1 苔藓植物标本的采集
自2018-07-05开始为期7 d的苔藓植物标本采集工作,对4个1 hm2典型森林群落的样地中所有苔藓植物进行调查。每个1 hm2样地划分为25个20 m×20 m的子样方,再进一步划分成4个10 m×10 m的小样方。以小样方为采样基本单位,对其中所有苔藓植物(石生苔藓植物、土生苔藓植物、腐木生苔藓植物以及树附生苔藓) 进行地毯式采集。样方中不同藓丛分别装袋,记录其所在样方号和生境信息[19]。本研究共采集到苔藓植物标本3 305份,在阴凉通风处晾干后存放在河南农业大学标本馆(HEAC)。
2.2 DNA的提取
将采集的不同种苔藓植物挑取相对干净的植株,蒸馏水漂洗,去除土壤和杂质。用镊子撕下苔藓的叶片,装入提前装好钢珠的2.0 mL的离心管中,用液氮浸泡后,使用组织研磨仪(JXFSTPRP,上海)磨样,磨成粉末状。使用百泰克植物DNA的提取试剂盒(百泰克BioTeKe,无锡),提取苔藓植物的DNA,用质量分数1%的琼脂糖凝胶进行电泳,条带明亮的苔藓物种DNA作为PCR的材料。
2.3 序列扩增和测序
采用rps4、trnL-F、rbcl基因共3个DNA片段进行分析。参照SAVOLAINEN等[20]的PCR反应体系进行优化和筛选。采用25 μL体系,包括模板10 μL,正反引物各1 μL,2×Es TaqMasterMix(Dye)12.5 μL,灭菌双蒸水0.5 μL。3个片段的长度不同所要求的变性温度、退火温度及循环次数不同,如表1、表2、表3所示。PCR 扩增后的产物经质量分数1%的琼脂糖凝胶电泳观察条带亮度,将有单一清晰条带的结果送至郑州瑞尤科技生物有限公司,测序引物为PCR反应正向引物。3个引物片段的序列如表4所示。
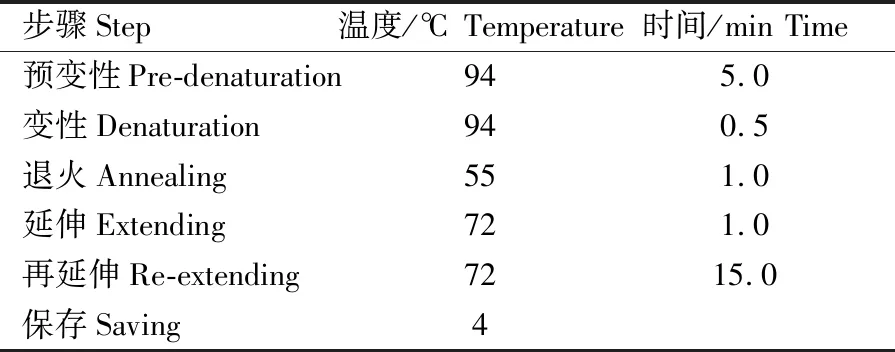
表1 rps4基因PCR反应步骤Table 1 rps4 gene PCR reaction steps

表2 rbcl基因PCR反应步骤Table 2 rbcl gene PCR reaction steps
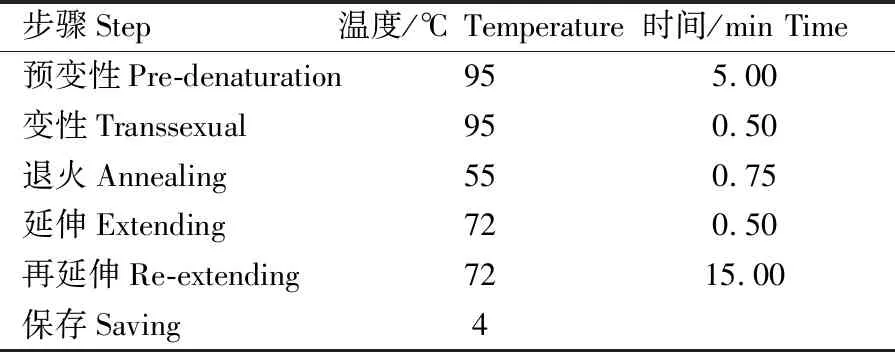
表3 TrnL-F基因 PCR反应步骤Table 3 TrnL-F gene PCR reaction steps
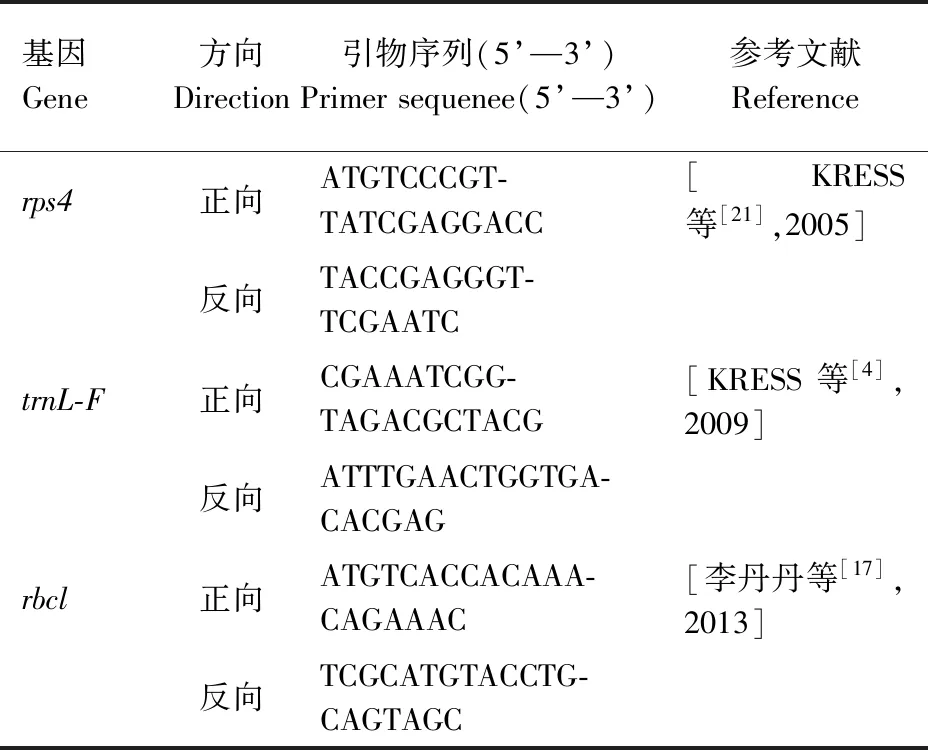
表4 引物序列片段Table 4 Primer sequence fragments
2.4 序列分析及系统发育进化关系树的构建
测序成功率是获得高质量序列数与苔藓植物总个体数的比例。物种识别率是识别成功的个体数与总个体数的比例[4]。综合成功率是苔藓植物测序成功率和苔藓植物匹配成功率的乘积[22]。根据测序结果,用MAGE7.0对序列进行整理、编辑后保存为FASTE格式文档,3个片段逐条Blast比对,确定该序列所属的科、属和种并记录鉴定成功率。BioEdit打开3个序列的DNA片段,对序列进行拼接,同种的序列自动连接在一起并调整,用最大似然法(maximum likelihood,ML)快速准确地获得苔藓植物的系统发育进化关系树。根据系统发育进化关系树上节点支持率的高低将进化枝分为3类:高支持率(支持率大于等于85%)、中支持率(支持率介于65%~84%)和弱支持率(支持率小于64%)[4]。使用R语言vegan包,用多重检验法比较样地内苔藓植物的测序成功率、物种识别成功率和综合成功率的差异。
3 结果与分析
3.1 样地内苔藓物种DNA条形码应用成功率
如图1、图2、图3所示,白云山国家森林公园动态监测样地苔藓植物DNA条形码片段rps4、rbcl、trnL-F、rps4+rbcl、rbcl+trnL-F、rps4+trnL-F、rbcl+rps4+trnL-F的测序成功率分别为98.59%、87.32%、94.89%、91.54%、90.14%、95.77%和91.54%;物种识别成功率分别为97.18%、77.64%、90.14%、87.32%、84.50%、91.54%和88.73%;综合成功率分别为95.81%、67.80%、85.53%、79.93%、76.16%、87.67%和81.22%。其中,rps4基因测序成功率,物种识别的成功率和综合成功率最高,且比其他条形码片段显著(P<0.05)。
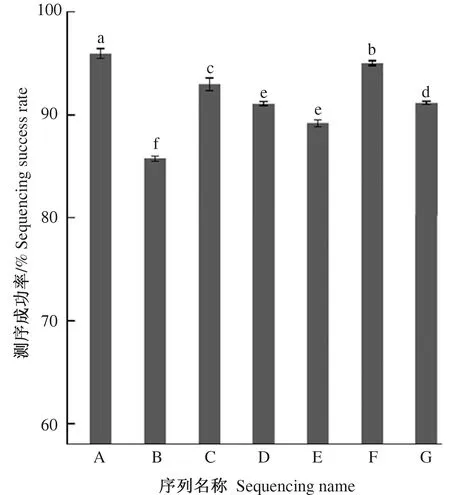
注:A~G分别代表rps4、rbcl、trnL-F、rps4+rbcl、rbcl+trnL-F、rps4+trnL-F、rbcl+rps4+trnL-F。不同字母表示结果差异性显著,P<0.05。下同。Note:A~G stands for rps4、rbcl、trnL-F、rps4+rbcl、rbcl+trnL-F、rps4+trnL-F、rbcl+rps4+trnL-F.Different letter indicate that the results are significantly different,P<0.05.The same as below.图1 白云山国家森林公园动态监测样地苔藓植物DNA条形码片段测序成功率Fig.1 Success rate of DNA barcoding fragment sequencing of bryophytes in the dynamic monitoring plots of Baiyun Mountain National Forest Park
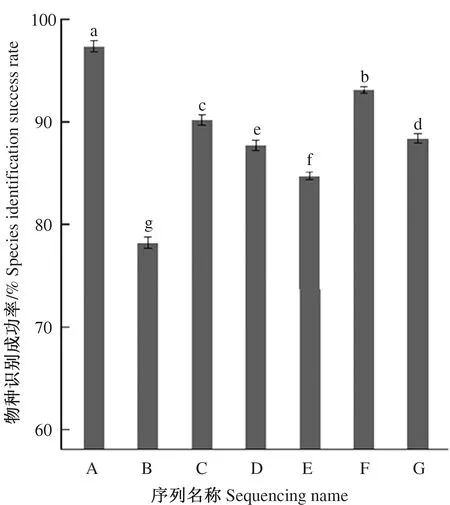
图2 白云山国家森林公园动态监测样地苔藓植物DNA条形码片段物种识别成功率Fig.2 The success rate of species identification of DNA barcoding fragments of bryophytes in the dynamic monitoring plots of Baiyun Mountain National Forest Park
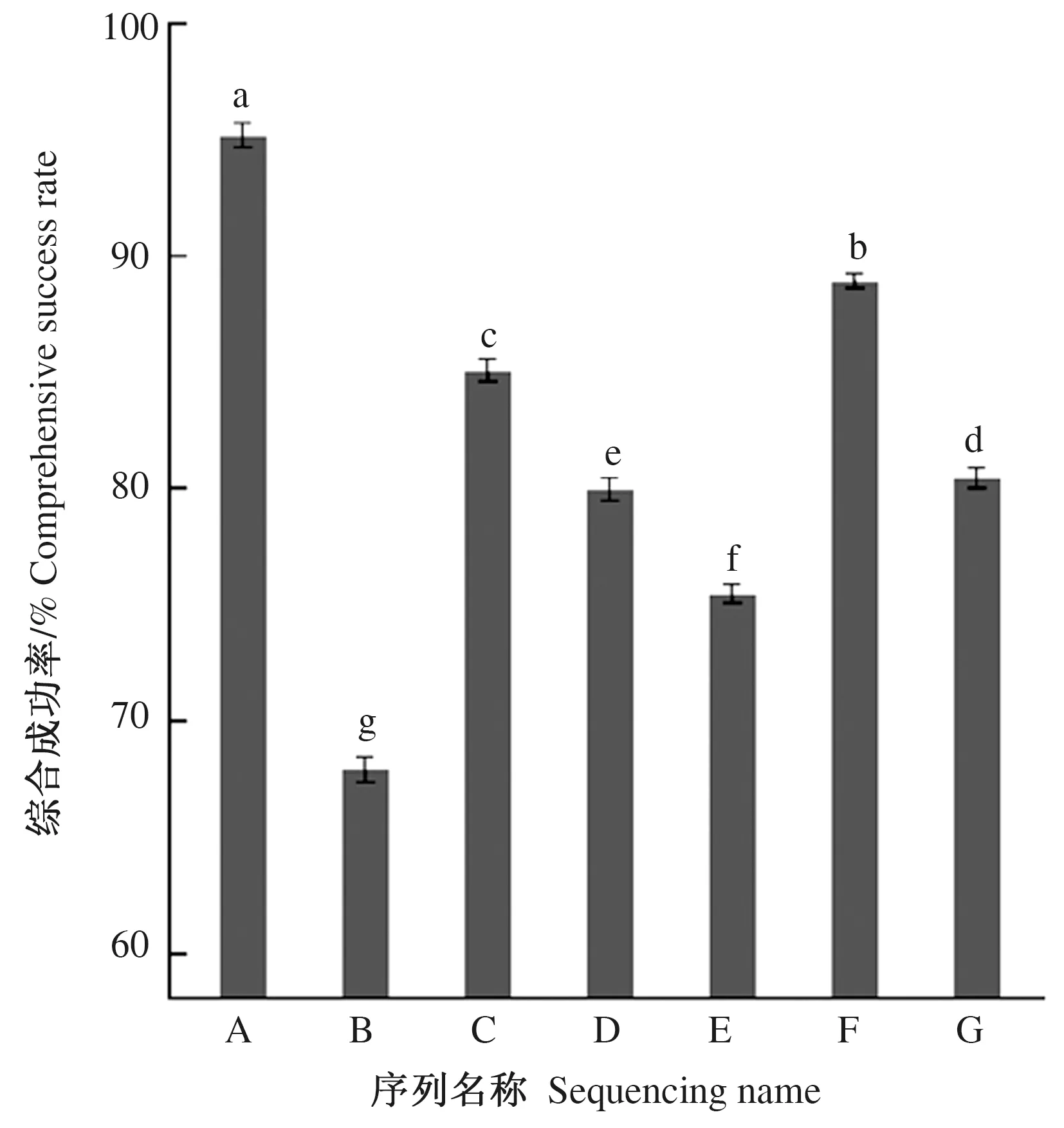
图3 白云山国家森林公园动态监测样地苔藓植物DNA条形码片段综合成功率Fig.3 Comprehensive success rate of DNA barcoding fragments of bryophytes in the dynamic monitoring plots of Baiyun Mountain National Forest Park
3.2 样地内苔藓植物系统发育进化关系树
利用Blast法对白云山国家森林公园监测样地苔藓植物DNA条形码片段进行搜索,结果共有25科45属71种,如表5所示。其中,藓类21科41属66种,占总物种数的92.96%;苔类4科4属5种,占总物种数的7.04%。优势科的属、种统计及多度排前4的物种分别为长肋青藓、短叶毛锦藓、尖叶匐灯藓和东亚毛灰藓,如表6所示。
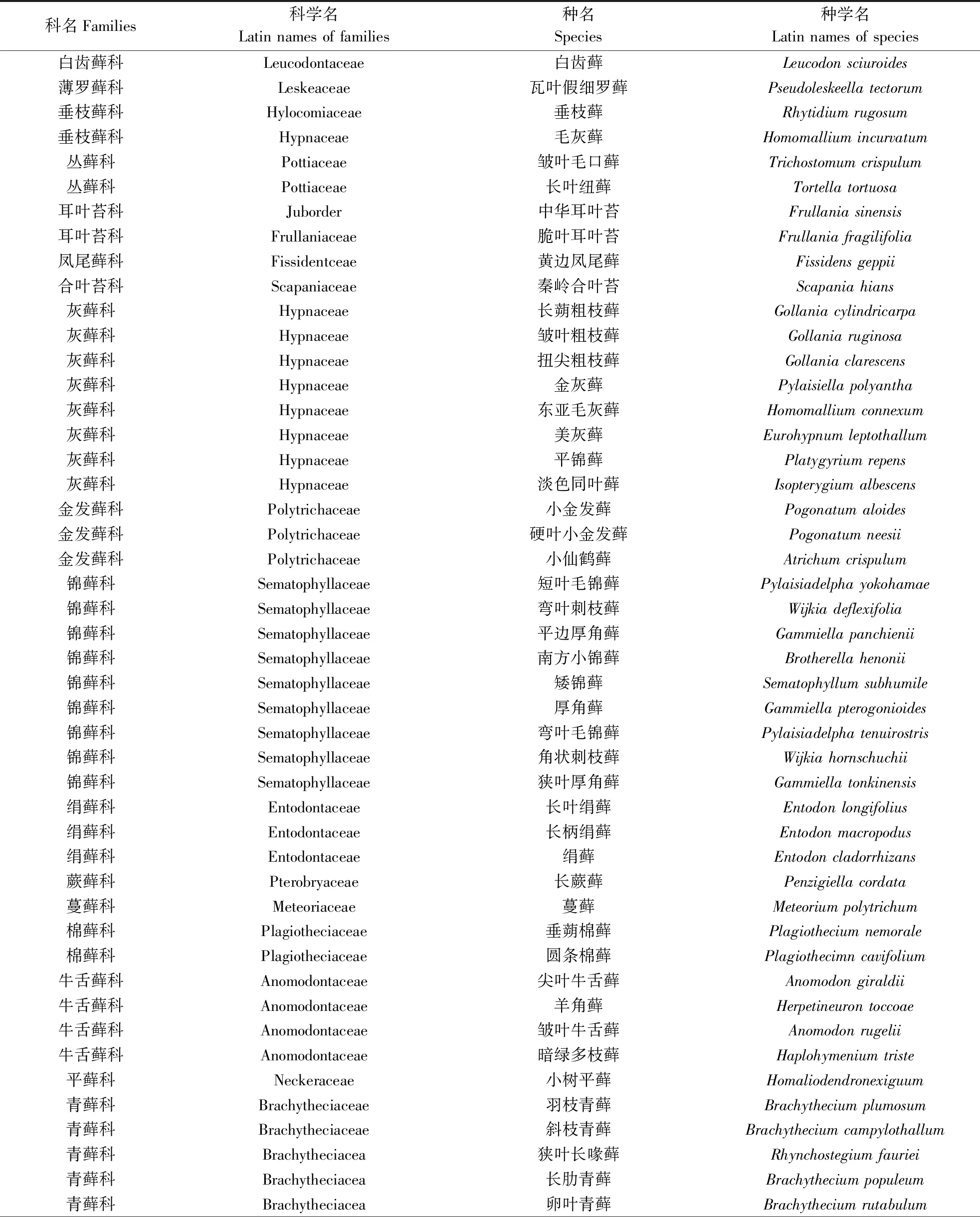
表5 白云山国家森林公园动态监测样地71种苔藓植物组成名录Table 5 List of 71 species composition of bryophytes in the dynamic monitoring plots of Baiyun Mountain National Forest Park
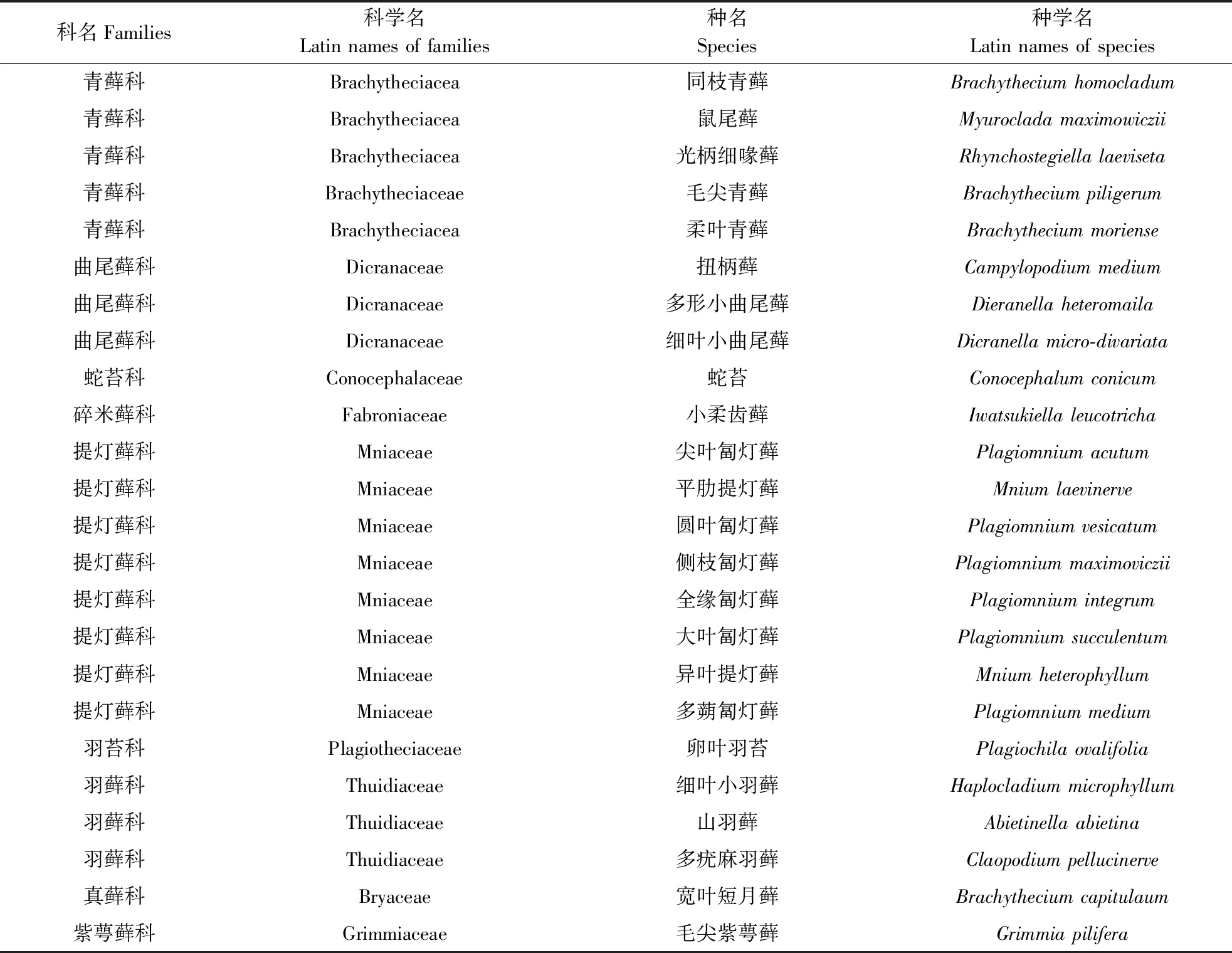
续表5 Continuing table 5
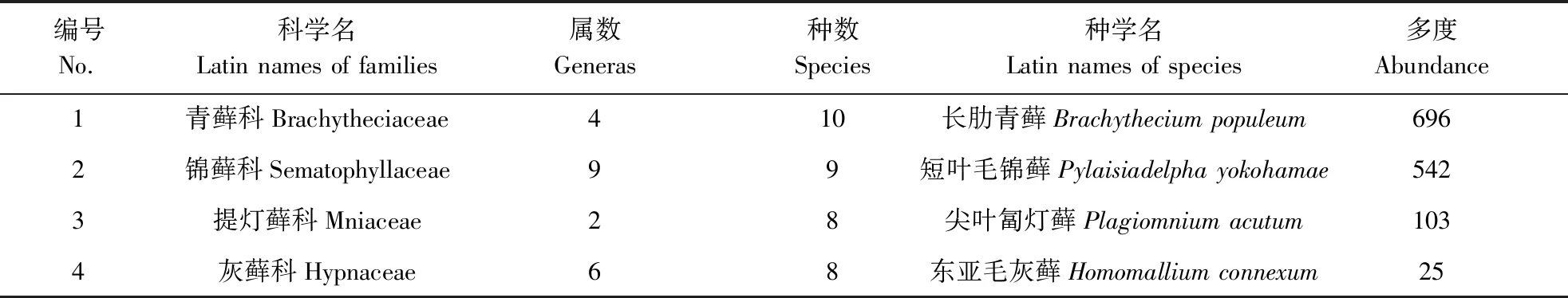
表6 动态监测样地71种苔藓植物优势科的属、种统计及多度排前4的物种Table 6 The top4 species in genera,species and abundance of dominant families of 71 species of bryophytes in the dynamic monitoring plots
基于rps4构建白云山国家森林公园动态监测样地71种苔藓植物的ML系统发育进化关系树,如图4所示。从系统发育进化关系树上可以看出,5种苔类和66种藓类形成2大分支,得到98%的支持率。其中,作为优势科的青藓科(Brachytheciacea)所有物种以93%的高支持率聚在1个大分支上;同属于羽藓科(Thuidiaceae)羽藓属(Abietinella)的山羽藓(Abietinellaabietina)、多疣麻羽藓(Claopodiumpellucinerve)和细叶小羽藓(Haplocladiummicrophyllum)以79%的中支持率聚在与青藓科相同的系统发育进化关系树分支上,说明物种之间亲缘关系相近,符合形态分类的理论。平边厚角藓(Gammiellapanchienii)、南方小锦藓(Brotherellahenonii)、厚角藓(Gammiellapterogonioides)、狭叶厚角藓(Gammiellatonkinensis)、弯叶刺枝藓(Wijkiadeflexifolia)、角状刺枝藓(Wijkiahornschuchii)、矮锦藓(Sematophyllumsubhumile)、弯叶毛锦藓(Pylaisiadelphatenuirostris)、短叶毛锦藓(Pylaisiadelphayokohamae)则以58%的弱支持率聚为另外1个大分支上。
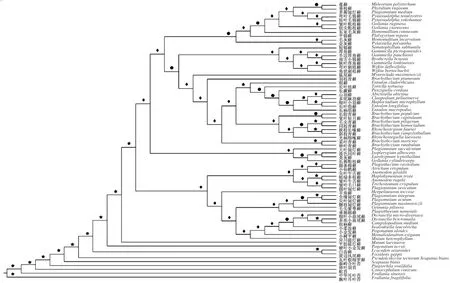
注:比例尺代表苔藓植物种间距离。节点支持率数值用星号★为高支持率、圆形●为中支持率、菱形◆为弱支持率。Note:Scale bar represents the distance between bryophytes.Point support value with asterisk ★ for high support rate,●for support rate,◆ for weak support rate.图4 基于rps4构建白云山国家森林公园动态监测样地71种苔藓植物的ML系统发育进化关系树Fig.4 ML phylogenetic relationship tree of 71 species of bryophytes in the dynamic monitoring plots of Baiyun Mountain National Forest Park constructed based on rps4
4 结论与讨论
测序成功率、物种识别成功率和综合成功率是评价DNA条形码片段是否适用于系统发育群落生态学研究的重要指标[22]。本研究中,rps4基因的测序成功率和物种识别成功率都明显高于rbcl和trnL-F,分别达到了98.59%和97.18%,推测是因为rps4基因在苔藓植物中的叶绿体基因组中属于偏保守的基因片段,在进化水平上变异速度较慢[23]。此外,尽管rps4基因进化速度慢,但是其序列内部的间隔区比例很小,能更好进行高级分类单元的序列比对,因此在苔藓植物的科级和属级水平具有较好的分辨率,所以被广泛应用到苔藓植物的系统重建和分析中[15]。本研究中trnL-F和rbcl基因的测序成功率和物种识别成功率都低于rps4基因。但依旧表现出适用于苔藓植物系统发育关系重建的潜力。trnL-F基因位于叶绿体的非编码区,功能编码区进化速度小,受外界因素变动压力小,在分类水平上用于系统关系的重建比较合适[16]。在苔藓植物的系统发育关系研究中,张安世等[17]曾基于叶绿体trnL-F基因序列对10种苔藓植物亲缘关系进行分析,构建出了可信度高的进化树。本研究中trnL-F和rbcl基因的测序成功率分别为94.89%和87.32%,物种识别成功率分别为90.14%和77.64%,表明二者依然是研究藓类物种系统重建时潜在的可参照的基因片段,即rps4、trnL-F和rbcl基因在苔藓植物中均具备了DNA条形码的通用性。相对于单一片段,在2个片段的不同组合中,rps4+trnL-F基因的测序成功率和物种识别率最高,分别达到了95.77%和91.54%。但与单一片段的rps4基因相比,苔藓植物的测序成功率和物种识别率都是降低的,这种现象在木本植物中鲜有报道,但是在苔藓植物中确是有过类似的现象。赵丽嘉等[18]研究发现2个基因组合片段时鉴别成功率最高,但当增加至3个基因组合片段时鉴别成功率反而有所降低。本研究结果为后续苔藓植物亲缘关系分析、谱系和演化以及遗传鉴定等方面的研究提供了理论基础。
此次苔藓植物DNA条形码研究是基于白云山国家森林公园动态监测样地数据,共鉴定出25科45属71种苔藓植物,其中藓类21科41属66种,苔类4科4属5种,序列分析结果和形态分类学中的结果基本一致。和以往研究不同,本研究是在单一科或者属内苔藓植物系统发育关系的重建和单一苔藓植物的DNA序列的鉴别。与典型的传统方法构建系统发育进化树相比,DNA条形码片段物种识别速度快,利用其构建的系统发育进化树能够为末端的低阶分类单元提供准确的位置,从而构建出精度更高的苔藓植物系统发育进化树[23]。本研究将白云山国家森林公园动态监测样地的苔藓植物划分成2个大分支,青藓科、锦藓科在样地中为优势科,科内高支持率的物种同聚在1个大分支上。相似的苔藓植物物种区分度高,亲缘关系近的苔藓物种聚集在一起,而亲缘关系远的苔藓物种相对比较分散,符合形态分类学的理论[24]。利用苔藓植物群落系统发育进化树有助于直观了解白云山国家森林公园动态监测样地内苔藓植物群落中物种亲缘关系。本研究表明,DNA条形码可以作为生态学研究的工具,为大样地研究苔藓植物的生态学提供新的发展方向,有助于大样地中苔藓植物研究的更深入开展,并且能够更加准确构建样地内植物群落的系统发育关系,区分开相似的苔藓植物物种,促进系统发育关系在生态学中的发展。以大样地为平台,在更大范围内研究苔藓植物的系统发育关系,有利于苔藓植物谱系结构和苔藓植物谱系生态学的研究,并为后期研究暖温带-北亚热带过渡区苔藓植物群落的共存机制和物种多样性的保护提供清晰的系统发育关系基础。
