白酒酿造窖泥未培养微生物菌群的可培养化策略
2020-10-22卢萌萌任聪聂尧徐岩
卢萌萌,任聪,2*,聂尧,2*,徐岩,2
1(江南大学 生物工程学院,酿造微生物学与应用酶学研究室,江苏 无锡,214122) 2(工业生物技术教育部重点实验室 (江南大学),江苏 无锡,214122)
从酿造方式来分,白酒可以分为窖泥依赖型与窖泥非依赖型。窖泥依赖型白酒主要包括浓香型、酱香型、凤香型、芝麻香型、兼香型和特香型白酒等;窖泥非依赖型白酒主要包括清香型、老白干香型、米香型和豉香型白酒等。窖泥中富含多种多样的微生物,如己酸菌、丁酸菌等,这些微生物多为厌氧菌,其代谢产生的脂肪酸、脂肪醇与酒醅中的乙醇、乳酸和乙酸等物质反应生成了种类繁多的酯类物质,赋予窖泥依赖型白酒窖香浓郁、绵甜等风味特征。在众多窖泥依赖型白酒中,浓香型白酒的质量与窖泥质量的相关性最为密切,优质窖泥是生产优质浓香型白酒的重要决定因素之一,而影响窖泥质量的根本因素为定植于其中生长的窖泥微生物。此前有研究表明,窖泥中存在的微生物可至少归类于225个属,其中至少31个属为主要的高丰度微生物[1]。然而,除部分己酸菌和丁酸菌外,其余众多窖泥微生物的具体物种信息及其在窖泥和酿造过程中的功能尚未可知。由ZHU等[2-3]分离于某浓香型白酒窖池中的梭菌纲微生物CPB6菌株,被鉴定为1株重要的产己酸菌;CHAI等[4-5]从窖泥中分离得到了多株梭菌纲下的产丁酸细菌。除梭菌纲微生物外,窖泥中还栖息着如拟杆菌纲、甲烷微菌纲和甲烷杆菌纲等丰度较高的微生物,其中产甲烷菌被推断为具有通过与产酸细菌进行互营代谢促进己酸生成的功能[6]。
对于窖泥微生物的结构和功能解析,可利用扩增子测序技术解析窖泥微生物结构,而广泛采用的二代高通量测序技术因读长限制,一般仅能将微生物种类精确鉴定到属水平;对窖泥微生物的酿造功能解析需借助基于全基因组解析的宏基因组学技术,应用该项技术的最大障碍在于提取完成的DNA较为困难,目前仅有少数报道通过宏基因组学技术手段对窖泥微生物结构和功能进行解析[7]。富集培养方法是实现窖泥微生物全基因组结构解析的重要途径。然而现有的研究表明,实验室人工条件下富集得到的微生物菌群组成大多结构单一,大量窖泥微生物仍然处于难以被富集和培养的状态。这对全面研究窖泥微生物菌群在浓香型白酒发酵过程中的作用造成较大阻碍。
富集培养通常被作为环境微生物可培养化研究的首要步骤。为尽可能多地实现未培养窖泥微生物在实验室环境中的可培养化,本研究采用经过配方改进的CGM[8]、NBM[9]以及MCI[10]三种培养基,对浓香型白酒窖泥菌群进行富集。其中CGM培养基属于富营养培养基,NBM和MCI属于寡营养培养基。随后结合16S rRNA基因扩增子测序技术,对富集菌群结构和动态变化进行分析,挑选出一种适合于最大程度还原窖泥样品原位菌群种类的富集培养基。本研究发现,寡培养条件对于最大程度富集获取窖泥中的未培养微生物菌群具有重要的作用,为实现更多种类的未培养窖泥微生物可培养化奠定了坚实基础。
1 材料与方法
1.1 实验材料
1.1.1 富集样品
富集用种泥来自于江苏省某浓香型白酒酒厂窖池。
1.1.2 培养基组成及相关试剂
CGM培养基(g):葡萄糖20,(NH4)2SO42,K2HPO41,KH2PO40.5,蛋白胨10,酵母粉10,MgSO4·7H2O 0.1,FeSO4·7H2O 0.15,CaCl20.01,MnSO40.01,CoCl2·6H2O 0.002,ZnSO40.002,1 g/L的刃天青溶液1 mL,加蒸馏水定容至1 L,灭菌前调pH至7.0±0.2,121 ℃灭菌20 min。
NBM培养基(g):KNO31,酵母粉3,蛋白胨5,牛肉膏3,1 g/L的刃天青溶液1 mL,加蒸馏水定容至1 L,灭菌前调pH至7.0±0.2,121 ℃灭菌20 min。
MCI培养基(mL):矿质元素溶液50,微量元素溶液1,B族维生素溶液5,NaHCO3溶液70,Na2SO4溶液20,三水合乙酸钠5 g,酵母粉1 g,蛋白胨1 g,1 g/L的刃天青溶液1 mL。灭菌前调pH至7.4±0.2,121 ℃灭菌20 min。灭菌后将维生素溶液、NaHCO3溶液和Na2S溶液在厌氧条件下按比例添加至培养基内,定容至1 L。
矿质元素溶液母液(g):KH2PO410,MgCl2·6H2O 6.6,NaCl 8,NH4Cl 8,CaCl21,添加蒸馏水定容至1 L。
微量元素溶液母液(g):ZnSO4·7H2O 0.1,MnCl20.03,硼酸0.3,CoCl2·6H2O 0.2,CaCl20.01,NiCl2·6H2O 0.02,钼酸钠0.03,FeCl2·4H2O 1.5,添加蒸馏水定容至1 L。
B族维生素溶液母液(mg):烟酸20,钴胺素20,盐酸硫胺10,对氨基苯甲酸10,盐酸吡哆醇50,泛酸钙5,添加蒸馏水定容至1 L,过滤除菌。
NaHCO3溶液:NaHCO35 g,添加蒸馏水定容至100 mL,过滤除菌。
Na2S溶液:Na2S 3 g,添加蒸馏水定容至100 mL,121 ℃单独灭菌20 min。
以上试剂中,酵母粉与蛋白胨购于Oxoid公司,其余常规试剂购于国药集团化学试剂有限公司。
1.1.3 主要仪器与设备
厌氧产气袋,日本三菱瓦斯化学株式会社;Forma 1029厌氧培养箱,美国Thermo Scientific公司;NanoDrop 8000蛋白核酸测定分光光度计,美国Thermo Fisher NanoDrop技术公司;FE 20型pH计,瑞士Mettler Toledo公司;DNeasy土壤微生物基因组DNA提取试剂盒,德国Qiagen公司。
1.2 实验方法
1.2.1 窖泥接种和菌群培养
CGM培养基:将培养基分装至50 mL离心管中,随后在厌氧条件下接种质量浓度100 g/L的窖泥样品,在37 ℃厌氧条件下进行静置培养。
NBM培养基:将培养基分装至50 mL离心管中,随后在厌氧条件下接种质量浓度100 g/L的窖泥样品,在37 ℃厌氧条件下进行静置培养。
MCI培养基:灭菌前将培养基等分至100 mL血清瓶内,灭菌后添加Na2S、NaHCO3和B族维生素溶液定容至100 mL,随后在厌氧条件下接种质量浓度100 g/L的窖泥样品,在37 ℃厌氧条件下静置培养。
1.2.2 宏基因组DNA提取
富集时序进程样品宏基因组DNA提取通过DNeasy土壤微生物基因组DNA提取试剂盒提取完成,操作步骤参考说明书。
1.2.3 PCR扩增及扩增子测序
本研究选取16S rRNA基因的V4区进行PCR扩增,对CGM、NBM和MCI的富集样品菌群结构进行分析,扩增引物为515FmodF(5′-GTGYCAGCMGCCGCGGTAA-3′)和806RmodR(5′-GGACTACNVGG-GTWTCTAAT-3′)[11-12]。对扩增的PCR产物进行建库,使用PE250策略在Illumina Miseq平台进行测序。测序数据的分析通过上海美吉生物制药有限公司的i-sanger云平台完成。使用FLASH进行高质量碱基和pair-end双端reads拼接[13]。短于50 bp的序列通过使包含在QIIME(ver.1.9.1)[14]中的Trimmomatic移除。之后,使用Usearch 7.0[15]中的UPARSE对97%相似水平下的操作分类单元(operational taxonomic units,OTU)进行聚类统计分析。将OTU代表序列与Silva数据库(ver.128)进行比对,得到每个OTU所对应的物种分类信息。
1.2.4 宏基因组测序
使用宏基因组测序技术对MCI培养基典型富集样品的菌群结构和功能信息进行分析。建库宏基因组的DNA由早期和中期富集样品的DNA混合而成。宏基因组DNA的文库构建和测序由上海美吉生物制药有限公司完成。通过NEXTFLXTM快速DNA-Seq试剂盒构建400 bp的文库,使用PE150策略在Illumina HiSeq 2500平台上进行测序。通过使用Seqprep和Sickle获得高质量pair-end双端reads。通过使用Megahit[16]对测序数据进行多重混合拼接组装,得到拼接效果最佳的序列。通过使用CDD-HIT[17]软件对样品中所有的预测基因序列进行聚类(默认参数:90% identity,90% coverage),取每个类别最长的基因作为代表序列。构建非冗余基因集。使用DIAMOND[18]中的BLASTP(BLAST Version 2.2.28+)[19]将非冗余基因集与NR数据库进行比对,通过对应的分类学信息数据库获得物种注释结果。
2 结果与分析
2.1 不同培养基富集条件下的菌群结构差异
本研究旨在通过使用不同的培养方法探究最适于培养和富集窖泥微生物菌群的培养基条件。首先使用了培养梭菌较为常用的CGM培养基,扩增子测序显示CGM富集条件下相对丰度>1%的细菌菌群仅归属到3个科,包括瘤胃菌科(Ruminococcaceae)、乳杆菌科(Lactobacillaceae)和Clostridiaceae_1(图1-a)。其中梭菌科微生物Clostridiaceae_1是此种富集培养条件下得到的相对丰度最高的类群,丰度达到了79.33%,菌群结构较为单一(图1-a)。自然条件下的微生物通常生活在营养缺乏,较为严苛的环境下,这些微生物在营养丰富,含有大量碳源、氮源的条件下则有可能难以生长,较为贫瘠的培养条件则有可能获得更多的物种多样性[20-21]。以此为基础,去除速效性碳源(葡萄糖)的NBM培养基被选择用来对窖泥菌群进行富集培养。NBM富集条件下相对丰度>1%的细菌菌群归属到6个科,包括消化链球菌科(Peptostreptococcaceae)、肠球菌科(Enterococcaceae)、瘤胃菌科(Ruminococcaceae)、Family_ⅩⅢ、Clostridiaceae_1和Family_Ⅺ_o_Clostridiales(图1-b),其中梭菌纲微生物在富集菌群中仍占主导地位,比如Clostridiaceae_1和Family_Ⅺ_o_Clostridiales相对丰度分别达到了21.60%和45.15%(图1-b)。结合之前的研究报道,酿造体系中的产己酸丁酸菌多属于梭菌[3-4, 22-23],可在营养丰富的条件下富集,而NBM培养基中含有共计11 g/L的速效氮源成分,这或许是梭菌纲微生物仍在菌群结构中占比较高的原因。随后,我们采用不含速效碳源葡萄糖且将氮源质量浓度(2 g/L)进一步降低的MCI培养基来富集窖泥菌群。如图1-c所示,MCI培养基富集培养条件下,科水平检测到的菌群数量明显增加,达到15个科,其中古菌2个科、细菌13个科。采用MCI培养基培养时,除常见梭菌科微生物外,其他窖泥微生物也明显富集,如甲烷鬃菌科(Methanosaetaceae)、理研菌科(Rikenellaceae)、纤维杆菌科(Fibrobacteraceae)和norank_c_BRH-C20a科是富集菌群中主要的主导菌群,相对丰度分别为10.90%、27.98%、15.10%和11.97%。上述结果表明,不含葡萄糖、降低氮源的寡营养条件有利于窖泥菌群的富集,且能同时富集出细菌和古菌。

a-CGM富集菌群科水平结构;b-NBM富集菌群科水平结构;c-MCI富集菌群科水平结构图1 不同培养基富集条件下科水平群落结构组成Fig.1 Microbial community structures at family level under different enrichment culture conditions 注:相对丰度<1%的科合并到未分类序列
2.2 MCI培养基富集时序进程中的菌群动态变化
由图1可知,在MCI培养基富集条件下,所得到的菌群结构中包括产甲烷古菌。为进一步分析MCI培养基富集过程中的菌群动态变化,分别接种来源于某浓香型酒厂不同窖池窖泥R1和R2,窖龄分别为4和10年,作为生物学重复。基于扩增子测序技术对2个生物学重复窖泥样本在时序进程中的菌群变化进行分析。在MCI富集培养下,2个生物学重复的富集样品中各自检测得到27和32个原核生物科。其中有17个科在2个样品中都能检测到。因富集培养的窖泥接种质量浓度100 g/L,在本研究中,某成功富集科(family)被定义为在任一生长阶段的富集体系中不低于其在对应窖泥中相对丰度的10%的科。如图2所示,在窖泥样品和任意富集样本中相对丰度为1%及以上的科被筛选显示出来。
对于生物学重复1,窖泥中相对丰度为1%及以上的科为16个,包括普雷沃氏菌科(Prevotellaceae)、理研菌科(Rikenellaceae)、Dysgonomonadaceae、Cloacimonadaceae、Family_XI_o__Clostridiales、Family_XIV、太阳杆菌科(Heliobacteriaceae)、瘤胃菌科(Ruminococcaceae)、互营单胞菌科(Syntrophomonadaceae)、Gracilibacteraceae、unclassified_c__Clostridia、Synergistaceae、Acholeplasmataceae、unclassified_Bacteria、甲烷八叠球菌科(Methanosarcinaceae)和甲烷杆菌科(Methanobacteriaceae)。除甲烷杆菌科、Cloacimonadaceae、Family_XIV和互营单胞菌科4个科外,其他科均被成功富集出来,富集有效性为75%(12/16)。窖泥中相对丰度<1%的6个科,包括Clostridiaceae_1、消化球菌科、Methanomethylophilaceae、Lachnospiraceae、norank_p__Armatimonadetes和氨基球菌科(Acidaminococcaceae),也被成功富集。此外,还有5个科包括芽孢杆菌科(Bacillaceae)、肠杆菌科(Enterobacteriaceae)、甲烷鬃菌科(Methanosaetaceae)、消化链球菌科(Peptostreptococcaceae)、Oligosphaeraceae,在窖泥中的丰度低于扩增子测序检测限,在窖泥中甚至无法检出,但仍可被富集出来,其中Oligosphaeraceae在富集中相对期样品中丰度甚至达到10.57%。

图2 窖泥菌群经MCI培养基富集后在不同阶段原核微生物菌群结构与种泥原核微生物菌群结构比较Fig.2 Comparation of microbial dynamics in typical enrichment cultures at different stages using MCI medium and the microbial community structure in pit muds at family level 注:相对丰度<1%的科合并到未分类序列。R1表示窖泥样本1;R2表示窖泥样本2;4、8和12 d等表示富集的培养天数;取样为连续取样
对于生物学重复2,窖泥中相对丰度为1%及以上的科为8个,包括Dysgonomonadaceae、norank_o__DTU014、动性球菌科(Planococcaceae)、互营单胞菌科(Syntrophomonadaceae)、Thermoanaerobacteraceae、太阳杆菌科(Heliobacteriaceae)、甲烷杆菌科(Methanobacteriaceae)和甲烷微菌科(Methanomicrobiaceae)。其中,Dysgonomonadaceae、互营单胞菌科和太阳杆菌科被成功富集,富集有效性为37.5%(3/8)。而窖泥中相对丰度<1%的13个科,包括理研菌科(Rikenellaceae)、芽孢杆菌科(Bacillaceae)、Clostridiaceae_1、Clostridiales_Incertae_Sedis、Family_XIII、Gracilibacteraceae、毛螺菌科(Lachnospiraceae)、消化球菌科(Peptococcaceae)、瘤胃菌科(Ruminococcaceae)、SRB2、Family_XI_o__Clostridiales、甲烷八叠球菌科(Methanosarcinaceae)和Methanomassiliicoccaceae,均被成功富集。此外,还有11个科为窖泥中几乎检测不到的低丰度微生物,包括Bacteroidales_UCG-001、Cloacimonadaceae、纤维杆菌科(Fibrobacteraceae)、norank_c__BRH-c20a、消化链球菌科(Peptostreptococcaceae)、肠球菌科(Enterococcaceae)、Oligosphaeraceae、norank_c__Berkelbacteria、norank_o__Candidatus_Moranbacteria、unclassified_Bacteria和Pedosphaeraceae,也被成功富集,其中norank_o__Candidatus_Moranbacteria在富集中期最高相对丰度达到15.06%。
上述富集分析表明,采用不同的窖泥作为种泥,使用MCI培养基进行培养均可富集到多样性较高的菌群,富集得到的厌氧菌类型与初始窖泥微生物种类有关。而且使用这种寡营养的培养方式不仅可以富集到窖泥中丰度较高的微生物,还能富集到窖泥中的低丰度微生物。这在其他环境微生物的富集培养研究中也得到类似印证,如MU等[24]通过使用海水为主要基质的寡营养培养基对海洋微生物进行培养过程中发现富集培养可有效提高难培养微生物的丰度,从而达到检测阈值,提高富集菌群的物种多样性。同时,应该注意到在富集过程中菌群结构发生的动态变化,如有些微生物在富集中期或后期相对丰度高于其他时期,比如生物学重复1富集样品中的Thermoanaerobacteraceae和甲烷微菌科(Methanomicrobiaceae)以及生物学重复2窖泥富集样品中的Cloacimonadaceae、纤维杆菌科(Fibrobacteraceae)和norank_o__Candidatus_Moranbacteria。这种现象也发生在其他类型环境微生物的富集培养研究中[24-25]。在富集过程中,微生物结构并非一成不变,说明在后续分离纯菌株的过程中,需要特别注意在不同生长阶段,目标菌可能具有不同的丰度,并关注富集过程中的时间依赖效应。
由图2可知,在MCI富集培养条件下,R1窖泥样品中甲烷菌群比例占原核菌群的19.26%,主导甲烷菌群为甲烷八叠球菌科(Methanosarcinaceae,占比2.31%)和甲烷杆菌科(Methanobacteriaceae,占比16.31%);而在富集培养体系中,甲烷菌群比例仅为7.95%~9.85%,且主导甲烷菌变为甲烷鬃菌科(Methanosaetaceae,占比4.62%~8.23%)。R2窖泥样品中甲烷菌群比例占原核菌群的62.76%,主导甲烷菌为甲烷杆菌科(34.31%)和甲烷微菌科(28.11%);而在富集培养体系中,甲烷菌群比例下降至3.30%~4.89%,主导甲烷菌是甲烷八叠球菌科(1.92%~3.17%)。富集样品的甲烷菌种类及丰度都与窖泥中的甲烷菌有差异,碳源类型与厌氧程度可能是导致这种差异的原因。本研究采用MCI培养基的最初目的为获得产甲烷菌群,但却意外发现采用该培养基同时可以富集得到丰富度较高的细菌菌群。之前的多项研究表明,梭菌纲微生物在窖泥微生物菌群中占据重要地位[26-27],传统的实验室培养方法也更倾向于得到高丰度的梭菌纲微生物,但得到的菌群结构较为单一[2-3]。而在本研究中,2个生物学重复富集样品中分别检测到11和15种梭菌纲微生物,包括太阳杆菌科(Heliobacteriaceae)、瘤胃菌科(Ruminococcaceae)、互营单胞菌科(Syntrophomonadaceae)、Lachnospiraceae、Clostridiaceae_1、Family_XI_o__Clostridiales、Gracilibacteraceae、消化球菌科(Peptococcaceae)、unclassified_c__Clostridia、消化链球菌科(Peptostreptococcaceae)、Family_XIV、norank_o__DTU014、Clostridiales_Incertae_Sedis、SRB2和Thermoanaerobacteraceae,所占比例范围分别为22.13%~25.43%(生物学重复1)、18.84%~51.63%(生物学重复2)。此外,在富集样品中也检测到了采用CGM培养基和NBM培养基未能成功富集的拟杆菌门(Bacteroidetes)微生物,如生物学重复1富集样品中的普雷沃氏菌科(Prevotellaceae)、理研菌科(Rikenellaceae)和Dysgonomonadaceae,所占比例达到16.55%~43.08%;生物学重复2富集样品中的Bacteroidales_UCG-001、Dysgonomonadaceae和理研菌科(Rikenellaceae),所占比例达到33.87%~42.71%。此前有研究表明,拟杆菌门属于对营养需求较低的菌群[28-29],与CGM(碳源20 g/L,氮源20 g/L)、NBM(氮源11 g/L)培养基相比,MCI培养基的有机营养成分(氮源2 g/L)较少,这或许符合了窖泥原位环境中某些寡营养菌群的营养需求。亦有研究表明,寡营养培养条件可培养出未培养的、多样性更丰富的微生物[30-31],或许正是因为这种寡营养条件抑制了优势生长菌,如利用葡萄糖等速效碳源生长的微生物,促使更多种类的难培养微生物得以被富集。
2.3 宏基因组学分析典型富集菌群组成
通过宏基因组测序技术对典型富集样品(生物学重复1)进行进一步的菌群结构分析。如图3所示,在域水平,相对丰度>1%的类群中,73.06%属于细菌,26.67%属于古菌。而扩增子分析显示,富集样品中古菌菌群比例仅为8.68%,这表明使用针对16S rRNA基因V4可变区的515FmodF/806RmodR引物进行扩增子分析可能低估了古菌的丰度。在门水平,扩增子测序可将菌群归属到拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、广古菌门(Euyarchaeota)、黏胶球形菌门(Lentisphaerae)、变形菌门(Proteobacteria)、互养菌门(Synergistetes)和未分类门unclassified_Bacteria,而宏基因组测序技术并未检测到黏胶球形菌门(Lentisphaerae)。在科水平,宏基因组学分析结果显示所得序列仅可归属到11个科,少于扩增子测序的分析结果(17个科)。其中,在宏基因组测序结果中,甲烷鬃菌科(Methanosaetaceae,20.09%)和紫单胞菌科(Porphyromonadaceae,17.53%)为主要的主导菌群;扩增子分析结果显示,富集样品中的主导菌群为Dysgonomonadaceae(25.15%)和理研菌科(Rikenellaceae,13.53%),而主导甲烷菌甲烷鬃菌科(Methanosaetaceae)最高相对丰度仅可达到6.76%,远低于宏基因组学分析结果,再次表明扩增子测序低估了古菌菌群的丰度。此外,值得注意的是,随着分类水平的提高,宏基因组和扩增子测序的未分类序列(others)的比例都越来越高,其中在科水平,宏基因组测序的未分类序列比例高达34.33%,这是由于窖泥中的大部分细菌和古菌尚未在全基因组水平得到鉴定,目前的宏基因组学数据库对于窖泥微生物基因组结构的解析度非常有限。
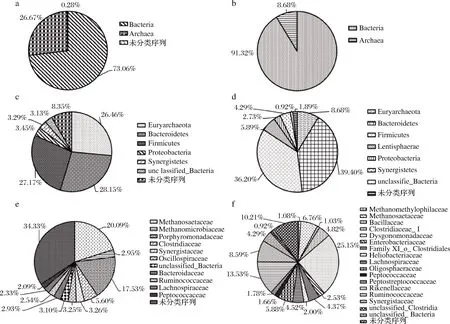
a-宏基因组域水平群落结构;b-扩增子测序域水平群落结构;c-宏基因组门水平群落结构; d-扩增子测序门水平群落结构;e-宏基因组科水平群落结构;f-扩增子测序科水平群落结构图3 富集样品扩增子测序与宏基因组域水平、门水平和科水平的菌群结构比对Fig.3 Comparison of the microbial compositions of a typical enrichment culture between 16S rRNA gene amplicon sequencing and metagenomic sequencing at domain, phylum and family levels 注:相对丰度<1%的菌群合并到未分类序列
用宏基因组数据对富集菌群物种信息进行注释,如表1所示。

表1 宏基因组学解析富集样品中微生物种水平结构Table 1 Microbial structure of a typical enrichment culture at species level by metagenomics
相对丰度>0.5%的菌群可注释到15个物种,其中有4种产甲烷古菌得到注释,占地22.34%,并可确定富集样品中的主体产甲烷菌为Methanosaetaconcilii(19.62%)。除此之外,得到注释的细菌比例仅为23.92%,而未得到分类注释的序列比例高达53.75%,包括部分未知的古菌和大量的细菌。因此依靠宏基因组技术虽然可以获得部分物种信息,但由于未培养微生物大量存在,而注释用的数据库信息不完善,对物种信息注释和功能研究不可避免地造成阻碍[7]。今后的窖泥微生物研究仍然需要重点分离与解析未培养古菌和细菌的基因组结构。本研究建立的寡培养方法为全面解析窖泥微生物的种类构建起从免培养技术到可培养技术的桥梁。
3 结论
本研究确立了寡营养方法为富集未培养白酒酿造微生物菌群的较优方式,这种培养方式不仅可以有效维持和培养窖泥原位菌群,而且使一些低丰度菌群也得到富集。本研究建立的寡培养方法适用于除己酸菌和丁酸菌以外的未培养窖泥微生物的可培养化研究。结合本研究建立的寡培养技术,对富集菌群进行宏基因组分析,可以得到部分准确的物种注释信息,提供了深入探索窖泥微生物酿造功能的技术手段。后续研究需进一步对窖泥微生物的可培养技术进行深入研究,以突破高通量测序的技术局限,更加准确地对白酒酿造窖泥微生物进行物种注释,从而解析窖泥厌氧菌的酿造功能和产风味功能,为提高窖泥依赖型白酒品质的厌氧微生物应用技术提供理论支撑。
