ITS2和psbA-trnH序列对海滩牵牛的分子鉴定研究
2020-10-19黄琛斐陈一村
黄琛斐,陈一村
(1.潮州市人民医院中药房,广东 潮州 521000;2.汕头大学医学院药理研究室,广东 汕头 515000)
海滩牵牛(Ipomoea stolonifera)是旋花科牵牛属植物假厚藤的全草,又名白花马鞍藤、小号马鞍藤,主要分布于台湾、福建、广东等沿海地区的高潮线上的沙滩或沙丘[1-2]。海滩牵牛是潮汕地区治疗风湿、类风湿关节炎的习用中药材[3],由于暂未作为药材收录于药典中,该药材暂无统一的质量标准,且市场上药材多经过修治,从植物形态进行鉴别存在困难,仅能根据传统的经验进行鉴别,存在一定的误差,因此深化海滩牵牛的分子鉴定研究对临床用药安全以及规范商品市场具有深刻意义。
目前海滩牵牛的研究多关于抗炎方面的研究[4-6],暂无DNA 条形码等相关的检测技术报道。常用的 DNA 条形码有 ITS、ITS2、psbA-trnH 及matK 等。其中,ITS2 序列是去除5.8S rRNA 序列和28S rRNA 序列的核糖体DNA 序列间隔区,psbA-trnH 片段是叶绿体DNA 的转录非编码序列,位于psbA基因和trnH基因之间。ITS2、psbA-trnH 等这些非编码区相对于编码基因在功能上的限制较少,变异程度较高,能提供较多的系统发育信息,且容易扩增和测序,现已被广泛应用于中药材的分子鉴定[7]。本研究运用ITS2 和psbA-trnH两个DNA条形码对11份海滩牵牛药材进行鉴定,并比较这两个序列的对海滩牵牛的鉴定能力,为海滩牵牛的质量标准研究提供理论支持。
1 材料与方法
1.1 材料
1.1.1 样品从广东潮州、揭阳、汕头、汕尾,福建南靖、莆田等地收集的海滩牵牛样品共11份,其中收集自福建南靖的8 号样本经汕头市中心医院药剂科罗梓河主任基源鉴定为海滩牵牛,作为本次实验对照组。通过BLAST 从GenBank 下载相似度高的同属植物厚藤(Ipomoea pes-caprae)的ITS2 序列和 psbA-trnH 序列各 2 条,甘薯(Ipomoea argillicola)的 psbA-trnH 序列 1 条,11 份海滩牵牛药材样品产地见表1。
1.1.2 主要试剂和仪器广谱性植物DNA 提取试剂盒(广州美基生物科技公司),ITS2、psbA-trnH序列正、反向引物由深圳华大基因科技有限公司合成,1 000×GeneGreen核酸染料(北京天根生化科技公司),琼脂糖(BIOWEST,西班牙),50×TAE缓冲液(上海索宝生物科技,中国),500 克摇摆式高速万能粉碎机(浙江屹立工贸公司),小型台式高速离心机(德国 Eppendorf 公司),Veriti96 PCR 仪(美国ABI公司),迷你核酸水平电泳仪(北京六一生物科技公司),凝胶成像分析仪(上海天能科技公司)。

表1 海滩牵牛样品信息
1.2 方法
1.2.1 海滩牵牛样品DNA提取将海滩牵牛药材进行粉碎处理,过4号筛后精密称取30 mg药材粉末于2 mL的离心管中,按植物DNA提取试剂盒的操作步骤提取海滩牵牛DNA。DNA于-20°C保存。
1.2.2 ITS2 和psbA-trnH 序列扩增及测序ITS2序列正向引物为5′-ATGCGATACTTGGTGTGAAT-3′,反向引物为5′-GACGCTTCTCCAGACTACAAT-3′。psbA-trnH 序列正向引物为 5′-GTTATGCAT GAACGTAATGCTC-3′,反向引物为 5′- CGCGCA TGGTGGATTCACAATCC-3′。PCR 反应体系:2×Taq PCRMix 酶 12.5 μL,正反向引物(2.5 μmol/L)各0.5 μL,模板DNA 1 μL,超纯水10.5 μL,总体积为 25 μL。ITS2 序列扩增程序:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸45 s,40 个循环;72 ℃延伸 10 min。psbA-trnH序列扩增程序:94 °C 预变性5 min;94 °C 变性1 min;55°C退火1 min,72°C延伸1.5 min;30个循环;72°C 延伸7 min。扩增产物用1%琼脂糖凝胶电泳检测。将显示清晰且单一的电泳条带对应的PCR扩增产物送深圳华大基因科技有限公司测序。
1.2.3 数据处理及分析使用BLAST 对测序结果进行比对,下载其中相似度高的序列作为后续分析数据。采用CodonCode Aligner V6.0.2 软件对ITS2、psbA-trnH 序列的测序峰图进行校对分析,除去低质量区,以MEGA 6.0 软件对所有的DNA条形码的序列进行分析比对,计算各序列的核酸组成以及K2P 遗传距离,构建邻接(neighbor-joining,NJ)进化树,以1 000次重复的步长设置检验各分支的支持率。
2 结果
2.1 序列分析
11份海滩牵牛样品ITS2引物的PCR扩增和测序成功率皆为100%,扩增特异性强,条带单一且清晰(图1)。ITS2 序列长度为349 bp,GC平均含量为60.2%,差异位点共48个(图2),其中1~7号样品的差异位点一致,共23个;8~11号样品差异位点一致,共25 个。psbA-trnH 序列扩增结果见图3,仅有1、3、4、8号样品显示清晰条带,PCR扩增成功率为36.4%;成功扩增并测序的仅有3和4号样品,测序成功率为18.2%,GC平均含量为13.9%。
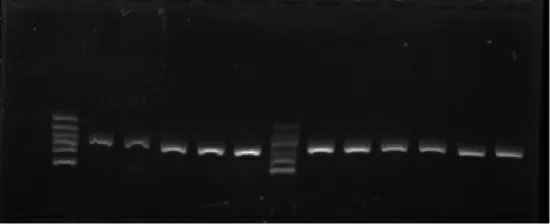
图1 海滩牵牛样品ITS2序列扩增

图2 海滩牵牛样品ITS2序列引物差异位点图

图3 海滩牵牛样品psbA-trnH序列扩增
2.2 遗传距离分析
通过对ITS2 区遗传距离的分析,发现海滩牵牛1~7号样品为1个种属,相互间的K2P遗传距离为 0.00,8~11 号样品为 1 个种属,相互间 K2P 遗传距离为 0.00,1~7 号样品与 8~11 号样品的种间遗传距离为0.13;同属植物厚藤的种间距离为0.01,与海滩牵牛11 个样品的种间遗传距离为0.34~0.55。种内遗传距离远小于种间遗传距离,即ITS2 序列能够区分海滩牵牛和厚藤。通过对psbA-trnH 区遗传距离的分析,发现海滩牵牛3~4号样品种内遗传距离为0.00,厚藤种内距离为0.78,种间距离为0.21~0.98,甘薯与厚藤2的种间遗传距离为0.00,即psbA-trnH序列无法把不同种的厚藤与甘薯区分开。
2.3 NJ树聚类分析
基于ITS2序列构建的NJ树(图4A)表明,不同来源的海滩牵牛样品聚为一支,能够跟同为牵牛属的植物(厚藤)区分开来。基于psbA-trnH 序列构建的 NJ 树(图4B)表明,海滩牵牛 3~4 号样品能够聚为一支,但厚藤的两个样品无法聚为一支,而是和不同种属植物(甘薯)聚为一支。

图4 基于不同序列构建的海滩牵牛、厚藤和甘薯的NJ树
3 讨论
本研究从7 个不同城市进行收集的11 份海滩牵牛实验样品,通过植物DNA 提取试剂盒提取总DNA,其ITS2 序列的PCR 产物测序成功率达100%,说明ITS2序列对于海滩牵牛的鉴定稳定性高,是有效鉴定海滩牵牛的DNA区段。
由于海滩牵牛目前在GenBank 上暂无相关的序列片段,因此仅从GenBank 上下载相似度高的同属植物(厚藤和甘薯)的ITS2和psbA-trnH序列共5条,与海滩牵牛样品进行比对。通过最小遗传距离法和基于ITS2 和psbA-trnH 序列构建的NJ 树都能区分海滩牵牛和其他种属的植物,但是psbA-trnH 序列无法明确分析牵牛属植物系统发育关系,而且psbA-trnH 序列在海滩牵牛中扩增和测序比较困难。本研究海滩牵牛1、8 号样品的psbA-trnH序列测序结果显示样本浓度低,加量测序仍无法成功测序。对样品DNA 提取进行了3 次重复实验,并对PCR反应体系进行多次调整(主要是调整模板DNA 含量),实验结果与前期结果相同。其原因可能是受药材的加工步骤、储存环境和储存时间的影响,导致了样品中的DNA 存在不同程度的降解。同时,psbA-trnH 序列GC 含量过低也可能是该序列测序成功率低的原因。
综上所述,本研究发现ITS2 序列能有效鉴定海滩牵牛及同属植物,而psbA-trnH 序列不适合作为海滩牵牛的DNA条形码技术鉴定序列。
