microRNA调控膜系统及离子转运介导青稞盐碱胁迫的应答
2020-10-09王玉林原红军扎桑杨春葆徐齐君
王玉林,原红军,扎桑,杨春葆,徐齐君
(省部共建青稞和牦牛种质资源与遗传改良国家重点实验室/西藏自治区农牧科学院农业研究所,西藏拉萨850032)
青稞(Hordeum vulgare var.nudum)是我国西部青藏高原常年种植的主要粮食作物和重要牲畜饲料来源[1],约占该地区粮食播种面积的80%左右。但是,由于对青稞食品需求量的增加以及气候变化的严重影响,青稞产量的提高对满足青藏高原地区粮食消费需求有着重要的意义[2]。青稞生长在极端环境,因此对非生物胁迫有着独特的适应机理。
土壤盐碱化是一种严重的非生物胁迫,影响作物生长并造成世界范围内作物产量损失[3]。据估计,在未来几十年中,盐碱胁迫可能会影响越来越多的耕地[4],并使植物遭到渗透胁迫、高pH胁迫和离子毒性。为了应对高盐碱胁迫,植物通过复杂的基因调控网络作出反应,改变基因表达水平并参与膜运输、信号转导和能量代谢相关的翻译后修饰[5-6]。但是,目前对植物盐碱反应的分子机制了解甚少,提高农作物的盐碱耐受性是一个重要的全球性问题。
MicroRNA(miRNA)属于一类小的单链RNA,它们在转录后水平上负调控基因表达[7]。许多研究表明,植物miRNA在应对非生物胁迫中起重要作用[8-9],如盐碱胁迫[10-12]。近年来,miRNA在模式植物和几种谷物中介导与胁迫适应相关基因的调控通路[13]。研究表明,不同植物物种在盐碱胁迫下可能具有不同的miRNA介导的调控策略。高通量测序技术已广泛用于miRNA鉴定[13-15],为青稞盐碱响应的miRNA检测及研究提供了很好的技术支持。
本研究的目的是在青稞中鉴定盐碱胁迫响应相关的miRNA,预测其靶标以及探索检测到的miRNA的功能,并揭示miRNA的表达和调控模式。研究结果可为进一步验证青稞在盐碱胁迫下的miRNA-mRNA调控模式提供重要的信息,并为解析青稞和其他农作物中盐碱胁迫响应的分子机理提供基础。
1 材料与方法
1.1 植物材料和盐碱处理
本研究以青稞地方品种0119和0226作为材料,其中0119对盐碱胁迫具有很高的抗性,而0226对盐碱胁迫敏感[16]。用2%H2O2将种子表面灭菌30 min,再用无菌水冲洗,放在25℃的黑暗环境下潮湿的薄纸中2 d。然后将发芽的幼苗转移至温室中的Hoagland营养液中,光温条件分别为14 h(光照)/10 h(黑暗)和22℃(光照)/20℃(黑暗)。盐碱胁迫处理,将2个材料的3周龄幼苗中的1/2移至添加了200 mmol/L Na+(NaHCO3和Na2CO3物质的量之比为1∶1)的培养液中72 h。其余1/2植物作为对照生长在Hoagland营养液中。
1.2 总RNA提取和miRNA文库的构建
分别取2个材料在盐碱胁迫(2、8、24、48、72 h)和对照(0、2、8、24、48、72 h)处理后的植株新鲜叶片,使用Trizol试剂(Invitrogen,Carlsbad,CA,美国)从中提取总RNA,并使用RNeasy Plant Mini Kit(Qiagen,Valencia,CA,美国)纯化。RNA样品的浓度和质量使用Nano Drop 2000分光光度计(Thermo Fisher Scientific,Inc.)通过测量A260/A280和A260/A230的比例来确定。将纯化的高质量RNA溶于无RNase的水中。取10μg总RNA样品,通过PAGE凝胶选择8~30个核苷酸的RNA片段。将RNA片段连接至5'接头和3'接头,通过PAGE凝胶再次过滤片段大小。使用One Step Primer Script miRNA cDNA合成试剂盒(Trans Gen)对连接的RNA进行RT-PCR,产生cDNA产物。cDNA文库通过凝胶纯化,并用安捷伦(美国,加利福尼亚州圣克拉拉)生物分析仪2100进行验证。最终使用HiSeq2000(Illumina)对文库进行深度测序。每个处理的每个时间点取3个生物学重复。本研究中使用的原始序列数据已提交GSA(Genome Sequence Archive)数据库[17]及中国科学院北京基因组研究所(BIG)生命与健康大数据中心(2019年会员),注册号为CRA001345,可在http://bigd.big.ac.cn/gsa上公开访问。
1.3 miRNA的鉴定和基因表达谱分析
对原始数据去接头[19]、过滤低质量读长序列之后,将miRNA读长检测序列与青稞参考基因组进行比对[18],使用miRDeep2(https://github.com/rajewskylab/mirdeep2)软件鉴定青稞miRNA。在所有样品中,miRNA读数均大于66,以此识别候选miRNA。使用BLAST将这些潜在的miRNA与Rfam数据库进行比对[20](版本:10.1),鉴定其他ncRNA(非编码RNA)。为了鉴定已知的miRNA,使用BLAST将潜在的miRNA标签与miRBase中存在的成熟miRNA进行比对[21],不超过2个错配的miRNA被认为是已知的miRNA。DESeq2 R包[22]用于鉴定差异表达的基因。显著性变化的阈值为fold-change(倍数变化)>2,校正后的P值<0.05。
1.4 靶基因预测和靶功能富集分析
使用psRobot(v1.2,http://omicslab.genetics.ac.cn/psRobot/downloads.php)软件在默认参数下预测miRNA的靶基因。使用Cytoscape 3.6.1可视化miRNA-mRNA调控网络[23]。使用Top GO R包[24]对显著差异表达的miRNA的靶基因进行功能分类和富集分析。
2 结果与分析
2.1 青稞miRNA的鉴定
对于所有66个miRNA文库,在将低质量读长序列和接头序列进行过滤后,共剩余897 112 430条净读长序列(clean reads),每个文库的读长总数范围为9 908 031~20 151 566条,唯一读长数目范围为1 059 453~5 097 449条。最丰富的miRNA的长度范围为20~24个核苷酸,最常见的miRNA的长度为21和24个核苷酸。读长比对率为58.01%~92.76%。在66个样品中,共13 757个潜在的miRNA有表达。在本分析中,与其他非编码RNA类型匹配的miRNA标签被去除,但是没有潜在的miRNA标签映射到Rfam数据库(http://rfam.xfam.org/)。分布在单个染色体上的miRNA数量在1 513(第4染色体)与1 996(第2染色体)之间,并且这些miRNA大致均匀地分布在整个染色体上。在本研究所有文库中,miRNA的相对表达水平分布为66~7 821 211个读长。
2.2 已知和新miRNA的BLAST比对
使用BLAST将潜在的miRNA序列与miRBase中公开的miRNA序列进行比较[13],鉴定已知的miRNA,结果显示:有225个miRNA在miRBase中是保守的,主要分布在hvu-miR5049和hvu-miR1436家族;其余的13 532个miRNA是推定的新型miRNA。长度在20~24个核苷酸的miRNA数量为11 759,其中28.3%长度为21个核苷酸,53.7%长度为24个核苷酸。miRNA前体的长度为34~113个核苷酸,平均长度为72个核苷酸。
计算所有miRNA的核苷酸组成,核苷酸中腺嘌呤(A)为23.5%,尿嘧啶(U)为26.4%,胞嘧啶(C)为18.6%,鸟嘌呤(G)为31.5%(图1)。
2.3 盐碱处理和对照青稞的miRNA表达模式
为了鉴定在盐碱胁迫下差异表达的miRNA,基于归一化表达水平的统计比较,对差异表达模式进行了分析。我们通过比较盐碱处理和对照样品分析了0226在5个应激阶段(2、8、24、48和72 h)中的miRNA表达模式,发现上调和下调的显著差异表达的miRNA在8和48 h阶段增加,在24 h阶段减少(图2-A)。对于0119发现了相同的表达模式(图2-B)。
比较了0119和0226品种中差异表达的miRNA(图3),大多数miRNA在0119或0226品种中被特异性上调或下调。
2.4 青稞miRNA的靶基因功能预测
miRNA的功能通常是靶基因的转录后调控,由于与特定mRNA的序列互补性,导致蛋白质翻译抑制或裂解[25],总共363个差异表达的miRNA具有潜在的靶标候选物,映射到了Swiss Prot数据库的靶标基因。miRNA预测的靶标数量差异很大,范围为1~2 107,总共为363个miRNA确定了7 608个唯一的目标候选物。大多数miRNA(224个)具有多个靶基因(2~100个),83个miRNA仅具有1个靶基因,56个miRNA每个具有100多个靶基因。miRNA及其靶基因的一些例子显示了相反的表达模式。该靶标预测分析表明,已鉴定的靶标基因调控了广泛的生物学过程。所有这些目标基因均按照基因本体(GO)数据库进行功能分类(表1)。在细胞成分(cellular component)类别中,前10个GO类别显示大 多 数 目 标 基 因 与 膜 相 关(GO:0016020,GO:0016021,GO:0031224,GO:0044425和GO:0012505等)。在生物过程(biological process)类别中,最富集的条目与运输相关(GO:0044765,GO:0006811,GO:0006810,GO:0006812,GO:0055085和GO:0030001)。在分子功能(molecular function)类别中,转运蛋白活性(GO:0005215)、离子结合(GO:0005507和GO:0030001)、过氧化物酶活性(GO:0004601)、氧化还原酶活性(GO:0016684)和抗氧化活性(GO:0016209)是显著富集。分析发现,与“膜”相关的miRNA-mRNA调控网络中,Thb-miRn724a-3p、Thb-miRn724-1-5p和Thb-miRn1158-3p位于网络的中心。KEGG通路富集分析表明,最富集的6个通路展示在表2中,其中“植物-病原体相互作用”和“植物激素信号转导”分别富集到248和149个靶向基因。此外,靶基因GO富集分析显示,目标基因在2 h阶段显著富集了氧化还原酶活性(GO:0016682,GO:0016491和GO:0016679)和阳离子结合(GO:0043169)。24 h时富集的GO条目是ATP酶活性(GO:0042623,GO:0042626和GO:0043492)和水解酶活性(GO:0016820)。在48 h的靶基因GO分析显示出金属簇结合(GO:0051540)、核苷酸结合(GO:0000166)和核苷三磷酸酶活性(GO:0017111)富集,而在72 h条件下的靶基因富集氧化还原酶活性(GO:0016682,GO:0016679和GO:0016491)和铜离子结合(GO:0005507)。
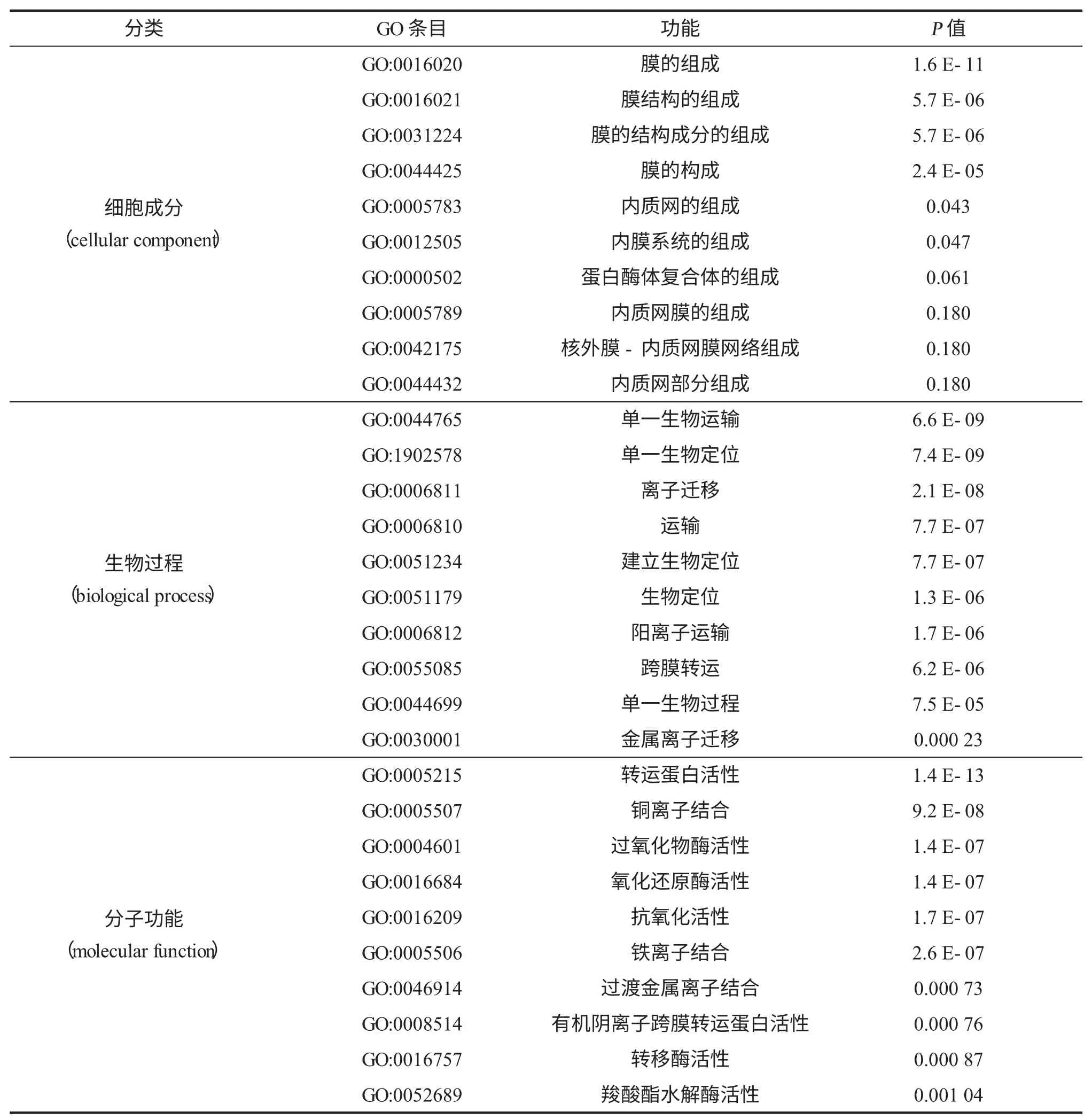
表1 所有盐碱反应性miRNA靶基因的GO功能分类

表2 所有盐碱反应性miRNA靶基因的KEGG通路富集的类别
3 讨论与结论
已有研究报道了对盐碱条件的两阶段生长反应[26-27],在玉米和小麦品种中已经证实了这种两阶段生长反应[28-30]。第一阶段主要的作用是水分胁迫或渗透作用,由于盐的积累,第二阶段的主要作用是离子性的。在本研究中,显著差异表达的miRNA数量在24 h下降。这种现象表明第一阶段可能发生在青稞用200 mmol/L Na+培养液处理的前24 h。GO富集分析表明,目标基因在2 h阶段主要为氧化还原酶活性相关的蛋白,随后在24、48及72 h,ATP酶活性、水解酶活性、核苷酸结合及铜离子结合相关的蛋白呈现显著的表达。这些结果与先前研究报道的生理现象[31-33]一致,这是对植物盐碱胁迫的反应。
本研究鉴定出的大部分miRNA是物种特异性的。在拟南芥[10]、玉米[11]和水稻[34]中,已鉴定出许多与植物对盐胁迫反应相关的miRNA。我们鉴定了13 757个miRNA,其中只有225个在大麦中保守,2 227个对盐碱胁迫有响应。这些miRNA可以与靶mRNA的非翻译区(UTR)或编码序列(CDS)互补结合,从而降低mRNA的表达水平[1,35]。尽管表达的miRNA可以负调控其靶标mRNA,但由于调控关系的复杂性,其靶标可能表现出多样化的表达模式。根据miRNA-mRNA调控网络的靶标基因的GO注释,我们推断“膜”的形成与盐碱胁迫应答有着密切的关系。在以前的研究中,为预测盐碱胁迫相关的靶标,发现许多靶标是SPL(类表面活性物质B蛋白)、MYB(myb域蛋白)、ARF(生长素应答因子)、AP2(花瓣分生组织发育蛋白APETALA2)、NAC(NAC结构域蛋白)和NF-Y(核转录因子Y)。NAC在盐度、寒冷、ABA和干旱胁迫下受到广泛调节[36-38]。在这里,盐和干旱胁迫广泛调节了一个NF-YA(核转录因子Y亚基A)靶标[39,34];确定了一组介导ARF调节的miRNA,ARF可能在植物对各种非生物胁迫(例如盐,冷和干旱胁迫)的反应中起重要作用[40-45];还鉴定了其他基因,包括调节植物生长和发育的SPL、MYB和AP2;认为许多编码重要酶和蛋白质的靶基因在盐胁迫反应中也起着重要作用。我们还发现,miRNA交叉调控了几个靶基因,且单个miRNA可以调控多个靶基因。灵活的调控模式表明,这些相互作用可以调控特定发育阶段和生活环境中的特定靶标。该研究可能有助于其他作物及植物盐碱逆境应答研究方法的完善,以提高农作物适应各种环境条件的能力。
