长链非编码RNA H19基因多态性与胃癌遗传易感性关系的研究
2020-09-15应伟王兴远江波周伟
应伟,王兴远,江波,周伟
广安市人民医院(四川大学华西广安医院)肿瘤科,四川 广安 638000
胃癌是常见的消化道恶性肿瘤,发病率及病死率均较高,发病率居恶性肿瘤第五位,病死率居恶性肿瘤第三位[1]。迄今为止,胃癌在中国、日本仍很普遍,但目前对胃癌发病机制的了解仍甚少[2]。证据显示遗传和环境因素与胃癌的发生密切相关,胃癌的发展是多种因素共同作用的结果,包含了多种表观遗传的改变[3-4]。长链非编码RNA(long non-coding RNA,lncRNA)长度约在200个核苷酸以上,被定义为在基因组中普遍转录的非蛋白质编码转录物[5]。其转录产物可以通过与染色质的修饰复合物结合,顺式和反式特异性沉默基因组位点,并参与细胞生长、凋亡、侵袭和转移[5-6]。H19是染色体11p15.5上的一个母系表达的印迹基因,编码一个带帽和剪接的RNA,并与癌症有关[7]。它是人类基因组中发现的第一个lncRNA,在哺乳动物的发育中起着至关重要的作用[8-11]。lncRNA的单核苷酸多态性(single nucleotide polymorphism,SNP)可影响基因表达和功能,并与人类多种疾病的易感性相关。因此,本研究拟通过对H19rs217727基因位点的SNP进行分析,研究其与胃癌之间的关联作用,现报道如下。
1 资料与方法
1.1 一般资料
收集2015年3月至2018年3月广安市人民医院收治的胃癌患者的病历资料。纳入标准:经病理检查确诊为胃癌。排除标准:有血液性或其他自身免疫性疾病和肿瘤病史;既往接受过放化疗。根据纳入、排除标准,共纳入128例胃癌患者,设为病例组,均抽取静脉血约5 ml。另选取广安市人民医院同期健康体检者130例为对照组。两组研究对象性别、年龄等临床特征比较,差异均无统计学意义(P>0.05)(表1),具有可比性。

表1 对照组与病例组研究对象的临床特征
1.2 主要试剂与仪器
lncRNA H19引物、2×Taq聚合酶链反应(polymerase chain reaction,PCR)Master Mix均购自Qiagen生物科技有限公司,标准酶试剂盒购自英国RNADOX公司,Gold ViewⅠ型核酸染色剂购自上海拜力生物科技有限公司,RsrⅡ限制性核酸内切酶购自NEB公司,核酸提取试剂盒购自北京庄盟国际生物基因科技有限公司。ChemiDoc XRC凝胶电泳成像系统购自美国Bio-Rad公司,DYY-6C型电泳仪购自北京六一公司,2-16K台式冷冻离心机购自德国Sigma公司,PTC200多通道PCR扩增仪购自美国MJ公司。
1.3 标本采集
采用真空抗凝管采集空腹静脉血5 ml,3000 r/min离心10 min,放于-80℃冷冻贮存、待检。
1.4 DNA 提取及基因分型
采用酚-氯仿抽提法提取DNA,通过Nanodrop检测浓度后进行SNP分型,用PCR-RFLP技术检测研究对象中lncRNA H19的基因多态性,rs217727 C>T基因引物:正义,5'-ACTCAGGAATCGGCTCTGGAAGGTG-3';反 义 ,5'-GATGTGGTGGCTGGTGGTCAACGGT-3'。PCR 反应体系 15 μl,含模板 DNA 1 μl,上游引物1 μl,下游引物1 μl,2×Taq PCR Master Mix 7 μl,去离子水 5 μl。PCR 反应条件:94 ℃预变性5 min,94 ℃ 30 s,63 ℃ 45 s,72℃45 s,35个循环;72℃延伸5 min。PCR产物分别用RsrⅡ酶、37℃恒温水浴箱进行酶切,2%琼脂糖凝胶100 V电泳40 min,溴化乙啶染色分析酶切结果。基因型的判读由2名研究人员进行,若出现判读不一致,则需重新检测。基因分型成功率达到100%。
1.5 统计学方法
采用SPSS 20.0统计软件进行数据分析,计数资料以例数及率(%)表示,组间比较采用χ2检验,计量资料以均数±标准差(±s)表示,组间比较采用t检验,单因素及多因素分析采用Logistic回归分析,以P<0.05为差异有统计学意义。
2 结果
2.1 凝胶电泳实验
lncRNA H19 rs217727 C>T被酶切为CC、CT、TT基因型,CC基因型显示为226 bp+21 bp,CT基因型为247 bp+226 bp+21 bp两条带,TT基因型为247 bp+21 bp;3种基因型中21 bp条带均不显示。(图1)
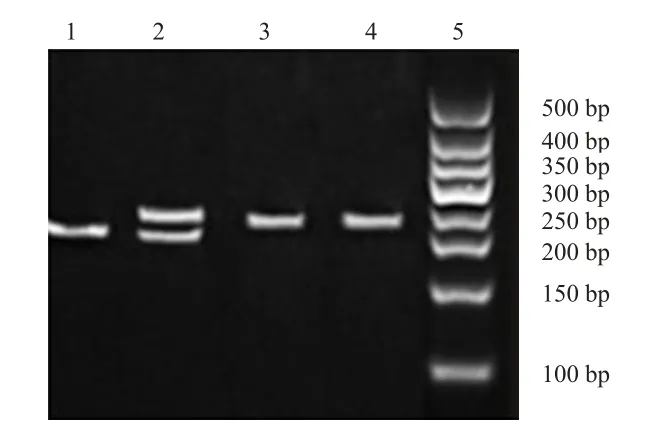
图1 rs 217727多态位点酶切图片
2.2 基因分型与测序
对照组中CC、CT、TT基因型分别为58例(44.6%)、58例(44.6%)、14例(10.8%),病例组中分别为 39例(30.5%)、63例(49.2%)、26例(20.3%)。随机抽取10%的样本进行DNA测序,其结果与PCR-RFLP分型结果一致(图2~图4)。Hardy-Weinberg遗传平衡检验提示P>0.05,对照组的基因型频率符合Hardy-Weinberg遗传平衡。
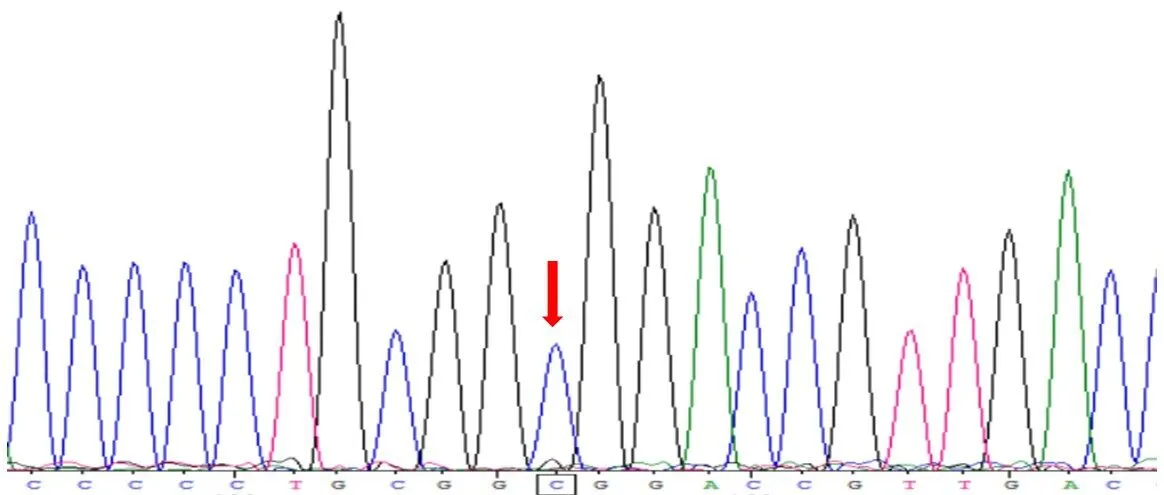
图2 lncRNAH19rs 217727CC基因型
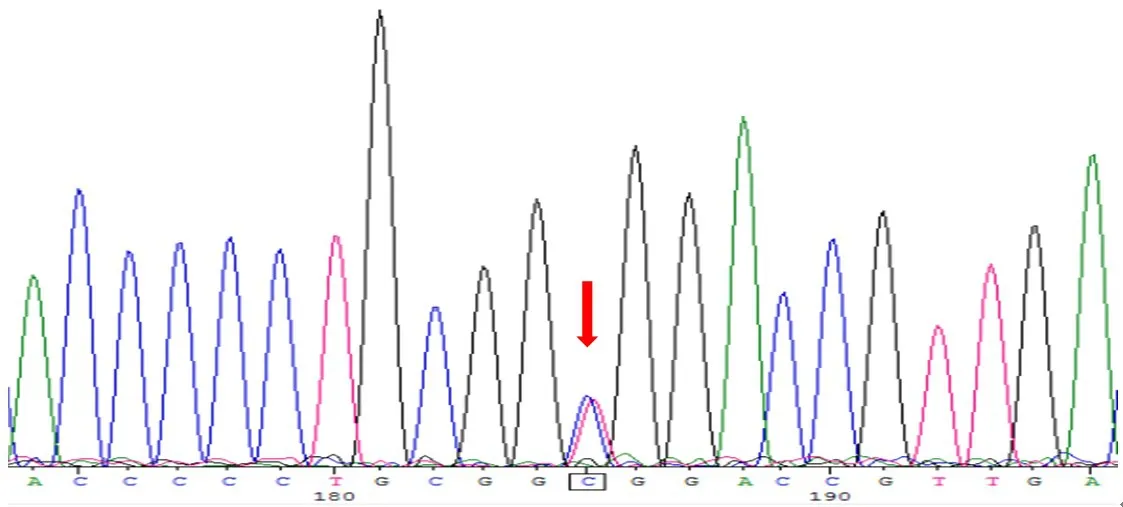
图3 lncRNAH19rs 217727CT基因型
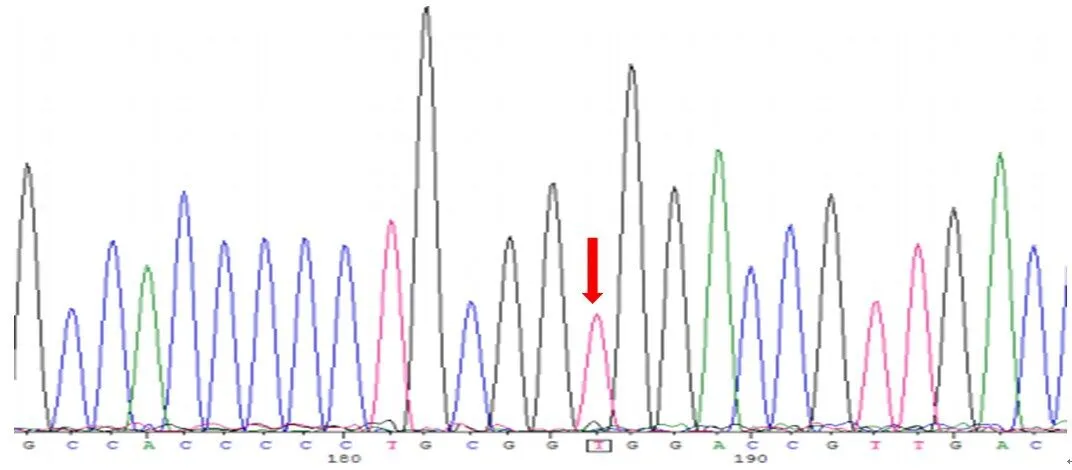
图4 lncRNAH19rs 217727TT基因型
2.3 lncRNAH19rs 217727C>T 与胃癌发生风险的关系
lncRNA H19 rs217727 C>T在对照组与病例组中进行不同基因模型下的Logistic回归分析结果显示,共显性基因模型,TT基因型与CC基因型比较,TT基因型增加胃癌的发病风险,OR=2.76,95% CI=1.28~5.94,P=0.009,校正混杂因素后OR=2.99,95% CI=1.37~6.56,P=0.006;显性基因模型 ,rs217727 CT+TT基因型使胃癌的发病风险增加1.84倍,OR=1.84,95% CI=1.10~3.09,P=0.020,校正混杂因素后 OR=1.84,95% CI=1.10~3.09,P=0.020;隐性基因模型,TT基因型胃癌发病风险是CC+CT基因型的 2.11倍,OR=2.11,95% CI=1.05~4.26,P=0.037,校正混杂因素后 OR=2.34,95% CI=1.13~4.83,P=0.022。此外,等位基因模型中,T等位基因显著增加胃癌的发病风险,OR=1.65,95% CI=1.16~2.36,P=0.006。(表2)
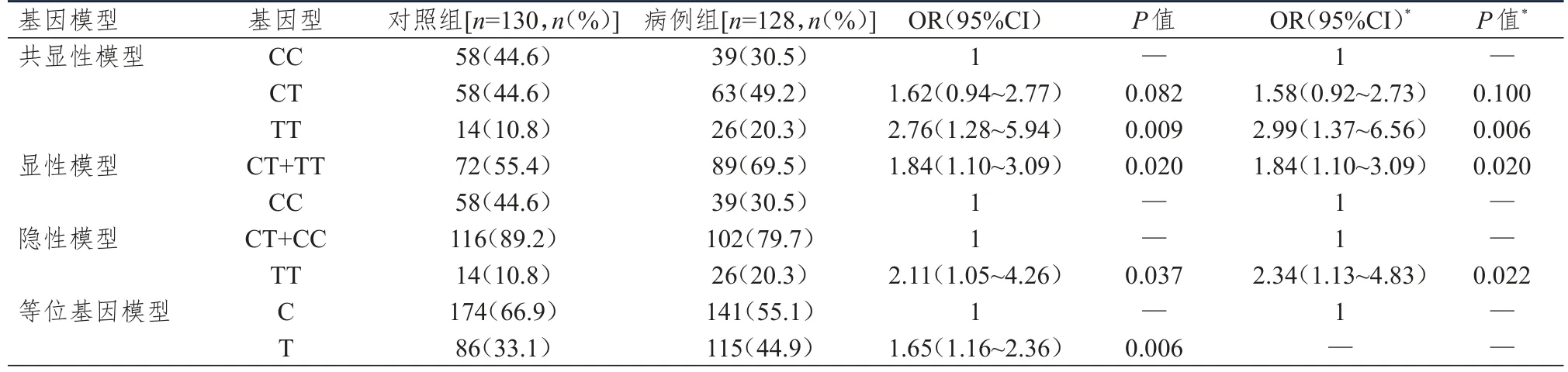
表2 lncRNA H19 rs217727与胃癌发生风险的关系
2.4 lncRNA H19rs 217727基因多态性与胃癌临床特征的关系
在病例组中,按照胃癌的分化程度、临床分期、淋巴结转移、远处转移进行分组,采用Logistic回归分析显性基因模型、隐性基因模型与胃癌临床特征的关系。结果显示,lncRNA H19 rs217727与胃癌分化程度、临床分期、淋巴结转移、远处转移均无关(P>0.05)。(表3)

表3 lncRNA H19 rs217727基因多态性与胃癌临床特征的关系
3 讨论
H19基因长约2.3 kb,位于11p15.5,包含5个外显子,3个内含子[12]。研究表明,lncRNA H19在肿瘤的发生、迁移、侵袭和转移中发挥重要的作用[13]。其作为一个lncRNA,缺乏开放阅读框翻译蛋白质,然而,其最终产物为RNA序列,并参与RNA的调节[14]。由于H19基因突变与癌症发生风险及预后的关系目前仍未明确,其基因多态性一直是近年来的研究热点[15]。迄今为止,已有许多研究显示H19单核苷酸多态性与肿瘤发生风险、预后密切相关,如胰腺癌、乳腺癌、肝癌等[16-18]。然而,lncRNA H19 rs217727 C>T基因多态性与胃癌的研究仍少有报道。
本研究通过对照研究旨在表明lncRNA H19 rs217727 C>T基因多态位点与广安地区人群胃癌遗传易感性的关系,采用PCR-RFLP技术检测其基因型,统计分析发现TT基因型能使人群罹患胃癌的风险增大,校正混杂因素后OR=2.99,95% CI=1.37~6.56,P=0.006;通过合并杂合子与突变型纯合子,组成显性基因模型,等位基因模型中,T等位基因与C等位基因相比可显著增加胃癌的发病风险,OR=1.65,95% CI=1.16~2.36,P=0.006。TT基因型、T等位基因增加人群罹患胃癌的风险,与Yang等[16]的研究结果一致。
SNP是最简单的DNA基因突变形式,它可以发生在群体内的个体之间,并且可能影响启动子活性(基因表达)、mRNA构象(稳定性)和翻译效率[19]。虽然lncRNA H19 rs217727 C>T基因多态性不影响H19 mRNA的表达水平,但突变可能改变翻译效率,可能导致H19结构的改变,最终影响H19的功能。研究表明,H19对胃癌的作用是通过直接上调ISM1介导的,ISM1是H19的结合蛋白,H19结构的改变可能影响H19与ISM1的结合[20]。然而,目前关于H19在肿瘤发生、发展中的具体机制仍尚不清楚。
本研究仍有局限性。首先,小样本量可能无法检测全面,人群基因受地域、环境、生活习惯等的影响可能表型不同,本研究样本来源单一。其次,纳入研究的对象无法避免选择偏倚。然而,由于缺少临床信息,如幽门螺杆菌感染的数据,使得没有对此因素进行分析。此外,由于吸烟、饮酒、个人生活习惯等数据是通过问卷收集的,仍可能存在偏倚。最后,本研究是基于广安地区汉族人群SNP与胃癌的关系研究,在数据应用上也谨慎民族、地域的差异。总之,本研究结果表明H19 SNP(rs217727 C>T)与广安地区汉族人群胃癌风险增加显著相关,未来仍需要多中心联合、大样本、分层研究更进一步进行验证。
