类乌齐牦牛C1QA、C1QB、C1QC基因克隆与组织表达分析
2020-08-29王吉坤柴志欣王嘉博武志娟信金伟张成福姬秋梅钟金城
钟 美,王吉坤,柴志欣,王 会,王嘉博,武志娟,信金伟,张成福,姬秋梅,钟金城
(1.西南民族大学 青藏高原研究院,青藏高原动物遗传资源保护与利用教育部重点实验室,四川省重点实验室,四川 成都 610041;2.省部共建青稞和牦牛种质资源与遗传改良国家重点实验室,西藏 拉萨 850000)
牦牛(Bosgrunniens) 耐粗耐劳,具有极强的生态环境适应性,在氧气稀薄、天气寒冷、牧草匮乏等恶劣环境条件下均可生存、繁衍后代,是极为宝贵的遗传资源基因库[1-3]。类乌齐牦牛主要生存于海拔3 700 m以上的高原,因其遗传多样性丰富,肉质鲜美,在2017年通过了国家畜禽资源委员会审定,评为优良的牦牛遗传资源[4]。
补体C1q(Complement 1)分子是由6个亚单位组成的异源六聚体,每个亚单位分别由C1QA、C1QB、C1QC基因编码的A、B、C 3条多肽链组成,即C1q 由18条多肽链构成[5]。链间靠3条链的N端半胱氨酸形成二硫键相连,构成同一个亚单位中的A-B 二聚体以及2个亚单位间的C-C二聚体,因此组成6个A-B二聚体和3个C-C二聚体。两A-B二聚体和一个C-C二聚体以非共价键形成含有2A、2B和2C的结构单位,3个这样的结构单位凭借非共价键构成完整的 C1q 分子[6]。补体系统是由存在于动物血清和组织液中的一组可溶性蛋白以及存在于其他细胞表面的一组膜结合蛋白和补体受体组成的,是一个复杂的蛋白质反应系统,在机体的抗感染免疫防御、维护内环境稳定、氧化应激、糖脂代谢中发挥重要作用。C1q是分子量为460 ku的糖蛋白,是C1的第一组成成分,对病原体的清除和细胞凋亡等重要生物过程至关重要[7],同时是启动补体系统经典途径的重要识别分子,在天然免疫和特异性免疫之间发挥连接作用[8]。
目前,C1QA、C1QB、C1QC基因在人[9]、牛[10]、猪[11]、鼠[12]等哺乳动物中均已得到广泛研究,多与疾病相关,在绵羊各个组织中也均有表达[13],该基因在牦牛中的研究鲜有报道。补体C1q蛋白与系统性红斑狼疮、免疫性肾病等免疫疾病[14]和缺氧条件下血管生成、促进缺血心肌新生血管形成等低氧适应有关[15-16]。如在Tohgi等[17]研究中发现小鼠PC12细胞在缺氧后立即表达C1q mRNA,然后在复氧过程中翻译出C1q蛋白的A、B、C链,导致缺氧细胞受损。Luo等[18]发现缺氧后C1q沉积是诱导阿尔茨海默病(AD)中神经元氧化应激和神经元死亡的重要因素,进一步证实了C1q是导致缺氧细胞受损伤的重要原因。Bossi等[19]研究表明,C1q与糖尿病患者慢性皮肤溃疡创面愈合及缺氧条件下脑损伤的血管生成有关。Fan等[20]研究结果显示,C1q可促进血管内皮生长因子VEGF的表达和分泌,通过LAIR-HIF1α-VEGF轴(重组人白细胞相关免疫球蛋白样受体1-缺氧诱导因子α-血管内皮生长因子)增强大脑动脉闭塞大鼠的血管新生以减轻脑水肿,由此可知,在缺氧状态下C1q确实能促进血管的生成。在维持机体的免疫耐受[21]方面,C1q 可识别许多自身结构发生改变的配体,如β淀粉样蛋白纤维[22]、凋亡细胞[23]、低密度脂蛋白修饰形式[24]和朊病毒蛋白病理形式[25],C1q 分子与这些配体结合可快速启动细胞的吞噬作用,从而清除凋亡细胞,抑制溶解效应和炎症。另一方面毋文静[26]研究发现CTRP6(一种脂肪分泌因子,属于补体C1q相关蛋白家族)通过Erk1/2和p38MAPK信号通路可调控猪肌内和皮下脂肪细胞的分化,以及皮下脂肪细胞的增殖。
缺氧组织中由于补体C1q沉积会导致细胞病变,研究表明神经元受β-淀粉样蛋白(Amyloid beta,Aβ)[27]、谷氨酰胺[28]、机械损伤[29]、超氧化物歧化酶(SOD)变异[30]以及单纯疱疹病毒感染[31]等因素刺激可产生氧化应激产物(ROS),而补体C1q蛋白可通过细胞表面受体促进巨噬细胞的吞噬作用并刺激中性粒细胞产生ROS, ROS过量会干扰细胞的正常功能[32]。赵杰等[33]研究显示肝脏器官中发生氧化应激时,含量过多的ROS会使细胞的抗氧化防御能力有所衰减。ROS可通过激活NF-κB、TGFβ、Nrf2等相关信号分子、细胞因子及其下游信号通路调控肝纤维化的发生和发展,如Nrf2作为调节氧化应激的重要转录因子,可调节肝脏中解毒和抗氧化防御相关基因的表达[34],在氧化应激的刺激下,Nrf2磷酸化后与Kelch样环氧氯丙烷相关蛋白-1(Kelchlike ECH-associated protein 1,Keap1)解离,进入到细胞核后与抗氧化反应元件(Antioxidant response elements,ARE) 序列结合,发挥抗氧化作用[35],所以C1q蛋白可以激活Nrf2,使得机体内源性抗氧化系统增强。
本研究以类乌齐牦牛为研究对象,通过克隆其C1QA、C1QB、C1QC基因的CDS区,并对CDS区进行生物信息学分析,预测其信号肽、亲水性/疏水性、跨膜区、理化性质、蛋白质二级及三级结构,实时荧光定量分析其在不同组织中的表达情况,为更进一步探讨C1QA、C1QB、C1QC基因在牦牛低氧适应性中的作用提供参考。
1 材料和方法
1.1 试验材料
1.1.1 采集样本 本试验样本取自西藏昌都市类乌齐县,挑选身体健康的6月龄牦牛3头,采集脾脏、心脏、肾脏、肝脏、肺脏组织,组织使用DEPC水预处理,放于液氮中保存备用。
1.1.2 主要试剂 pMDTM19-T Vector cloning Kit、YB GreenTMPremix ExTaqTMⅡ,购自TaKaRa公司;DNA 纯化回收试剂盒、DL-2000 Marker、大肠杆菌 DH5α 感受态细胞,购自天根生化科技(北京)有限公司。
1.2 试验方法
1.2.1 引物设计与合成 参照GenBank中牦牛C1QA(XM_005905182.20)、C1QB(XM_005905180.2)、C1QC(XM_005905183)基因mRNA 的预测序列,用Primer Premier 5.0软件设计引物。引物(表1)由擎科生物工程(上海)股份有限公司合成。

表1 引物序列信息Tab.1 Primer sequence information
1.2.2 样品RNA提取及检测 将各组织样本,利用液氮于研钵中研磨至粉末状后移入1.5 mL无RNase离心管中,TRIzol(Invitrogen)试剂1 mL溶解,根据TRIzol法提取总RNA。使用NanoDropTMOne 2000分光光度计检测RNA样品的纯度和浓度,用1.0%琼脂糖凝胶电泳检测RNA的完整性。
1.2.3 反转录 根据反转录试剂盒PrimeScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time) (TaKaRa)使用说明,将提取总RNA反转录成cDNA,于-20 ℃保存备用。
1.2.4C1QA、C1QB、C1QC基因PCR扩增 反应体系(25 μL):cDNA模板1 μL,上、下游引物(10 μmol/L)各1 μL,ddH2O 9.5 μL,TaqGreen PCR Master Mix(2×)12. 5 μL。反应程序参见文献[36]。PCR产物用1.0%凝胶电泳检测后根据DNA纯化试剂盒使用说明纯化回收DNA。
1.2.5 克隆及测序 pMD19-T载体与纯化后的PCR产物连接,将连接产物转化至DH5α感受态细胞中,涂布于含氨苄的LB 固体培养基表面,37 ℃恒温培养箱中倒置培养10~14 h;选取白色菌落置于液体培养基中,37 ℃/(180 r/min)摇床中培养;用菌液PCR鉴定阳性克隆,送擎科生物工程(上海)股份有限公司测序。
1.2.6 生物信息学分析 利用在线软件(表2)对C1QA、C1QB、C1QC蛋白进行生物信息学分析。
1.3 实时荧光定量PCR(Real-time PCR)
利用YB GreenTMPremix ExTaqTMⅡ试剂(TaKaRa),实时定量PCR检测C1QA、C1QB、C1QC基因在心脏、肝脏、脾脏、肺脏和肾脏等组织的相对表达量。
反应体系 (10 μL):YB GreenTMPremix 5 μL,上下游引物(10 μmol/L)各0.4 μL,cDNA模板0.8 μL,无RNAase水3. 4 μL。反应程序参见文献[36]。利用熔解曲线来鉴定引物特异性,每个样品设置3个生物学重复,3个技术重复。以牦牛β-actin为内参基因,记录每个样品的Ct值,利用 2-ΔΔCt法得到各个样本的相对表达量。

表2 在线分析软件Tab.2 Online analysis software
利用GraphPad Prism 5绘制C1QA、C1QB、C1QC基因在类乌齐牦牛心脏、肝脏、脾脏、肺脏、肾脏组织中的相对表达柱状图,根据SPSS 22.0软件分析得到各组织间的差异显著性,其中P<0.05表示差异显著,P<0.01表示差异极显著。
2 结果与分析
2.1 牦牛C1QA、C1QB、C1QC基因克隆
牦牛C1QA、C1QB、C1QC基因PCR扩增结果见图1,分别获得835,945,850 bp的扩增产物,与预期结果(表1)相符。

M.DL2000 DNA Marker;1-3.C1QA、C1QC、C1QB基因。M.DL2000 DNA Marker;1-3.C1QA,C1QC,C1QB genes,respectively.
2.2 同源性分析及构建系统进化树
利用DNAMAN软件将克隆获得牦牛C1QA、C1QB、C1QC基因CDS区与普通牛、水牛、野猪、人、卡氏小鼠和高原地山雀基因的CDS区进行比较,结果显示,牦牛C1QA基因与普通牛(XM_001014945.2)、水牛(XM_006063784.2)、野猪(XM_001003924.1)、人(XM_001347465.2)、卡氏小鼠(XM_021160797.2)、高原地山雀(XM_065528281.2)的序列一致性分别为99.0%,95.9%,30.7%,67.3%,60.4%和28.3%;牦牛C1QB基因与普通牛(XM_001046599.2)、水牛(XM_006063788.2)、野猪(XM_001244283.1)、人(XM_001371184.1)、卡氏小鼠(XM_021160680.2)、高原地山雀(XM_005528279.2)的一致性分别为99.3%,97.2%,28.3%,34.7%,29.3%和34.2%;牦牛C1QC基因与普通牛(XM_001206396.1)、水牛(XM_025278760.1)、野猪(XM_001244286.1)、人(XM_001114101.3)、卡氏小鼠(XM_021160901.2)、高原地山雀(XM_014257216.1)的一致性分别为82.7%,84.1%,81.3%,100.0%,77.9%和63.9%。
利用ClustalX和MAGA 5.2构建类乌齐牦牛(Lwq yak)与水牛(Bubalusbubalis)、普通牛(Bostaurus)、人(Homosapiens)、黑猩猩(Pantroglodytes)、卡氏小鼠(Muscaroli)、野猪(Susscrofa)、藏羚羊(Pantholopshodgsonii)、非洲爪蟾(Xenopuslaevis)、青藏高原蛙(Nanoranaparkeri)、斑马鱼(Daniorerio)和高原地山雀(Tibetan ground-tit)C1QA、C1QB、C1QC基因核苷酸序列的系统进化树。结果(图2-4)表明,类乌齐牦牛C1QA、C1QB基因最先与普通牛、水牛、藏羚羊聚为一类,其次是卡氏小鼠、人、黑猩猩等其他动物,而类乌齐牦牛C1QC基因却最先与人、长尾猕猴、野猪聚为一类,其次是水牛、普通牛等其他动物,说明类乌齐牦牛C1QA、C1QB基因与普通牛亲缘关系最近,类乌齐牦牛C1QC基因则与人亲缘关系最近,类乌齐牦牛C1QA基因与野猪、高原地山雀亲缘关系最远,类乌齐牦牛C1QB、C1QC基因与斑马鱼亲缘关系最远。
2.3 序列分析结果
使用NCBI ORF Finder对克隆所得牦牛C1QA、C1QB、C1QC基因序列进行开放阅读框分析,可知C1QA基因开放阅读框长735 bp,共编码244个氨基酸,起始密码子为ATG,终止密码子为TGA;C1QB基因开放阅读框长744 bp,共编码247个氨基酸,起始密码子为ATG,终止密码子为TGA;C1QC基因开放阅读框长732 bp,编码243个氨基酸。

图2 11个不同物种基于C1QA基因的系统进化树Fig.2 Phylogenetic tree based on C1QA gene sequences of 11 species

图3 12个不同物种基于C1QB基因的系统进化树Fig.3 Phylogenetic tree based on C1QB gene sequences of 12 species

图4 10个不同物种基于C1QC基因的系统进化树Fig.4 Phylogenetic tree based on C1QC gene sequences of 10 species
2.4 牦牛C1QA、C1QB、C1QC蛋白结构和功能预测
2.4.1 理化性质 根据在线软件ExPASy Prot-Param 程序预测C1QA、C1QB、C1QC蛋白的理化性质。预测结果(表3)显示,C1QA、C1QB和C1QC蛋白所含氨基酸残基以甘氨酸(Gly)和脯氨酸(Pro)最多;不稳定系数分别为34.11,18.79和28.77,均属于稳定蛋白;脂肪系数分别为66.68,72.59和67.74,总平均亲水系数分别为-0.386,-0.353和-0.298,依据氨基酸分值越低亲水性越强或分值越高疏水性越强的规律分析可知, C1QA、C1QB和C1QC蛋白整体表现出较强的亲水性,属亲水性蛋白。
2.4.2 蛋白结构预测分析 利用软件SignalP 5.0、TMHMM、Interpro和NetPhos 2.0在线工具分别预测C1QA、C1QB和C1QC蛋白质的信号肽、跨膜结构、蛋白结构域和磷酸化位点。结果显示,C1QA、C1QB和C1QC蛋白均含有信号肽和C1Q结构域,不含跨膜结构域;蛋白氨基酸序列中分别存在18,21,15 个潜在的磷酸化位点,其中C1QA编码的氨基酸含有12个Ser磷酸化位点、4个Thr磷酸化位点、2个Tyr 磷酸化位点;C1QB包含10个Ser磷酸化位点、8个Thr磷酸化位点、3个Tyr磷酸化位点;C1QC检测到8个Ser磷酸化位点、5个Thr磷酸化位点、2个Tyr磷酸化位点。

表3 C1QA、C1QB、C1QC蛋白序列分析Tab.3 Analysis of C1QA,C1QB and C1QC genes sequences
利用ExPaSy中SOPMA、SWISS-MODEL和PSORTⅡPrediction工具预测C1QA、C1QB和C1QC蛋白的二、三级结构和亚细胞定位,结果显示C1QA、C1QB和C1QC蛋白二级结构主要以无规则卷曲为主,比例分别为61.85%,63.97%,66.67%,其中在蛋白结构相似性比对中C1QA与5hkj.1结构具有75.71% 的相似性,C1QB、C1QC与5hkj.1结构相似性分别可达73.33% 和74.81%;三级结构由无规则卷曲、α-螺旋、延伸链和β-转角构成(图5-7);蛋白质的亚细胞定位预测显示,C1QA、C1QB、C1QC位于细胞外(包括细胞壁)的比例分别为55.6%,66.7%,66.7%,C1QA与C1QB位于内质网、高尔基体各占11.1%,C1QB、C1QC位于高尔基体所分泌的囊泡中分别占11.1%和22.2%,C1QA位于细胞骨架、细胞核内各占11.1%,C1QC还有11.1%位于线粒体。

图5 牦牛C1QA蛋白的三级结构预测Fig.5 The tertiary structure prediction of C1QA protein in Bos grunniens

图6 牦牛C1QB蛋白的三级结构预测Fig.6 The tertiary structure prediction of C1QB protein in Bos grunniens

图7 牦牛C1QC蛋白的三级结构预测Fig.7 The tertiary structure prediction of C1QC protein in Bos grunniens
2.4.3 功能预测 通过蛋白功能预测分析,C1QA、C1QB和C1QC蛋白的主要功能包括细胞表面受体信号通路的调控、基因表达调节、代谢过程的调节等(表4)。

表4 C1QA、C1QB、C1QC蛋白功能分析Tab.4 The functional analysis of C1QA, C1QB and C1QC protein in Bos grunniens %
2.4.4 蛋白网络互作分析 C1QA、C1QB和C1QC蛋白是组成C1Q蛋白的3条链,均含有C1Q结构域,相关性较高,以C1QC蛋白为例,C1QA和C1QC的相关性为0.976,C1QB与C1QC间的相关性为0.996,补体系统第一成分的S亚成分(C1S)和C1QC的相关性可达0.910,SERPING1是丝氨酸蛋白酶抑制剂家族的G1成员,与C1QC的相关性为0.864,蛋白酪氨酸激酶结合蛋白(TYROBP)与C1QC间的相关性可达0.815,组织蛋白酶S(CTSS)为木瓜蛋白酶超家族成员,在抗原递呈中起重要作用,并可水解多种蛋白,参与多种疾病的病理过程,与C1QC的相关性达到了0.805(图8)。

图8 C1QA、C1QB、C1QC蛋白网络互作分析图Fig.8 C1QA,C1QB,C1QC protein network interaction analysis
2.5 C1QA、C1QB、C1QC基因在类乌齐牦牛各组织中的差异表达分析
实时荧光定量PCR对类乌齐牦牛心脏、肝脏、脾脏、肺脏和肾脏组织中C1QA、C1QB、C1QC基因mRNA表达水平进行检测,用2-ΔΔCt法计算C1QA、C1QB、C1QC基因表达量,结果(图9-11)表明:C1QA、C1QB、C1QC基因在所检测组织中均有表达,在肺脏中C1QB基因的表达量最高,C1QA次之,C1QC基因的表达量最低,其中类乌齐牦牛C1QA、C1QB基因在脾脏、肺脏中的表达量极显著高于心脏、肝脏和肾脏中的表达量(P<0.01);C1QC基因在肺脏中的表达量极显著高于心脏、肝脏、脾脏和肾脏中的表达量(P<0.01),在心脏中的表达量极显著低于脾脏和肝脏(P<0.01),显著高于肾脏(P<0.05);C1QA、C1QB基因在心脏、肝脏和肾脏中的表达量差异不显著(P>0.05)。
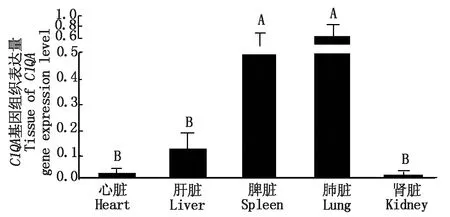
不同大写字母表示差异极显著(P<0.01);不同小写字母表示差异显著(P<0.05);相同字母表示差异不显著。图10-11同。
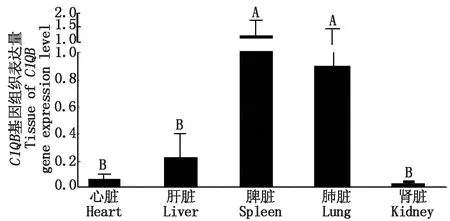
图10 牦牛C1QB基因在不同组织中的相对表达量Fig.10 Tissue expression of C1QB gene in different tissues in Bos grunniens

图11 牦牛C1QC基因在不同组织中的相对表达量Fig.11 Tissue expression of C1QC gene in different tissues in Bos grunniens
3 讨论与结论
全世界约有牦牛1 600万头,中国是世界上拥有牦牛数量和品种类群最多的国家,约占世界牦牛总数的95%以上,占中国牛只总数的1/6。牦牛作为一种“全能”家畜,对人类有着不可忽视的社会及经济意义。本研究通过对类乌齐牦牛C1QA、C1QB、C1QC基因的克隆、生物信息学分析与组织表达分析,为今后补体C1q蛋白在牦牛低氧适应研究提供依据,同时也为开发和利用类乌齐牦牛遗传资源奠定基础。将类乌齐牦牛与水牛、普通牛、人、黑猩猩、卡氏小鼠、野猪、藏羚羊、非洲爪蟾、青藏高原蛙、斑马鱼和高原地山雀的核苷酸序列进行比对,发现牦牛C1QA、C1QB、C1QC基因在反刍动物间具有高度保守性。
本研究显示牦牛C1QA、C1QB和C1QC蛋白甘氨酸含量为15.2%,13.4%,16.5%。甘氨酸含量越高,机体抗氧化应激的能力越强;另一方面,由于缺氧会导致体内C1q的沉积,C1q增多促使体内ROS增加,而甘氨酸作为内源性抗氧化剂还原性谷胱甘肽的组成氨基酸,对活性氧(ROS) 有很强的清除作用,因此,牦牛体内甘氨酸的含量影响其对高海拔低氧环境的适应。脯氨酸亲水性极强,可降低凝固点防止细胞脱水从而能稳定生物大分子结构及组织内的代谢过程,同时作为能量库调节细胞的氧化还原;在低温条件下,植物组织中脯氨酸增加,可提高植物的抗寒性,因此,脯氨酸可作为抗寒育种的生理指标[37]。本研究表明,牦牛C1QA、C1QB和C1QC蛋白脯氨酸含量为9.8%,9.3%,10.7%,表明类乌齐牦牛体内脯氨酸的含量可能是导致其适应高海拔低温环境的重要因素。
信号肽是指导新合成的蛋白质的跨膜转移(定位)的短肽链,可与膜上蛋白受体结合,指引蛋白质被运输到特定位置,而结构域是生物大分子中具有特异结构和独立功能的区域。本试验预测到牦牛C1QA、C1QB和C1QC蛋白含有C1Q结构域与一条信号肽,说明补体C1q蛋白的功能较保守,牦牛在补体系统经典途径的免疫调节中与其他物种是一致的。胞内的信号蛋白主要分为两大类:一类在蛋白激酶的作用下磷酸化,共价结合ATP所提供的磷酸基团;另一类则在信号作用下结合GTP,使得GTP取代GDP,这2种胞内信号蛋白会在信号达到时获得磷酸基团而被激活,在信号减弱时去除这些基团,从而失活。在信号中继网中,这种某个信号蛋白磷酸化从而造成下游蛋白依次发生磷酸化的过程称为磷酸化级联反应。而牦牛C1QA、C1QB和C1QC蛋白磷酸化位点为18,21,15个且均有信号肽,说明牦牛C1QA、C1QB和C1QC蛋白是信号蛋白,在细胞内和细胞间进行信号传递,协调和控制相关代谢和生命活动。近年来磷酸化在低温胁迫上的研究数不胜数,Zhao等[38]利用蛋白磷酸化技术探究了低温胁迫下植物生长发育的相关机制;杨梅燕[39]研究表明在冻融胁迫下,PM18蛋白可有效降低乳酸脱氢酶的活性丧失比率,且磷酸化后的PM18蛋白对乳酸脱氢酶的保护能力比非磷酸化的PM18蛋白更好,牦牛作为生存在高原低温、低氧等恶劣环境下的优势物种,探讨牦牛低温胁迫的调控机制有利于牦牛遗传资源的开发与利用。
蛋白功能预测分析显示,牦牛C1QA、C1QB和C1QC蛋白的功能包括细胞表面受体信号通路的调控、基因表达调节、代谢过程的调节、细胞信号转导、细胞定位等,这些功能预测与本研究对C1QA、C1QB和C1QC蛋白的生物信息学分析所得结果相对应。牦牛由于对高原环境的适应,在机体代谢上势必与其他物种存在差异,牦牛C1QA、C1QB和C1QC蛋白对于代谢过程的调节几率达到了84.7%,84.0%和81.7%,但关于其对牦牛代谢过程的调控机制需进一步研究。蛋白质互作分析中,由于C1QA、C1QB和C1QC蛋白是补体C1q的构成蛋白,三者相关性达到了0.90以上,C1QC与补体第一成分的S亚成分(C1S)的相关性亦达到了0.901,是因为C1q的胶原样区与两分子的补体第一成分的r亚成分(C1R )和 C1S 结合而形成C1大分子(C1QC1R2C1S2),再通过C1q球状结构域识别与Ig G或IgM免疫复合体结合,激活C1R和C1S,进而启动补体经典途径。蛋白酪氨酸激酶是一类催化ATP上γ-磷酸转移到蛋白酪氨酸残基上的激酶,能催化多种底物蛋白质酪氨酸残基磷酸化,在细胞生长、增殖、分化中具有重要作用;蛋白质互作显示蛋白酪氨酸激酶结合蛋白(TYROBP)与C1QC间相关性达0.815,蛋白质磷酸化位点预测发现牦牛C1QA、C1QB和C1QC蛋白酪氨酸磷酸化位点为2,3,2个,表明牦牛C1QA、C1QB和C1QC蛋白有可能是通过酪氨酸磷酸化参与调控牦牛细胞生长、增殖与分化。
实时荧光定量PCR结果显示,C1QA、C1QB基因在肺脏和脾脏中表达量最高,心脏、肝脏与肾脏三者间表达量差异不显著(P>0.05),与Jiang等[13]发现绵羊脾脏中表达量最高,其次是肺脏的研究结果基本一致;与Yue等[40]发现小鼠脾脏中表达量最高,在肺脏与肝脏、心脏和肾脏中的表达量差异不显著的研究结果存在差异,说明C1QA、C1QB基因的组织表达具有物种特异性,反刍动物中C1QA、C1QB基因在肺脏中的表达量要高于肝脏、心脏和肾脏;而脾是重要的淋巴器官,可以制造免疫球蛋白、补体等免疫物质,发挥免疫作用,也是人体的“血库”,当人体休息、安静时贮存血液,当处于运动、失血、缺氧等应激状态时又将血液排送到血循环中,以增加血容量,所以C1QA、C1QB基因在不同物种的脾脏中表达量都比较高;在牦牛肺脏组织中表达量与其他物种存在差异的原因可能是C1QA、C1QB基因参与调控牦牛肺部低氧适应机制。C1QC基因在肺脏中表达量极显著高于脾脏和肝脏,在心脏与肾脏中表达量较低,与Jiang等[13]发现绵羊脾脏中表达量最高,其次是肺脏,及Yue等[40]发现小鼠脾脏中表达量最高,在肺脏与肝脏、心脏和肾脏中的表达量差异不显著的研究结果存在差异,说明C1QC基因的组织表达也具有物种特异性,但C1QC基因在脾脏与肾脏中的表达量与在C1QA、C1QB基因中的相比,肺脏显著高于脾脏,可能是因为C1QC基因在调控牦牛肺部低氧适应机制中承担着不同的作用。
本研究首次从类乌齐牦牛肺脏组织中克隆出牦牛C1QA、C1QB、C1QC基因,其CDS区全长分别为735,744,732 bp,分别编码244,247,243个氨基酸;系统进化树结果表明,C1QA、C1QB基因与普通牛、水牛亲缘关系最近,C1QC基因则与人的亲缘关系最近;荧光定量结果表明,C1QA、C1QB基因在脾脏、肺脏中的表达量极显著高于心脏、肝脏和肾脏中的表达量(P<0.01),C1QC基因则是肺脏中的表达量极显著高于心脏、肝脏、脾脏和肾脏中的表达量(P<0.01)。试验结果为深入研究C1QA、C1QB、C1QC基因在牦牛高原适应中的分子机制提供了基础数据。
