EBPR聚磷菌种群结构及其分支研究进展
2020-08-23谢朝新王涛龙向宇唐然陶光建王嘉粤
谢朝新 王涛 龙向宇 唐然 陶光建 王嘉粤

摘 要:强化生物除磷是经济有效去除污水中磷(P)的重要方法,已广泛应用于水环境保护领域。EBPR系统成功运行的关键在于聚磷菌(PAOs)通过聚磷代谢(PAM)富集聚磷酸盐(poly-P),获得碳源竞争优势,成为优势菌种。其中Accumulibacter菌是EBPR系统实现生物除磷的重要菌群,且主要存在Type I型和Type II型两种类型的进化枝。总结了EBPR系统中常见的PAOs,并对Accumulibacter为主的进化枝进行了鉴别。从碳源、温度和电子受体3个方面探讨了Accumulibacter进化枝的代谢特性。总结了现有认知的不足和对未来研究方向进行了展望。
关 键 词:强化生物除磷;聚磷菌;进化枝;代谢特性
中图分类号:X524 文献标识码: A 文章编号: 1671-0460(2020)07-1513-06
Research Progress in Community Structure and Branching of
Polyphosphate Accumulating Organisms in EBPR System
XIEChao-xin1, WANGTao1, LONG Xiang-yu1*, TANG Ran2, TAO Guang-jian1, WANG Jia-yue1
(1. Army Logistic University of PLA, Chongqing 401331, China;
2. Chongqing University of Science & Technology, Chongqing 401331, China)
Abstract: Enhanced biological phosphorus removal is an important method to remove phosphorus (P) from wastewater economically and effectively. The key to the successful operation of EBPR system is that polyphosphate accumulating organisms (PAOs)can accumulate polyphosphate ( poly-P) through polyphosphate metabolism to obtain carbon source competitive advantage and become the dominant bacterial. Accumulibacter bacteria is an important bacteria group in EBPR system to realize biological phosphorus removal, and there are mainly type I and type II evolutionary strains. In this paper, the common PAOs in EBPR system were summarized, and the evolvement branch of Accumulibacter was differentiated. Then the metabolic characteristics of Accumulibacter evolvement branch were discussed from three aspects of carbon source, temperature and electron acceptor. Finally, the existing cognitive deficiencies were summarized, and the future research direction was prospected.
Key words: Enhanced biological phosphorus removal; PAOs; Clade; Metabolic characteristics
目前,我國湖泊(水库)富营养化形势依然严峻,磷(P)元素是导致湖泊(水库)富营养化最为关键的限制性因素,为此国家和地方政府对污水处理厂出水磷浓度制定了严格的排放标准。强化生物除磷(EBPR)是一种处理成本低和环境友好的技术,已广泛用于实际污水处理厂。普遍认为,EBPR过程是基于聚磷菌(PAOs)厌氧释磷、好氧过量吸磷的原理,通过排放剩余富磷污泥实现良好的除磷效果。厌氧条件下,PAOs分解胞内poly-P和糖原,吸收挥发性脂肪酸(VFAs,如乙酸和丙酸)并合成PHAs储存;缺氧/好氧条件下分解胞内PHAs提供能量用于糖原恢复、poly-P的合成以及维持微生物生长代谢。
EBPR系统微生物种群具有多样性和复杂性,采用分子生物学技术(如,原位荧光杂交(FISH)、实时荧光定量PCR(qPCR)、16SrRNA和宏基因组测序等)能够有效的促进PAOs的识别[1-2]。目前,PAOs被广泛定义为利用poly-P提供厌氧阶段摄取碳源能量的所有微生物[3],其中β变形菌中与红环菌属相关的β-2亚组(Candidatus_Accumulibacter_phosphatis)是广泛存在于EBPR系统的典型PAOs[2-3]。基于Accumulibacter的ppk1基因的差异,Accumulibacter又可以分为两种类型(Type I型和Type II型),并且Type I型又可以进一步分为进化枝IA-E共5个分支,Type II型又可以进一步分为进化枝IIA-G共7个分支。此外,大量研究表明不同的进化枝的PAOs常常表现出不同的生理特性。因此,有必要进一步了解Accumulibacter进化枝的代谢特性,提高EBPR系统运行的稳定性和可靠性[4]。
1 EBPR系统内的聚磷菌
Fuhs和Chen[5]在1975年利用纯培养技术最早将Acinetobacter鉴定为EBPR中主要的除磷微生物菌种,并且Acinetobacter长期都被认为是唯一的PAO。然而,Wagner 等[6]在全规模污水处理厂中采用专一FISH探针研究发现,与β-变形菌和Actinobacteria等其它系统发育菌群相比,Acinetobacter的除磷作用相对较低。
尽管PAOs分离培养困难,近十年来,分子技术在PAOs菌群鉴定上的应用取得了一定的进展。Bond 等[7-8]采用了基因库系统发育分析技术(16S rRNA)和荧光原位杂交技术(FISH)研究了EBPR污泥和非EBPR污泥中菌群结构的差异,他们发现β-变形菌亚纲2下的Rhodocyclus属是除磷菌群中的主要微生物。与Rhodocyclus紧密相关的β-变形菌亚纲2被命名为“CandidatusAccumulibacter phosphatis”,并简称为“Accumulibacter”[9]。研究人员报道了几种FISH探针根据16SrRNA上不同区域来鉴定Accumulibacter[9-10]。本文对EBPR系统中PAOs的常用探针总结如表1所示。
研究者通过FISH发现Accumulibacter均大量富集于实验室规模的EBPR系统中,其丰度高达55%~95%[10-11]。然而,在实际污水处理厂中,由于进水水质差异较大,导致其微生物种群结构也存在明显差异。Saunders等[15]对澳大利亚污水处理厂的研究发现,Accumulibacter在污水处理厂污泥微生物中同样具有相当高的丰度(占细菌总数的4%~22%),且EBPR系统除磷性能与污泥中Accumulibacter比例呈正相关。然而,部分研究者研究表明,Accumulibacter菌群中并非所有与FISH探针结合的细胞都能观察到贮存有poly-P[16-17]。Stokholm-Bjerregaard等[18]对丹麦的18个全规模污水处理厂微生物种群结构进行评价,发现Tetrasphaera也是一种常见的PAOs,且具有较高丰度(8.9%)。同时与Accumulibacter菌相比,Tetrasphaera有更多的生理学功能,包括厌氧发酵和累积发酵副产物和氨基酸等。此外,虽然Actinobacteria菌生理性状与大多数PAOs生化模型描述的性状存在较大差异,但Actinobacteria菌也能够在好氧阶段超量吸磷并实现良好的除磷性能,是一种潜在的PAOs。为此,Wong等[19]用特定菌群探针研究测定污泥菌群种类,结果表明:β-变形菌是所研究的特定菌群中的主导PAOs菌群,而Actinobacteria、α-变形菌和γ-变形菌丰度较低。
2 Accumulibacter进化枝的鉴别及分布特征
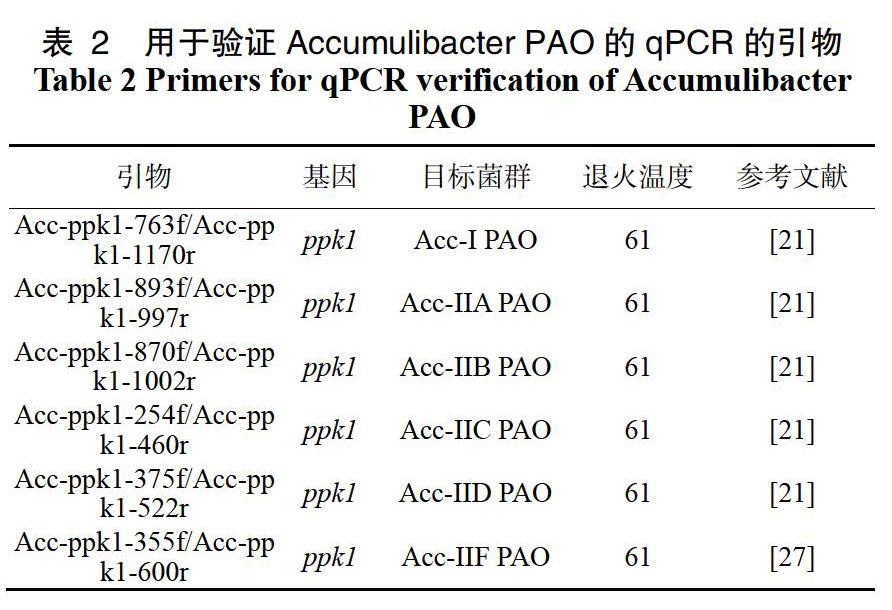
如前所述经大量研究报道,Accumulibacter是EBPR中的优势PAOs。McMahon等[20]在2002年首次利用PCR分析实验室规模SBR反应器中微生物的ppk基因。经ppk1-PCR扩增实验发现Accumulibacter包含两种类型(Type I型和Type II型)[21],Type I型被细分为进化枝IA~ IE。Type II型被细分为进化枝IIA~ II-G。后来He等[21]利用实时定量聚合酶链反应(qPCR)来鉴别Accumuli-bacter进化枝,研究者利用系列引物并采用宏基因分析对污水处理厂污泥种群结构进行测定,所用引物如表2所示。
然而后来的大量研究表明,在不同的污水处理厂或者实验室规模的反应器条件下,不同外界条件的EBPR系统中占主导地位的Accumulibacter分支种类和数量也不同。Slater等[22]发现良好的EBPR性能与进化枝IA有關,而进化枝IIC导致EBPR恶化,同时指出,进化枝IIF属于进化枝IIC。采用定量ppk1-PCR技术对丹麦28家污水处理厂中的Accumulibacter种群结构进行分析,结果发现几乎所有污水处理厂都存在Accumulibacter Type IA和Type IIC,而仅在少数污水处理厂存在Type IIA 和 Type IID[23]。由于操作运行条件和废水水质成分的复杂性,全规模污水处理厂中Accumulibacter进化枝种群结构与实验室规模EBPR系统相比明显不同。有研究发现,进化枝IA、IC、IIA和IID是实验室规模反应器的优势Accumulibacter。Kim 等[24]发现,与Type I型相比,实验室规模反应器中进化枝IIC只占很小的百分比,与之相反,进化枝IIC在污水处理厂广泛存在,甚至是优势微生物菌种。Zeng
等[25]采用ppk1基因作为系统发育标记研究了Accumulibacter的群落结构、丰度与北京四个代表性污水处理厂(WWTPs)操作运行的相关性,研究发现EBPR过程中存在有Accumulibacter的5个进化枝(IA、IIA、IIB、IIC和IID)。进化枝IIC占有最大比例,进化枝IIC和IIF存在紧密的遗传关系。上述结论与Slater等[22]和 Kim 等 [24]的结论一致。通过上述研究发现我们可以得出结论,实验室规模EBPR和实际污水处理厂的Accumulibacter在进化枝水平上的种群结构明显不同并且污水处理厂的Accumulibacter主要与Type II型有关,然而,进化枝多样性与运行条件的相关性尚不清楚。
近年来,研究者开展了Accumulibacter进化枝多样性与EBPR系统操作运行条件的相关性的研究。此外,挥发性脂肪酸(VFA)、COD/P比、溶解氧(DO)浓度与地理区域等因素似乎也会影响Accumulibacter的丰度与种群结构[26]。迄今为止,Accumulibacter表现出的种群结构变化主要控制因素在很大程度上尚不清楚。上述内容需要进一步研究。
3 Accumulibacter各进化枝的代谢特性
3.1 对碳源的选择性
在实际的EBPR过程中,废水中存在不同的碳源类型会影响聚磷菌的种群结构,从而影响EBPR的性能。在绝大多数实验室规模的EBPR采用挥发性脂肪酸-VFAs(乙酸或丙酸)作为碳源,因为在实际污水中VFAs是占较大比例,并且VFAs对EBPR具有促进作用。Qiu等[27]对新加坡三个具有EBPR性能的全规模污水处理厂的活性污泥进行取样然后采用5类29种有机化合物作为碳源进行培养比较(4种糖、3种醇、3种挥发性脂肪酸(VFAs)、8种氨基酸和6种其他羧酸),结果发现挥发性脂肪酸(VFAs)能够诱导最大的厌氧释磷量(乙酸为12.0~18.2 mgP·g-1MLSS、丙酸为9.4~18.5 mg P·g-1 MLSS),这也证明了选择VFAs作为碳源能够对EBPR产生促进作用。
Martin等[28]对EBPR系统中微生物的酶及代谢途径进行了详细的研究 , 结果发现不同的碳源可能导致 PAOs在亚种上存在差异, 而且代谢途径可能也会受到影响。Nan Shen等[29]在30 ℃条件下分别用乙酸和丙酸作为碳源运行两个SBR,结果发现在两个反应器中TypeⅡC是优势进化枝。A.B.Lanham等[30]以丙酸为碳源下运行SBR,可富集到大量Type Ⅰ型Accumulibacter,几乎没有Type Ⅱ型。Brenda[31]等采用乙酸作为碳源并调整进水P/C比例,结果发现长期运行的PAO培养物中PAO Type I型成为了优势进化枝。而当L.Welles等[32]分别运行3个SBR,第一个SBR用乙酸和丙酸作混合碳源,结果发现均包含Type I和Type II型Accumulibacter。另外两个SBR反应器以乙酸为碳源并调整进水P/C,发现Type I型和 Type Ⅱ型Accumulibacter分别在两个反应器中占主导地位。Saad 等[33]以乙酸∶丙酸为3∶1 作为混合碳源的条件下,成功地将Type IC富集至 99%,而没有观察到 Type II型或聚糖菌(GAO)。并且Camejo等[34]在厌氧-微好氧模式下以乙酸为碳源,也成功地富集到Type IC(占Accumulibacter的99%)。然而,Carvalhod等[35]发现,将A/O(厌氧/好氧)变为A/A(厌氧/缺氧)系统,并用丙酸代替乙酸作为碳源,更有利于缺氧吸磷过程,研究者认为这是由于以丙酸为碳源Type I型更倾向于较Type Ⅱ型取得竞争优势。
以上结果说明,以丙酸为碳源时Type I型似乎更倾向于成为EBPR系统中的优势菌种,而以乙酸为碳源时Type I型和Type Ⅱ型都有可能成为系统的优势菌种。然而,对于上述研究者的研究结果又启示我们:虽然不同的进化枝对碳源的选择性不同,而一些其他因素(比如P/C比、碳源比例、工艺流程、温度等)对Accumulibacter群落结构也可能起着决定性的作用。这或许能够解释为什么不同的研究者在同样的碳源条件下检测出的优势Accumulibacter分支不同。所以研究者们还需进一步深入研究完善其他因素对于Accumulibacter进化枝对碳源的选择性的影响作用。而Carvalhod的研究结果又给优化Accumu-libacter种群结构和提高EBPR性能和提供了新的研究思路。
3.2 溫度的影响
大量的研究报道,温度是影响EBPR除磷效果的关键因素,且就目前已有的研究结果来看,由于低温能够抑制GAOs的生长,研究者们对低温(5~20 ℃)有利于PAOs的生长并能够提高EBPR的除磷效果已经达成了共识[36-38]。Tian等[37]采用10℃和16天SRT的条件研究发现,Type I型Accumulibacter是占主体PAOs,而GAOs受到了抑制。相关研究也印证了低温有利于PAOs,当温度高于30 ℃,除P效率大幅度降低[25,39-40]。在低温下运行的EBPR系统(季节性)稳定性和P去除效率是非常有价值的(例如在冬季或北方国家,其平均年温度往往低于15 ℃)。然而,由于该实践可能涉及的相当高的能量成本,人工降低当前实际或全规模应用中的温度似乎不可行。所以对于夏季或热带国家温度在25 ℃甚至更高的条件下,EBPR仍然是这些地区的污水处理厂除磷采取的主要工艺,所以研究EBPR在高温下的稳定运行是非常有必要性的。
Flowers等[41]研究发现进化枝IIA的丰度与温度变化呈正相关性,而进化枝IA与温度变化呈负相关性。Ong等[26]采用两组SBR反应器先后在24、28和32 ℃的温度下运行,并采用FISH探针和qPCR分别描述PAO种群数量和PAOs种群动态,结果发现,IIF是高温条件下EBPR系统中的主要进化枝。其中当反应器稳定运行后Accumulibacter丰度在24 ℃时为51%,在28 ℃时为33%,在32 ℃时为12%,虽然温度升高与PAO的增殖呈负相关,然而他们发现三种温度下的除磷效果均为95%~99%,这说明仅从PAO的丰度来揭示EBPR的稳定性可能并不全面,需要更深入的探讨温度对EBPR稳定性的影响机制。然而,Nan Shen等[29]指出在30 ℃的条件下分别用乙酸和丙酸运行两个反应器并利用qPCR检测Accumulibacter支系的丰度,结果发现在两个反应器中IIC进化枝均为优势进化枝,这与Zeng[25]对实际的污水处理厂取样结果一致。然而,这个结果与Ong等[26]的研究结果相冲突,说明在高温条件下的EBPR中的Accumulibacter优势进化枝还需进一步研究确定。值得注意的是,在Nan Shen等[30]的研究中观察到呼吸硝酸盐的减少,硝酸盐的存在可能在一定程度上改变Accumulibacter种群结构。
3.3 电子受体的影响
具有反硝化除磷功能的聚磷菌一般被研究者统称为DPAOs,在对PAOs的研究初期,有研究者认为DPAOs和PAOs分别是两种不同的微生物[42-43]。但随着Accumulibacter是EBPR系统中的主导PAOs所形成共识以及发现Accumulibacter进化枝也具有反硝化除磷功能,于是有Carvalhod[35]提出DPAOs和PAOs都属于Accumulibacter,本节也基于这样的观点,着重讨论电子受体对Accumulibacter进化枝群落结构的影响。
Oehmen[4]等认为Type I型Accumulibacter可以利用硝酸盐(NO3--N)和亚硝酸盐(NO2--N)为电子受体进行反硝化吸磷,但Type II型只能利用亚硝酸盐为电子受体进行反硝化吸磷。进一步研究发现进化枝IA能利用硝酸盐为电子受体吸收P,而进化枝IIA只能利用亚硝酸盐[13]。García等[44]通过宏基因组学研究认为Type II型缺少硝酸盐还原酶(nar),这似乎是上述Type I型和Type II型不同区别的原因
然而,李博晓[45]的研究结果与上述原因有不一致的地方,他采用硝酸盐(NO3--N)、亚硝酸盐(NO2--N)作为单一电子受体并且采用乙酸和丙酸作为碳源分别运行4组SBR反应器,从反应器开始运行至运行稳定期间采用qPCR定量分析并监测反应器的菌群变化。SBR1运行条件为乙酸、NO3--N,结果发现Type I型的比例增速最快。SBR2运行条件为丙酸、NO3--N,结果发现PAOs的总比例有很大提升,这也证明了对于Accumulibacter的富集丙酸作为碳源更有优势,而Type I型的比例一直降低,Type II D的比例却一直升高。SBR3运行条件为乙酸、NO2--N,结果发现Type I型和Type IIA的比例先快速上升然后降低,进化枝IIB的数量增加但其所占比例却降低,进化枝IID的比例却一直升高。SBR4运行条件为丙酸和NO2--N,结果发现Type I型大幅增加,随后EBPR性能恶化。采用硝酸盐作为电子受体似乎有利于Type I型较Type II型取得竞争优势,但在大多数情况下,它们倾向共存于EBPR系统中。然而值得注意的是4组反应器进化枝IID虽然比例有所波动但一直都是比例最高的菌种,SBR3和SBR4中IIC均被淘汰。上诉结果这说明不能简单地以Type I和Type II类型来判断Accumulibacter进化枝对电子受体的选择性,其他影响因素(比如碳源、温度等)也可能起着决定性的作用。
4 存在的问题和展望
本综述简要总结了聚磷菌的分类及Accumulibacter进化枝的鉴别和从碳源、温度和电子受体三个方面阐述了Accumulibacter进化枝的代谢特性及种群变化。然而,目前就对Accumulibacter进化枝的代谢特性和种群变化的认知还存在缺陷,在进化枝层面的影响因素的研究还不够完善,特别是在多因素的条件下,群落结构会受到较大影响,同时检测手段也限制了对Accumulibacter进化枝的进一步认识。
为了更好的理解Accumulibacter进化枝的代谢特性,进一步完善和提高EBPR性能,在未来还需要进一步完善在多因素條件下种群结构的变化研究以及在分子水平上深入研究进化枝的代谢活动和种群结构的动态变化。
參考文献:
[1]OEHMEN A. Anaerobic and aerobic metabolism of glycogen- acc-umulating organisms selected with propionate as the sole carbon source[J]. Microbiology, 2006, 152(9):2767-2778.
[2]STOKHOLM-BJERREGAARD M, MCLLROY S J, NIERYCHLO M, et al. A critical assessment of the microorganisms proposed to be important to enhanced Biological phosphorus removal in full-Scale wastewater treatment systems [J]. Frontiers in Microbiology, 2017, 8: 1-18.
[3]SEVIOUR R J,MCILROY S. The microbiology of phosphorus removal in activated sludge processes-the current state of play[J]. J. Microbiol., 2008, 46: 115-124.
[4]OEHMEN A, LEMOS P C, Carvalho G, et al. Advances in enhanced biological phosphorus removal: From micro to macro scale[J]. Water research, 2007, 41 (11): 2271-2300
[5]FUHS G W, CHEN M. Microbiological basis of phosphate removal in the activated sludge process for the treatment of wastewater[J]. Microb. Ecol,1975,2 (2):119-138.
[6]WAGNER M, ERHART R, MANZ W, et al. Development of an ribosomal-RNA-targeted oligonucleotide probe specific for the genus Acinetobacter and its application for in-situ monitoring in activated-sludge[J]. Appl. Environ.Microbiol,1994,60 (3):792-800.
[7]BOND P L, HUGENHOLTZ P, KELLER J, et al. Bacterial community structures of phosphate-removing and non-phosphate- removing activated sludges from sequencing batch reactors[J]. Appl. Environ. Microbiol,1995, 61(5):1910-1916.
[8]BOND P L, ERHART R, WAGNER M, et al. Identification of some of the major groups of bacteria in efficient and nonefficient biological phosphorus removal activated sludge systems[J]. Appl. Environ. Microbiol. 1999, 65(9):4077-4084.
[9]HESSELMANN R P X, WERLEN C, HAHN D, et al. Enrichment, phylogenetic analysis and detection of a bacterium that performs enhanced biological phosphate removal in activated sludge[J]. Syst. Appl. Microbiol. 1999, 22 (3):454-465.
[10]CROCETTI G R, HUGENHOLTZ P, BOND P L, et al. Identification of polypho-sphate-accumulating organisms and design of 16S rRNA- directed probes for their detection and quantitation. Appl[J]. Environ. Microbiol, 2000, 66 (3):1175-1182.
[11]SAUNDERS A M. The physiology of microorganisms in enhanced biological phosphorus removal[D]. Ph.D. Thesis, University of Queensland, St. Lucia, Australia, 2005.
[12]ZILLES J L, PECCIA J, NOGUERA D R. Microbiology of enhanced biological phosphorus removal in aerated-anoxic orbal processes[J]. Water Environ. Res, 2002b, 74 (5):428-436.
[13]FLOWERS J J, HE S, YILMAZ, et al. Denitrification capabilities of two biological phosphorus removal sludges dominated by different ‘CandidatusAccumulibacter clades[J]. Environ. Microbiol. Rep, 2009, 1: 583-588.
[14]KONG Y H, NIELSEN J L, NIELSEN P H. Identity and ecophy- siology of uncultured actinobacterial polyphosphate-accumulating organisms in full-scale enhanced biological phosphorus removal plants[J]. Appl. Environ Microbiol, 2005, 71 (7):4076-4085.
[15]SAUNDERS A M, OEHMEN A, BLACKALL L L, et al. The effect of GAOs (glycogen accumulating organisms) onanaerobic carbon requirements in full-scale Australian EBPR(enhanced biological phosphorus removal) plants[J]. Water Sci. Technol, 2003, 47 (11):37-43.
[16]ZILLES J L, PECCIA J, KIM M W, et al. Involvement of Rhodocyclus-related organisms in phosphorus removal in full-scale wastewater treatment plants[J]. Appl. Environ. Microbiol, 2002, 68 (6):2763-2769.
[17]WONG M T, MINO T, SEVIOUR R J, et al. In situ identification and characterization of the microbial community structure of full-scale enhanced biological phosphorous removal plants in Japan[J]. Water Res, 2005, 39 (13): 2901-2914.
[18]STOKHOLM-BJERREGAARD M, MCILROY S J, NIERYCHLO M, et al. A critical assessment of the Microorganisms proposed to be important to enhanced biological phosphorus removal in full-scale wastewater treatment systems.Front[J]. Microbiol, 2017, 8:718.
[19]WONG M T, MINO T, SEVIOUR R J, et al. In situ identification and characterization of the microbial community structure of full-scale enhanced biological phosphorous removal plants in Japan[J]. Water Res, 2005, 39 (13): 2901-2914.
[20]MCMAHON K D, DOJKA M A, PACE N R, et al. Polyphosphate kinase from activated sludge performing enhanced biological phosphorus removal. Appl[J]. Environ. Microbiol, 2002, 68 (10): 4971-4978.
[21]HE S, GALL D L, MCMAHON K D.“CandidatusAccumulibacter” population structure in enhanced biological phosphorus removal sludges as revealed by polyphosphate kinase genes[J].Applied and Environmental Microbiology, 2007, 73: 5865-5874.
[22]SLATER F R, JOHNSON C R, BLACKALL L L, et al. Monitoring associations between clade-level variation, overall community structure and ecosystem function in enhanced biological phosphorus removal (EBPR) systems using terminal-restriction fragment length polymorphism (T-RFLP)[J]. Water Res,2010,44:4908-4923.
[23]MIELCZAREK A T, NGUYEN H T T, NIELSEN J L, et al. Population dynamics of bacteria involved in enhanced biological phosphorus removal in Danish wastewater treatment plants[J]. Water Res, 2013, 47: 1529-1544.
[24]KIM J M, LEE H J, KIM S Y, et al. Analysis of the fine-scale population structure of “CandidatusAccumulibacterphosphatis” in enhanced biological phosphorus removal sludge, using fluorescence in situ hybridization and flow cytometric sorting. Appl[J]. Environ. Microbiol , 2010, 76 (12): 3825-3835.
[25]ZENG W, ZHANG L M, FAN P C, et al. Community structures and population dynamics of “CandidatusAccumulibacter” in activated sludges of wastewater treatment plants using ppk1 as phylogenetic marker[J]. Journal of environmental sciences, 2018, 67: 237-248.
[26]ONG Y H, ChUA A S M, FUKUSHIMA T, et al. High-temperature EBPR process: the performance, analysis of PAOs and GAOs and the fine-scale population study of Candidatus “Accumulibacterphosphatis”[J]. Water Res, 2014,64:102-112.
[27]Qiu, Guanglei,Zuniga-Montanez, Rogelio,Law, Yingyu,Thi, Sara Swa,Thi Quynh Ngoc Nguyen,Eganathan, Kaliyamoorthy,Liu, Xianghui,Nielsen, Per H.,Williams, Rohan B. H.,Wuertz, Stefan.Polyphosphate-accumulating organisms in full-scale tropical wastewater treatment plants use diverse carbon sources[J].Water Research,2019,149(FEB.1):496-510
[28]MARTIN H G , Ivanova N, Kunin V, et al . Metagenomic analysis of two enhanced biological phosphorus removal (EBPR) sludge communities [ J] .NatBiotechnol , 2006, 29(10):1263-1269.
[29]SHEN N, CHEN Y, ZHOU Y. Multi-cycle operation of enhanced biological phosphorus removal(EBPR) with different carbon sources under high temperature[J]. Water Research, 2017,114: 308-315.
[30]LANHAM A B, MOITAR, LEMOS P C, et al. Long-term operation of a reactor enriched in Accumulibacter clade Ⅰ DPAOs : performance with nitrate, nitrite and oxygen[J].Water Science & Technology, 2011, 63 (2 ):352.
[31]BRENDA A, M?NICA M, LUIS B, et al. New insights in the metabolic behaviour of PAO under negligible poly-P reserves[J]. Chemical Engineering Journal, 2017,311: 82-90.
[32]LWELLESL,TIAN W D, SAAD S, et al. Accumulibacter clades Type Ⅰ and Ⅱ performing kinetically different glycogen-accumulating organisms metabolisms for anaerobic substrate uptake[J]. Water Research ,2015, 83:354-366.
[33]SAAD S A, WELLESL, ABBASB, et al. Denitrification of nitrate and nitrite by ‘Candidatus,Accumulibacterphosphatisclade IC[J]. Water Research, 2016,105: 97-109.
[34]CAMEJO P Y, OWEN BR, MARTIRANO J, et al. CandidatusAccumulibacterphosphatis clades enriched under cyclic anaerobic and microaerobic conditions simultaneously use different electron acceptors[J].Water Research, 2016,102: 130-137.
[35]CARVALHOD G, LEMOS P C, OEHMEN A, et al. Denitrifying phosphorus removal: Linking the process performance with the microbial community structure[J]. Water Research, 2007, 41 ( 19): 4383-4396.
[36]ERDAL U G,ERDAL Z K, RANDALL C W. The competition between PAOs and GAOs in EBPR systems at different temperatures and the effects on system performance[J]. Water Sci. Technol, 2003,47 ( 11):1-8.
[37]TIAN W, LOPEZ-VAZQUEZ C M, LI W , et al. Occurrence of PAOI in a low temperature EBPR system[J]. Chemosphere, 2013, 92 (10):1314-1320.
[38]TIAN W D, LOPEZ-VAZQUEZ M, LI W G, et al. Occurrence of PAOI in a low temperature EBPR system[J].Chemosphere, 2013, 92: 1314-1320. http://dx.doi.org/10.1016/j.chemosphere,2013-05:009.
[39]PANSWARD T, DOURGCHAI A, ANOTAI J.Temperature effect on microbial cmmunity of EBPR dystem[J].Water Res, 2003, 37 (2): 409-415.
[40]WANG L M,PARK J K.Ompetition between polyphosphate and glycogen or ganismsinbiological phosphorus removal systems-effect of tmperature[J].Water Sci Technol,2002,46(1-2):191-194.
[41]FLOWERS J J, CADKIN T A, MCMAHON K D. Seasonal bacterial community dynamics in a full-scale enhanced biological phosphorus removal plant[J]. Water Res, 2013, 47: 7019-7031.
[42]Kerrn-Jespersen J P , Henze M . Biological phosphorus uptake under anoxic and aerobic conditions[J]. Water Research, 1993, 27(4):617-624.
[43]MCINHOLD J, FILIPC C, DAIGGER G T , et al. Characterization of the denitrifying fraction of phosphate accumulating organisms in biological phosphate remival[J]. Water Science and Technology, 1999, 39(1):31-42.
[44]GARC?A M H, IVANOVA N, KUNIN V, et al. Metagenomic analysis of two enhanced biological phosphorus removal ( EBPR ) sludge communities [J ].Nature Biotechnology, 2006,24(10):1 263-1 269.
[45]李博曉. 反硝化除磷系统中聚磷菌菌群功能与群落分布研究[D].北京:北京工业大学, 2013.
基金项目:国家自然科学基金(项目编号:21577174);重庆市基础科学与前沿技术研究专项(项目编号:CSTC2015jcyjBX0012、CSTC2014jcyjA20004、CSTC2015jcyjA1286)。
收稿日期:2019-10-16
作者简介:谢朝新(1968-),男,博士,教授,主要从事环境工程教学科研工作。E-mail:a86909304@163.com。
通讯作者:龙向宇(1979-),男,博士,副教授,致力于废水处理理论与技术研究。E-mail:2002longxy@sina.com。
