甜瓜circRNA及其蔓枯病抗性靶基因鉴定
2020-07-28陈静鲁秀梅任琴琴孙亚亭包卫红陈雯倩钱春桃
陈静,鲁秀梅,任琴琴,孙亚亭,包卫红,陈雯倩,钱春桃*
(1.南京农业大学园艺学院/作物遗传与种质创新国家重点实验室,江苏 南京 210095;2.海门市农业科学研究所,江苏 南通 226100)
circRNA(circular RNA)是生物体内广泛存在的环状非编码RNA,由外显子或内含子序列剪切产生,大量存在于真核细胞的细胞质中[1]。近年来研究表明,在生物体内,circRNA可以作为竞争性内源RNA(ceRNA)结合miRNA,剪接和转录的调节因子以及亲本基因表达的修饰因子在细胞分化和发育的过程中发挥重要作用[2]。在动物中,研究发现circRNA在疾病发生[3]、细胞分化[4]等方面具有重要作用。在植物上的研究主要围绕组织特异性[5]、果实发育[6]等方面,且大多集中在拟南芥[7]、水稻[8]等模式植物。
在植物抗病育种方面,Ghorbani等[9]认为玉米接种伊朗花叶病毒前、后差异表达的circRNA可能影响植物的代谢和发育。Wang等[10]研究表明猕猴桃circRNA的表达具有组织特异性和物种特异性,并在植物免疫应答中发挥重要作用。Wang等[11]在番茄黄叶卷曲病毒侵染前、后的番茄植株中分别筛选出32个和83个特异表达的circRNA,认为其靶基因沉默导致番茄黄叶卷曲病毒积累减少。李冰冰等[12]在泡桐中鉴定出50个circRNA,且在健康泡桐苗和感丛枝病泡桐苗中差异表达,表明circRNA在泡桐抗丛枝病中发挥重要作用。
蔓枯病是甜瓜主要病害之一,其严重影响甜瓜的产量及品质。目前,关于甜瓜抗蔓枯病的研究主要集中于分子标记的筛选和等位性检测[13]等方面,而在非编码RNA的转录调控水平方面尚未见报道。本研究以抗病材料PI420145和感病品种‘白皮脆’自交系作为材料,在接种蔓枯病菌前、后取样,构建circRNA库,对其进行双端测序,筛选差异表达的circRNA,并进行抗蔓枯病基因验证及靶基因结构域分析,以获得甜瓜抗蔓枯病相关circRNA,揭示circRNA在甜瓜抗蔓枯病中的作用,旨在为甜瓜抗病育种研究提供新的思路。
1 材料与方法
1.1 试验材料
蔓枯病抗性材料PI420145(A)由南京农业大学葫芦科作物遗传育种与种质创新实验室提供;蔓枯病感病品种‘白皮脆’(B)由新疆农业科学院提供,经本实验室连续多代自交留种。
蔓枯病菌采用A型菌株,由本实验室提供。采用李英等[14]的方法分离纯化菌株,采用PDA培养基在(25±1)℃黑暗条件下培养7 d,然后在紫外条件(光照/黑暗时间为12 h/12 h)下培养4 d,使其产生大量分生孢子。
1.2 蔓枯病菌分生孢子悬浮液配制和蔓枯病接种
在显微镜下利用血球计数板将分生孢子配成浓度为5×105mL-1的孢子悬浮液,加乳酸将pH值调至3.5~4.0,再加入20滴Tween 20增加表面活性。参照Zhang等[15]的方法,待植株长至3叶1心时,使用手持喷雾器,进行人工喷雾接种,茎部及叶片正、反面均喷到滴水为止。接种后置于相对湿度和相对温度分别为95%和28 ℃左右的人工气候室中。
1.3 RNA文库构建和测序
在接种蔓枯病菌前(0 d)和接种后(2 d)对PI420145和‘白皮脆’取样,取植株全部完全展开的功能叶,每个样品3次重复。采用Trizol试剂法提取叶片总RNA。根据说明书,取5 μg Ribo-Zero rRNA Removal Kit(Epicentre)移除核糖体RNA,Rnase R(Epicentre)移除线性RNA,TRIzol Reagent(Invitrogen)纯化RNA,使用NEBNext®Ultra Directional RNA Library Prep Kit(NEB)构建RNA文库。Agencourt AMPure XP Magnetic Beads(Beckman)筛选长度为350~550 bp的片段,在Hiseq X10 平台上机测序,策略为PE150,即双端测序(pair-end)测序模式,即从最左端测150 bp,再从最右端测150 bp。
1.4 circRNA的鉴定
使用inhouse Perl脚本过滤fastq格式的原始reads,以删除包含adapter或ploy-N和低质量的reads。将所得到的reads利用TopHat算法比对到甜瓜参考基因组DHL92(ftp://ftp.ncbi.nlm.nih.gov/genomes/Cucumis_melo/)。使用CIRCexplorer识别circRNA,并对其中没有比对到基因组的reads用TopHat-Fusion再次比对,寻找 back-spliced reads,最后将比对到相同位置的 read count>2 的 junction reads 作为新鉴定的 circRNA。
1.5 差异表达circRNA的筛选及分析
使用TPM(Transcripts PerMillion)对circRNA的表达进行归一化。差异表达circRNA的输入数据为readcount数据。使用基于负二项分布的DESeq2分析带有生物重复的样本。
利用火山图推断差异表达circRNA的分布。通过评估差异倍数(fold change)和校正后的显著水平(padj)2个方面来筛选差异表达的circRNA。对于有生物重复的样本,筛选条件为padj<0.05。对差异表达的circRNA进行聚类分析。
1.6 circRNA靶基因的富集分析
根据circRNA与其靶基因间的对应关系,对每组差异表达circRNA的靶基因集合分别进行Gene Ontology 和KEGG富集分析。其中GO富集分析方法为GOseq,经校正后统计GO注释的候选靶基因数,并以柱状图的形式展示。根据KEGG数据库(http://www.genome.jp/kegg/)对靶基因进行注释,将P值小于0.05的Pathway定义为在候选靶基因中显著富集的Pathway。
1.7 靶基因的筛选及分析验证
筛选差异表达的circRNA,利用NCBI Conserved Domain Search Service(CD Search)平台(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行靶基因保守结构域的预测。
使用Primer Premier 5.0设计引物,SYBRPremixExTaqTM(TaKaRa)试剂盒及CFX 96荧光定量PCR仪(Bio-Rad)进行Real-time PCR。总反应体系为20 μL,其中Template cDNA 1 μL,SYBR(-20 ℃)10 μL,前引物0.5 μL,后引物0.5 μL,ddH2O 8 μL。反应程序为:95 ℃30 s,95 ℃5 s,55 ℃30 s,72 ℃30 s,共 40个循环。每个样品3次重复。以甜瓜Actin基因为内参基因,采用2-ΔΔCT方法计算基因的相对表达量,并根据单因素方差分析法进行差异显著分析。引物见表1。

表1 本研究所用引物序列Table 1 Primer sequence used in this study
2 结果与分析
2.1 甜瓜抗性表型鉴定
由图1可见:接种蔓枯病菌后3 d,抗病材料PI420145无明显症状,而感病品种‘白皮脆’表现典型的蔓枯病感染症状,即接种叶片已经出现褪绿变黄,病斑中心出现轮纹状病斑,所以选择接种后2 d的无病症叶片用于文库制备和RNA测序。
2.2 甜瓜circRNA的鉴定
经过滤,PI420145接种蔓枯病菌前(A0)和接种后(A2)分别得到102 710 802和92 350 890 条clean reads;‘白皮脆’接种前(B0)和接种后(B2)分别得到105 019 117 和95 093 825条clean reads。同时,A0、A2、B0和B2分别保留了30.81、27.71、31.51和28.52 Gb的clean bases。本试验共鉴定出来自12个文库的1 511个circRNA。在A0和B0中分别鉴定出626个和609个circRNA,在A2和B2中分别鉴定出 499个和374个circRNA。在A0和B0中,相同的circRNA有158个;在A2和B2中,相同的circRNA有105个(图2)。与A0相比,A2的circRNA数量减少20.29%;与B0相比,B2的circRNA数量减少38.59%。
2.3 甜瓜差异表达circRNA的筛选及分析
在PI420145和‘白皮脆’接种蔓枯病菌前、后的12个文库中筛选差异表达的circRNA并进行聚类分析,结果(图3)发现A0和B0共鉴定出64个差异表达的circRNA,其中21个表达上调,43个表达下调,表明 circRNA 在不同品种中存在明显差异。A2和A0中有30个差异表达circRNA,4个circRNA表达上调,26个circRNA表达下调,只有2个circRNA在两者中均有表达;B2和B0中有47个差异表达的circRNA,其中3个表达上调,44个表达下调,在两者间都有表达的circRNA只有3个,说明接种蔓枯病会引起甜瓜circRNA的变化,但在抗感品种中的反应有差异。A2和B2有9个差异表达circRNA,其中4个表达上调、5个表达下调,只有1个circRNA都有表达,说明在甜瓜不同品种中,病害的侵染会引起circRNA不同的表达。
2.4 差异表达circRNA靶基因的功能分析
对接种蔓枯病菌前、后差异表达的circRNA的靶基因进行GO富集和基因数统计。结果显示,在 A0 vs B0 中,共发现38个差异表达的circRNA靶基因,这些靶基因主要富集于3-羟基酰基辅酶α脱氢酶、氧化还原酶、钙调蛋白结合和络合物合酶等条目,其中分子功能(molecular function)类型中富集于氧化还原酶活性调控的基因数最多。在A2 vs A0中,共获得48个差异表达circRNA的靶基因,主要功能涉及应激反应、小分子代谢、脂肪酸代谢、钙调蛋白结合以及络合物合酶和氧化还原酶活性调控,其中在生物学过程(biological process)类别中响应刺激的基因数最多。B2 vs B0中,有53个差异表达circRNA的靶基因被富集到芳香族氨基酸生物合成代谢、双糖生物合成、络合酶活性调控、异构酶活性调控和催化活性调控等条目,其中催化活性调控的基因数最多。在A2 vs B2中,共发现45个差异表达的circRNA靶基因,这些靶基因的主要功能与芳香氨基化合物的生物合成、信号转导、有机环化合物的生物合成、细胞通路、细胞对刺激的反应以及生物和细胞的调控相关(图4)。
进一步用KEGG信号通路对circRNA的靶基因进行注释分类,发现在A0 vs B0中,差异表达的circRNA靶基因富集在20个通路中,主要体现在次生代谢产物合成,苯丙氨酸、酪氨酸和色氨酸的生物合成,甘油磷脂代谢,氨基酸生物合成和代谢途径。在A2 vs A0中,只有在植物激素信号转导通路中富集。B2 vs B0 中有4条通路富集,分别为苯丙氨酸、酪氨酸和色氨酸的生物合成,氨基酸的生物合成,次生代谢物的生物合成和代谢途径。在A2 vs B2中,差异circRNA靶基因在5个通路中富集(表2)。

表2 候选靶基因的KEGG信号通路分析(A2 vs B2)Table 2 KEGG signaling pathway analysis of candidate target gene(A2 vs B2)
2.5 靶基因筛选及分析验证
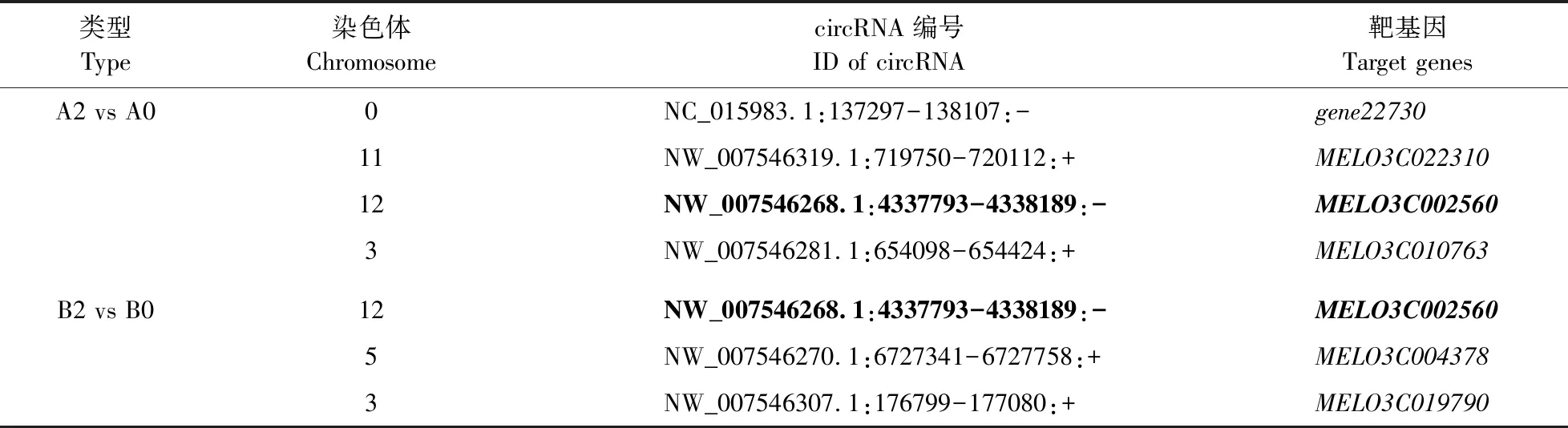
2.5.1 靶基因筛选通过比对发现,在PI420145中4个circRNA在接种蔓枯病菌前低表达或不表达,而在接种后表达上调(图3)。这些circRNA在基因组上的ID分别为NC_015983.1:137297-138107:-、NW_007546319.1:719750-720112:+、NW_007546268.1:4337793-4338189:-和NW_007546281.1:654424:+,分别位于0、11、12和3号染色体上。NC_015983.1:137297-138107:-为新鉴定的circRNA,其靶基因命名为gene22730,基因序列如图5所示,位于叶绿体基因组。同时,‘白皮脆’中有3个circRNA在接种前低表达或不表达,而在接种后表达上调(图3),分别为NW_007546268.1:4337793-4338189:-、NW_007546270.1-6727758:+和NW_007546307.1:176799-177080:+,分别位于12、5和3号染色体上。ID为NW_007546268.1:4337793-4338189:-的circRNA在PI420145和‘白皮脆’接种后都有表达(表3)。
表3 PI420145和‘白皮脆’接种蔓枯病菌前、后特异表达的circRNA
Table 3 The circRNA of differential expression in PI420145 and‘Baipicui’
before and after inoculation ofD.bryoniae
类型Type染色体ChromosomecircRNA编号ID of circRNA靶基因Target genesA2 vs A00NC_015983.1:137297-138107:-gene2273011NW_007546319.1:719750-720112:+MELO3C02231012NW_007546268.1:4337793-4338189:-MELO3C0025603NW_007546281.1:654098-654424:+MELO3C010763B2 vs B012NW_007546268.1:4337793-4338189:-MELO3C0025605NW_007546270.1:6727341-6727758:+MELO3C0043783NW_007546307.1:176799-177080:+MELO3C019790
2.5.2 基于RT-qPCR的靶基因验证由图6可知:在PI4201245中,靶基因gene22730、MELO3C022310、MELO3C002560和MELO3C019790在接种蔓枯病菌前低表达,接种后表达上调,其中基因gene22730、MELO3C022310和MELO3C002560均显著上调。在‘白皮脆’中,靶基因MELO3C002560、MELO3C004378和MELO3C010763在接种前低表达,接种后表达上调,其中基因MELO3C004378和MELO3C010763显著上调,这与测序结果一致,说明测序结果可靠。由于circRNA的表达与靶基因呈线性相关,因此基因gene22730、MELO3C022310、MELO3C002560和MELO3C010763可能为甜瓜PI420145抗蔓枯病相关基因。
2.5.3 基于保守结构域的靶基因验证利用NCBI conserved domain在线预测保守结构域,发现基因MELO3C022310编码组氨酸激酶,在516~803和1 073~1 606核苷酸处有CHASE 家族结构域,该结构域是存在于受体样蛋白的胞外部分,编码丝氨酸/苏氨酸激酶和腺苷酸环化酶等酶;在2 349~2 402核苷酸处存在HisKA结构域,其与信号转导、反应调节相关;在3 477~3 758核苷酸处有HATPase 家族,包含组氨酸激酶样ATP酶(HATPase)结构域,同样与信号转导有关。靶基因MELO3C002560编码棉纤维表达蛋白,存在DUF761结构域。基因MELO3C010763编码液泡加工酶样蛋白,在268~321、2 431~2 805、3 432~3 533这3个核苷酸区域含有CASc家族,该家族编码半胱氨酸依赖的天冬氨酸导向蛋白酶。MELO3C019790基因编码分支酸合酶,该酶与芳香族氨基酸的合成有关。
3 讨论
本研究发现2种材料接种蔓枯病菌后circRNA的数量均比接种前减少,且‘白皮脆’中的circRNA变化更大,这与Xiang等[16]的研究结果一致。聚类分析发现,接种前、后PI420145和‘白皮脆’中均产生种类和数量存在差异的circRNA,其中PI420145中差异表达的circRNA数少于‘白皮脆’,但上调表达的 circRNA 较多,说明二者的防御机制不同,这与番茄接种晚疫病病菌后相关circRNA的变化一致[17]。
差异表达circRNA靶基因功能分析的结果说明,PI420145可以激活更多的有效途径来应对蔓枯病菌的侵入。A2 vs A0中,靶基因MELO3C022310和MELO3C010763参与响应外界刺激、植物激素信号转导途径,其中MELO3C022310基因的KEGG 富集通路显示与玉米素的生物合成有关,这与高粱感染炭疽病的代谢组学研究结果一致[18]。在B2 vs B0中,MELO3C004378基因与DNA和RNA的合成相关。研究表明,MELO3C022310基因中保守结构域与植物抗病过程中信号转导有关[19]。MELO3C002560基因的DUF761结构域编码的DUF蛋白在拟南芥中参与植物的防御反应[20]。MELO3C010763基因编码的蛋白酶介导细胞程序性死亡[21]。MELO3C019790基因与芳香族氨基酸的合成有关,而芳香族氨基酸与植物抗病过程相关[22]。NC_015983.1:137297-138107:-的靶基因gene22730定位于甜瓜叶绿体基因组上,拟南芥叶绿体基因组转录产生的circRNA含量较高,可能是circRNA产生的热区之一。根据现有研究还不能判断其是否与蔓枯病抗性有关,还需要对其合成过程、发挥位点及作用进一步研究。
本研究中,circRNA在调控甜瓜PI420145对蔓枯病侵入的防御反应过程中发挥重要的作用。借鉴番茄芝麻斑病侵染过程的研究[23],推测蔓枯病菌侵染后,甜瓜的第1道防线是物理防御,即蜡质层;当蔓枯病菌穿透蜡质层后,PI420145中的circRNA启动响应外界刺激的基因,激活茉莉酸和水杨酸介导的防御信号和相关途径[24],促进芳香族氨基酸化合物合成。尽管蔓枯病菌侵入时,‘白皮脆’和PI420145都启动了防御反应,但可能PI420145对蔓枯病的识别时间更短,防御反应更迅速持久,避免进一步的伤害;而‘白皮脆’对蔓枯病的识别反应较慢,信号较弱,从而导致蔓枯病菌的繁殖和转移,最终使植株坏死。下一步研究将在PI420145对蔓枯病抗性基因克隆和表达分析的基础上,从组织解剖结构学、转录组学、代谢组学和蛋白组学等不同层面,系统地研究甜瓜PI420145对蔓枯病的防御特征,准确揭示其抗性形成机制。
