基于数据挖掘分析LUM基因在胃癌中的表达及意义*
2020-07-23彭帅田敏秀王珊珊李慧敏沈磊
彭帅,田敏秀,王珊珊,李慧敏,沈磊
430060武汉,武汉大学人民医院 消化内科
胃癌是最常见的恶性肿瘤之一,早期缺乏特异性症状与体征,故检出率较低, 影响患者预后,目前仍是癌症死亡的第二大原因[1-2]。在中国,大多数患者被诊断时已为晚期胃癌,只有10%的患者为早期胃癌,5年生存率仅为10%~30%[3]。胃癌的发生发展是由多种基因异常积累导致的多步骤复杂过程,侵袭性和转移性是导致胃癌高死亡率的主要原因[4]。蛋白聚糖作为细胞外基质的重要成分,参与癌症的发生和侵袭。富含亮氨酸的低分子聚糖(small leucine-rich proteoglycan,SLRP)不仅调节细胞外渗透压,并且在肿瘤生长、增殖、粘附、迁移和调节生长因子活性中起显著作用[5]。作为SLRP的重要成员,基膜聚糖(Lumican,LUM)被认为可以修饰纤维组织并诱导肿瘤特异性细胞外基质[6];LUM在多种组织中表达,包括皮肤、动脉、椎间盘、肾脏、骨骼、主动脉、角膜、肺和结直肠癌等[7]。LUM的表达与多种恶性肿瘤的生长和转移密切相关。最近的研究表明,在乳腺癌中,LUM基因的高表达水平与肿瘤分级、低雌激素受体表达和年龄有关[8];但在黑色素瘤中,LUM抑制黑色素瘤的发展,并起到抑癌基因的作用[9]。LUM在肿瘤组织中的异常表达表明其可能在癌症的发病机制和进程中起作用,但LUM促进胃癌发生的具体机制尚未完全阐明[7]。本研究通过数据库挖掘的方法,分析LUM在胃癌发病中的作用及其对患者预后的影响。
1 资料与方法
1.1 胃癌中的基因表达差异性分析
利用Oncomine数据库(https://www.oncomine.org)分析胃癌组织与癌旁正常组织中mRNA的差异性表达。筛选条件:①“Analysis Type: Cancer VS Normal‘’;②“Cancer type: Gastric cancer”;③“Data Type: mRNA”;④设定条件:over-expression。P<0.05为差异有统计学意义。
1.2 LUM基因在肿瘤组织中的表达差异分析
通过Oncomine数据库分析不同肿瘤间的差异性表达。设置的基因数据提取条件为:“Gene:LUM”。
1.3 LUM基因在胃癌中基因表达荟萃分析
对Oncomine数据库中所有有关胃癌与LUM基因表达相关的数据进行荟萃分析。
1.4 基于MethHC数据库的甲基化分析
MethHC数据库(http://methhc.mbc.nctu.edu.tw)主要整理了TCGA数据库中肿瘤基因组的甲基化信息,基于此分析LUM基因甲基化与胃癌的相关性。筛选条件限定于:①search by: gene;②cancer:STAD;③Select a gene region:Promoter;④Select a methylation level evalution method:Average;⑤Gene:LUM。
1.5 基于GEPIA数据库的生存分析(http://gepia.cancer-pku.cn)
通过GEPIA数据库进行胃癌患者生存分析。筛选条件:①Methods:overall survival;②Group cutoff:median;③Hazards Radio:Yes;④95% Confidence Interval:Yes;⑤Axis Units:Months;⑥Datasets Selection:STAD。
1.6 基于TCGA数据对LUM在胃癌中的表达及预后进行验证
UALCAN(http://ualcan.path.uab.edu)是一个基于TCGA数据集,肿瘤基因表达与生存分析的在线数据库。在UALCAN数据库中验证LUM在胃癌中的表达及预后。筛选条件:①“gene:LUM”;②“cancer type:stomach adenocarcinoma”;③“Split patients by:Auto select best cutoff”。P<0.05为差异有统计学意义。
1.7 蛋白质相互作用网络分析(https://string-db.org)
通过String数据库预测LUM蛋白质的上下游的调控蛋白及蛋白质间的相互调控作用。筛选条件:①Protein Name:LUM;②Organism:auto-detect。
2 结 果
2.1 胃癌中LUM转录水平的差异性分析
通过Oncomine数据库分析胃癌中LUM基因转录水平,我们发现LUMmRNA在胃癌中高表达(P<0.001),中位秩为226.5(图1)。

图1 Oncomine数据库中在胃癌中的基因差异性表达
2.2 LUM在不同肿瘤类型中的差异性表达
在Oncomine数据库中共收集了457项不同类型的研究结果,其中关于LUM表达并有统计学差异的研究结果有50项,LUM表达增高的研究有36项,表达降低的研究有14项。在胃癌组织中,LUM高水平表达的研究有5项,低水平表达的研究有0项(图2)。

图2 Oncomine数据库中LUM在不同肿瘤中的差异性表达
2.3 LUM基因在胃癌组织中表达的荟萃分析
Oncomine数据库中LUM在不同胃癌组织芯片中的表达结果显示,共有5项符合条件的研究并且每项研究中胃癌组织中LUM基因表达均高于正常对照组织(图3)。进一步对这5项研究进行荟萃分析发现,在胃癌组织中LUM表达明显高于正常对照组织(P<0.001)(图4)。
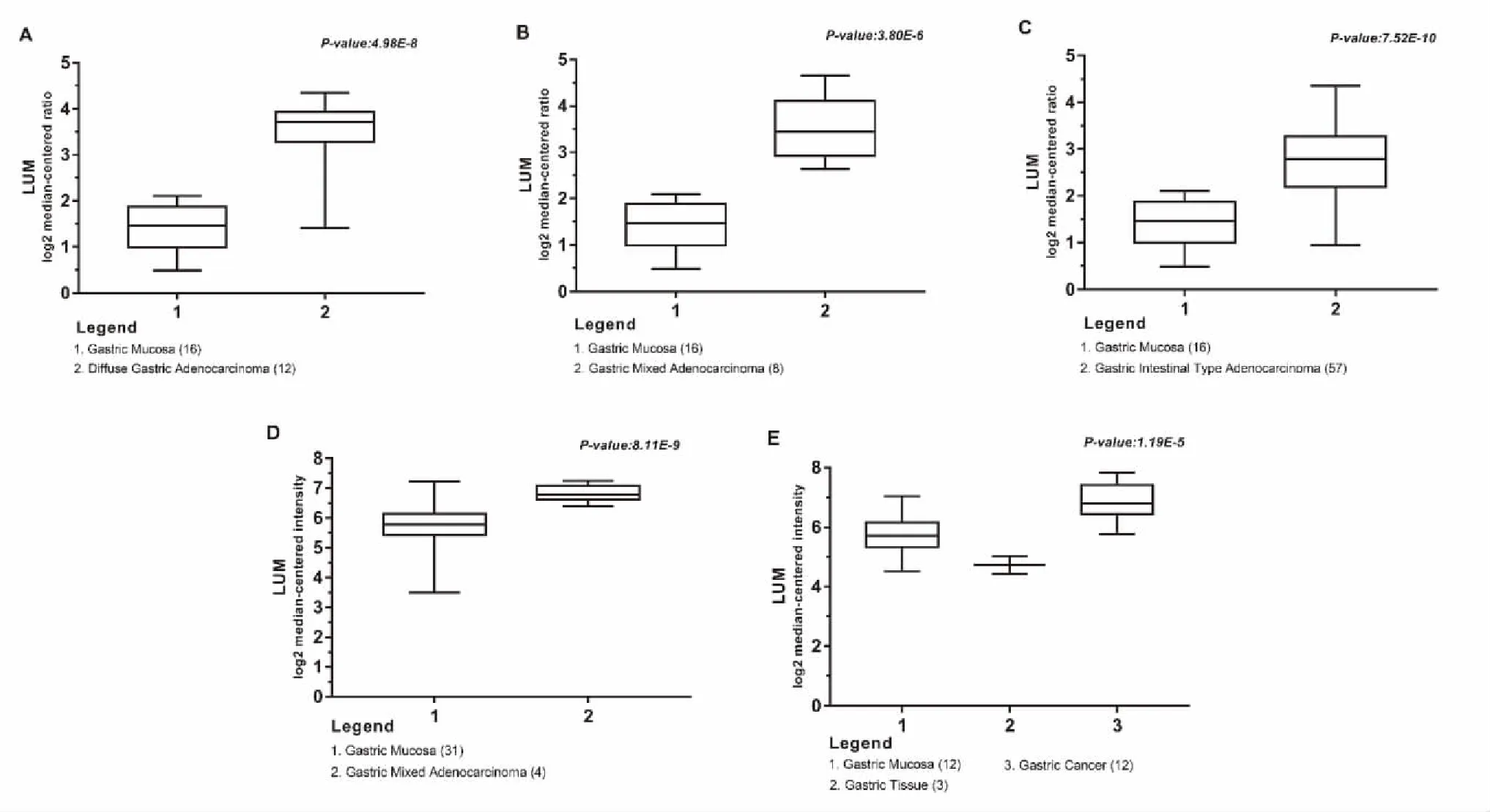
图3 Oncomine数据库中LUM在不同胃癌中的表达
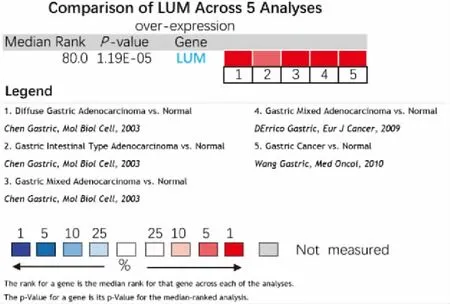
图4 Oncomine数据库中LUM在胃癌组织与正常胃组织中的表达差异
2.4 胃癌与正常组织中LUM甲基化水平
在MethHC数据库中对LUM甲基化水平差异性分析发现,胃癌组织相对正常对照组织的甲基化水平明显增高(P<0.005)(图5)。

图5 MethHC数据库中LUM在胃癌与正常组织中的甲基化水平
2.5 基于GEPIA数据库的生存曲线
通过GEPIA在线数据库对LUM在胃癌组织中的表达水平对患者生存周期的影响的分析发现,高表达和低表达组各有192例,且LUM高表达组患者的生存周期较低表达组缩短(P<0.05)(图6)。
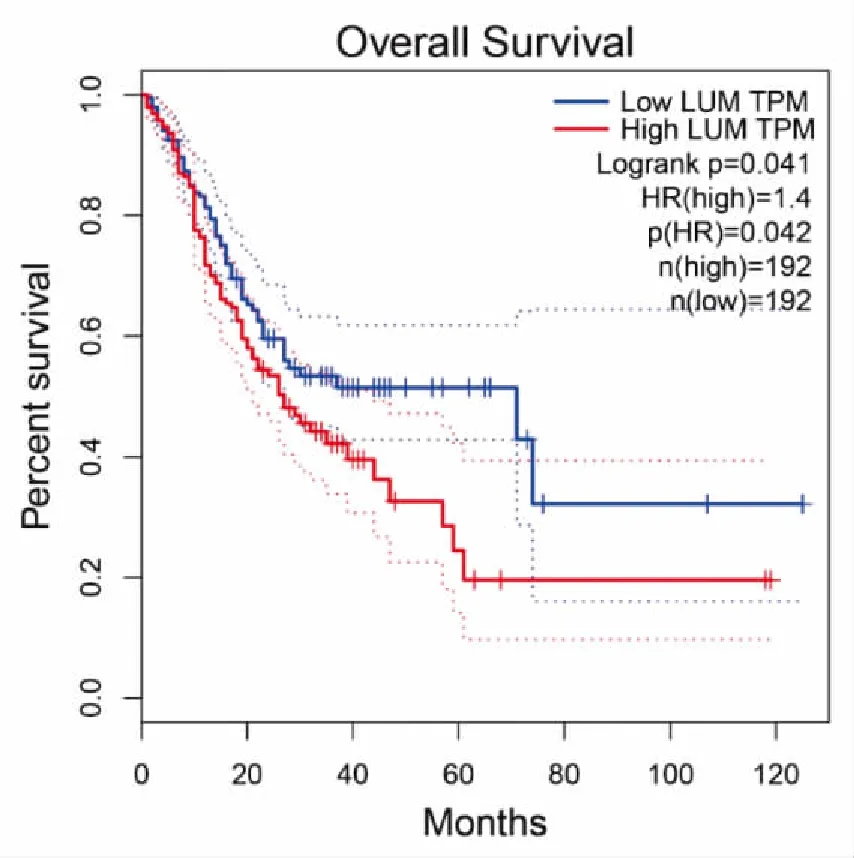
图6 LUM不同表达水平的胃癌患者总体生存曲线
2.6 基于TCGA数据集对LUM在胃癌中的表达及预后进行验证
通过UALCAN数据库在线分析发现,与正常组织相比,LUM在胃癌组织中高表达(图7);与此同时,LUM高表达组患者相较于低表达组患者总生存时间缩短(P<0.05),这与我们此前进行分析的结果一致(图8)。
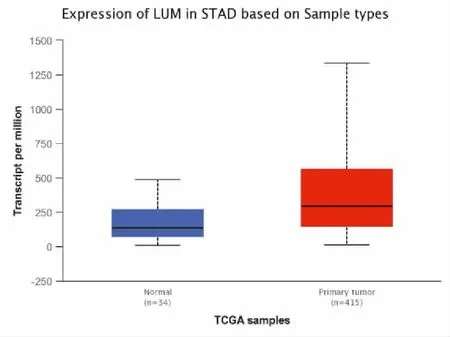
图7 LUM在TCGA数据集胃癌患者中的表达情况
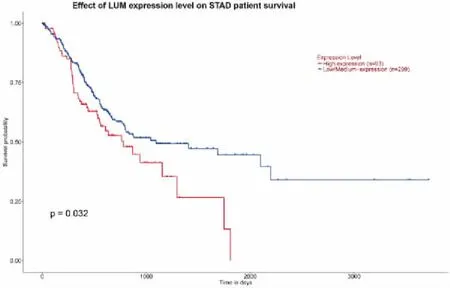
图8 TCGA数据集中LUM表达水平与胃癌患者生存时间的相关性分析
2.7 LUM基因上下游调控关系
通过String网站分析可得到LUM蛋白相互作用网络(图9),PPI富集P值为1.8×10-8,且其相互作用节点为11个,分别为OGN、CHST1、CHST5、CHST6、DCN、COL1A1、COL1A2、COL3A1、COL4A2、ACAN。同时我们发现,LUM广泛存在于人体的间质中,在正常上皮细胞和间质细胞均有不同程度表达,调节细胞外基质的水合作用,间质细胞分泌基质蛋白,及胶原纤维的形成,维持组织间质结构的完整性等。

图9 LUM基因与上下游基因调控关系
3 讨 论
胃癌的发生发展是一个受多种因素影响的复杂过程,随着人类基因组学、蛋白组学等多组学以及精准医疗的发展,人们对胃癌的认识越来越深入[10]。尽管研究者们在胃癌的病因研究中取得了重大突破,但早期胃癌的诊断和治疗仍是研究的重点,并且胃癌的侵袭转移和复发仍是胃癌预后差的主要原因[11-13]。因此,寻找与胃癌转移、复发的相关基因,以其作为临床监测、预后判断指标或精准治疗的靶点,是目前研究的重要方向。
LUM属于富含亮氨酸的SLRP家族的一员,研究发现其在多种肿瘤中均有表达,比如胰腺癌、结直肠癌、乳腺癌以及子宫颈癌等[14-15]。然而,LUM可能与肿瘤增殖呈正相关或负相关。在前列腺癌细胞中已经证明,LUM核心蛋白对癌细胞增殖没有影响,但会损害其迁移和侵袭能力[16]。相反,细胞外基质分泌的LUM蛋白可通过重塑肌动蛋白细胞骨架来促进结肠癌细胞的迁移[17],也可通过调节TGF-β2下游信号通路抑制骨肉瘤细胞的粘附[18]。LUM基因的差异表达模式和糖基化模式的差异均可能解释LUM在血管入侵、分化、增殖和侵袭中的不同作用,具体情况取决于组织类型[19]。本研究通过Oncomine数据库收集有关胃癌的相关研究,通过差异性分析发现,LUM基因在胃癌中高表达。为了增加结论的真实性和可靠性,进一步对相关研究进行Meta分析,结果与上述一致。与此同时,利用MethHC数据库分析LUM基因甲基化程度可得出在胃癌中LUM基因甲基化程度明显升高,甲基化水平与基因表达呈正相关。GEPIA数据库可基于不同数据库进行生存分析,在本研究中发现LUM高表达组生存周期明显短于低表达组,这可为胃癌患者的精准治疗提供依据。LUM在蛋白质相互作用网络中也起着重要作用,本研究通过String数据库发现LUM可能与OGN、CHST1、CHST5、CHST6、DCN、COL1A1、COL1A2、COL3A1、COL4A2、ACAN等蛋白相互作用,这可为LUM基因表达调控研究提供新的思路。最后,为了进一步验证研究的准确性,本研究使用基于TCGA数据集的UALCAN在线数据库对LUM在胃癌患者中的表达及预后进行验证[20],结果证明LUM在胃癌组织中呈现高表达,且其表达水平与胃癌患者总体生存相关,可作为胃癌诊疗的生物学标志物。
本研究基于多种数据库分析发现了基因LUM与胃癌之间的相关性,揭示了LUM参与胃癌发生发展的调控。随着对LUM研究的深入,本研究或将有助于进一步了解胃癌的发生机制,为胃癌的诊疗提供新的方向。LUM与胃癌发生发展关系的复杂性值得进一步试验验证。
作者声明:本文全部作者对于研究和撰写的论文出现的不端行为承担相应责任;并承诺论文中涉及的原始图片、数据资料等已按照有关规定保存,可接受核查。
学术不端:本文在初审、返修及出版前均通过中国知网(CNKI)科技期刊学术不端文献检测系统的学术不端检测。
同行评议:经同行专家双盲外审,达到刊发要求。
利益冲突:所有作者均声明不存在利益冲突。
文章版权:本文出版前已与全体作者签署了论文授权书等协议。
