基于2.5D级联卷积神经网络的CT图像胰腺分割方法
2020-07-10周芳芳王一达宋阳杨光
周芳芳,王一达,宋阳,杨光
华东师范大学物理与电子科学学院/上海市磁共振重点实验室,上海200062
前言
胰腺癌近年来在全球范围内呈上升趋势,难以治疗且死亡率很高[1]。精确的自动器官分割是定量评估和计算机辅助诊断的前提,但是胰腺体积小、个体差异性大、形状不规则、背景场的干扰很大,精确分割十分困难[2-3]。近年来,随着深度学习的发展,研究者们越来越多地使用自然图像语义分割模型来解决医学图像的分割任务[4],如全卷积网络(FCN)[5]和UNet[6]。其中UNet 通过跨层连接,其编码器进行特征提取时可以检测整个图像中目标组织的更多特征,且解码器进行特征融合时,可以获取到比FCN 更精确的细节信息。在卷积神经网络中加入深度监督,能够减轻训练过程中潜在的梯度消失问题,且能够有效地提高模型性能[7-8]。
传统的图像分割,常常在二维图像上进行分割[9-10],这种方法忽略了器官的3D 信息,往往达不到最佳的效果,所以很多研究人员将整个三维图像作为输入[11-14],这种方法对计算资源特别是GPU 内存的要求很高,高内存消耗限制了网络的深度和特征图的个数,而这又恰恰是性能改进的两个关键性因素[15]。为解决上述问题,一些研究者开始尝试两步分割[10,16-17]。例如,Zhou 等[10]为减小背景区域的干扰,使用了先粗分割再细分割的方法。首先应用一个2D FCN 对整个CT 图像进行胰腺的粗分割,然后根据粗分割结果定位胰腺区域并对图像进行裁剪,得到包含胰腺区域的较小的图块,再将其输入另一个2D FCN 对胰腺进行细分割。此方法在一定程度上减小了背景区域的干扰,提升了分割效果,但由于没有实现端到端的训练,因而容易失去全局信息,影响分割精度。Cai 等[16]提出了一种端到端的堆叠的CNN-RNN 分割模型,其训练过程是首先对2D CNN进行胰腺分割的预训练,然后用训练好的CNN 对多层相邻的胰腺区域进行分割,再将分割结果输入RNN,RNN通过整合其相邻层的信息,进一步得到精确平滑的分割结果。此方法对于胰腺的分割精度有明显的提升效果,但是需要对CNN 进行预训练。Yu等[17]利用两个级联的2D FCN 对胰腺进行分割。首先用一个FCN 对整个CT 图像进行胰腺区域的分割,得到一个粗分割概率图,然后将粗分割概率图通过一个显著性转换模块将其转换成空间权重,与原图相乘得到一个包含粗分割信息的图像,接着根据粗分割概率图将此图像裁剪成包含胰腺区域的较小的图像,然后输入另一个FCN 进行细分割,最后对粗分割和细分割的网络的能量函数进行联合优化。
本文提出了一种结合概率图的2.5D 级联深度监督UNet 分割模型。此网络利用两个深度监督UNet分别进行粗分割与细分割,且将粗分割结果与细分割结果通过一个简单的卷积进行概率融合。训练时通过联合优化这3 个部分的能量函数,使3 个部分的分割结果相互纠正,进一步提升了分割效果。
1 材料与方法
1.1 实验数据与计算机工作平台
本实验使用美国国立卫生研究院(NIH)的胰腺分割公开数据集(https://wiki.cancerimagingarchive.net/display/Public/Pancreas-CT),数据集中包含对82名被试者进行腹部对比增强3D CT扫描(门静脉静脉造影注射后约70 s)。CT 扫描图像大小为512×512×L,且L∈[181,466],其空间分辨率为1.5~2.5 mm,采用飞利浦和西门子MDCT扫描仪(120 kVp管电压)。进行本实验的硬件系统包含两颗INTEL 至强E5 2678 V3 CPU,64 G 内存,4 块TITAN XP 显卡;软件系统使用Ubuntu Server 16.04 操作系统,CUDA9.0 和cuDNN7.0,以及TensorFlow1.13.1深度学习框架。
1.2 数据预处理
将此82例CT数据随机分成训练集(56例)、验证集(9例)和测试集(17例)。数据预处理的流程为:(1)挑选含有胰腺的层和随机在胰腺边缘20 层内选取8 层图像作为训练数据集;(2)将原图结合其自身相邻的上两层和下两层图像变成5 层图像Ii(i=2,…,n-2,其中n为每例CT 数据中图像的层数)作为输入;(3)训练过程中,对训练数据在层内随机进行5°以内的旋转、形变系数0.1 以内的拉伸和5 个像素内的平移操作来进行数据扩增;(4)最后将数据的像素值截断在[-100,240]之间,并进行标准化,即减均值并除以标准差。
1.3 训练网络
1.3.1 训练过程对提出的CSNet,即2.5D 级联深度监督UNet 网络进行训练,得到胰腺的分割模型。训练时使用Adam 优化器,初始学习速率设置为0.001,迭代次数设为15 000,同时设置当验证集能量函数输出的能量值在1 000 次迭代中不再下降时,整个网络训练过程自动停止,每次迭代随机输入网络的样本数为6。
CSNet 模型训练流程图如图1所示。首先,将经过数据预处理的图像[5 层邻近的原始图像Ii-2,Ii-1,Ii,Ii+1,Ii+2,其中i代表第i层,结合而成3 层的图像(Ii-2,Ii-1,Ii)、(Ii-1,Ii,Ii+1)和 (Ii,Ii+1,Ii+2)]输入到CSNet的第1 个深度监督UNet网络SNet1 中,得到中间3 层对应的粗分割概率图(Pc(i-1),Pci,Pc(i+1));然后将概率图Pc(i-1),Pc(i+1)与原始图像Ii相结合(按Pc(i-1),Ii,Pc(i+1)的顺序结合)输入到第2 个UNet 网络SNet2 中,得到对应图像Ii的精细分割结果Pfi。最后粗分割概率图Pci与精细分割概率图Pfi结合输入到只含有1 层卷积核为1×1 的卷积层的概率融合网络Fuse 中,进行概率融合,得到最终输出结果。图1中的SNet 的结构如图2所示。网络由4 层下采和4 层上采组成,网络的第1 层特征数为32,卷积层中的卷积核为3×3,步长为1,池化层采用最大池化,步长为2,且每一次用激活函数Relu 激活之前,均先对卷积的输出做归一化。对于深度监督部分,第1层卷积核为5×5,第2 层卷积核为3×3,不同的卷积核大小便于提取不同的特征。最后一层卷积核为1×1,主要是为了在实现跨通道的交互及信息整合的同时降维。
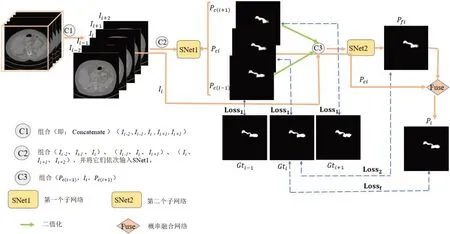
图1 CSNet流程图Fig.1 Flow chart of CSNet
本文使用的基本能量函数为交叉熵,如方程式(1)所示,L表示能量值代表金标准图的数据表示预测概率图数据。
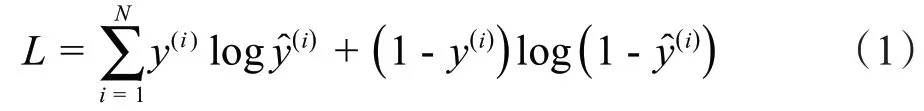
为了得到更好的分割结果,本文对能量函数进行了联合优化,即两个子网络和概率融合后的输出均与金标准做交叉熵,集成在一个能量函数中,但对3 个函数设置不同的权重。因为网络的目标是分割输入样本的中间层的胰腺,所以为SNet2和概率融合网络的交叉熵函数设置更大的权重。参考Yu等[18]和Xie 等[19]的研究结果,以及自己的实验,遍历λ=[1,2,3,4],最后经实验λ= 2,模型表现最好。如方程式(2)所示:其中Loss 代表整个CSNet 网络的总的能量函数,Loss1是第1 个子网络SNet1 的输出与其金标准的交叉熵,Loss1是第2个子网络SNet2的输出与金标准的交叉熵,Lossf是概率融合网络的输出与金标准的交叉熵。SNet1 和SNet2 两个子网络的Lossj(j=1,2)中,包含了lm和ls,如图2所示。
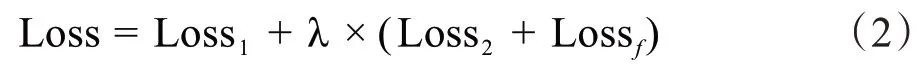
1.3.2 预测过程第一步,将预处理过的测试集数据,输入分割模型进行预测。对网络输出的分割概率图,设置阈值P=0.45,得到二值图。第二步,因为胰腺在三维空间上是一个整体,所以将分割的结果重组成三维图像,取其最大连通区域,得到最终分割的胰腺区域。测试时分割每例胰腺数据平均用时约50 s。
1.3 评价指标

图2 2.5D深度监督UNet网络SNet的结构Fig.2 2.5D deep supervision UNet used for sub-networks
本文使用2017LiTS 挑战赛评估体系中的5 种评价指标戴斯相似性系数(Dice Similarity Coefficients,DSC)、精确率(Precision)、召回率(Recall)、体素重叠误差(Volumetric Overlap Error,VOE)、体素相对误差(Relative Volume Difference,RVD)对分割模型进行评估。
2 结果
2.1 对胰腺分割的视觉结果
图3展示了分割模型对测试集中1例数据的部分分割结果。每一行包括了同一例测试数据中不同层的分割结果。从不同层中分割结果中的DSC值变化可以看出,分割模型对胰腺中央层的分割结果优于边缘层;图3中的第1列代表CSNet分割模型中子网络SNet1的分割结果;第2列代表子网络SNet2的分割结果;第3列代表整个网络的分割结果。从第1列和第2列图中的箭头处可以看出,第2个子网络SNet2和最后概率融合后的分割结果,相比于第1个子网络SNet1的分割结果都有明显提升,说明在训练分割网络时加入概率图,能够明显地改善分割效果。从第3列图中的箭头所指的区域可以看出,SNet1 的分割结果效果要优于SNet2;SNet2的分割结果在左边箭头所指的地方,分割效果要优于SNet1,而将两者概率融合后,在两个箭头处,取长补短,都达到了更优结果。
2.2 对胰腺分割的量化评估结果
为评价本文网络的分割效能,使用DSC、精确率、召回率、VOE 和RVD 作为评价指标,在17 个测试集上对2D UNet、2.5D UNet(图2的网络结构中去掉深度监督层后的网络模型)、深度监督2.5D UNet 即SNet(图2的网络模型)和CSNet 进行了比较。结果见表1,其中DSC、精确率和召回率值越接近于1,VOE 和RVD 值越接近于0,证明分割模型性能越好。由表1可以看出,本文模型在评价指标DSC、召回率和VOE 上,表现优于其它UNet 模型,但在精确率和RVD上与其他模型相近。
3 讨论
3.1 视觉效果
从图3可以看出,CSNet 对胰腺中央层的分割效果优于边缘。由于中央层中的胰腺结构与相邻层更相似,因此在结合了相邻层的粗分割结果后,分割模型能够利用相邻层的分割结果对当前层的胰腺区域进行约束,取得更好的分割结果。对于胰腺的边缘层,由于邻接层可能不包含胰腺,或者胰腺区域较小,所以相邻层信息的帮助就没这么大。但是从图3中箭头处可以看出,不论是中间层还是边缘层,与SNet1 和SNet2 相比,CSNet 网络对于胰腺边界模糊区域的分割都取得了更好的效果。
3.2 量化评估

图3 测试集中1例第59、92、102层的测试结果Fig.3 Test results for the 59th,92nd and 102nd slices in one case from test set
表1 不同分割模型在DSC、VOE、RVD、精确率和召回率上的评价结果(± s,%)Tab.1 Evaluation of DCS,VOE,RVD,precision rate and recall rate of different segmentation models(Mean±SD,%)
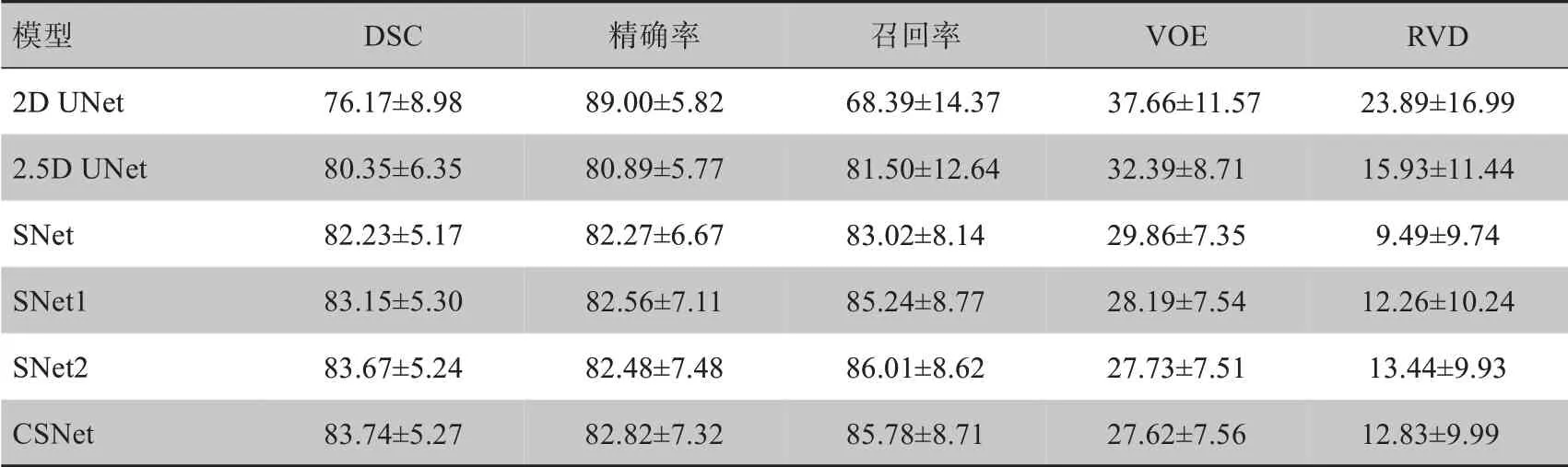
表1 不同分割模型在DSC、VOE、RVD、精确率和召回率上的评价结果(± s,%)Tab.1 Evaluation of DCS,VOE,RVD,precision rate and recall rate of different segmentation models(Mean±SD,%)
模型2D UNet 2.5D UNet SNet SNet1 SNet2 CSNet DSC 76.17±8.98 80.35±6.35 82.23±5.17 83.15±5.30 83.67±5.24 83.74±5.27精确率89.00±5.82 80.89±5.77 82.27±6.67 82.56±7.11 82.48±7.48 82.82±7.32召回率68.39±14.37 81.50±12.64 83.02±8.14 85.24±8.77 86.01±8.62 85.78±8.71 VOE 37.66±11.57 32.39±8.7129.86±7.35 28.19±7.54 27.73±7.5127.62±7.56 RVD 23.89±16.99 15.93±11.44 9.49±9.74 12.26±10.24 13.44±9.93 12.83±9.99
从实验结果中的表1可以看出,本文提出的CSNet在5个评价指标上都取得了较好的结果。2.5D UNet网络模型的分割结果优于2D UNet,这是因为2.5D的输入更有效地利用了胰腺三维结构信息;深度监督2.5D UNet网络模型SNet是在特征组合时,在最后一次反卷积之前加一个深度监督层,通过下采样层的监督,使网络能够保留更多的结构信息;同时,在深度监督层上使用与主网络不同大小的卷积核(图2),这样能够得到不同视野的组合特征,从而利用更多的细节信息[19-20],最终获得比2.5D UNet更好的分割结果。CSNet及其两个子网络SNet1和SNet2的分割结果均优于其余传统的UNet分割模型,即使相比于单独的深度监督2.5D UNet网络模型SNet,也明显有很大提升。在训练过程中,同时训练两个子网络和概率融合网络,对3个部分的能量函数进行联合优化,与只训练最后的概率融合结果相比,这样的训练方式使3个部分在各自优化的同时可以相互促进,而又不会因为只注重全局优化而失去能够进一步整个网络的分割结果的细节信息。
从表1中最后3 行可以看出,CSNet 的分割结果在多数评价标准上,优于两个子网络的分割结果。这是因为SNet1 与SNet2 输出的概率图所包含的信息并不完全相同,当进行概率融合时,通过对卷积核权重的优化就可以保留两者中有利于分割的信息。最终使得CSNet 的分割结果优于粗分割网络SNet1和细分割网络SNet2。
3.3 本文工作的局限与展望
本文的研究工作可以从几个方面进行改进:(1)本实验只使用NIH 数据集中的82例腹部CT 数据训练网络,数据来源单一;(2)本实验的数据预处理中,为了减小周围背景区域的影响,简单裁剪图像的中央部分输入网络,未来的工作中可以尝试与目标检测网络进行联合,先检测再分割;(3)实验过程中,输入网络中的只是5 层图像,损失了胰腺的全局特征,使模型对于胰腺边缘的分割效果并不是很好,且对于边界模糊区域不能做出精准判断。未来我们会尝试在CSNet网络中加入注意力机制或者与RNN 联合使用,进一步提高分割效果,且收集更多不同机构的数据进行研究。
4 结论
本文提出一种结合概率图的深度监督的2.5D UNet网络模型(CSNet)对胰腺进行分割,实验表明此方法优于传统的UNet分割方法,DSC值为(83.74±5.27)%。后续工作将尝试将此分割模型推广到其他组织器官的分割中。
