基于Group-Depth U-Net的电子显微图像中神经元结构分割
2020-07-10李玉慧梁创学李军
李玉慧,梁创学,李军
华南师范大学物理与电信工程学院,广东广州510006
前言
神经元结构分割是目前生物医学图像分析的重要课题之一[1]。然而,通过重建神经元结构来获得对人类或动物神经系统功能连接性的深入了解是神经解剖学者们面临的重大挑战[2]。电子显微成像(Electron Microscope,EM)是神经元结构成像的重要设备,但其成像数量巨大,采用人工分割的方式虽然能获得很好的分割效果,但这却是一项枯燥、繁琐且耗时耗力的任务[3]。Helmstaedter 等[4]使用20 000 h的人工仅从小鼠视网膜重建了大约1 000 个神经元。自U-Net 在神经元结构分割中取得当时最好的效果后,基于深度学习的分割算法成为医学图像分割算法的主流方法[5-6]。因此,设计一种基于深度学习的精确、快速、有效的神经元结构分割算法对于研究神经系统功能有着重大意义。
自AlexNet 在2012年的ImageNet 竞赛 中取得冠军后,卷积神经网络(Convolutional Neural Network,CNN)逐渐成为图像特征提取的主流工具[7-8]。CNN的典型使用是在图像分类任务上,一些优秀的模型如AlexNet[7]、VGG[9]、GoogleNet[10]、ResNet[11]等都会在网络的最后加入一层全连接层,然后经过softmax后获得图像的类别信息。在生物医学图像分割任务中,期望的输出应当是每个像素对应的类别。Long等[12]提出了全卷积网 络(Fully Convolutional Network,FCN),主要思想是用卷积层代替全连接层,使得任意图像大小的输入成为可能,开启了一种端到端的训练模式。但是这种模型进行语义分割时有一个主要的问题:下采样操作(比如pooling 层)会降低空间分辨率,削弱了空间“位置”信息。这个问题在编码器-解码器的结构(Encoder-Decoder)中得到有效解决。编码器经过下采样逐渐减小空间维度提取高维信息,而解码器逐渐恢复细节信息和空间维度。可利用shortcut conncetions 连接编码解码器不断补全空间细节信息。U-Net 是生物医学分割中的一种非常流行的编码器-解码器结构[5]。在大规模数据集情况下,随着网络层数的加深,网络的表达能力以及抽象能力也会随之提高,并且已经有实验证明,对于时间复杂度相同的两种网络结构,深度较深的网络性能会有所提升[13]。比如ResNet 在图像识别中,ResNet101 的识别效果要优于 ResNet34 和ResNet18[11]。同理,在语义分割任务中,一定情况下,模型越深分割效果越佳,DeepLab 利用ResNet101 作为骨架网络,在PASCAL VOC 2012 以及MS-COCO数据集中取得了最好的结果[14-16]。然而,医学图像有一个比较严重的问题:数据量小,深层模型在小样本的情况下训练时容易出现过拟合的问题。有研究者们发现,分组卷积能够使过拟合问题得到有效解决[17]。MobileNet 和Xception 成功地借鉴了这种思想,在图像分类和目标检测中能够达到较好的实时性和性能[18-19]。
在医学图像分割中,损失函数的设计与选用尤为重要,常用的损失函数包括像素级交叉熵损失、Dice coefficient[20]、MSEloss 等。如何针对特定的样本选用合适的损失函数,是目前研究的重点问题之一。
基于此,本研究提出一种用于EM 图像中神经元结构的分割深度学习模型:Group-Depth U-Net(G-D U-Net),实验结果表明该学习模型取得了比U-Net更好的分割效果。
1 方法与原理
1.1 G-D U-Net模型结构
本研究的模型结构见图1,主要由4部分组成,即分组卷积、下采样、特征图融合和上采样。

图1 G-D U-Net模型示意图Fig.1 Schematic representation of G-D U-Net model
1.1.1 分组卷积本研究的模型起始端分3 组卷积,因为过多的分组会增加卷积层的乘累加运算(Multiply Accumulate,MAC)[17],导致GPU 运算量过大。第一组由输入图片直接进行两次下采样,得到128 张feature map;第二组开始由输入图片进行卷积,得到32 张feature map,具体过程如图1c 所示,然后进行两次下采样,得到128 张feature map;第三组和第二组类似,只是开始时由输入图卷积得到64 张feature map。
MAC公式如下:

其中,h、w分别表示输出特征图的高度与宽度;c1、c2分别表示输入的通道数和输出的通道数;g表示分组卷积数。
1.1.2 特征图融合模型起始端的3 组卷积得到了384 不同的feature map,将其进行融合为下一步下采样过程做准备。
1.1.3 下采样模型的下采样过程是在原来的U-Net下采样改进的,原始的U-Net 一共下采样4 次,由于前面进行了分组卷积,为防止模型过深而过拟合,所以本研究增加了3 次下采样,一共7 次下采样。每次下采样都是由3×3 的卷积核、ReLU 激活以及最大池化(Maxpooling)组成,具体过程见图1a 所示。ReLU激活函数表示为:

1.1.4 上采样为了恢复细节信息和空间维度,本研究的模型进行了7 次上采样,用的是双线性插值(Bilinear Interpolation)[21],实现端到端的训练,见图1b 所示。同时利用shortcut conncetion 连接上采样和下采样,不断补全空间细节信息,插值公式如下:

1.2 算法流程
提出的算法框图如图2所示,主要分两大部分:(1)训练过程。为了训练本研究的分割模型,使用提供的标准EM 神经元细胞图像以及对应的标签同时对训练集进行适当的数据增强,以提高模型的鲁棒性。输入训练的神经元图像,然后经过G-D U-Net模型,得到一个预测分割图像,与相对应的标签做loss,然后经过优化器不断优化,直到loss 降到最低,模型训练完成,保存训练好的权重。(2)测试过程。输入需要测试的EM 神经元图像,加载步骤1 训练好权重,然后经过已经训练好的G-D U-Net 模型,从而得到预测的分割图像。
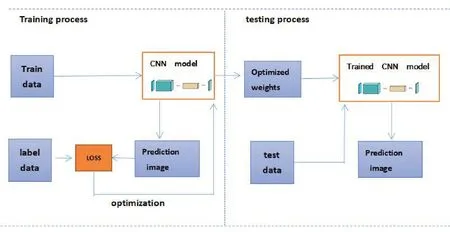
图2 算法流程框图Fig.2 Flowchart of the algorithm
2 结果
2.1 实验数据
本研究采用的数据集为ISBI 2012 提供的数据集[22-23],训练数据是来自果蝇第一龄幼虫腹侧神经索(VNC)的连续部分透射电子显微镜(ssTEM)数据集的30 个部分,共30 张图片。微管尺寸约为2.0 μm×2.0 μm×1.5 μm,分辨率为4×4×50 nm/像素。相应的标签以二进制的方式提供,即分割对象的像素为白色,其余像素为黑色(主要对应于细胞膜)。为达到实验需求,本研究将其中的25 张图像作为训练集,剩下的5张图像作为测试集。为了避免过度拟合,本研究采用旋转、翻转和弹性变形等增强策略来扩充数据集。经过数据扩充,本研究的训练数据达到800 张,其中90%作为训练集,10%作为验证集。同样,将测试集扩充为30 张。模型框架的实现是基于开源软件库pytorch 0.4 版本,并在NVIDIA GeForce 1080Ti GPU 上采用端到端的方式训练。
2.2 模型评估指标
本研究采用近年来比较常用的医学图像分割评价指标,其中包含HD 系数、Dice 系数、Conformity 系数[24]、Sensitivity 系数和RECALL 系数。为了与UNet进行对比,同样使用了Warping Error[25]、Rand Error[26]、Pixel Error 这3 个评价指标。每种指标的介绍见表1。

表1 医学图像分割评价指标介绍Tab.1 Introduction to the evaluation indexes of medical image segmentation
2.3 实验结果分析
在ISBI 2012 数据集中的测试效果如图3所示。从视觉感受上可以看出,本研究的模型在细节处理方面表现优于原始的U-Net,对于神经结构内部的一些不明确区域和复杂区域的分割效果更好。

图3 分割结果的可视化Fig.3 Visualization of segmentation results
具体定性分析见表2~表4。表2为ISBI 2012 旧的评分指标,从表2可知,G-D U-Net 模型在Rand Error 和Pixel Error 两项指标中优于U-Net 模型,这是因为G-D U-Net模型不仅加深了网络层数,而且增加了分组卷积,从而补全了细节信息。表3为ISBI 2012 新的评分指标,从表3可知,G-D U-Net 模型在新的评分指标中也有优异的表现。同样,从表4可知,G-D U-Net 模型在5 种常用的医学图像分割指标中表现比U-Net 模型好,从而验证了模型设计的有效性。

表2 不同模型分割效果的对比(a)Tab.2 Comparison of results of segmentation by different models(a)

表3 不同模型分割效果的对比(b)Tab.3 Comparison of results of segmentation by different models(b)

表4 不同模型分割效果的对比(c)Tab.4 Comparison of results of segmentation by different models(c)
2.3.1 模型的不同深度对分割性能的影响为了进一步验证模型的深度对于分割效果的影响,本研究在G-D U-Net 模型原来7 次上下采样的基础上进行改进,得到两个较浅的模型,一个是5 次上下采样的模型:G-D U-Net1;另一个是6 次上下采样的模型:G-D U-Net2。然后训练相同的次数对其进行测试,得到的分割指标如表5所示。从表5可知,模型的深度对分割效果有一定的影响,在一定范围内模型越深,分割效果会更好,但是网络过深训练时容易过拟合,且容易出现计算机显存不足的情况。

表5 不同深度的模型分割效果的对比Tab.5 Comparison of results of segmentation by different deep models
2.3.2 分组卷积对分割性能的影响同理,为了验证分组卷积对分割性能的影响,本研究在G-D U-Net的基础上,移除第一组,得到G-D U-Net3;移除第一组和第二组卷积,保留第三组,得到新的模型G-D UNet4。测试效果如表6所示。模型起始端加入分割后,能够在一定程度上提高分割效果。

表6 分组卷积对分割性能的影响Tab.6 Effect of group convolution on segmentation performance
2.3.3 不同的损失函数对分割性能的影响实验发现使用不同的损失函数对分割性能有一定的影响。本研究提出的G-D U-Net模型采用的是L1 loss,为了进行对比,本研究把loss换成MSE loss和交叉熵损失函数(Cross Entropy loss,BCE loss),得到两个新的模型,记为G-D U-Net5、G-D U-Net6。3 种loss 的分割效果图如图4所示,从视觉感受L1 loss最接近理想效果,其他两种loss 分割后图比较模糊。3 种损失函数的公式如下:

其中,lN表示第N个样本中预测值与标签的误差。

图4 不同的损失函数对分割性能的影响Fig.4 Effect of different loss functions on segmentation performance
从以上的损失函数表达式可以看出,采用BCE loss 单独评估每个像素矢量的分类预测,然后对所有像素求平均值,基本上假定同等地对待图像中每个像素点。如果各种类在图像分布不平衡时,这可能会是一个问题,因为训练过程将受到最多的类所支配。MSE loss 同样也是利用预测的像素值与标签的像素值的loss 的平方求平均,所以也会出现上述问题。因为此分割任务是预测一张二值图(细胞膜像素值为1,非膜像素为0),如果采用BCE loss 或者MSE loss,预测结果就会出现如图4c 和4d 上的“阴影”,而L1 loss 是将预测值与实际值误差的绝对值作为loss,这样预测值就能够不断接近真实值,不会出现“阴影”,因此L1 loss将更适用于此分割任务。
本研究对3 种不同损失函数的模型进行测试,得到的分割指标如表7所示。其中,G-D U-Net模型(采用L1 loss)在神经元结构的分割中表现最佳。

表7 不同的损失函数对分割性能的影响Tab.7 Effect of different loss functions on segmentation performance
3 总结与讨论
本研究提出一种用于EM 图像中神经元结构的分割深度学习模型G-D U-Net,在EM 神经细胞的分割实验中,相比于原始的U-Net 模型,该模型获得了较好的分割效果。同时本研究经过大量的实验,验证了分组卷积、加深模型深度以及使用L1 loss 对分割效果的提升,为研究神经结构的生物医学专家提供了一定的参考,也为其他生物图像图像分割领域提供一定的方法借鉴。
