基于转录组分析筛选牛心朴子响应低温胁迫的转录因子家族
2020-07-07马晓闻周思泓王丹玉贠桂玲侯玉霞
马晓闻 周思泓 王丹玉 贠桂玲 侯玉霞
摘要:【目的】从转录水平分析牛心朴子在低温胁迫下的差异表达基因,筛选响应低温胁迫的转录因子家族,鉴定出牛心朴子低温胁迫响应的关键调控基因,为全面解析逆境胁迫响应分子调控网络及有效挖掘关键调控基因提供理论参考。【方法】通过高通量测序技术对低温胁迫(CT组)和常温处理(对照,CK组)的牛心朴子cDNA文库进行转录组测序分析,对差异表达基因进行功能注释和富集,鉴定出响应低温胁迫的转录因子家族,并选取4个差异表达的转录因子基因进行实时荧光定量PCR检测,以验证转录组测序结果的可信度。【结果】从CK组和CT组共获得30.50 Gb的原始数据,Cycle Q20平均值在96%以上,经数据过滤及去冗余后,拼接组装获得100006条Unigenes,但其功能注释率较低,有47082条至少在一个数据库中被功能注释,占Unigenes总数的47.07%;而在NR、NT、KO、SwissProt、PFAM、GO和KOG数据库中均被注释的Unigene有7070条,仅占Unigenes总数的7.06%。GO功能富集分析筛选到5545个差异表达基因,其中,上调表达基因2039个,下调表达基因3506个,分别富集到生物过程、细胞组分和分子功能三大类别中。从牛心朴子Unigene中共鉴定到83个转录因子家族的1826个转录因子,其中,以MYB转录因子家族成员数目最多,为136个(占7.45%)。从差异表达基因中筛选到与低温胁迫有关的66个转录因子家族的550个转录因子,其中MYB、C3H、bHLH、AP2-EREBP、C2H2、NAC、bZIP、CCAAT和WRKY等转录因子家族均有大量转录因子能被低温胁迫诱导表达。基于实时荧光定量PCR的牛心朴子低温胁迫下转录因子基因表达水平检测结果与转录组测序分析结果基本一致。【结论】MYB、C3H、bHLH、AP2-EREBP、C2H2、NAC、bZIP、CCAAT和WRKY等转录因子家族成员在牛心朴子响应低温胁迫时发挥主导作用,同时各家族转录因子间存在共表达性或协同作用,通过复杂的转录调控网络发挥重要调节作用,进而提高牛心朴子对低温胁迫的耐受性。
关键词: 牛心朴子;转录组;转录因子;低温胁迫;差异表达基因
中图分类号: S567.239 文獻标志码: A 文章编号:2095-1191(2020)05-0995-09
Cold-responsive transcription factor family in Cynanchum komarovii based on transcriptome data
Abstract:【Objective】In order to identify the cold-responsive transcription factors and screen the key regulatory genes of Cynanchum komarovii in response to cold stress,the transcriptome analysis on the differentially expressed genes(DEGs) in C. komarovii was analyzed,which provided a theoretical reference for the comprehensive analysis of the stress response molecular regulation network and the effective mining of key regulatory genes. 【Method】High-throughput sequencing technology was applied to detect the transcriptome sequencing data of the cDNA library of C. komarovii under cold stress treatment(CT) and normal atmospheric temperature treatment(control, CK). The DEGs were functionally annotated and enriched,and the cold-responsive transcription factor families were identified. Fourdifferentially expressed transcription factor genes were selected using real time fluorescent quantitative PCR for the determination to check the reliability of transcriptome sequencing. 【Result】30.50 Gb of sequencing data were obtained from CK and CT groups with the average Cycle Q20 over 96%. A total of 100,006 uni-genes were assembled after data filtering and elimination of redundancy, but the functional annotation rate was low. 47082 uni-genes had functional annotation in at least one database, accounting for 47.07% of total unigenes. By comparing the unigenes with all the public protein databases of NR,NT,KO,SwissProt,PFAM,GO,and KOG,only 7,070(7.06% of the total) were functionally annotated. GO enrichment analysis was performed and 5,545 differentially expressed genes were identified in three main GO terms including biological process,cellular component,and molecular function,among which 2,039 were up-regulated and 3,506 were down-regulated under cold stress conditions. A total of 1,826 transcription factors belonging to 83 families were identified in C. komarovii unigenes. The most abundant transcription factors families detected in the transcriptome was the MYB(136),accounting for 7.45%. Moreover,550 transcription factors in 66 transcription factor families related to cold stress were selected from differentially expressed genes, MYB,C3H,bHLH,AP2-EREBP,C2H2,NAC,bZIP,CCAAT,and WRKY families had abundant transcription factors that could be induced to express under cold stress. The transcription factor gene expression detection results of C. komarovii under cold stress based on real time fluorescent quantitative PCR was consistent with that by transcriptome sequencing analysis. 【Conclusion】The transcription factors families MYB,C3H,bHLH,AP2-EREBP,C2H2,NAC,bZIP,CCAAT,and WRKY in C. komarovii play a leading role in response to cold stress. There might have co-expression or synergistic effects among transcription factors of these family members, they regulate through complex transcription regulatory network,which can improve the tolerance of C. komarovii to cold stress.
Key words: Cynanchum komarovii; transcriptome; transcription factor; cold stress; differentially expressed genes
Foundation item: National Key Research and Development Program(2017YFD0201900)
0 引言
【研究意义】低温是影响植物生长与分布的重要环境因素之一,严重威胁农作物产量的形成,制约着农业生产的发展(张健等,2019)。牛心朴子(Cynanchum komarovii)是一种荒漠草本植物,对低温强光、干旱高温的荒漠环境具有极强的适应性,拥有潜在的抗逆基因资源待挖掘,是研究植物抗逆机制的良好材料(马晓闻,2016)。在逆境胁迫响应过程中,转录因子作为关键调控蛋白控制着下游大量抗逆相关功能基因的表达,是一类在转录调控过程中发挥重要作用的蛋白(朱玉贤等,2013)。因此,研究抗逆相关转录因子的功能及特性,并筛选出优良候选基因,利用其综合改良作物的抗逆性已成为农作物抗逆育种的重要途径(王薇,2009)。通过转录组测序分析牛心朴子在低温胁迫下的差异表达基因,鉴定响应低温胁迫相关转录因子,发掘出耐低温关键调控基因,对培育农作物耐冷新品种及减少生产损失具有重要意义。【前人研究进展】基于高通量测序技术的转录组测序已成為发掘缺乏基因组信息物种功能基因的重要手段,被广泛应用于筛选植物抗逆关键基因(唐玉娟等,2018)。目前,已有较多针对植物低温逆境胁迫转录组分析的研究报道。Zhao等(2012)基于高通量测序平台对冷胁迫下具有极强耐冷能力的高山冰缘植物高山离子芥(Chorispora bungeana)进行转录组分析,结果发现MYB(v-myb avian myeloblastosis viral oncogene homolog)、AP2/ERF(APETALA2/ethylene-responsive factor)、WRKY和NAC等转录组因子家族成员参与低温胁迫响应时的表达量差异显著。Xu等(2014)研究低温环境下正常生长的山葡萄(Vitis amurensis)抗冷相关基因,通过转录组测序鉴定出38个与抗冷相关的转录因子家族,推测这些转录因子与该物种独特的耐冷能力有关。Wang等(2014)对我国东北地区严寒条件下正常生长的卷丹百合(Lilium lancifolium)在低温胁迫不同时段的基因转录水平进行Solexa测序分析,初步解析了钙信号和脱落酸(ABA)依赖与非依赖途径的差异性表达,结果发现冷诱导相关基因ICE、钙依赖而钙素不依赖蛋白激酶基因CDPK、干旱应答元件的结合蛋白基因DREB、AP2/EREBP基因等表达显著上调,推测这些基因是响应低温胁迫的主要调控基因。Wang等(2015)通过高通量测序技术分析耐寒植物枳[Poncirus trifoliate(L.) Raf.]在低温胁迫下的应答基因,结果发现5177个基因受低温胁迫诱导发生差异表达,其中有60个与冷胁迫应答相关的转录因子,且上调表达基因主要参与植物激素信号转导、次生代谢产物生物合成途径。Wei等(2015)研究发现,抗冷植物结缕草(Zoysia japonica)在低温胁迫处理后,其应答基因数量最多的是DREBs转录因子家族成员,且参与淀粉水解、脯氨酸合成、抗坏血酸代谢、超氧化物歧化酶(SOD)激活的基因均显著上调表达。Yang等(2015)利用高通量测序技术对水稻抗冷品种TNG67与冷敏感品种TCN1幼苗进行转录组测序分析,结果表明,TNG67和TCN1在冷应答转录因子基因表达量、激素相关基因表达量及胁迫响应相关基因应答时间等方面均存在明显差异,可能是造成品种间抗冷性差异的主要原因。Tan等(2016)对低温胁迫下豇豆(Vigna unguiculata subsp. sesquipedalis)的抗冷品种和冷敏感品种进行转录组测序分析,结果显示冷应答DREBs转录因子家族与ICE1-CBF3-COR冷诱导信号间存在级联交互,且其在豇豆应答低温胁迫时发挥重要调控作用。Gong等(2018)对低温胁迫处理的橡胶树进行转录组测序分析,并筛选响应低温胁迫相关的调节候选基因,结果发现9个转录因子家族的239个转录因子参与低温胁迫响应。【本研究切入点】目前,针对牛心朴子响应逆境胁迫的研究主要集中在植株生理生化水平的适应性调节(陈翠云等,2012),而鲜见有关牛心朴子在低温胁迫下的分子机理和转录组学的研究报道。【拟解决的关键问题】通过高通量测序技术对低温处理和常温处理(对照)的牛心朴子RNA文库进行转录组测序分析,对差异表达基因进行功能注释和富集,分析牛心朴子低温胁迫相关转录因子的表达特性,以期筛选出牛心朴子低温胁迫响应的关键调控基因,为全面解析逆境胁迫响应分子调控网络及有效挖掘关键调控基因提供理论参考。
1 材料与方法
1. 1 试验材料
供试植物材料为牛心朴子(C. komarovii)种子,采自宁夏灵武市瓷窑堡镇,由中国农业大学理学院分子生物学实验室提供。TRIzol植物RNA提取试剂盒购自Invitrogen公司;文库构建试剂盒(NEBNext UltraTM RNA Library Prep Kit)购自NEB公司;实时荧光定量PCR试剂盒[SYBR? Premix Ex TaqTM(Tli RNa-seH Plus)]购自TaKaRa公司。主要仪器设备:HITACHI高速低温离心机、THERMO -86 ℃超低温冰箱、Agilent 2100、Qubit Fluorometer 2.0、Illumina HiSeqTM 2000高通量测序仪、ABI 7500 Thermocycler荧光定量PCR仪、光照培养箱和无菌操作超净工作台。
1. 2 样品处理及采集
牛心朴子种子经4%次氯酸钠和75%乙醇浸泡消毒10~20 min,无菌水冲洗3次,播种于混合土(营养土∶蛭石=2∶1)中,在16 h光照/8 h黑暗,温度25 ℃/22 ℃的条件下培养3周。将长势一致的牛心朴子幼苗从土壤中转移至Hoagland营养液中继续培养,在4 ℃低温培养箱中进行低温胁迫处理(CT组),以常温为对照(CK组),每处理设3个生物学重复,在处理72 h后收集幼苗植株,-80 ℃超低温冰箱保存备用。
1. 3 测序样品制备及高通量测序
采用TRIzol植物RNA提取试剂盒分别提取CT和CK组样品总RNA,并进行RNA浓度、纯度和完整性检测。待检测合格后,参照NEBNext UltraTM RNA Library Prep Kit说明构建cDNA文库,最后使用Illumina 2000测序平台(HiSeq/MiSeq)进行测序。
1. 4 转录组数据分析
测序所得的原始序列(Raw reads)经检查过滤后获得待分析数据(Clean reads),利用Trinity软件对其进行组装拼接以获得转录本序列,取每个基因最长的转录本为Unigene,与公共数据库比对分析后进行基因功能注释(Moriya et al.,2007;Grabherr et al.,2011)。应用iTAK软件预测植物转录因子家族成员,利用database中分类定义好的转录因子家族及其规则,通过hmmscan鉴定转录因子家族(Zheng et al.,2016)。鉴于试验设有生物学重复,采用DESeq进行差异表达基因分析,筛选阈值为padj<0.05(Anders and Huber,2010)。
1. 5 实时荧光定量PCR检测
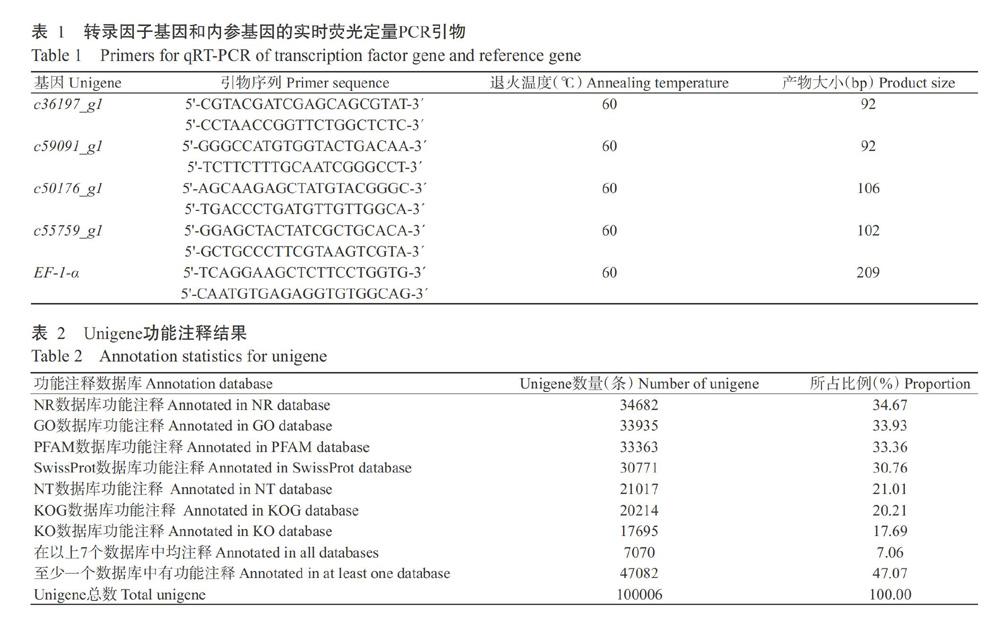
选取4个差异表达转录因子基因进行实时荧光定量PCR检测,以EF-1-α为内参基因,按照SYBR? Premix Ex TaqTM(Tli RNaseH Plus)试剂盒说明配制反应体系,扩增反应在ABI 7500 Thermocycler上进行(Wang et al.,2011)。扩增程序:95 ℃预变性30 s;95 ℃ 5 s,60 ℃ 34s,进行40个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。采用2-ΔΔCt法换算目的基因的相对表达量。4个差异表达转录因子基因的实时荧光定量PCR扩增因引物如表1所示。
2 结果与分析
2. 1 测序分析及Unigene功能注释结果
从CK组和CT组共获得30.50 Gb的原始数据,Cycle Q20平均值在96%以上,表明测序数据质量较好,可用于后续分析。经数据过滤及去冗余后,对高质量Clean reads进行拼接组装后获得转录本序列,取每个基因最长的转录本作为Unigene,最终获得100006条Unigenes,平均长度为649 bp,N50为1129 bp。由表2可知,100006条Unigenes在NR、GO、PFAM、SwissProt、NT、KOG、KO等数据库中注释的Unigenes分别为34682(占34.67%)、33935(占33.93%)、333363(占33.36%)、30771(占30.76%)、21017(占21.01%)、20214(占20.21%)和17695条(占17.69%)。有47082条Unigenes至少在一个数据库中得到功能注释,占Unigenes总数的47.07%;而在以上7个数据库均被注释的Unigenes仅有7070条,仅占Unigenes总数的7.06%。可见,牛心朴子Unigenes功能注释率较低。
2. 2 牛心朴子响应低温胁迫的基因差异表达分析结果
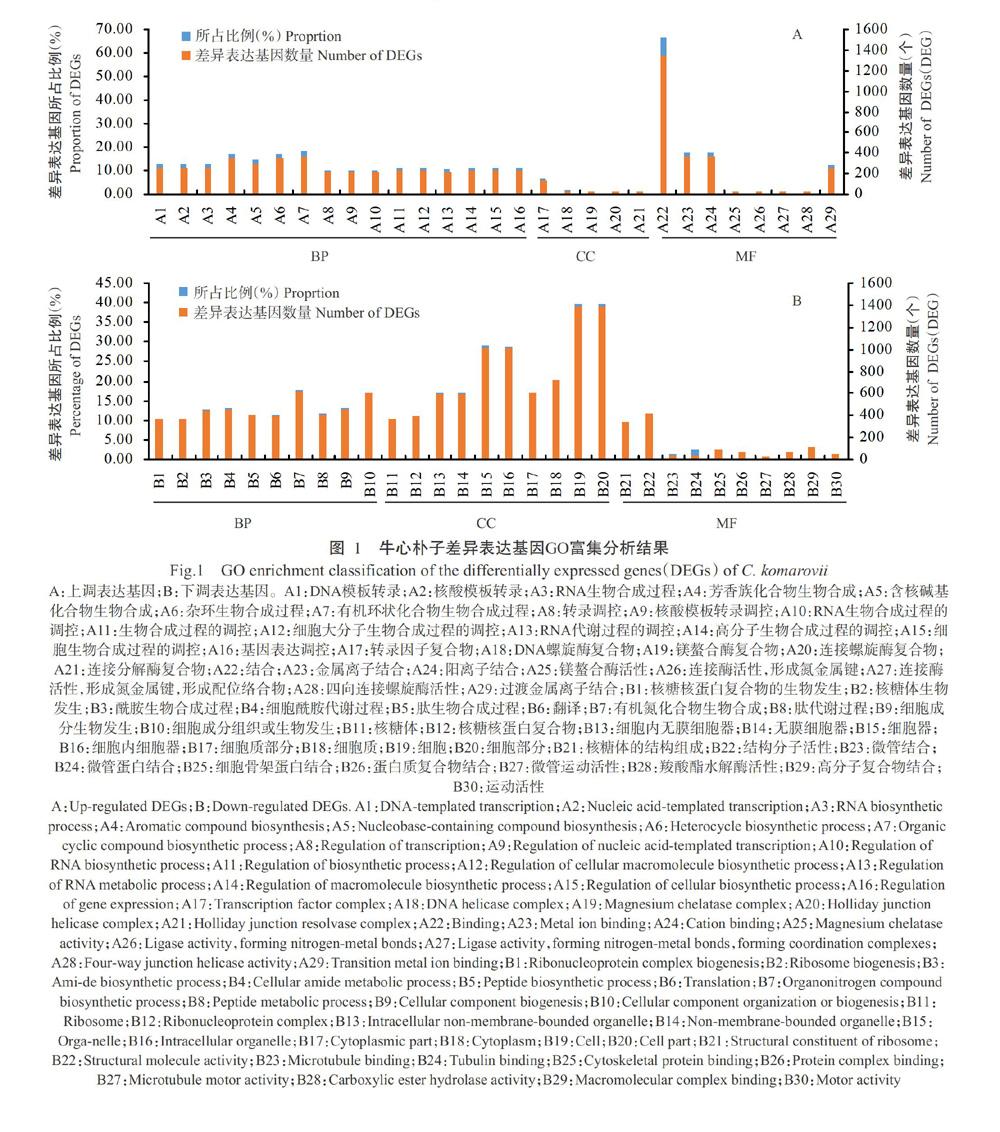
采用DESeq进行差异表达基因分析,共获得7742个差异表达基因。将这些差异表达基因进行GO富集分析,最終筛选得到5545个差异表达基因,其中,上调表达基因2039个,下调表达基因3506个,可富集到生物过程(Biological process,BP)、细胞组分(Cellular component,CC)和分子功能(Molecular function,MF)三大类别中。将牛心朴子响应低温胁迫时差异表达基因按照不同的功能进行归类和注释,可为后续分析差异表达基因中的转录因子家族功能提供参考。
对上调表达基因进行GO富集分析,结果(图1-A)发现,富集在MF的上调表达基因主要分子功能为结合、金属离子结合和阳离子结合;富集在BP的上调表达基因主要参与环状化合物、杂环化合物和芳香族化合物的生物合成过程,且参与各化合物生物合成过程的差异表达基因数量为200~400个;富集在CC的上调表达基因中,有132个基因富集于转录因子复合物,推测这些基因差异表达是响应低温胁迫,参与调节合成代谢。
对下调表达基因进行GO富集分析,结果(图1-B)发现,下调表达基因主要富集在BP和CC两大类别中。其中,富集在BP的下调表达基因主要参与有机物生物合成过程(有机氮化合物及酰胺生物合成等)、细胞生物合成和蛋白代谢过程、有机物代谢过程;富集在CC的下调表达基因中以富集于细胞和细胞部分的基因最多,推测这些基因表达被低温胁迫所抑制,且低温胁迫使植物的细胞成分和结构、生物合成及代谢受到一定程度的影响。
2. 3 牛心朴子转录因子家族分析结果
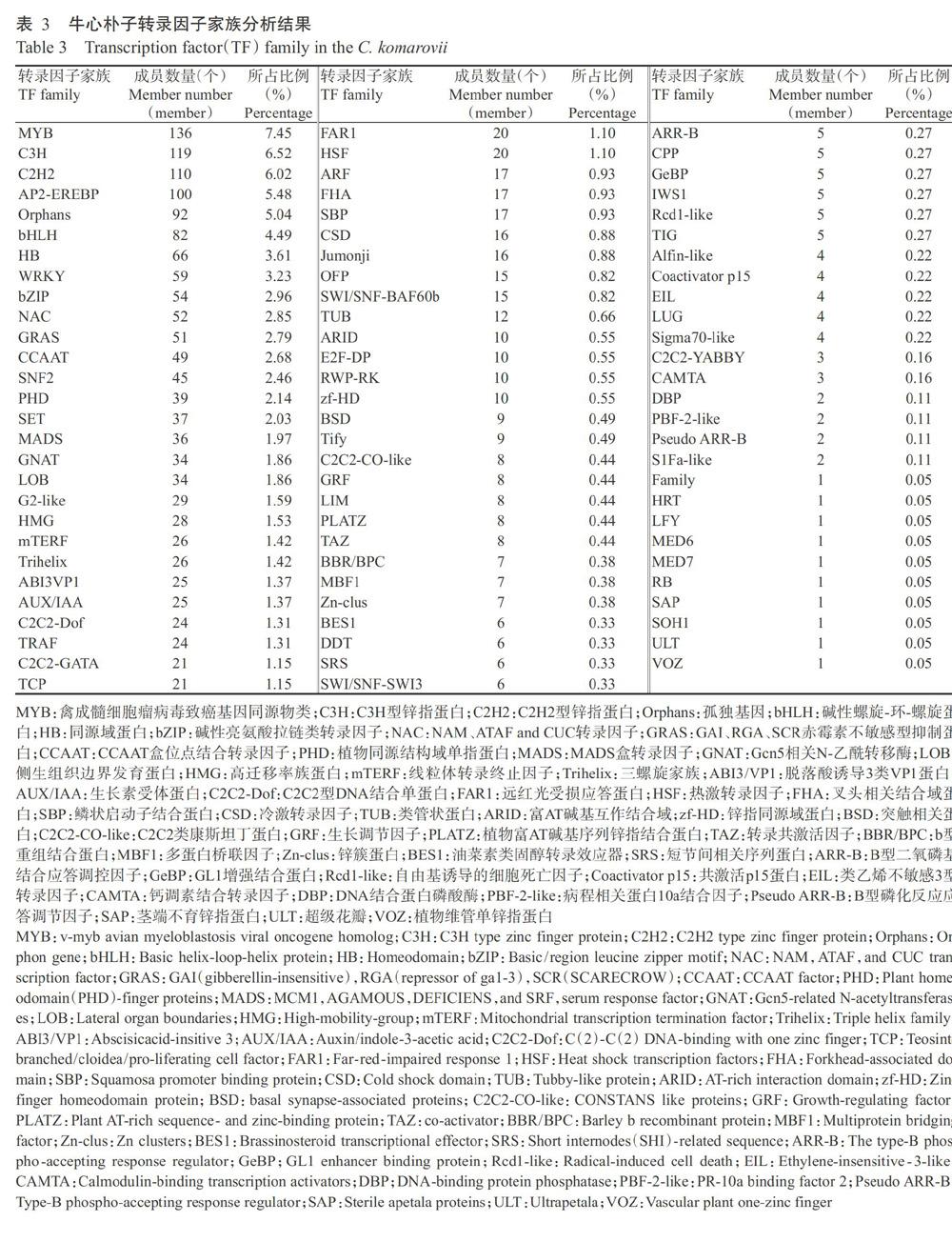
使用iTAK软件对牛心朴子100006条Unigenes进行转录因子家族鉴定,结果共鉴定出1826个转录因子,隶属于83个转录因子家族(表3)。其中,以MYB转录因子家族成员数量最多,为136个(占7.45%),成员数量排前15位的家族共有1091个转录因子,占转录因子总数的59.75%。
2. 4 牛心朴子响应低温胁迫的转录因子分析结果
从低温胁迫牛心朴子转录组已鉴定且注释功能的差异表达基因中,共鉴定出550个转录因子,隶属于66个转录因子家族,其中有15个转录因子家族成员数量较多(表4),占转录因子总数的58.91%,尤其是MYB、C3H、bHLH、AP2-EREBP、Orphans、HB、C2H2、NAC、bZIP、CCAAT和WRKY等家族均有大量转录因子被低温胁迫诱导,是研究牛心朴子低温胁迫响应机制中调控功能较多、参与代谢通路调控较多的转录因子家族,将是后续研究工作的重点。
2. 5 实时荧光定量PCR检测结果
为验证转录组测序结果的可信度,随机选取4个差异表达的转录因子基因,以EF-1-α基因为内参基因进行实时荧光定量PCR检测,结果如表5所示。虽然因方法学上的差异导致结果略有偏倚,但实时荧光定量PCR检测出的基因表达模式与转录组测序分析获得的基因表达趋势基本一致,表明转录组测序数据结果可信度较高。
3 讨论
本研究发现,高通量测序技术可有效挖掘牛心朴子中的大量转录信息及差异表达基因,但牛心朴子Unigene功能注释率较低,仅有47.07%的Unigene至少被一个数据库进行功能注释。Long等(2014)对荒漠地区极为抗旱、耐热的超旱生固沙植物梭梭(Haloxylon ammodendron)进行干旱胁迫转录组分析,结果发现仅有37.52%的Unigene至少被一个数据库功能注释。Dang等(2013)、Wu等(2014)、满玲娟等(2019)也研究发现,强抗逆植物长叶红砂、沙冬青和梭梭分别有41.91%、59.3%和53.9%的Unigene至少在一个数据库中得到功能注释,其原因是这些植物属于无基因组测序数据公布的非模式植物,公共数据库中亲缘关系相近物种遗传信息缺乏或不足,无法与基因组已测序公布的拟南芥、玉米、棉花、水稻和烟草等物种相对比。因此,本研究通过高通量测序技术对未知基因进行组装拼接及功能注释,为亲缘关系相近植物提供了大量参考数据,对研究植物逆境下的基因表达调控和抗逆育种具有重要意义。
转录因子在植物逆境信号转导和基因表达调控中均发挥重要作用。大量研究证实,MYB、ZFP、bHLH、NAC、AP2/EREBP、bZIP、WRKY、CAMTA、EIN3、HSF、YABBYs、GATA、GRAS和TCP等转录因子家族参与响应低温胁迫应答及调控相关功能表达基因(刘潮等,2017;周丽霞和曹红星,2018;张健等,2019)。Pang等(2013)对冷驯化条件下的沙冬青进行转录组测序分析,结果鉴定出720个差异表达基因参与转录调控,其中数量最多的转录因子家族为ERF、bHLH、C2H2、HD-ZIP和WRKY。Wu等(2014)利用转录组测序技术分析沙冬青在低温胁迫下差异表达基因的表达情况,结果发现,隶属于AP2/EREBP、NAC、WRKY、bHLH、C2H2、C3H、bZIP、GRAS和TCP等转录因子家族的差异表达基因在低温胁迫下显著上调表达。Chen等(2014)使用Solexa测序技术对4 ℃和-4 ℃的胡杨叶片进行转录组测序分析,结果发现AP2/ERF、WRKY、NAC、C3H、bHLH和HD-ZIP等转录因子家族的基因差异表达。本研究发现,牛心朴子响应低温胁迫的差异表达基因大多数隶属于MYB、C3H、bHLH、AP2-EREBP、C2H2、NAC、bZIP、CCAAT和WRKY等转录因子家族,其与植物抗性密切相关。尽管与沙冬青、胡杨逆境胁迫转录组研究结果在转录因子家族数量和排序上存在差异,但均鉴定到相同类型的抗逆相关功能转录因子家族成员,说明不同荒漠植物在受到低温胁迫时具有共同的响应调节机制,从而增强植物的耐受能力。
在提高作物抗性的分子研究中,与导入个别单一功能基因以提高某种抗性相比,导入转录因子基因提高作物抗性更有效,其原因是通过转录因子能促使多个功能基因发挥作用,从而实现供试植株性状获得综合改良的效果。Pang等(2013)研究发现,逆境胁迫处理下,转沙冬青AmDREB2C和AmNAC11基因的植株表现出较强的低温、干旱和高温耐受能力。这些强抗逆植物的相关研究初步探明轉录因子在抗逆中的功能,为改良植物抗逆性状提供了新基因和新思路。前人对牛心朴子的研究主要集中于单一功能基因鉴定,对具有调节功能的转录因子研究鲜见报道。马晓闻(2016)对牛心朴子进行干旱转录组测序分析,并克隆CCAAT家族的CkNF-YB1基因,结果发现该基因参与植物对渗透胁迫的应答反应,其过表达不仅会增强植物的抗氧化能力,减轻膜损伤程度,还能提高光合速率、降低自身水分散失,致使植物对逆境具有更强的耐受能力。此外,过表达拟南芥AtNF-YB1(Nelson et al.,2007)、玉米ZmNF-YB2(Nelson et al.,2007)、胡杨PdNF-YB7(Han et al.,2013)、大豆GmNFYA3(Ni et al.,2013)及青扦NF-YB3(Zhang et al.,2015)等基因,均能提高转基因植株对逆境的抗性。本研究从牛心朴子差异表达基因中鉴定出17个响应低温胁迫CCAAT转录因子家族成员,表明可能还存在大量与CkNF-YB1抗逆功能相似的其他家族成员基因。可见,CCAAT转录因子家族成员具有多种抗逆功能,可从综合抗逆性强的种质资源中发掘该家族抗逆基因。本研究为探究植物逆境胁迫调控机制提供了庞大的转录因子基因资源,随着研究的深入,将鉴定出更多植物抗逆相关的转录因子,为进一步培育农作物耐冷新品种打下理论基础。
4 结论
MYB、C3H、bHLH、AP2-EREBP、C2H2、NAC、bZIP、CCAAT和WRKY等转录因子家族成员在牛心朴子响应低温胁迫时发挥主导作用,同时各家族转录因子间存在共表达性或协同作用,通过复杂的转录调控网络发挥重要调节作用,进而提高牛心朴子对低温胁迫的耐受性。
参考文献:
陈翠云,赵昕,李新荣. 2012. 干旱胁迫下牛心朴子的渗透调节机制研究[J]. 中国沙漠,32(5):1275-1282. [Chen C Y,Zhao X,Li X R. 2012. Osmotic adjustment mechanism of Cynanchum komarovii under drought stress[J]. Journal of Desert Research,32(5):1275-1282.]
刘潮,韩利红,宋培兵,王德琴. 王海波,唐利洲. 2017. 桑树WRKY转录因子的全基因组鉴定及生物信息学分析[J]. 南方农业学报,48(9):1691-1699. [Liu C,Han L H,Song P B,Wang D Q,Wang H B,Tang L Z. 2017. Genome-wide identification and bioinformatics analysis for mulberry WRKY transcription factors[J]. Journal of Southern Agriculture,48(9):1691-1699.]
马晓闻. 2016. 牛心朴子草逆境胁迫转录组分析与CkNF-YB1,CkEDS1的功能研究[D]. 北京:中国农业大学. [Ma X W. 2016. Transcriptome analysis of stress response in Cynanchum komarovii and functional study of the genes CkNF-YB1 and CkEDS1[D]. Beijing:China Agricultural University.]
满玲娟,张桦,姚正培,宗兴风,李志强. 2019. 梭梭同化枝及其叶苞状虫瘿的转录组差异表达分析[J]. 南方农业学报,50(8):1657-1664. [Man L J,Zhang H,Yao Z P,Zong X F,Li Z Q. 2019. Differential expression analysis of transcriptome of assimilating branches and leafy-bracted galls of Haloxylon ammodendron[J]. Journal of Southern Agriculture,50(8):1657-1664.]
唐玉娟,黄国弟,罗世杏,周俊岸,莫永龙,李日旺,赵英,张宇,宋恩亮,宁琳. 2018. 芒果2个不同花芽分化时期转录组分析[J]. 南方农业学报,49(7):1257-1264. [Tang Y J,Huang G D,Luo S X,Zhou J A,Mo Y L,Li R W,Zhao Y,Zhang Y,Song E L,Ning L. 2018. Transcriptome of Mangifera indica L. in two different flower bud differentiation stages[J]. Journal of Southern Agriculture,49(7):1257-1264.]
王薇. 2009. 抗逆相關转录因子基因GmDREB3启动子分析及兼抗白粉病、条锈病小麦分子标记检测[D]. 乌鲁木齐:新疆农业大学. [Wang W. 2009. Promoter analysis of stress-related transcription factor gene,GmDREB3 and characterization of wheat line resistant to powdery mildew and stripe rust by molecular markers[D]. Urumqi:Xinjiang Agricultural University.]
张健,唐露,冉启凡,黄德均. 2019. 植物响应低温胁迫转录组测序研究进展[J/OL]. 分子植物育种. http://kns.cnki.net/kcms/detail/46.1068.S.20190418.0935.023.html. [Zhang J,Tang L,Ran Q F,Huang D J. 2019. Advances in RNA sequencing in response to low temperature stress in plants[J/OL]. Molecular Plant Breeding. http://kns.cnki.net/kcms/detail/46.1068.S.20190418.0935.023.html.]
周丽霞,曹红星. 2018. 低温胁迫下油棕WRKY转录因子基因的表达特性分析[J]. 南方农业学报,49(8):1490-1497. [Zhou L X,Cao H X. 2018. Expression characteristics of WRKY transcription factor genes in oil plam under low temperature[J]. Journal of Southern Agriculture,49(8):1490-1497.]
朱玉贤,李毅,郑晓峰,郭红卫. 2013. 现代分子生物学[M]. 第4版. 北京:高等教育出版社. [Zhu Y X,Li Y,Zheng X F,Guo H W. 2013. Modern molecular biology[M]. The 4th Edition. Beijing:Higher Education Press.]
Anders S,Huber W. 2010. Differential expression analysis for sequence count data[J]. Genome Biology,11(10):R106-R110.
Chen J H,Tian Q Q,Pang T,Jiang L B,Wu R L,Xia X L,Yin W L. 2014. Deep-sequencing transcriptome analysis of low temperature perception in a desert tree,Populus euphratica[J]. BMC Genomics,15(1):1-15.
Dang Z H,Zheng L L,Wang J,Gao Z,Wu S B,Qi Z,Wang Y C. 2013. Transcriptomic profiling of the salt-stress response in the wild recretohalophyte Reaumuria trigyna[J]. BMC Genomics,14(1):29.
Gong X X,Yan B Y,Hu J,Yang C P,Li Y J,Liu J P,Liao W B. 2018. Transcriptome profiling of rubber tree(Hevea brasiliensis) discovers candidate regulators of the cold stress response[J]. Genes & Genomics,40(11):1181-1197.
Grabherr M G,Haas B J,Yassour M,Levin J Z,Thompson D A,Amit I,Chen Z. 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology,29(7):644-652.
Han X,Tang S,An Y,Zheng D C,Xia X L,Yin W L. 2013. Overexpression of the poplar NF-YB7 transcription factor confers drought tolerance and improves water-use efficiency in Arabidopsis[J]. Journal of Experimental Botany,64(14):4589-4601.
Long Y,Zhang J W,Tian X J,Wu S S,Zhang Q,Zhang J P,Dang Z H,Pei X W. 2014. De novo assembly of the de-sert tree Haloxylon ammodendron(CA Mey.) based on RNA-Seq data provides insight into drought response,gene discovery and marker identification[J]. BMC Genomics,15(1):1111-1122.
Moriya Y,Itoh M,Okuda S,Yoshizawa A C,Kanehisa M. 2007. KAAS:An automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Research,35:182-185.
Nelson D E,Repetti P P,Adams T R,Creelman R A,Wu J,Warner D C,Anstrom D C,Bensen R J,Castiglioni P P,Donnarummo M G,Hinchey B S,Kumimoto R W,Maszle D R,Canales R D,Krolikowski K A,Dotson S B,Gutterson N,Ratcliffe O J,Heard J E. 2007. Plant nuclear factor Y(NF-Y) B subunits confer drought tolerance and lead to improved corn yields on water-limited acres[J]. Proceedings of the National Academy of Sciences of the United States of America,104(42):16450-16455.
Ni Z Y,Hu Z,Jiang Q Y,Zhang H. 2013. GmNFYA3,a target gene of miR169, is a positive regulator of plant tole-rance to drought stress[J]. Plant Molecular Biology,82(1-2):113-129.
Pang T,Ye C Y,Xia X L,Yin W L. 2013. De novo sequen-cing and transcriptome analysis of the desert shrub,Ammopiptanthus mongolicus,during cold acclimation using Illumina/Solexa[J]. BMC Genomics,14(1):488-503.
Tan H Q,Huang H T,Tie M M,Tang Y,Lai Y S,Li H X. 2016. Transcriptome profiling of two asparagus bean(Vigna unguiculata subsp. sesquipedalis) cultivars differing in chilling tolerance under cold stress[J]. PLoS One,11(3):e0151105.
Wang J M,Yang Y,Liu X H,Huang J,Wang Q,Gu J H,Lu Y M. 2014. Transcriptome profiling of the cold response and signaling pathways in Lilium lancifolium[J]. BMC Genomics,15(1):203.
Wang M,Zhang X N,Liu J H. 2015. Deep sequencing-based characterization of transcriptome of trifoliate orange (Poncirus trifoliata(L.) Raf.) in response to cold stress[J]. BMC Genomics,16(1):555.
Wang Q H,Li F G,Zhang X,Zhang Y A,Hou Y X,Zhang S R,Wu Z X. 2011. Purification and characterization of a CkTLP protein from Cynanchum komarovii seeds that confers antifungal activity[J]. PLoS One,6(2):e16930.
Wei S J,Du Z L,Gao F,Ke X,Li J X,Liu J X,Zhou Y J. 2015. Global transcriptome profiles of ‘Meyer Zoysiagrass in response to cold stress[J]. PLoS One,10(6):e0131153.
Wu Y Q,Wei W,Pang X Y,Wang X F,Zhang H L,Dong B,Xing Y P,Li X G,Wang M Y. 2014. Comparative transcriptome profiling of a desert evergreen shrub,Ammopiptanthus mongolicus,in response to drought and cold stresses[J]. BMC Genomics,15(1):671.
Xu W R,Li R M,Zhang N B,Ma F L,Jiao Y T,Wang Z P. 2014. Transcriptome profiling of Vitis amurensis,an extremely cold-tolerant Chinese wild Vitis species,reveals candidate genes and events that potentially connected to cold stress[J]. Plant Molecular Biology,86(4-5):527-541.
Yang Y W,Chen H C,Jen W F,Liu L Y,Chang M C. 2015. Comparative transcriptome analysis of shoots and roots of TNG67 and TCN1 rice seedlings under cold stress and following subsequent recovery:Insights into metabo-lic pathways,phytohormones,and transcription factors[J]. PLoS One,10(7):e0131391.
Zhang T,Zhang D,Liu Y J ,Luo C B,Zhou Y N,Zhang L Y. 2015. Overexpression of a NF-YB3 transcription factor from Picea wilsonii confers tolerance to salinity and drought stress in transformed Arabidopsis thaliana[J]. Plant Physiology and Biochemistry,94:153-164.
Zhao Z G,Tan L L,Dang C Y,Zhang H,Wu Q B,An L Z. 2012. Deep-sequencing transcriptome analysis of chilling tolerance mechanisms of a subnival alpine plant,Chorispora bungeana[J]. BMC Plant Biology,12(1):222-222.
Zheng Y,Jiao C,Sun H,Rosli H G,Pombo M A,Zhang P,Zhao P X. 2016. iTAK:A program for genome-wide prediction and classification of plant transcription factors,transcriptional regulators,and protein kinases[J]. Molecular Plant,9(12):1667-1670.
(責任编辑 陈 燕)
