实时PCR 技术在羊亚科肉类鉴定中的应用
2020-07-04梁恒兴段庆梓
尚 柯,梁恒兴,张 彪,段庆梓,王 巍,张 玉
(成都市食品药品检验研究院,四川 成都 610045)
羊肉作为一种优良的畜肉,属于高蛋白、低脂肪、低胆固醇类营养保健食品,有着悠久的食用历史[1]。我国的肉类生产以猪肉为主,随着生活水平的提高,猪肉产量的比例逐年下降,而牛、羊等高价格肉类产量逐年上升,在2015年羊肉产量达到了肉类产量的7%[2]。目前,我国的羊肉市场以绵羊、山羊为主,同时部分地区,如贵州、云南等地也生产与食用麻羊等[3]。羊肉中的赖氨酸、精氨酸、组氨酸含量明显高于猪、牛、鸡等肉类,且胆固醇含量仅有猪、牛的1/3甚至1/4,同时羊肉易于消化吸收,深受消费者喜爱[4-5]。
近年来,政府及消费者对肉及肉制品的产品质量和食用安全越来越重视,但不法商贩在肉制品生产(加工)中进行肉质掺伪、造假的现象仍屡有发生,这一掺假造假现象在羊肉制品中表现较为突出[6]。掺假的方式越来越多,形式越来越复杂[7]。目前,肉制品的掺假主要表现为原料肉掺假、组织替换等[8],鸭肉冒充羊肉事件、欧洲马肉门事件、巴西肉类丑闻、阿胶用猪皮制作、欧洲鱼肉事件层出不穷[9-10],“挂羊头卖狗肉”的肉类掺假掺杂事件近年来屡被曝光。这些食品问题严重地侵害了消费者的合法利益,给肉制品食用安全带来潜在危害,同时也对民族宗教信仰带来较大的风险[11]。
DNA杂交、免疫扩散和等电点聚焦等方法曾经被用于肉类品种鉴定[12],但这些方法大都存在成本高、基质单一、对检测对象要求高、工作量大、无法检测深加工肉类等缺点[13]。随着分子生物学技术的发展,目前从基因角度检测物种的真实属性已经成为主流检测技术,规避了性状检测、蛋白检测、化学成分检测所存在的诸多局限[14]。Nataliav等[15]利用鸡尾酒引物实现了物种的通用扩增,采用条形码技术鉴定肉类的成分;Guan Feng等[16]采用八重聚合酶链式反应(polymerase chain reaction,PCR)方法,可区分山羊、绵羊、鹿、水牛、牛、牦牛、猪和骆驼;冯海永等[17]采用七重PCR检测方法,可区分猪、牛、绵羊、山羊、鸡、马和牦牛7 种动物;杨冬燕等[18]对国外现有方法进行优化与完善,建立了猪、马、牛、鸭多重实时PCR(real-time PCR)检测体系,用于检测羊肉制品掺假情况;熊蕊等[19]采用限制性片段长度多态性PCR技术,可从牛羊肉中检测猪源性成分。但这些方法均存在一定的局限性,普通PCR方法及DNA条形码技术由于操作繁琐,准确度与灵敏度不高,对样本及实验设备要求高,易造成污染等问题,不适于检测技术发展的需要[20];目前已建立的实时荧光检测技术大多仅针对某一种羊肉进行检测,无法确定羊肉的具体种类[21]。
为提高肉制品中羊源性成分检测方法的可操作性,建立建全羊肉具体种类的检测方法,本实验针对山羊、绵羊、麻羊3 种食用羊的基因序列,分别建立绵羊、山羊、羊亚科的专属性real-time PCR检测方法,对羊肉制品中的羊源性成分进行检测,以期为监管部门提供技术手段与数据支持。
1 材料与方法
1.1 材料与试剂
本实验所用样品包括确定基原的对照物种20 种,以及市场采购样品20 种。样品采自养殖厂、研究所、农贸市场、电商、超市等。所有样品通过合成引物[22-23]进行线粒体基因测序验证,保证样品的真实性。样品的具体信息见表1。
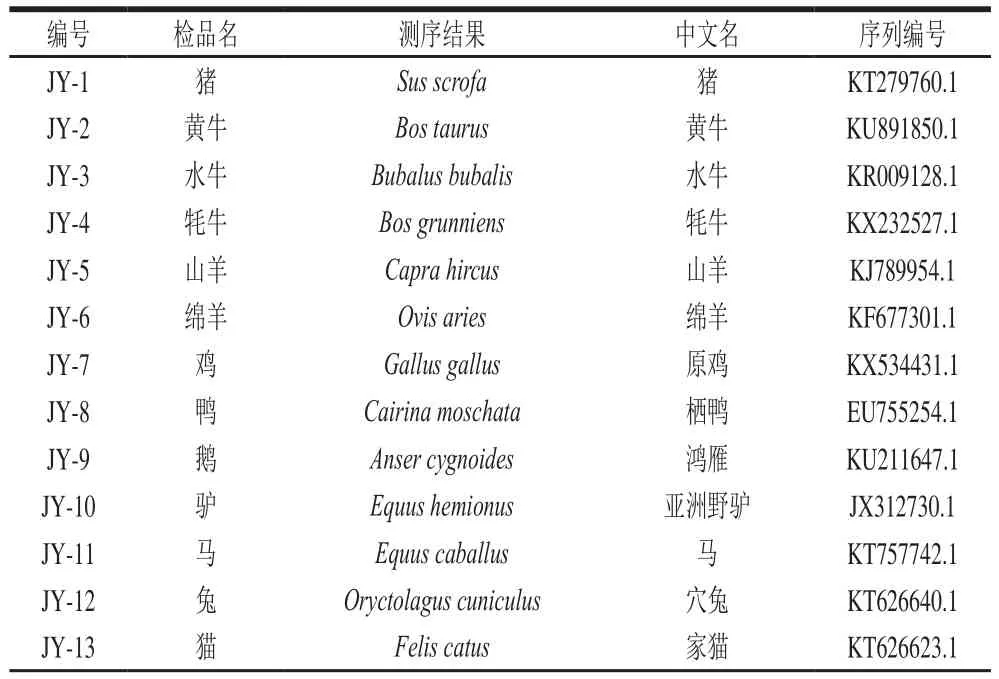
表1 样品信息Table 1 Detailed information about the samples tested in this study
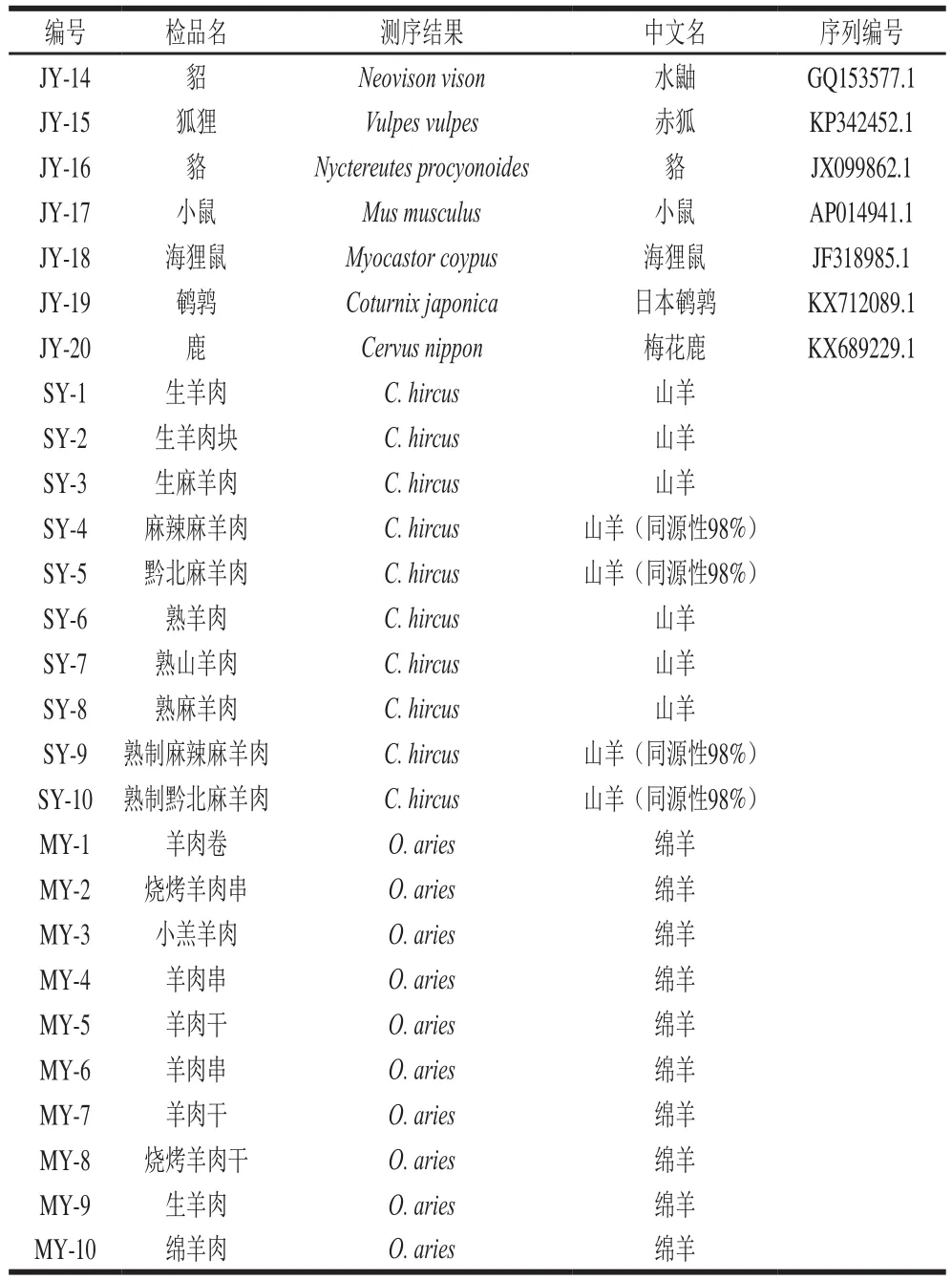
续表1
动物基因组提取试剂盒 上海捷瑞生物工程有限公司;实时荧光PCR扩增试剂盒:Bio-Rad SsoAdvancedTMUniversal Probes Supermix、Tiangen SuperReal PreMix(Probe)、TransStart®Probe qPCR SuperMix、TaKaRa Premix ExTaqTM(Probe qPCR)。
1.2 仪器与设备
Applied Biosystems Veriti II PCR仪 美国Thermo Fisher Scientific公司;CFX96 Touch实时荧光PCR仪、PowerPac电泳仪 美国Bio-Rad公司;GelDoc-It凝胶成像仪 美国UVP公司;Centrifuge 5427 R高速冷冻离心机 德国Eppendorf公司;ME2002E电子天平 瑞士Mettler Toledo公司;MK3涡旋振荡器 德国IKA公司;P330核酸蛋白定量仪 德国Implen公司;MM400行星球磨仪 德国Retsch公司;Milli-Q Academic实验室超纯水仪美国Millipore公司;Thermo-Shaker BG-100干式恒温器杭州瑞诚仪器有限公司;SW-CJ-FD型单人单面净化工作台 苏州净化设备有限公司。
1.3 方法
1.3.1 引物及探针
将GenBank数据库中山羊、绵羊以及其他18 种动物的cyt b基因序列运用生物软件进行比对分析,选择山羊(EU130780.1)、绵羊(KP228916.1)、羊亚科(MF503655.1)的基因序列进行引物和探针的设计。引物及探针由生工生物工程(上海)股份有限公司合成。探针荧光基因为FAM,淬灭基团为BHQ1,最终确定的引物组具体序列见表2。
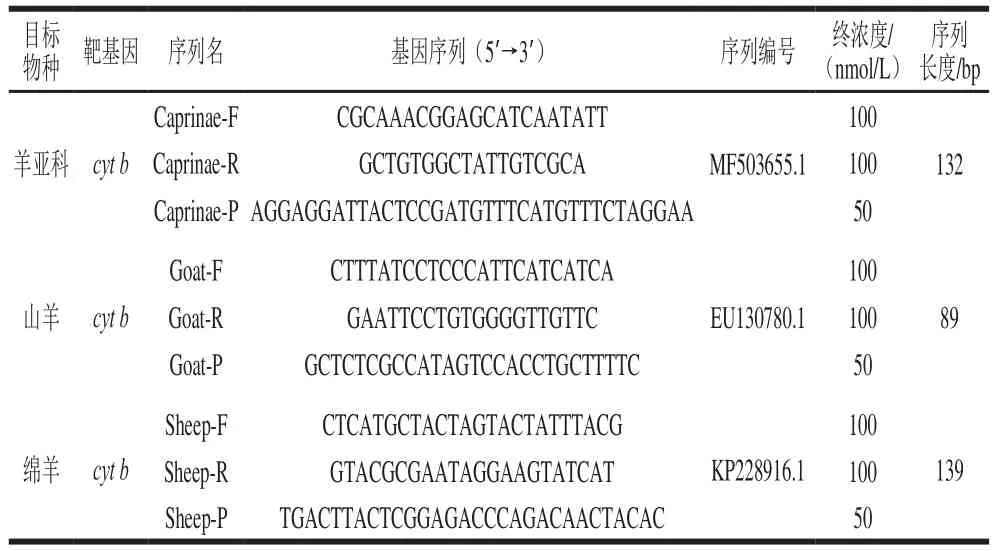
表2 引物信息Table 2 Sequences of PRC primers and probes
1.3.2 real-time PCR方法建立
按照试剂盒说明书对样品进行基因组提取,用核酸蛋白定量仪确定DNA提取的效率与纯度,进行OD260nm/OD280nm值测定,测得值均在1.7~2.0之间,表明可用于PCR实验。real-time PCR包括10 µL 2×SsoFast Advanced Supermix real-time PCR预混液、DNA模板、可变引物和探针量以及去离子水,其体积为20 µL。通过改变引物浓度、探针浓度和退火温度进行方法的筛选与建立。引物终浓度分别为25、50、100 nmol/L和150 nmol/L,探针终浓度分别为5、12.5、25 nmol/L和50 nmol/L,退火温度分别为56、58、60、62 ℃和64 ℃。
1.3.3 特异性实验
以20 种基原动物基因组DNA(JY-1~JY-20)为模板,用建立的real-time PCR方法进行扩增,评价特异性。阳性、阴性和空白对照在实验中同步进行,进行质量控制。
1.3.4 灵敏度实验
对模板DNA进行倍比稀释,质量浓度设定为10-4、10-3、10-2、10-1、100、101ng/μL。作为反应模板进行扩增反应,确定模板DNA质量浓度范围与Ct值线性关系,同时根据原理,参考文献[24]建立扩增效率计算公式:扩增效率/%=(lg稀释倍数/lg2)/|标曲斜率|×100,并建立本方法的判定原则。
1.3.5 检出限实验
分别将山羊、绵羊肉样品以1%、0.1%、0.01%三个水平掺入鸡肉样品中,充分混匀后,提取基因组DNA,作为检测模板,分别使用山羊、绵羊、羊亚科检测体系进行肉样水平检出限检测。
1.3.6 试剂一致性考察
考察不同公司实时荧光预混液对检测结果的一致性,分别进行常见品牌的实时荧光预混液比对实验,确定不同试剂盒对检测结果的影响。检测体系仅替换real-time PCR预混液,其他条件均一致。
1.3.7 加工肉制品Ct值变化
确定肉制品影响DNA质量的主要是杀菌环节。目前,国内肉制品企业的杀菌方式主要为高温杀菌、巴氏杀菌、辐照杀菌等。据此,采用对DNA影响最大的高温杀菌方式对样品进行处理,确定杀菌工艺对DNA质量的影响,以及本方法是否适用于加工后的肉制品。联系肉制品生产企业获取加工前绵羊、山羊生肉原料及最终成品肉制品,提取DNA后,依照建立的方法分别进行检测。
1.3.8 方法适用性考察
对不同产地、不同基质的肉及肉制品(表1)进行检测,确定3 种检测方法的适用性。1.3.9 方法验证
为了保证方法的一致性和可重复性,起草组制定验证工作方案,邀请了山东省食品药品检验研究院、中国检验检疫科学研究院、北京市食品安全监控和风险评估中心、河北省食品检验研究院、国家副食品质量监督检验中心5 家检测机构进行方法验证,由验证单位独立完成从DNA提取到结果判定的全过程验证实验。
1.4 数据统计及图表绘制
使用Bio-Rad CFX Manager 3.0.1224.1015软件制作real-time PCR结果图谱,使用Microsoft Excel 2007软件对real-time PCR数据进行统计分析,并绘制相关的数据分析图表。
2 结果与分析
2.1 引物探针的设计筛选

图1 山羊目标序列比对分析Fig. 1 Target sequence alignment analysis of Capra
根据引物设计原则,利用DNAStar软件设计山羊、绵羊、羊亚科通用的引物探针,并对各物种的引物及探针进行序列高级结构分析比对,以确保其高度的特异性,防止出现二聚体、茎环及发卡等影响检测效率的结构。具体信息见图1~3。

图3 羊亚科目标序列比对分析Fig. 3 Target sequence alignment analysis of Caprinae
2.2 real-time PCR方法建立

表3 real-time PCR体系筛选与建立(Ct值)Table 3 Screening and establishment of real-time fluorescence PCR
分别进行引物浓度筛选、探针浓度筛选和退火温度筛选,具体信息见表3。最终确定的real-time PCR方法为:PCR预混液(2×)10 μL,5’端、3’端引物(10 μmol/L)各0.2 μL,探针(10 μmol/L)0.1 μL,DNA模板(1~100 ng/μL)1 μL,用无菌双蒸水补足20 μL。PCR扩增程序为:95 ℃预变性5 min;35 个循环(95 ℃变性15 s,58 ℃退火及延伸1 min,收集荧光)。
2.3 特异性结果
特异性实验结果显示,相应动物源性成分有典型扩增曲线且Ct值均小于25,其他动物源性成分均未检出,实验结果理想,均能达到检测要求,其中羊亚科源性检测可同时检测绵羊、山羊、麻羊。具体检测情况见图4。
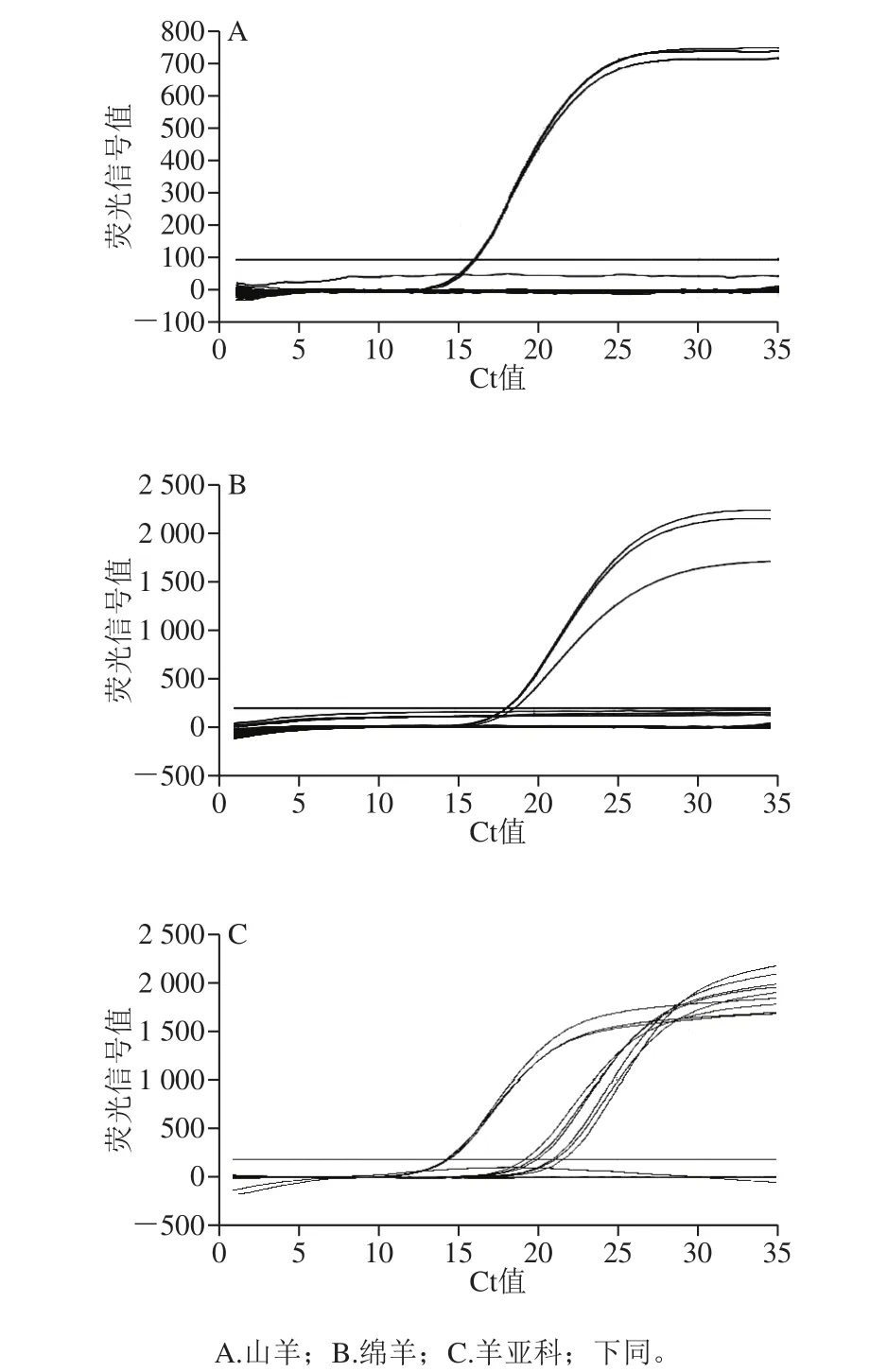
图4 特异性检测图谱Fig. 4 Specificity evaluation
2.4 灵敏度结果
在Ct值小于或者等于30时,灵敏度均可达10-3ng/μL,满足多种检测要求,在线性范围内均有良好的线性关系,山羊、绵羊、羊亚科检测方法的R2分别为0.999、0.997、0.999,扩增效率分别为99.37%、92.48%、96.32%。具体检测情况见图5。
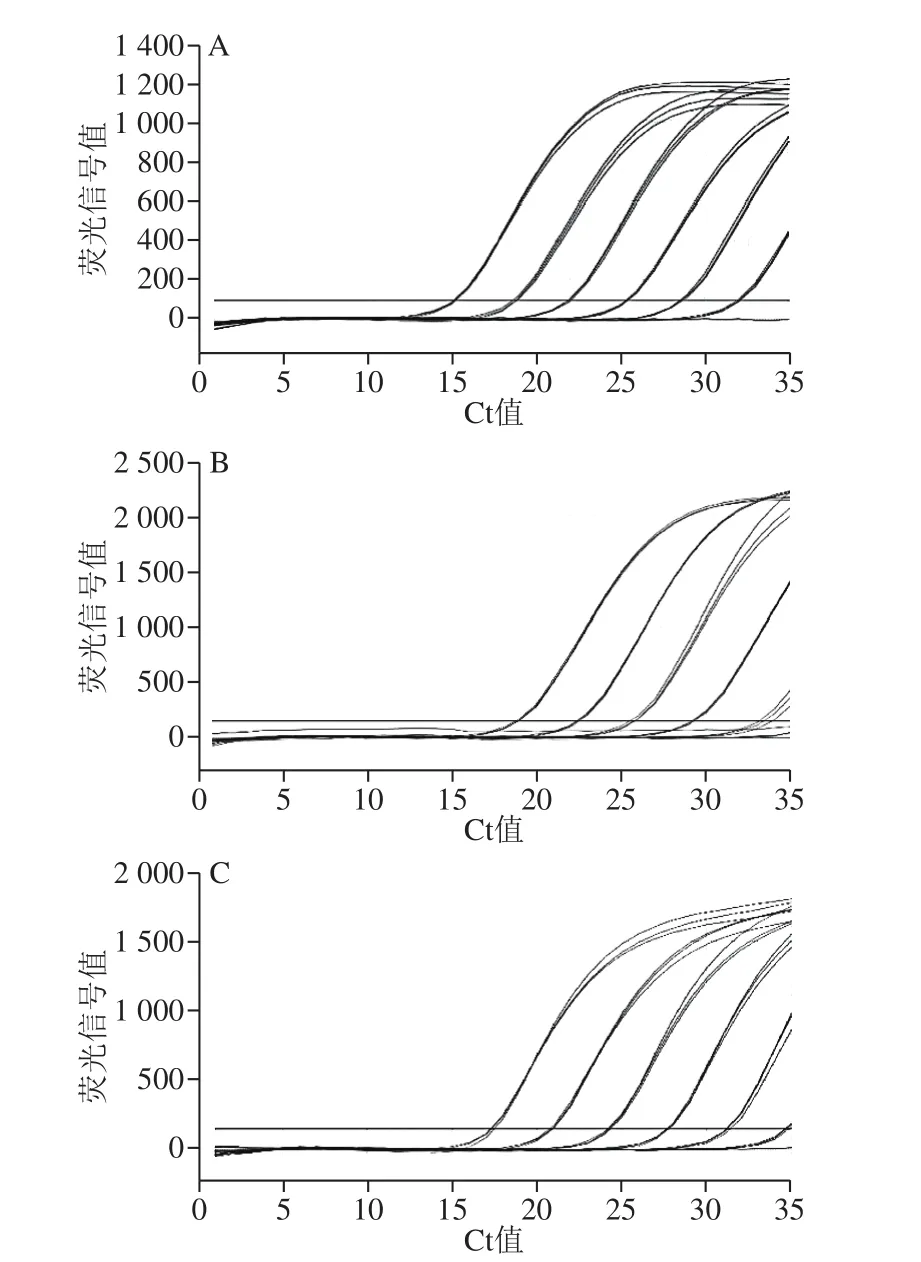
图5 灵敏度实验图谱Fig. 5 Sensitivity evaluation
2.5 检出限结果
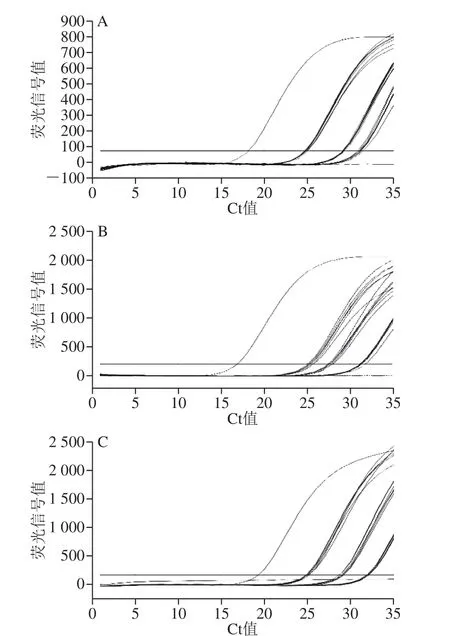
图6 检出限实验图谱Fig. 6 Detection limit evaluation
图6 及表4显示,在鸡肉中掺入1%、0.1%的山羊、绵羊肉时,本方法检测Ct值均小于30。而在0.01%的掺入比例实验时,相应Ct值接近甚至超过30 个循环,对其判定可能会出现偏差。根据以上验证结果,本方法的检出限为0.1%。
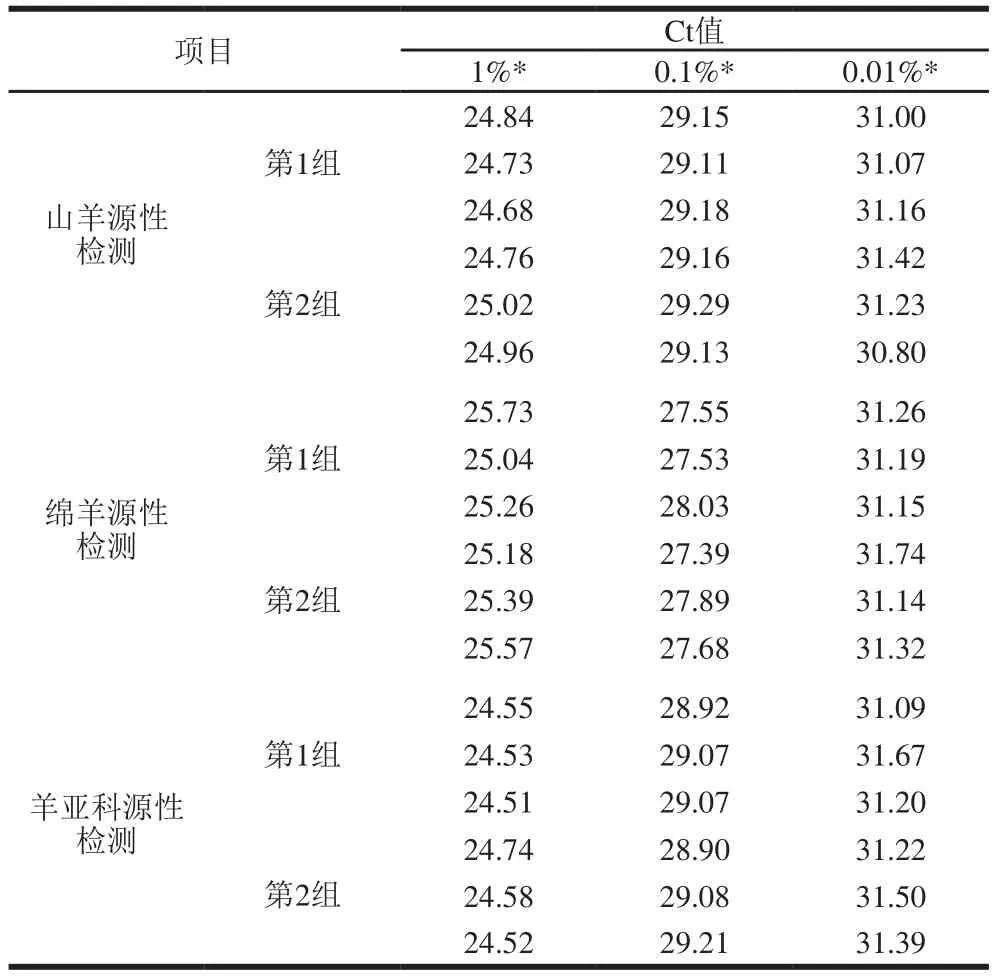
表4 检出限检测结果Table 4 Detection limit validation
2.6 试剂一致性分析
选择Bio-Rad SsoAdvancedTMUniversal Probes Supermix、Tiangen SuperReal PreMix(Probe)、TransStart®Probe qPCR SuperMix、TaKaRa Premix ExTaqTM(Probe qPCR) 4 种real-time PCR预混液对方法进行重复性考察,结果显示,不同试剂表现出一致的检测结果,表明本方法的试剂一致性较好,常用实时荧光预混液均适用于本方法的检测。具体情况见图7及表5。
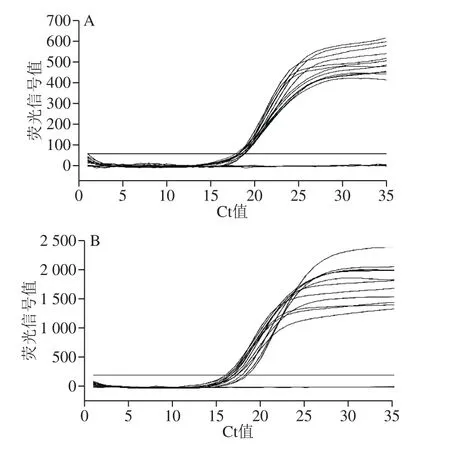
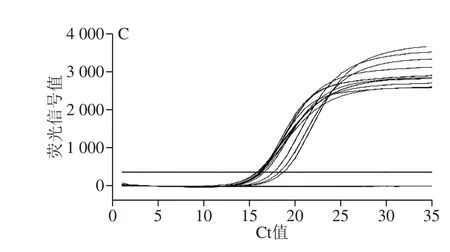
图7 试剂一致性实验图谱Fig. 7 Reagent consistency evaluation
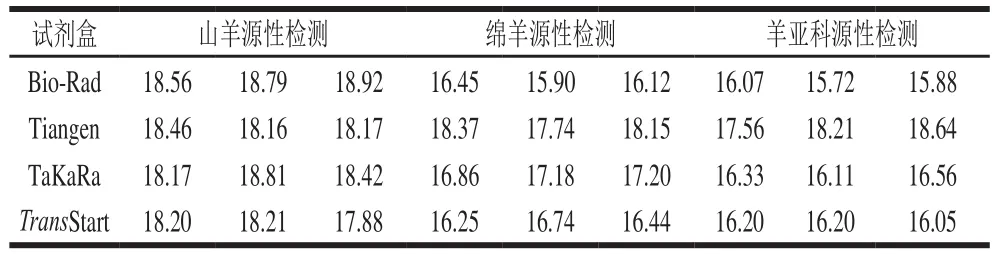
表5 试剂一致性结果(Ct值)Table 5 Reagent consistency validation
2.7 加工肉制品Ct值变化情况分析
如图8所示,原料肉与最终产品检测结果存在一定的差异,但加工后的成品与原料肉相比Ct值差异在3以内,且Ct值均在30以下,有典型扩增曲线,不影响结果的判断。因此,本方法适用于生鲜肉及肉制品的检测。
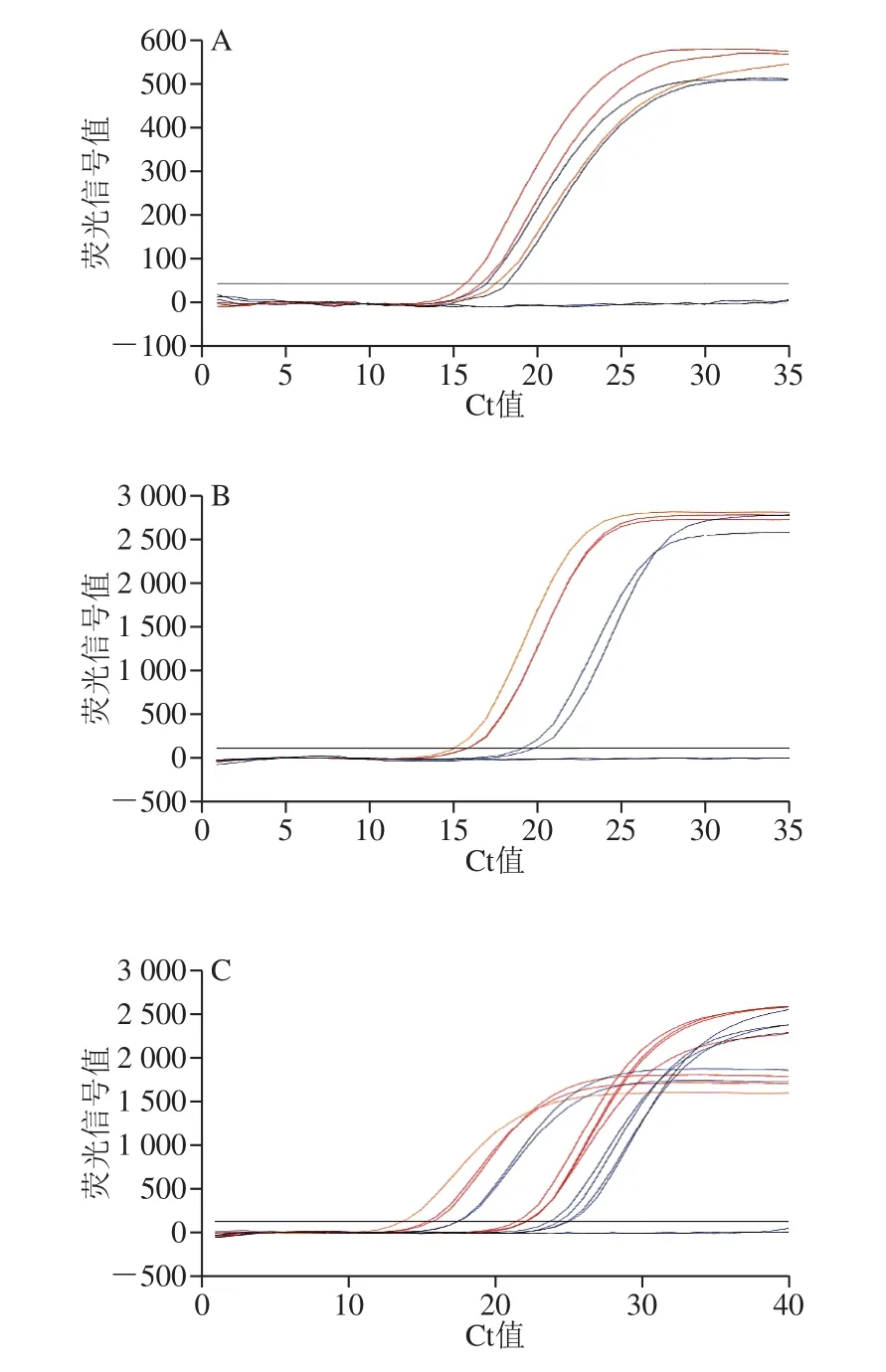
图8 生鲜肉与加工肉制品real-time PCR结果比较Fig. 8 Comparison of real-time PCR results of raw meat and processed meat
2.8 方法适用性分析
山羊、绵羊、羊亚科源性成分检测的10 个样品3 个浓度水平Ct值均小于30,且具有典型扩增曲线,适用性实验结果符合预期,满足检测要求,见图9及表6。
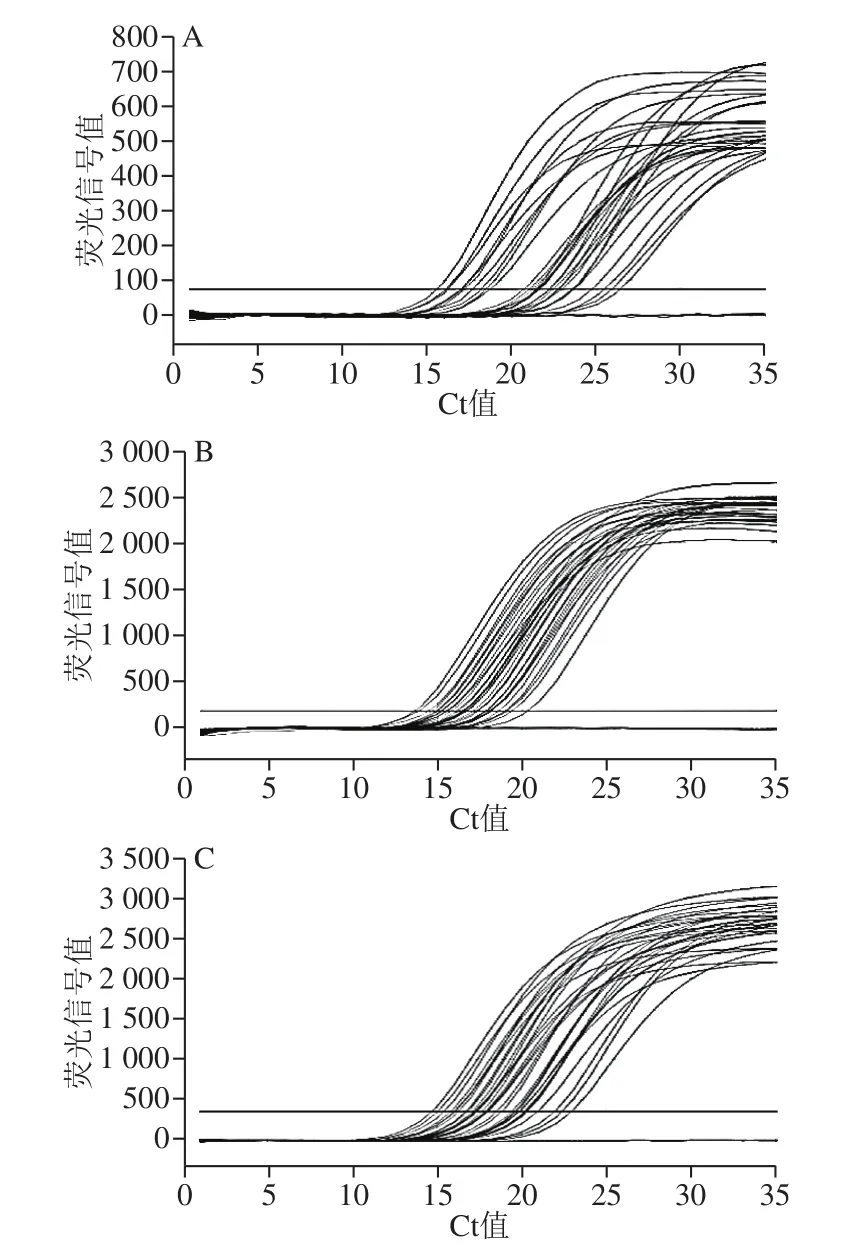
图9 适用性实验图谱Fig. 9 Applicability evaluation

表6 适用性检测结果(Ct值)Table 6 Applicability validation
2.9 方法验证
由成都市食品药品检验研究院提供验证样品580 个,其中特异性验证样品100 个,适用性验证样品300 个,灵敏度验证样品90 个,检出限验证样品90 个;由验证单位独立完成从DNA提取到结果判定的全过程验证试验。5 家验证单位均获得了满意的验证结果,验证汇总结果见表7。
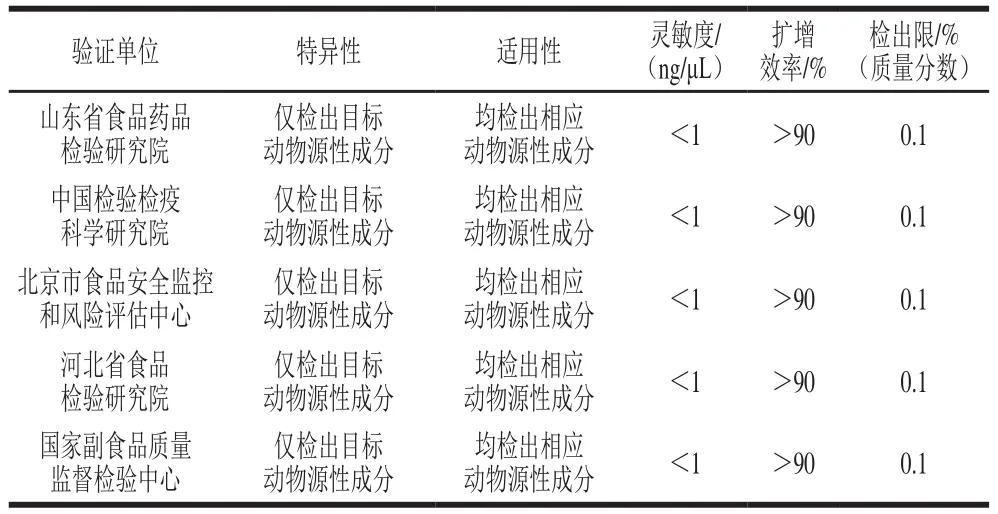
表7 验证结果汇总Table 7 Summary of validation results from five domestic authoritative institutions
3 结 论
鉴于肉类掺假现象的出现,基于DNA的肉类鉴定方法已成为食品真实性鉴定的首选方法[25]。本研究开发并验证了羊亚科3 种real-time PCR检测普通肉制品的方法,分别为羊亚科通用检测方法、山羊检测方法及绵羊检测方法。这些方法特异性强、灵敏度高、适用性强,可用于羊肉类原料及加工肉的鉴别,以及肉制品中羊源性成分的检测。3 种方法的反应体系及检测步骤一致,可在一次real-time PCR实验中同步完成3 种方法的检测。结果表明,该方法是一种高效、灵敏、特异、高实用性的羊亚科专属性鉴别方法。
本方法以real-time PCR探针法为基础,从基因水平对肉类DNA进行检测。real-time PCR技术利用荧光基团的淬灭与激发[26],在反应过程中可以通过荧光信号反映DNA扩增情况,在实验过程中对反应情况进行实时监测[27],扩增反应结束,直接获得检测结果。整个过程闭管操作,不需要进行酶切、染色、电泳、测序等传统PCR方法所需的繁琐步骤,缩短了检测时间,提高了检测效率,减少了后续实验的污染以及染色试剂对操作人员造成的潜在危害[28]。
本方法的成功依赖于以下3 个方面:适当的靶基因位点、专属性极强的引物和探针以及DNA真实性考察。首先,根据在动物物种中高度保守且母体遗传的cyt b基因[29],并对20 个物种进行了目的基因序列比对,以确保该方法的有效性。其次,用DNAStar v7.1.0.44软件[30]设计了针对目标物种特定DNA序列的引物和探针,并对高级结构进行分析,以确保引物与探针不存在茎环及发夹等影响扩增效率的结构[31]。通过猪、牛、绵羊、鸡、鸭、鹅等20 个物种的特异性鉴定,验证了所有引物和探针的特异性,保证了方法的特异性。最后,本实验所用的动物肉类样品和PCR产物均经过测序验证,以确保样品的真实性[32],整个方法依据分子生物学标准流程进行设计、优化与验证[33]。
本研究的目的是建立一种快速、高通量的real-time PCR方法,以鉴定市场上销售的肉制品的羊源性成分。因此,在建立了羊亚科3 种检测方法后,本实验邀请了5 家中国权威的检测机构对该方法进行系统、科学地验证。在验证之前,实验设计了方法验证的内容和步骤,包括方法的特异性、适用性、灵敏度、扩增效率和检出限。联合验证的结果表明,该方法的特异性满足预期,仅能扩增目标物种,不能扩增其他非目标物种;方法的扩增效率达90%以上,检出限均可达0.1%;此外,对真实的市售产品进行检测,检测准确率达100%,方法适用性满足检测要求。鉴于这些方法是基于real-time PCR原理进行开发,希望增加定量检测方法,为肉制品的真实性鉴定和掺假提供了更加准确和科学的检测方法,为监管部门提供技术标准与检测依据。
