谷子SBP蛋白基因家族的鉴定及表达分析
2020-07-01曹晓宁王海岗王君杰乔治军刘思辰
宋 健 曹晓宁 王海岗 陈 凌 王君杰 乔治军 刘思辰
(1山西大学生物工程学院,山西 太原 030006;2山西省农业科学院经济作物研究所,山西 汾阳 032200;3山西省农业科学院农作物品种资源研究所,山西 太原 030031)
Squamosa 启动子结合蛋白(squamosa promoter binding protein,SBP)是植物生长发育过程中非常重要的一类转录因子,主要参与花的形成[1-6]、叶的发育[1-2,6]、植物育性[2-4]、发育阶段的转变[2,6]、赤霉素(gibberellins,GA)响应[2,4]、光周期响应[2,4]、果实成熟[2-3,6]、调控器官大小和产量[3-5]、应对胁迫应答[1,5-6]等多个方面。SBP 蛋白具有一个高度保守的DNA 结合区域,该区域由80 个左右的氨基酸残基组成。Yamasaki 等[7]通过核磁共振法,在AtSPL4 和AtSPL7 蛋白中均发现2 个锌指结构,这2 个锌指结构均由8 个氨基酸残基和Zn2+组成,8 个氨基酸残基包括组氨酸和半胱氨酸。2 个锌指构成分别为(Cys3HisCys2HisCys)和(Cys6HisCys),Zn2+分别结合在前后4 个氨基酸残基上。
Huijser 等[8]最早在金鱼草(Antirrhinum majus)中发现SBP基因。目前已经在水稻、小麦、玉米、大豆、番茄、高粱等作物中分离鉴定出SBP基因。SBP基因家族成员较多,其中,在拟南芥中共有17 个SPL基因[9-11];水稻中有20 个OsSBPs基因,且其SBP基因主要在花和愈伤组织中表达[10-11];二倍体小麦具有19个SPL基因,六倍体小麦中国春中具有58 个SPL基因[12]。研究表明,水稻[13]、小麦[14-16]中SBP基因具有调控光合产物积累,增加产量的作用。近些年,对玉米[17-19]、大豆[20-22]、番茄[23]、高粱[24-25]、水稻[26-28]、拟南芥[29]等作物中的SBP基因的研究显示,SBP基因具有激素和逆境响应元件,说明SBP基因是与抗旱和耐逆相关的一类重要基因。
谷子(Setaria italica)是很强的抗旱耐瘠薄作物,其富含类胡萝卜素、维生素B1 和维生素B2,具有非常高的营养价值,是目前杂粮作物中播种面积较大的作物之一,也是干旱丘陵地区种植的首选农作物之一。但由于北方降雨量稀少,尤其在谷子生长发育的关键时期缺水少雨,导致谷子的生长发育受阻,严重影响谷子产量。因此,挖掘与谷子抗旱性和耐逆性相关的基因,通过分子育种,提高谷子的抗旱耐逆性对农业生产具有十分重要的意义。虽然已有各种作物的SBP基因研究成果,但鲜见有关谷子SBP基因功能的研究报道。谷子全基因组测序的完成,为谷子功能基因的挖掘研究提供了有利支撑。本试验以谷子基因组数据为基础,采用生物信息学方法,对谷子SBP 基因家族进行鉴定和分析;同时结合RT-qPCR 分析谷子SBP基因在PEG 胁迫及脱落酸(abscisic acid,ABA)诱导后的表达情况,以期为进一步研究谷子SBP基因的功能奠定基础。
1 材料与方法
1.1 试验材料
以谷子品种豫谷1号为试验材料,由河南省安阳农科所选育并提供。
1.2 谷子SBP 基因家族的鉴定
在JGI(Setaria italicav2.2)数据库下载谷子的蛋白序列,用文献中已公布的拟南芥、水稻和玉米的SBP蛋白序列作为种子文件,在谷子蛋白数据库中搜索BLAST 具有SBP 结构域的侯选序列;同时使用HMMsearch 搜索谷子基因组中具有SBP 保守结构域的蛋白序列。将查找到的所有谷子SBP 蛋白序列,使用 CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)和Pfam(http://pfam.xfam.org/search)分析是否具有保守的SBP 蛋白结构域,删除比对结果保守结构域小于50%的SBP 序列,最终确定谷子的SBP 蛋白家族成员。下载对应的核酸序列,对鉴定结果进行基因注释。使用ProtParam tool(https://web.expasy.org/protparam/)分析谷子SBP 蛋白的氨基酸组成、等电点、分子量、亲疏水性指数等。使用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP-4.1/)和PSORT Prediction(http://psort1.hgc.jp/form.html)分别进行信号肽和亚细胞定位的预测。
1.3 谷子SBP 蛋白系统进化树及SiSBPs 基因结构分析
将鉴定出的谷子SBP 氨基酸序列通过MEGA 6中MUSCLE 进行序列比对(参数默认)。同时利用phylogeny 中的NJ 邻接(neighbor joining,NJ)法构建发育树,Bootstrap 参数设定为1 000[30],Gaps 处理方法为成对删除。在JGI 官网下载谷子Setaria italicav2.2中的SBP基因组序列和CDS 序列,使用ClustalX 对19个SiSBPs 蛋白序列进行比对,将比对结果保存为nwk格式。利用GSDS 2.0[31]的Sequence(FASTA)Format绘制SBP基因结构图。
1.4 不同作物间SBP 蛋白的系统发育分析
分别下载拟南芥、水稻、高粱、玉米的SBP 蛋白家族,通过Muscle 对各物种的SBP 蛋白进行完全序列比对,使用MEGA6.0 中的NJ 法(bootstrap 参数设定为1 000)构建系统发育树[30]。
1.5 基因定位
在NCBI 谷子数据库下载谷子的染色体组数据,在JGI(Setaria italicav2.2)数据库确定谷子SBP 家族成员的位置信息及其在染色体上的分布情况。并使用MapGene2Chromosome 绘制SiSBPs基因在谷子9 条染色体上的分布图。
1.6 启动子分析
使用Plantscare 对SiSBPs进行启动子分析。在phytozome 中下载谷子(Setaria italicav2.2)的全基因组序列和GFF3 文件,然后通过TBtools 批量提取谷子基因组序列,最后利用TBtools 的Fasta Extractor 将SBP序列全部提取出来,分析SiSBPs编码区起始密码子上游2 000 bp 的启动子情况。
1.7 胁迫处理试验
将饱满的谷子种子放入含有均衡营养液的保湿水培盒并置于通风较好的30℃光照培养箱中进行育苗(16 h 光照+8 h 黑暗),幼苗长至4~5 叶时分别进行10% PEG 6000 和100 μmol·L-1ABA 溶液胁迫处理,分别于胁迫0、2、4、6、12、18 h 时,取谷子幼苗根、茎、叶,立即放入液氮速冻,于-80°冰箱保存,用于提取RNA。
1.8 谷子幼苗根茎叶总RNA 提取及反转录
分别将不同处理的根、茎、叶组织充分磨碎,使用植物总RNA 提取试剂盒提取RNA,琼脂糖凝胶电泳检测RNA 完整性。将完整的RNA 总量统一定为1 000 ng,使用cDNA 第一链试剂盒对总RNA 进行反转录。所用试剂盒均为天根生化科技北京有限公司产品。
1.9 PEG 6000 胁迫和外源ABA 诱导后SiSBPs 基因的RT-qPCR 分析
根据荧光定量引物设计的原则,使用Premier 5 设计荧光定量引物,产物片段设定为100~300 bp 之间,将设计好的引物先通过琼脂糖凝胶电泳进行特异性筛选,根据结果选用具有特异性的RT-qPCR 引物。以25S为内参基因。使用Applied Biosystems QuantStudio 6 & 7 Flex Real-Time PCR Software,对经过10% PEG 6000 和ABA 胁迫后幼苗不同组织部位中SiSBP15、SiSBP19 的表达量进行分析。反应体系为20 μL,包括1 μL cDNA、0.6 μL 上游引物、0.6 μL 下游引物、10 μL mix、7.8 μL H2O。程序设定为:95℃预变性15 min;95℃变性10 s,60℃退火25 s,共40 个循环。运行结束后将结果以Excel 格式导出。
2 结果与分析
2.1 SiSBPs 基因家族成员鉴定结果
通过NCBI Blast 搜索出19 个SiSBPs基因,通过Hmmer search 查找出42 个可能的SBP 家族成员,利用CDD 去除不含有SBP 蛋白保守域的成员,最终确定有19 个SiSBPs 蛋白(表1)。该结果与phytozome中通过Pfam 蛋白ID(PF03110)关键词在谷子全基因组数据库中比对的结果基本一致。通过CDD 分析谷子SBP 蛋白家族的结构域,结果显示该蛋白家族的保守结构域都含有74~76 个氨基酸残基。除SiSBP12的结构域有74 个氨基酸残基构成,SiSBP8 的结构域有76 个氨基酸残基构成外,其余家族成员的保守结构域均由75 个氨基酸残基构成。SiSBPs 蛋白所包含氨基酸数目范围为199~1 118 aa,等电点为5.41~10.41,分子量介于20.49~122.18 kDa 之间,从蛋白亲水性指数来看,SBP 蛋白家族均为亲水性蛋白,其中SiSBP6 蛋白的亲水性最强。SignalP 分析结果显示,19个SiSBPs 蛋白均无信号肽。PSORT 对谷子19 个SBP蛋白进行亚细胞定位预测,发现除SBP7 蛋白在细胞核中无信号外,其余18 个SBP 蛋白在细胞核中均有信号,即存在于细胞核中,其中SBP2、SBP5、SBP11、SBP16、SBP17 蛋白仅存在于细胞核中,而SBP1、SBP6、SBP7 蛋白也存在于线粒体中,SBP3、SBP4、SBP10、SBP13 蛋白也存在于过氧化物酶体中,SBP6、SBP7、SBP8、SBP14、SBP15、SBP18 蛋白也存在于在叶绿体中,SBP9、SBP12、SBP15 蛋白也存在于内质网膜上,SBP19 蛋白也存在于细胞质膜上。
2.2 谷子SBP 蛋白的系统进化、基因结构及蛋白结构域的分析
为研究SiSBPs 蛋白的系统进化关系,对谷子全基因组中的19 个SiSBPs 蛋白进行系统发育树构建,进化树的分支结果显示(图1),19 个SiSBPs 蛋白可以分为7 个分支,除SiSBP9 蛋白单独为一个分支外,其余分支均包含2~4 个SBP 蛋白。谷子SBP 基因家族结构的绘制结果显示(图2),SiSBP7 和SiSBP16 基因无upstream 序列,SiSBP17 既没有upstream 序列,也无downstream 序列。其余16 个SiSBPs基因都包含上游序列、CDS 区、内含子、下游序列,其中,SiSBP6 和SiSBP7均仅含有一个内含子,SiSBP1、SiSBP4、SiSBP5、SiSBP8、SiSBP10、SiSBP13、SiSBP14、SiSBP16、SiSBP17、SiSBP18 均含有2个内含子,SiSBP2 和SiSBP11 均含有3 个内含子,SiSBP3 均含有4 个内含子,SiSBP9 和SiSBP15 均含有9 个内含子,SiSBP12 和SiSBP19 均含有10 个内含子。从SiSBPs 蛋白结构域的分析结果可知,SiSBP19 除了SBP 蛋白结构域(PF03110)外,还有一个Ank-2(Ankyrin repeats 3 copies)蛋白的结构(PF12796),而其余18 个SiSBPs蛋白只含有SBP 蛋白结构域(图3)。
2.3 不同作物SBP 蛋白的系统进化分析
根据染色体的分布,对谷子全基因组获得的19 个SiSBPs 蛋白进行重新命名。同时在phytozome 中下载拟南芥(17 个)、水稻(19 个)、高粱(19 个)、玉米(36个)的SBP 蛋白,同样根据染色体分布对这几个物种的SBP 蛋白重新命名。利用MEGA6 中的NJ 法对5个物种的SBP 蛋白构建系统进化树。由图4可知,这110 个SBP 蛋白大致分为9 个分支,谷子的19 个SBP蛋白分布在其中7 个分支,其中,SiSBP5、SiSBP8、SiSBP13、SiSBP16、SiSBP18 聚在第Ⅰ分支,SiSBP1、SiSBP10 聚在第Ⅱ分支,SiSBP4、SiSBP6、SiSBP7、SiSBP14、SiSBP17 聚在第Ⅲ分支,SiSBP2、SiSBP3、SiSBP11 聚在第Ⅳ分支,SiSBP9、SiSBP15 分别聚在第Ⅵ和第Ⅷ分支,SiSBP12、SiSBP19 聚在第Ⅸ分支。而AtSBP6 单独为第Ⅴ分支,AtSBP5 和AtSBP11 聚在第Ⅶ分支。总体来看,谷子的SBP 蛋白家族与高粱和水稻SBP 蛋白家族的亲缘关系较近,与玉米的亲缘关系次之,与拟南芥的亲缘关系最远。此外,第Ⅰ分支中SiSBP5、SiSBP8 都是单独为一个亚分支,第Ⅲ分支中SiSBP6 和SiSBP7 聚为一个小分支,说明这2 个蛋白进化关系更近。
2.4 谷子SBP 基因家族的染色体定位分析
根据SBP 基因家族鉴定结果中所在的染色体位置信息,使用Map Gene 2 Chromosome 工具绘制19 个SiSBPs基因在谷子染色体上的分布图。由图5可知,谷子SBP基因家族成员在谷子的9 条染色体上均有分布。其中,在2 号染色体上分布最多,包含4 个SBP基因,在1号和6 号染色体上各有3 个SBP基因,在1号、2 号和6 号染色体上的SBP基因占该基因家族的一半以上,在3 号、4 号和5 号染色体上各有2 个SBP基因,在7 号、8 号和9 号染色体上都只有1 个SBP基因分布。分析发现,SBP基因在染色体上的分布大都集中在染色体的端部,仅3 号和8 号染色体上的SBP8和SBP18 位于染色体中部,3 号染色体的SBP9 和9 号染色体的SBP19 位于近端部。

表1 SiSBPs 基因家族信息Table 1 SiSBPs gene family information
2.5 谷子SBP 基因家族的启动子分析
通过Plantscare 对19 个SiSBPs基因编码区起始密码子上游2 000 bp 序列进行顺式作用元件分析,结果显示,谷子SBP 基因家族启动子区域主要涉及的顺式作用元件包括以下几类:干旱响应元件,高温、低温及渗透胁迫响应元件,光反应元件,植物激素类(生长素、脱落酸、赤霉素、MeJA、水杨酸、乙烯)响应元件,病原菌及损伤类响应元件等(表2)。19 个SiSBPs基因启动子区基本都存在的响应元件有TATA-box、CAATbox、MYB、MYC、STRE、as-1、ABRE 和G-Box。
2.6 谷子SBP 基因家族的表达分析
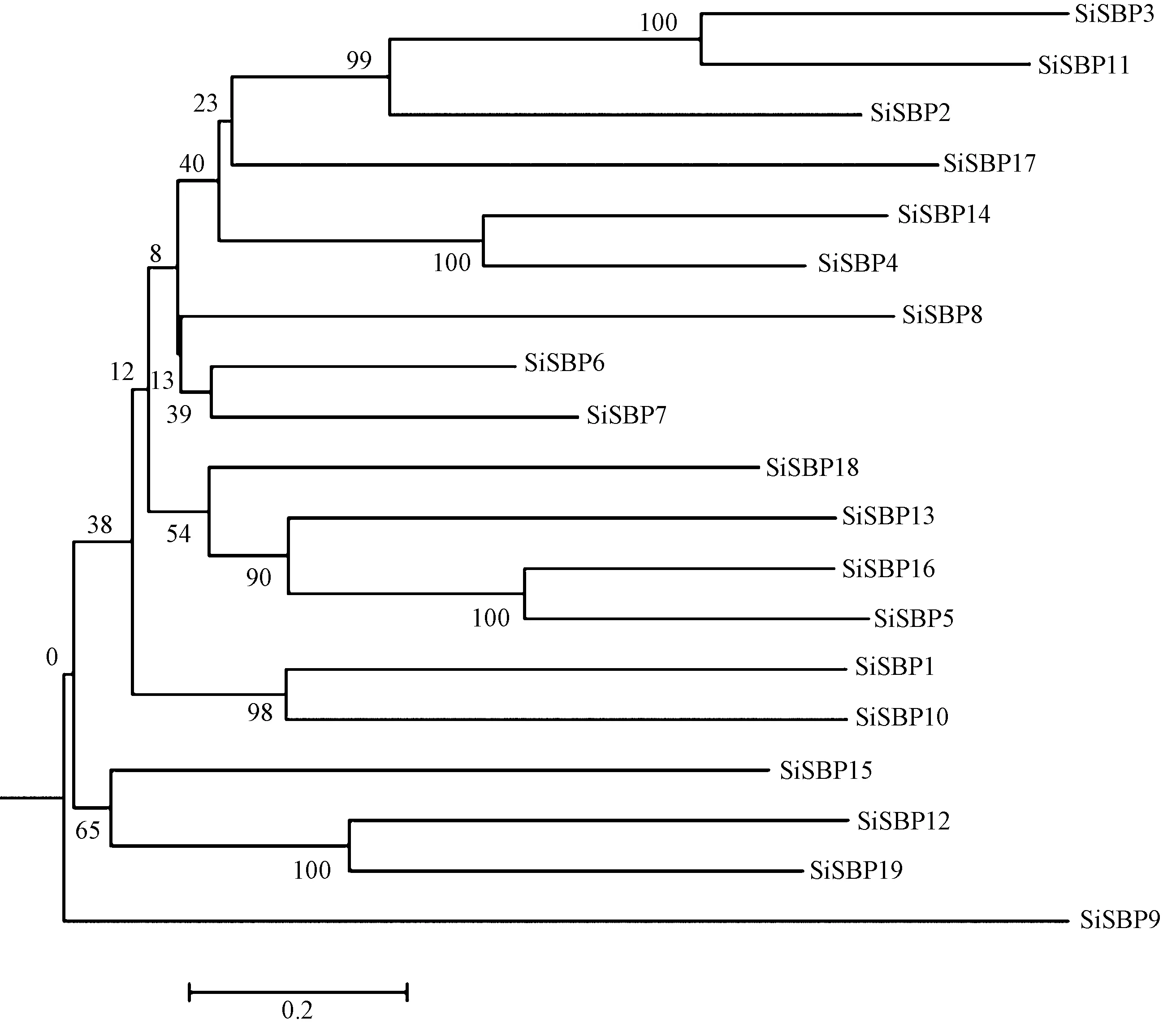
图1 谷子SBP 蛋白系统发育树Fig.1 Phylogenetic tree of SBP proteins in foxtail millet

图2 谷子SBP 基因家族结构图Fig.2 The structure of SBP genes family in foxtail millet
2.6.1 PEG 6000 胁迫对SiSBPs基因相对表达量的影响 经10% PEG 6000 胁迫处理后,SiSBP2、SiSBP3、SiSBP8、SiSBP9、SiSBP11、SiSBP12、SiSBP13、SiSBP15 和SiSBP19 在谷子茎、叶中的表达量与未胁迫时相比均呈上调趋势(图6),且其在叶中表达量的增幅均大于茎。PEG 6000 胁迫后,SiSBP2、SiSBP3 在叶和茎中的表达都呈现先上调后下调再上调的趋势;叶中SiSBP8 表达呈先上调后下调再上调的趋势,而在茎中呈先上调后下调的趋势;SiSBP9、SiSBP11、SiSBP13、SiSBP15 和SiSBP19 在茎和叶中的表达都呈先上调后下调的趋势;SiSBP12 在叶和茎中的表达为上调后下调、再上调最后下调。PEG 胁迫后各SiSBPs基因在叶中的表达量是未胁迫时的10~180 倍,而在茎中的表达量是未胁迫时的4~15 倍。其中,SiSBP3 表达量的上调最小,胁迫18 h 后其在叶中表达量约是未胁迫时的10 倍,胁迫4 h 后在茎中的表达量是未胁迫时的5 倍。与未胁迫相比,表达量上调增幅最大的基因为SiSBP13,胁迫4 h 后在叶和茎中的表达量同时达到最高峰,叶中的上调最显著,是未胁迫的180 倍,茎中的上调较小,是未胁迫的15 倍以上。综上,这9 个SiSBPs基因表达明显受10%PEG 6000 胁迫的调控。
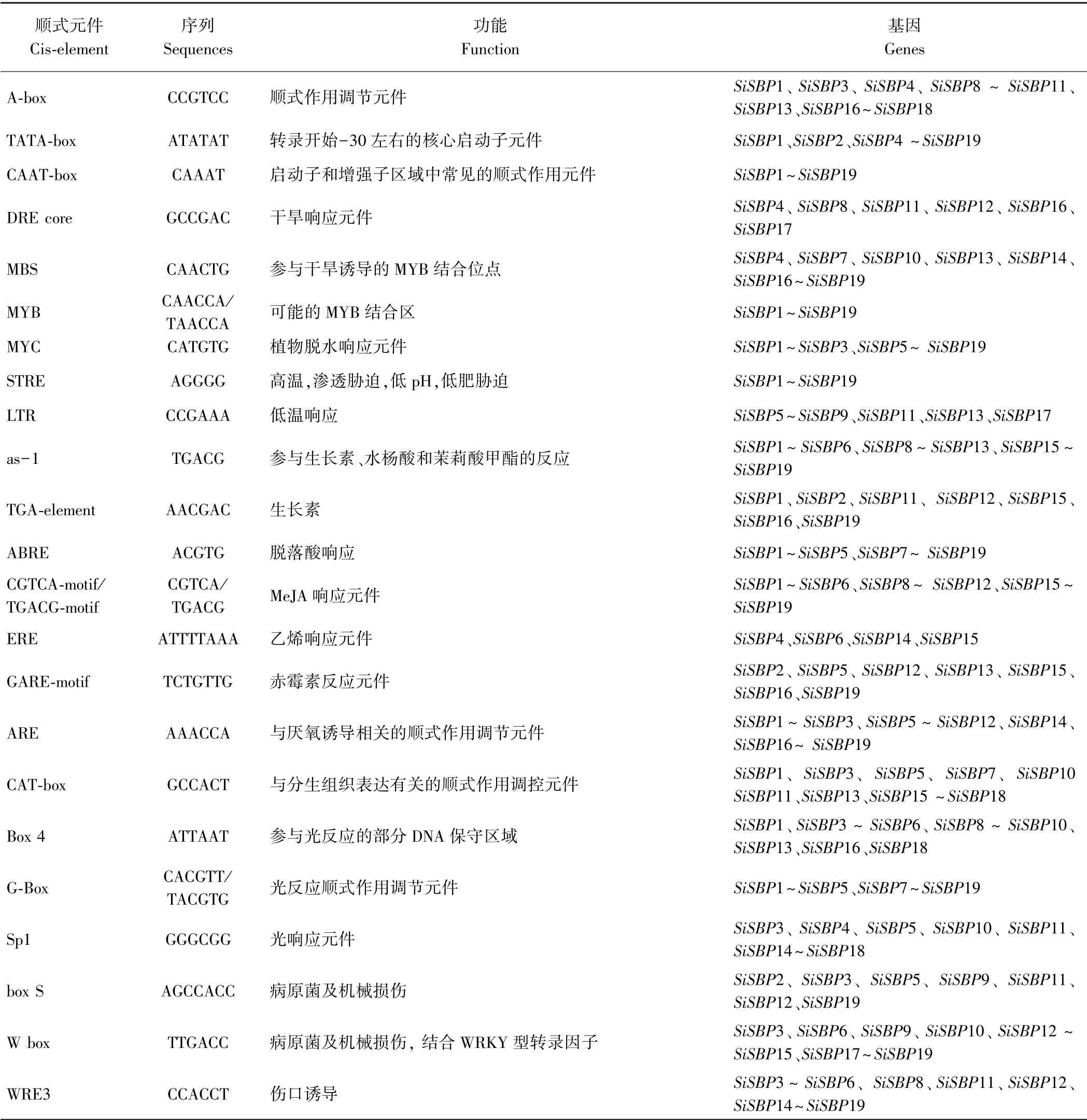
表2 SiSBPs 基因家族启动子区顺式作用原件Table 2 Cis-acting regulatory elements in promoter of SiSBPs gene family

图3 谷子SBP 蛋白的结构域分析Fig.3 Domain analysis of SBP proteins in foxtail millet

图4 谷子、拟南芥、水稻、高粱和玉米的SBP 家族蛋白系统发育树Fig.4 SBP family protein phylogenetic tree of millet, Arabidopsis, rice, sorghum and maize
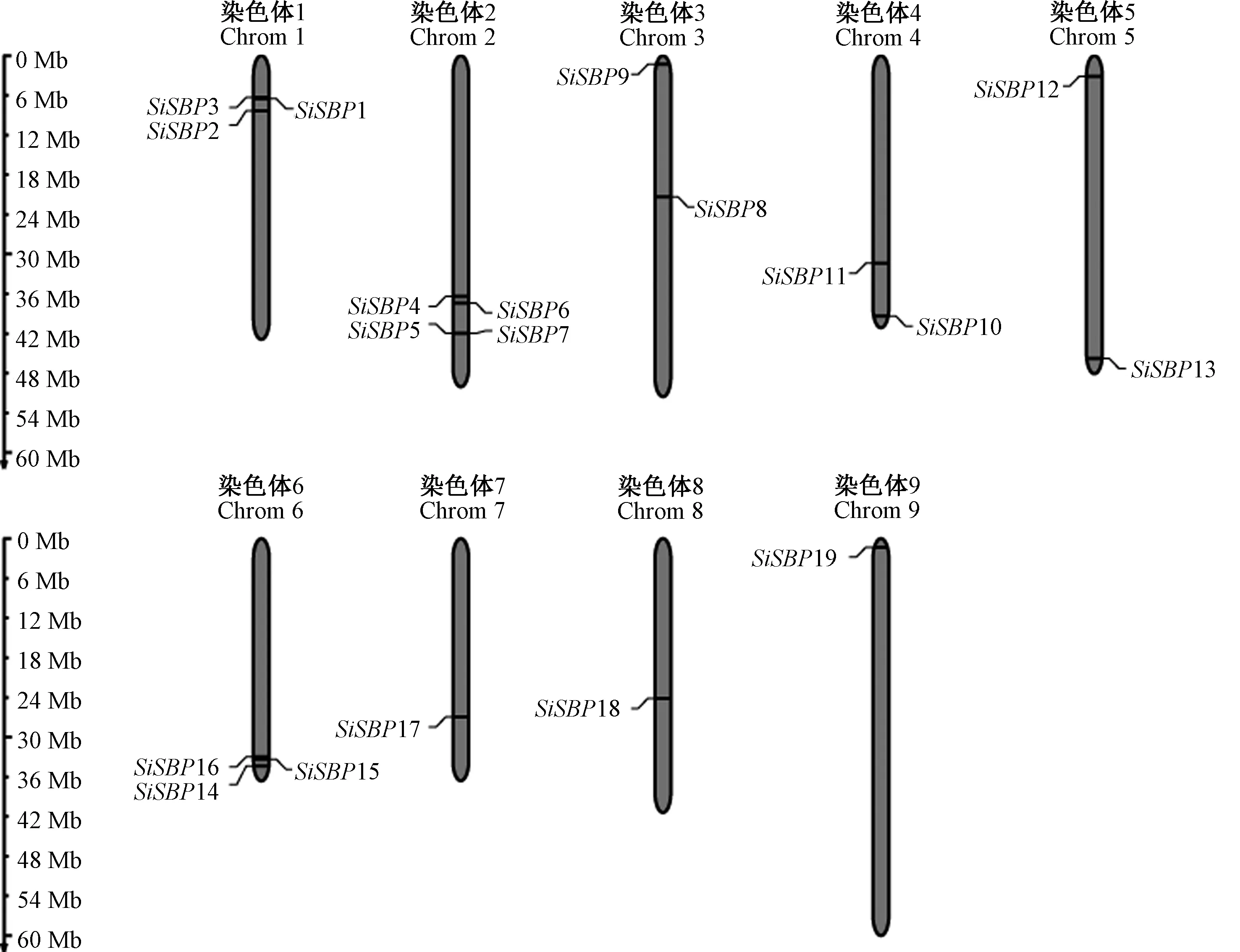
图5 谷子SBP 基因在染色体上的定位Fig.5 The location of the SBP genes on different chromosomes of millet
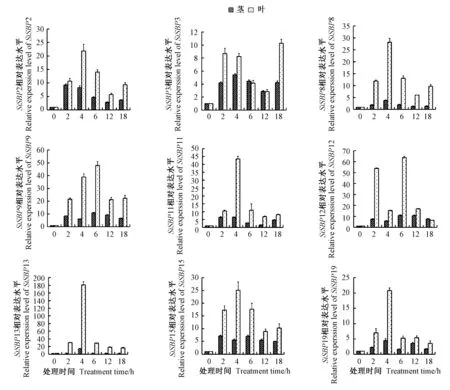
图6 PEG 6000 胁迫下SiSBPs 基因的相对表达量Fig.6 Relative expressions of SiSBPs gene under the PEG 6000 treatments

图7 外源激素ABA 胁迫下SiSBPs 基因的相对表达量Fig.7 Relative expressions of SiSBPs gene under stress treatment of exogenous hormone ABA
2.6.2 外源ABA 胁迫对SiSBPs基因相对表达量的影响 外源100 μmol·L-1ABA 胁迫谷子幼苗后,SiSBP2、SiSBP3、SiSBP8、SiSBP9、SiSBP11、SiSBP12、SiSBP13、SiSBP15 和SiSBP19 在谷子茎、叶中的表达量与未胁迫相比均呈上调趋势(图7)。外源ABA 胁迫后,SiSBP2 在茎和叶中的表达量峰值均出现在胁迫4 h 时;SiSBP3 在叶中出现2 个表达高峰,分别是胁迫2、12 h 时,胁迫12 h 时在茎中的表达量最高;SiSBP8在茎和叶中出现表达高峰的时间,分别是胁迫4、12 h时;SiSBP9 在叶中的表达呈先上调后下调再上调的趋势,而在茎中呈先上调后下调的趋势;在胁迫4 h 时,SiSBP11 在叶中的表达量最高,胁迫6 h 时,SiSBP11在茎中的表达量最高;SiSBP12 在茎和叶中出现表达高峰的时间,分别是胁迫2、4 h 时;SiSBP13 在茎部的表达量于胁迫4 h 时最高,在叶中的表达量持续上调;胁迫4 h 时,SiSBP15 在茎部的表达量最高,胁迫12 h时,SiSBP15 在叶中的表达量达到峰值;胁迫4 h 时,SiSBP19 在茎和叶中表达量均达到峰值。与未胁迫相比,ABA 胁迫后,在叶中表达量上调较高的基因为SiSBP11 和SiSBP13,分别是未胁迫的19.6 倍、28.1倍,在茎中表达量上调较高的基因为SiSBP9、SiSBP11、SiSBP13,分别是未胁迫的6.4 倍、6.7 倍、8.6 倍。9 个SiSBPs基因的表达均受外源ABA 的调控,说明SiSBPs基因是谷子生长过程中较为重要的一类与逆境胁迫相关的基因家族。
3 讨论
SBP 是植物中重要的转录因子,通过结合下游基因启动子区的顺式作用元件,调控下游基因的表达[6]。不同物种之间SBP 基因家族成员数目差异较大,拟南芥[9,11]有17 个、玉米[18]有42 个、高粱[23]有18 个、水稻[10-11]有20 个,这可能与全基因组的复制相关。高粱、水稻与谷子的SBP 基因家族成员相差不大,说明这3 个物种在分离之后全基因组未发生大规模复制。SiSBPs 蛋白所包含氨基酸数目、等电点以及分子量都存在较大差异,可能是由于各家族成员在生长发育过程中的功能不同。
系统发育树结果显示,谷子SBP 蛋白分布于7 个分支。在第1 分支中包含高粱的SbSBP3、SbSBP14,与这两个蛋白关系较近的谷子SBP 蛋白有SiSBP5 和SiSBP16。常建忠等[24]研究显示,高粱的SbSBP3、SbSBP14 基因与高粱的籽粒发育、干物质积累和产量构成有关,推测SiSBP5 和SiSBP16 可能是产量相关的候选基因。同时,位于第1 分支中的SbSBP13、OsSBP8与SiSBP8 的同源关系较近,以及位于第3 分支中的SbSBP16 与SiSBP4、SiSBP14 的同源关系较近。研究表明OsSBP8 是控制植株叶舌发育的关键基因[29],预测SiSBP4、SiSBP8、SiSBP14 可能是谷子中与株型发育相关的基因,植株叶片上冲,可增加光照的吸收,提高光合产物的积累,为后续株型育种提供一定的目标基因。在第9、第8 和第6 分支中还有与胁迫应答相关的3 个SbSBPs基因,分别是SbSBP1、SbSBP15、SbSBP17,与这3 个同源关系较近的基因为SiSBP9、SiSBP15、SiSBP19,推测这3 个基因是谷子SBP 基因家族中与逆境胁迫相关的重要基因成员,可能会提高植物对逆境环境的耐受性。同时,谷子SBP 基因家族成员的启动子区有各种响应元件,如干旱响、高温、低温及渗透胁迫响应元件、光反应元件、植物激素类(生长素、脱落酸、赤霉素、MeJA、水杨酸、乙烯)响应元件、病原菌及损伤类响应元件等,说明SiSBPs基因的表达受诸多因素的调控,涉及非生物胁迫、生物胁迫和生长发育等方面。
本研究重点对SiSBPs基因与抗旱、耐逆方面的相关性做了进一步分析。结果表明,经过10% PEG 6000胁迫和外源ABA 诱导之后,9 个SiSBPs基因在茎和叶中的表达都上调,且9 个SiSBPs基因在叶中的上调表达与茎中相比较显著。这些基因的启动子区都包含脱水响应元件(MYC)、脱落酸响应元件(ABRE),进一步证明了启动子区分析结果的可靠性。其中,SiSBP11和SiSBP13 上调表达的增幅最大,推测这2 个SBP基因可能是参与植物逆境胁迫应答的主要基因,为下一步研究抗旱耐逆的相关基因提供了备选基因。谷子作为禾本科C4作物,其抗旱、光合能力都非常强,但本研究只针对SiSBPs基因响应干旱胁迫及ABA 诱导的应答做了初步研究,对SiSBPs基因对光合产物积累方面的功能还未深入探究,下一步需要对SiSBPs 家族成员在光合产物积累过程中所起的作用进行分析,为挖掘谷子抗旱高产相关的候选基因提供参考。
4 结论
本研究结果表明,谷子SBP 基因家族共有19 个成员,这19 个基因家族成员启动子区的响应元件主要为光照、激素类等元件,SBP 蛋白在细胞的大部分细胞器中均有分布。在10%PEG 6000 胁迫和100 μmol·L-1ABA 诱导后,谷子SBP基因在茎和叶中的表达量显著上调,证实SBP基因对胁迫应答的响应调控具有重要作用,也说明SBP基因涉及植物生长发育过程中的多方面调控。同时,在谷子SBP 基因家族中筛选出了与干旱胁迫最相关的基因SiSBP13,以及与ABA 胁迫相关的3 个重要基因,即SiSBP9、SiSBP11、SiSBP13。但尚未对这些相关基因做进一步验证,未对这些关键基因所涉及的其他重要功能作分析。本研究结果为揭示谷子SBP 蛋白功能和发掘谷子的抗逆基因提供了一定的理论依据。
