桑葚转录组SNP/Indel位点的挖掘及功能注释
2020-07-01王晖谢岩高玉军李季生王彬彬高妍夏
王晖,谢岩,高玉军,李季生,王彬彬,高妍夏
(承德医学院蚕业研究所/河北省高校特产蚕桑应用技术研发中心,河北 承德 067000)
桑树是一种分布于世界各地的商品树种,其经济效益主要来自于桑叶,而桑叶主要用于养蚕业。由于养蚕成本的增加和“东桑西移”战略的实施,东中部地区养蚕业规模锐减,导致桑树产业亟待转型。桑葚作为桑树的果实,含有矿质元素、花青素、风味物质等多种有益成分。近些年桑葚作为一种新型水果受到了极大的关注,整个产业得以迅速发展,果用桑树新品种的选育工作也取得了一定进展。目前通过传统育种手段选育出的果桑新品种主要有大10、白玉王、珍珠白、大马牙等[1]。随着多种分子标记如RAPD、AFLP、SSR、SNP/Indel的不断涌现,利用分子标记进行新品种的选育逐渐成为趋势。相比其它类型的分子标记,SNP/Indel作为一种新型分子标记,其优势在于基因组及转录组数据中存在着大量该标记位点。从茄子[2]、西葫芦[3]、向日葵[4]、武昌鱼[5]、碧桃[7]、松萝凤梨[8]、辣椒[9]、陆地棉[10]、大菱鲆[11]、花叶海棠[12]等多种动植物的基因组或转录组数据库,已成功挖掘出众多SNP/Indel位点。SNP/Indel标记可应用于品种选育、基因型分型、种间亲缘关系分析等。Vivek等[6]用桑树査尔酮合酶基因作为候选基因开发出SNP分子标记,但只分析了査尔酮合酶活性与分子标记分离方式的相关性。目前桑树的基因组测序已经完成,但针对桑树SNP/Indel标记的开发并不充分,更缺乏适宜于果桑的SNP/Indel标记。本研究拟利用RNA-seq技术对不同发育时期的桑葚进行测序,获得桑葚SNP/Indel标记并进行生物信息学分析,将含有SNP/Indel标记的基因导入到多个数据库中进行功能注释分析,获得有重要价值的SNP/Indel分子标记,以期为果桑种质资源评价、品种选育、鉴定等工作的开展提供参考依据。
1 材料与方法
1.1 材料
分别于绿果期、红果期、黑果期收集果桑品种“安葚”的果实,清理表面后迅速投入液氮中,-80 ℃保存。转录组测序工作由北京康普森生物公司完成。
1.2 数据分析方法
将Clean reads通过Trinity软件进行拼接,获得Unigene,将其在GO、COG、KEGG数据库进行比对,获得其注释信息。把各个样品的数据与已组装好的Unigene进行比对,统计每个位点的碱基分布情况,然后结合GATK和SAMtools从头挖掘SNPs和INDELs位点。按照质量值≥20、测序深度≥2、SNP间距≥5、50 bp内SNP数<5的过滤原则,统计基因的SNP分布情况。Excel 2010进行数据分析,Sigmaplot 12.5软件作图。
2 结果与分析
2.1 桑葚转录组数据质量评估
3个时期桑葚的转录组数据共获得51 895条Unigene,各时期桑葚均含有特定表达的Unigene。桑葚转录组的序列数及碱基(A、T、C、G)数目由高到低依次为绿果期、黑果期、红果期,序列平均长度为300 bp。3个时期总GC含量差值较小,且总AT含量均高于总GC含量;测序质量值Q20均大于97%、Q30均大于93%(表1)。表明测序结果良好,可以进行后续的SNP/Indel数据挖掘、分析工作。
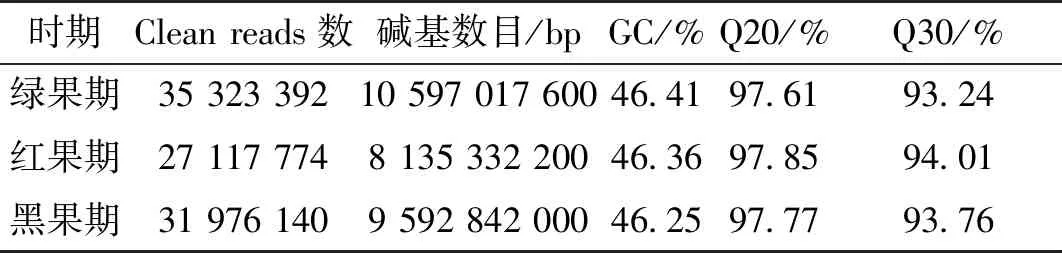
表1 测序产生数据质量评估
2.2 桑葚转录组SNP/Indel类型分析
绿果期13 086条Unigene含有35 024个SNP/Indel位点,平均每条Unigene 2.68个SNP/Indel位点;红果期11 001条Unigene含有28 856个SNP/Indel位点,平均每条Unigene 2.62个SNP/Indel位点;黑果期10 666条Unigene含有26 956个SNP/Indel位点,平均每条Unigene 2.53个SNP/Indel位点。绿果期桑葚转录组的SNP类型中,转换、颠换分别占61.75%、38.25%;红果期分别为61.37%、38.63%;黑果期分别为61.23%、38.77%。绿果期与黑果期转换类型中A/G型最多,颠换类型中A/T型最多。红果期转换类型C/T型最多,颠换类型中A/T型最多;3个时期转换与颠换类型之比均在1.6左右(表2)。
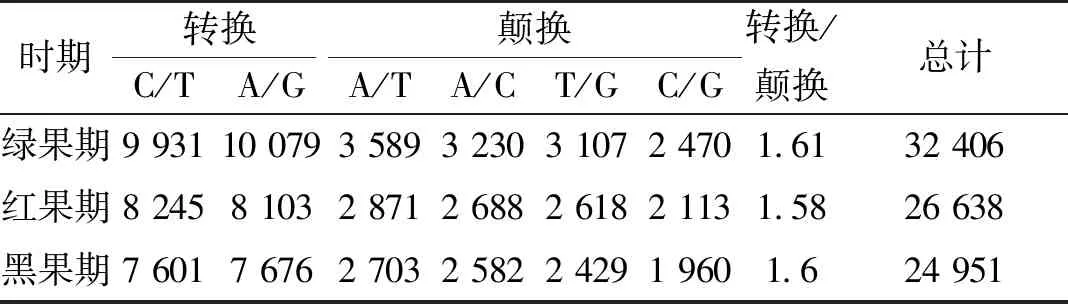
表2 SNP类型统计
桑葚3个时期转录组基因序列中,每1 000 bp的基因序列均以包含1、2、3个SNP位点的基因数目最多,分别占各自时期含有SNP位点的基因总数目的86.99%、88.51%、87.84%(图1)。
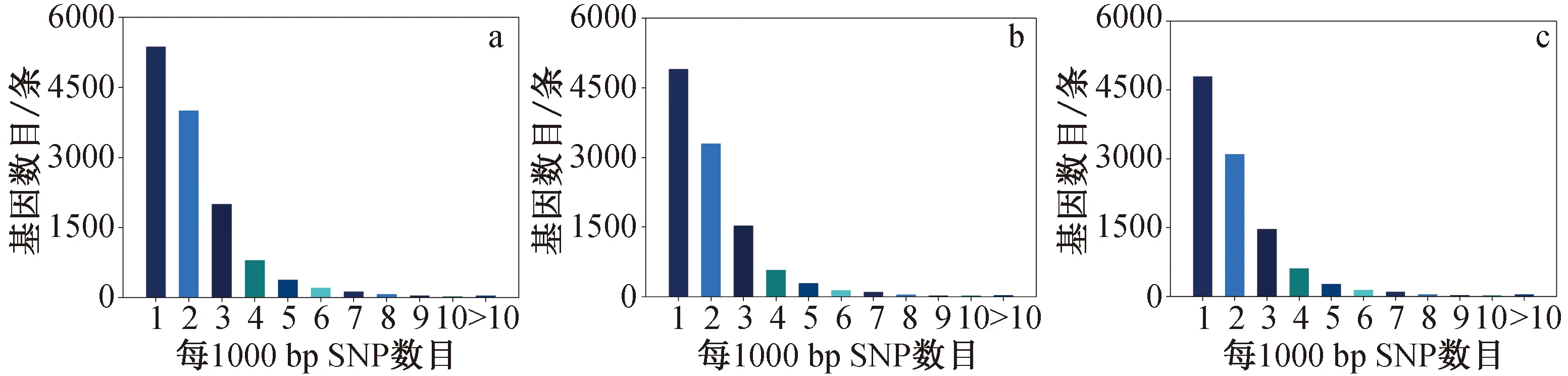
图1 绿果期(a)、红果期(b)、黑果期(c)桑葚转录组SNP位点密度分布频率
桑葚转录组序列中插入/缺失片段从1到10 bp,Indel数量逐渐减少,仅在6 bp时小幅增加;插入/缺失片段长度以1、2、3 bp为主;绿果期3种长度类型的插入、缺失片段数目占总插入、缺失片段数目的76.53%、57.98%;红果期为71.07%、58.15%;黑果期为77.31%、61.21%。桑葚转录组序列中大于10 bp的缺失突变数量远大于插入突变数量(图2)。
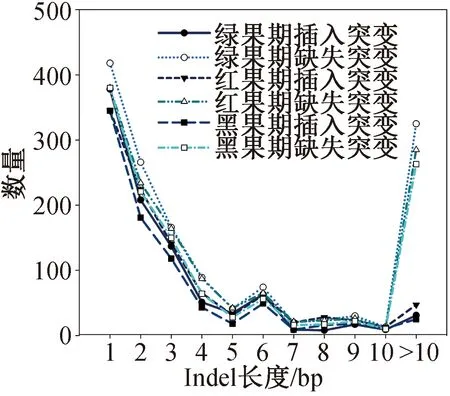
图2 Indel类型统计
2.3 含有SNP位点Unigene GO功能注释
BLAST结果显示,3个时期桑葚转录组中含有SNP/Indel位点的Unigene共有28345条序列注释到GO数据库。在注释后的3个主要通路中,占比最高的是细胞组分(50.55%),其次为分子功能(26.64%),最后为生物过程(22.81%)。细胞组分通路分为12个小类,细胞器部分和细胞通路的基因数目最多;分子功能通路分为7个小类,催化活性和结合通路的基因数目最多;生物过程通路分为21个小类,细胞过程和生殖过程通路的基因数目最多(图3)。
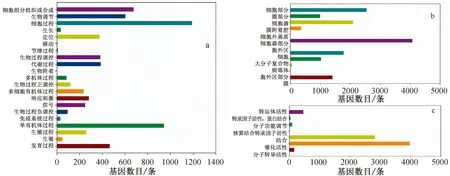
图3 桑葚转录组序列中SNP/Indel基因的GO功能类别:生物过程(a)、细胞组分(b)、分子功能(c)
2.4 含有SNP位点Unigene KOG注释
将含有SNP/Indel位点的序列在KOG数据库中进行功能注释,共6 299条Unigene得到注释,分为4个大类通路:细胞进程与信号、信息存储与加工、代谢及表征不明显。可进一步分为23个通路。功能预测通路1501条基因得到注释,所有亚类中最多;其次为翻译后修饰、蛋白转换、分子伴侣通路606条、信号转导机制通路504条等;最少为细胞核结构通路,5条(图4)。
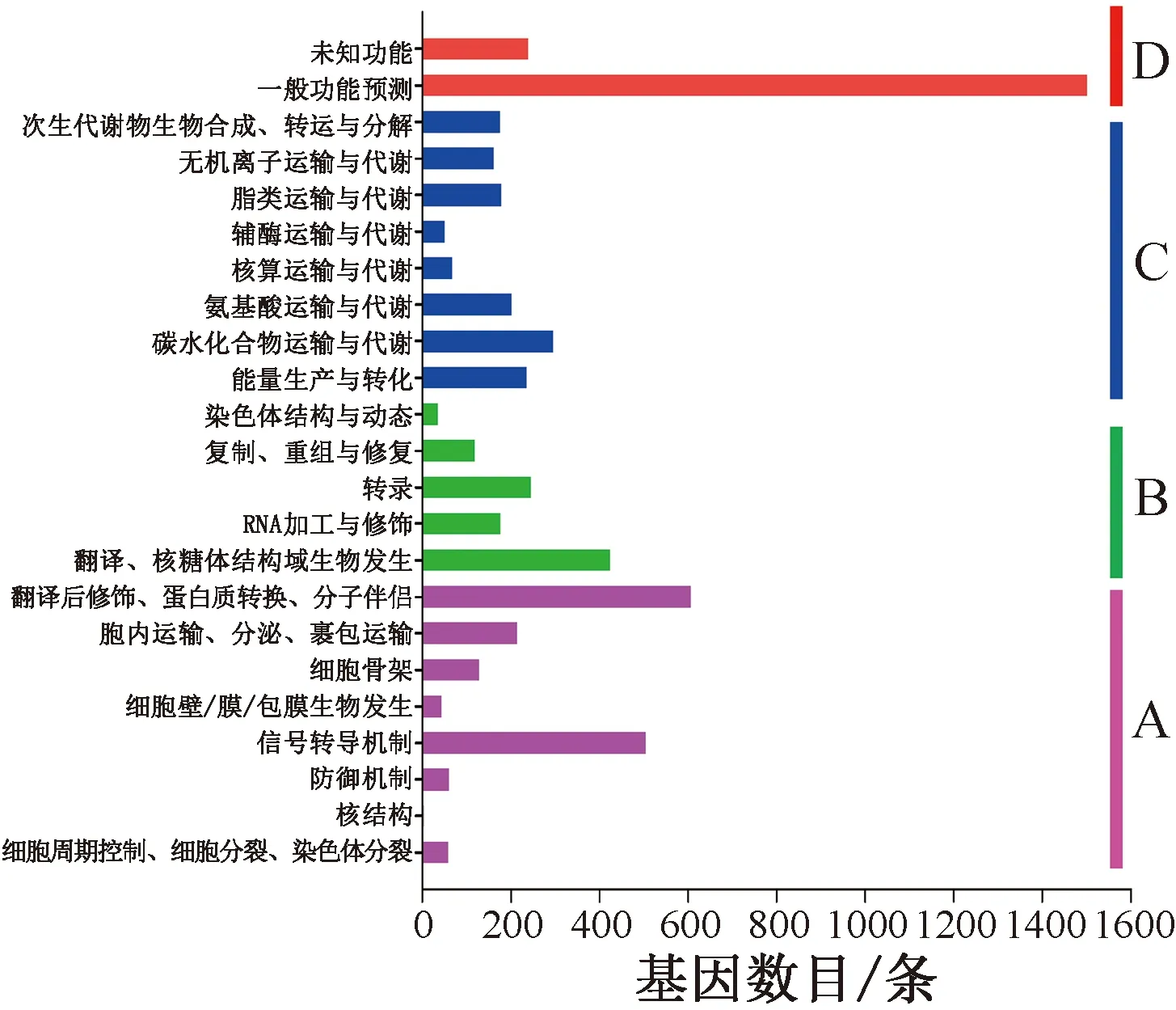
A—细胞进程与信号;B—信息存储与加工;C—代谢;D—表征不明显。图4 桑葚转录组序列中SNP/Indel基因的KOG功能通路
2.5 含有SNP位点Unigene KEGG注释
通过比对分析,共有5737条序列在KEGG数据库中得到注释,共包括5个第一层级通路:细胞过程、环境信息处理、遗传信息处理、代谢、有机系统。5个第一层级通路包含19个第二层级通路,主要以碳水化合物代谢、翻译为主(图5)。
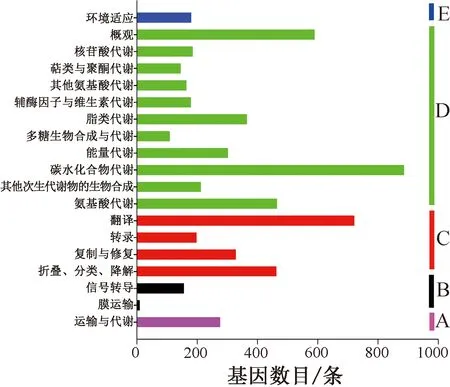
A—细胞过程;B—环境信息处理;C—遗传信息处理;D—代谢;E—有机系统。图5 桑葚转录组序列中SNP/Indel基因的KEGG功能类别
再进一步可分为129个通路,其中注释到核糖体、碳代谢、氨基酸生物合成、内质网蛋白质加工等通路的基因最多(图6)。重要次生代谢物的生物合成通路包含黄酮生物合成、异黄酮生物合成、花青素生物合成、苯丙素生物合成、黄酮与黄酮醇生物合成等通路,上述通路分别有45、1、5、60、11条基因序列得到注释(图7)。
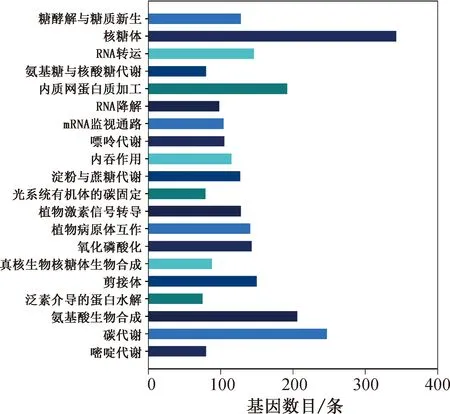
图6 注释到KEGG数据库含有SNP/Indel基因数目最多的20个通路
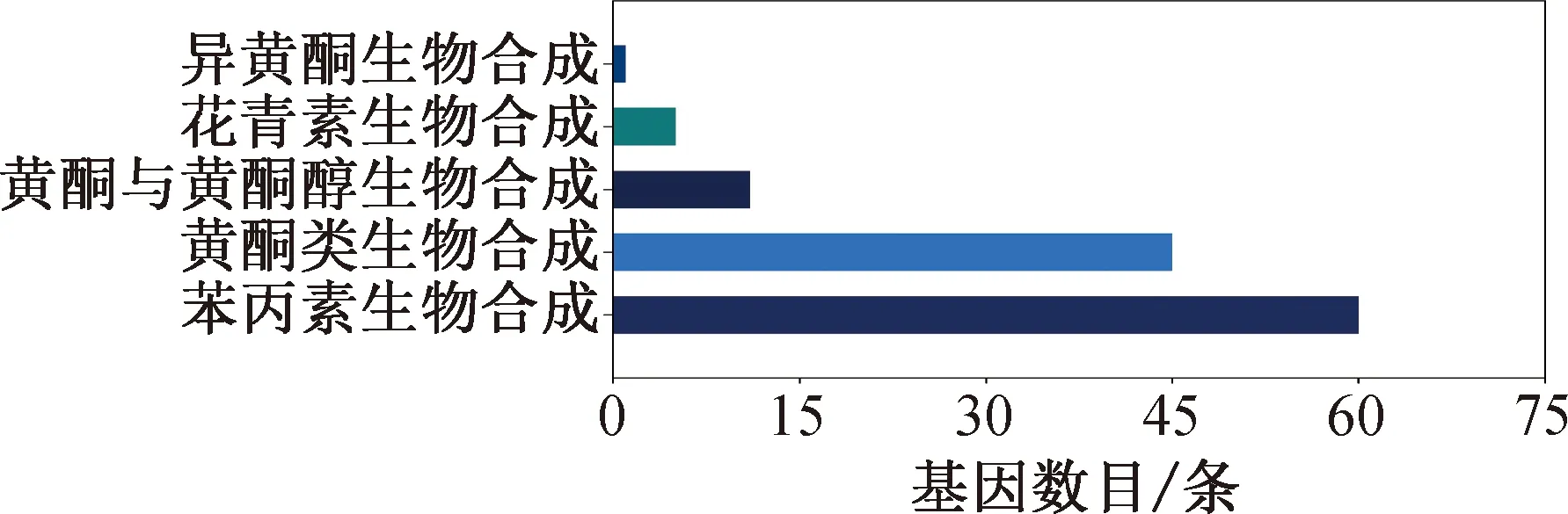
图7 含有SNP/Indel位点的黄酮类与花青素成分合成相关基因
3 讨论
目前,NCBI数据库中约有6 000多万条EST序列,尚无桑葚的EST序列数据库,这不利于理解桑葚的发育进程及重要营养物质的积累过程,制约利用分子标记快速开展优良果桑品种的选育工作。随着测序技术的不断成熟及成本的降低,利用RNA-seq技术已建立了多个物种的EST数据库[13]。本研究对桑葚进行转录组测序,共获得51 895条Unigene序列,重点分析了Unigene中的SNP/Indel位点特征。3个时期桑葚转录组基因序列中SNP位点的出现频率依次为1个/462 bp、1个/482 bp、1个/478 bp,低于桉树基因组(1个/192 bp)[14]、苹果基因组(1个/149 bp)[15]、葡萄基因组(1个/117 bp)[16]、柿树转录组(1个/253 bp)[17];说明不同物种基因组、转录组数据中SNP的出现频率各不相同,推测其具有物种特异性。即使同一物种,SNP出现频率也有所不同。橡胶树转录组数据中SNP位点出现频率,有报道是1/1.5 kb[18];也有报道是1/5.2 kb[19],可能是由测序材料、深度、检测软件的版本及参数设置等多种因素所引起。SNP类型及各类型间比例在不同植物基因组间则相对稳定。桉树转录组数据中,转换、颠换SNP位点分别占60%、占40%[20];在太平洋白虾中,分别为66.8%、33.2%[21];在桑葚中,分别为61%、38%左右(表2)。在自然选择过程中,转换突变在蛋白编码序列中会产生同义突变,因此通常情况下SNP的转换类型出现频率都高于颠换类型[22]。
利用现已建立的多种数据库可以对已获得的基因组、转录组数据进行注释,通过分析,在黄花鱼[23]、树鼩[24]、褐色砂梨[25]、马铃薯[26]、向日葵锈菌[27]、菜豆[28]、玉米[29]等多个物种中筛选出与重要性状相关的SNP/Indel分子标记。桑葚转录组数据库中含SNP/Indel标记基因经KOG数据库注释,发现翻译后修饰、蛋白转换、分子伴侣、信号转导机制、翻译、核糖体结构与生物发生、碳水化合物运输与代谢、转录、能量生产与转化、胞内运输、分泌、囊泡运输、氨基酸运输与代谢等类别中的基因数有3 019条,占所有注释基因数的47.93%(图4),说明随着桑葚的成熟,一系列复杂的分子机制发挥作用,引起桑葚颜色、味道、口感发生剧烈的变化。从KEGG数据库注释结果分析,含有SNP/Indel标记基因数目最多的通路依次为核糖体、碳代谢、氨基酸生物合成、内质网蛋白质加工、剪接体、RNA转运等(图6),这些通路与物质、能量代谢的分子机制紧密相关,进一步验证了KOG数据库的注释结果。KEGG注释结果中共筛选出122个与花青素、黄酮类合成有关的含有SNP/Indel位点的基因(图7);这些SNP/Indel位点可能与桑葚品质形成的关键基因相连锁,将成为开发选育优良果桑品种的分子标记的重要来源。
4 结论
本研究通过RNA-seq技术建立了桑葚转录组数据库,证实桑葚SNP/Indel位点比较丰富。通过对获得的SNP/Indel标记特征、基因功能注释结果进行详细研究,有助于下一步筛选出鉴别力强的分子标记及开发出高密度SNP基因分型技术,加快果用桑树的品种选育、种质资源鉴定等工作,更好地服务桑葚产业发展。
