基于Adaboost 的脑肿瘤患者存活周期分析
2020-06-17张桂凤
张桂凤
(黑龙江科技大学电气与控制工程学院 哈尔滨 150022)
脑肿瘤是指发生于颅腔内的神经系统肿瘤,又称颅内肿瘤,其主要发生于成年人,特别是老年人,具有复发率高、病死率高以及治愈率低3 大特点[1]。脑肿瘤产生的常见危害是造成脑内组织水肿和压迫中枢神经等脑内正常组织。脑肿瘤伴随的脑水肿会导致颅内压升高,在临床上脑肿瘤患者多表现为头痛、呕吐或瞳孔病变等。脑肿瘤的生长会直接压迫正常脑内功能结构,诱发认知能力下降、偏瘫、失语症、反应迟钝等局部神经症状的发生。
脑部磁共振成像(magnetic resonance images,MR)是脑肿瘤疾病检查的主要手段之一,具有实时成像、无创伤性成像、无辐射性成像、成像分辨率高等优点。MR 利用原子核自旋运动的特点,成像设备对脑组织进行外加磁场干预,并用探测器检测射频脉冲激发脑内原子后所产生的信号,再由计算机对探测器检测的信号处理成图像[2]。脑肿瘤疾病诊断普遍使用的MR 是结构性磁共振影像(structural magnetic resonance imaging, sMRI),sMRI 能够定性地展现大脑中肿瘤组织和正常组织的位置、形状和大小,为医生把握患者的病情提供有效参考。sMRI 在成像过程中可以通过调节参数生成T1、T2、T1ce(T1 增强)、Flair(液体衰减反转恢复)等多种序列,以不同方式呈现组织特性,为临床诊断提供多角度、多方位的准确信息。如,肿瘤和纤维组织在T1 序列中均多呈低信号,而在T2 序列中肿瘤多呈现为高信号,纤维组织呈现低信号。这样,就可以通过T1、T2 序列的结合来对目标组织进行判断。通常脑肿瘤在多序列MR 中呈现出的肿瘤区域由3 个子区域组成,分别为水肿区域(edema tumor, ED)、增强区域(GD-enhancing tumor, ET)、非坏死区域(necrotic and non-enhancing tumor, NCR)。
基于MR 的脑肿瘤患者存活周期预测研究,能够为临床诊断效果的判别和医师相关治疗计划的调整提供相应的辅助参考信息。因此,基于MR 的脑肿瘤患者存活周期预测在临床中也极具意义。但是,基于MR 的脑肿瘤患者存活周期分析是一项极具挑战的工作,因为临床中保留下的可供分析的完备且有效的样本数量相对较少,增加了分析难度。随着机器学习等人工智能技术的不断成熟,基于MR 的脑肿瘤患者存活周期预测的研究也取得了一些进展。文献[3]提出了一种基于图像特征和非图像特征的线性回归模型的分析方法,其从脑肿瘤MR 的Groundtruth 中提取了ED、ET 以及NCR 部分的体素数目和表面积等6 种图像特征以及患者的年龄和手术状态2 种非图像特征,最后将8 种特征输入到线性回归(linear regression, LR)[4]模型中进行学习训练以进行存活周期分析。文献[5]提出了一种基于纹理特征和多层感知机(multi-layer perception, MLP)[6]模型的分析方法,其通过计算肿瘤MR 的一阶统计、形状特征、灰度共生矩阵和灰度运行长度矩阵特征,并从ED、肿瘤核区域(tumor core, TC)及ET 区域中提取了468 个特征,并将提取的468 种特征输入到MLP 中学习训练,经过相关模型调优后完成脑肿瘤患者存活周期分析的工作。文献[7]提出了一种基于脑肿瘤患者年龄和LR 的分析方法,其直接将患者年龄输入到LR 模型中训练以进行存活周期分析。以上3 种方法在Brats2018 Challenge[8]的脑肿瘤患者存活周期分析赛道中取得了前3 名的成绩。除了以上方法外,相关的机器学习回归方法,如支持向量机模型(support vector machine, SVM)[9]、K 近邻(K-nearest neighbor,KNN)[10]、随机森林(random forest, RF)[11]等模型,也在此领域中有着相关应用。
虽然脑肿瘤患者存活周期分析领域已经取得了一些进展,但是仍存在一些问题待解决。首先,一些方法在前期特征工程阶段存在特征提取针对性不强和特征选择缺失的问题。现行系统中的许多方法在特征提取阶段针对性不强,没有根据分析目的进行特征提取。并且相关方法没有对所提特征集进行去冗余和去相关处理,在特征提取阶段提取的原始特征集中可能存在大量的冗余特征和相关特征。如果直接利用原始特征集进行模型的训练不仅增大模型的训练代价而且可能会限制模型泛化能力的提升。其次,分析模型选择目的性不明确。由于临床中可用的训练样本较少,所以在进行分析时就应该尽可能地选取泛化能力高的分析模型。虽然MLP、LR 等模型具有简单、可操作性高等优点,但是通过优化目标的分析可以了解到这些模型的泛化能力与基于MR 的脑肿瘤患者存活周期分析的要求还相距较远。为解决以上问题,本文提出了一种基于Adaboost 的脑肿瘤患者存活周期分析系统。
1 脑肿瘤患者生存周期分析
图1 为本文提出的基于Adaboost 的脑肿瘤患者存活周期分析系统图。为增强影像样本中有关信息的可检测性、最大限度地简化数据以及改进特征提取和识别的可靠性,首先对脑肿瘤患者的多序列功能性磁共振样本进行时间层校正、头动校正、图像配准、空间配准、空间标准化等预处理。之后通过读取肿瘤区域标注的Groundtruth 进行肿瘤感兴趣区域(region of interest, ROI)的确定和肿瘤目标区域的分割。通过以上预处理等操作,本文对脑肿瘤多序列MR 影像提取纹理特征以获取原始特征集。在特征提取之后,为减少模型训练过程中的时间代价、资源占耗和提高模型预测的结果,利用前向分布算法以互信息(mutual information, MI)[12]为评价准则对原始特征集进行去冗余和去相关性等降维处理,并获取原始特征集的有益特征子集。在特征选择之后,利用特征子集和对应样本的存活情况对Adaboost.R2[13]进行学习训练,并在经过调优后输出相关样本的存活周期的分析结果。
1.1 特征提取
由于MR 样本的体素数目巨大,直接利用MR进行学习分析就变得十分困难。为了减少学习代价和最大化模型的泛化效果,必须从脑肿瘤MR 中提取具有信息量和非冗余性的派生值,这样不仅能够尽可能无差性地描述原始数据,而且能够简化学习过程。纹理是一种反映图像中同质现象的视觉特征,它体现了物体表面具有缓慢变化或者周期性变化的表面结构组织排列属性[14]。通过像素及其周围空间邻域的灰度分布展现的纹理可以了解肿瘤等病变组织的发展区域和恶化程度,从而了解脑部肿瘤的本质属性。同时,纹理特征具有旋转不变性和对于噪声有较强的抵抗能力,因此,本文采用纹理特征作为进行脑肿瘤患者存活周期分析的主要特征。

图1 基于Adaboost 的脑肿瘤患者存活周期分析系统图
对脑肿瘤患者的MR 影像进行多序列、多重分形的纹理特征提取。首先,要获取应用于肿瘤分析的感兴趣区域(ROI),采取的方法是读取脑肿瘤患者影像的分割标签(groundtruth),这些分割标签是由脑肿瘤影像分析专家逐层勾画的,能够精准表示肿瘤的区域。脑肿瘤患者MR 影像的标注、分割、三维ROI 示例如图2 所示。其中图2a 为脑肿瘤患者的MR 影像,左上曲线为肿瘤边界,图2b 为分割结果,图2c 为ROI 示意图。

图2 脑肿瘤MR 标注、分割、ROI 示意图
在获取脑肿瘤MR 的ROI 之后, 通过Pyradiomics[15]分别计算脑肿瘤MR 的三维灰度共生矩阵(gray-level co-occurrence matrix,GLCM)[16]、灰 度 游 程 矩 阵 (gray-level run-length matrix,GLRLM)[17]、灰 度 级 带 矩 阵(gray-level size zone matrix, GLSZM)[18]、灰度空间依赖矩阵(gray level dependence matrix, GLDM)[19]及 其 相 关 矩 阵 属 性值,提取70 种纹理特征。最后,按照以上特征提取方法在脑肿瘤患者的T1、T2、T1ce、Flair 4 个模态以及ED、ET、NCR 3 个肿瘤子区域中,分别提取了以上70 种特征。综上,本文共提取了490(7×70)种纹理特征。
1.2 特征选择
本文在特征提取阶段提取了490 种特征,这些特征为后续预测模型的学习提供了学习依据。由于所提取的原始特征集可能存在冗余特征和相关特征,如果这些特征全部用于预测模型的建立,必然会增大预测模型训练代价和降低学习模型的预测效果。因此,在预测模型学习之前需要进行特征选择将提取的原始特征集进行去冗余性和去相关性操作,即进行特征选择。本文使用前向逐步算法以MI,如式(1)为评价准则提取的原始特征集选出了一个包含70 个特征的特征子集。式(1)互信息描述的是两个特征的分布相似度。
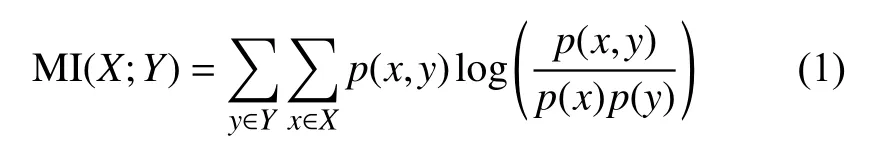
式中, p(x,y)是 特征X 和特征 Y 的联合概率分布函数; p(x)和 p(y)分别是特征X 和特征 Y 的边缘概率分布函数。
对原始特征集进行特征选择的步骤如下:1) 计算原始特征集中每一个特征与其他特征的互信息之和,将原始特征集中与其他特征互信息之和最小的特征放入特征子集中;2) 更新原始特征集和特征子集;3) 依次迭代,直到特征子集中包含70 个特征为止。具体算法结构如算法1 所示,算法1 中index 表示每次迭代时选择的特征的序号。
算法1 特征选择

1.3 Adaboost.R2
Adaboost.R2 算法是一种前向分布算法,即此模型是由基本分类器组成的加法模型:

式中, Bt(x) 是基学习器; αt是基学习器的系数,其预测结果是各个基学习器预测结果的加权和,所以在训练过程中,依次迭代训练各个学习器及其系数。在这一训练过程中,为了增大学习器的收敛效果,采用平方误差以监督模型的学习,即:
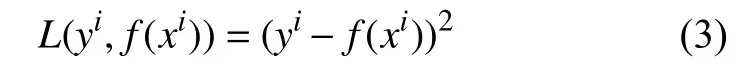
为简化计算,用归一化因子D 将以上损失函数进行归一化:
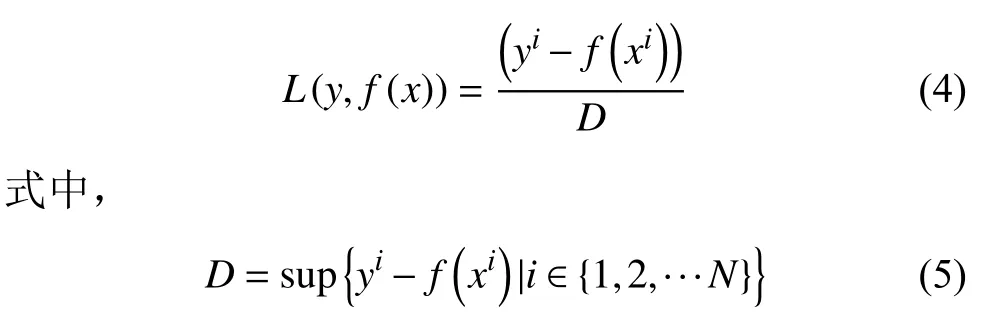
根据Adaboost.R2,假设经过t−1 轮迭代训练生成的预测学习器
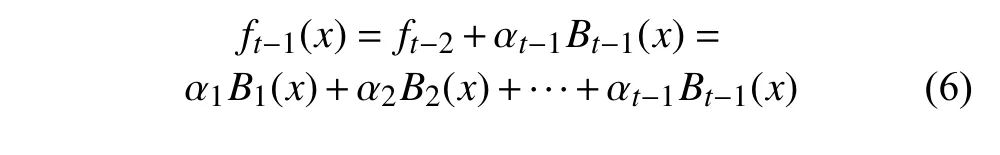
则在第t 轮迭代学习训练中得到 αt、 Bt(x)和ft(x) 为: ft(x)= ft−1(x)+αtBt(x),其中:

Adaboost.R2 的算法伪代码如算法2 所示。
算法2 Adaboost.R2
输 入:训 练 集(x1,y1), (x2,y2),···,(xN,yN)。其 中xi为训练样本;yi为回归标签;最大迭代次数 T;当前迭代指数 t;学习损失 L;T 次迭代生成的学习器映射权重 Wt。

2 实验与分析
2.1 实验数据
Brats2018 数据集是一个开源的多序列的脑肿瘤MR 数据集,由训练数据(train data)、验证数据(validation data)、测试数据(test data)3 个部分组成。其中,只有训练部分公开了相关样本的存活情况,所以本文使用Brats2018 的训练数据进行相关实验验证。Brats2018 的训练数据包括210 个高级别受试MR 和75 个低级别受试MR。每例受试MR 中分别包含T1、T2、T1ce、Flair 4 个序列的sMRI 和肿瘤区域标注Groundtruth。在肿瘤区域标注Groundtruth 中,Brats2018 对脑肿瘤的ED、ET 和NCR 的不同区域用不同标签进行了标记。除此之外,Brats2018 训练数据集还提供了163 例高级别受试MR 对应的生存周期、年龄、手术情况信息。本文利用Brats2018 训练数据中拥有存活周期信息的160 例受试MR 进行训练和验证。图3 为Brats2018 训练数据集中某一受试样本的T1、T2、T1ce、Flair 及分割Groundtruth像。其中图3a 为T1 序列;图3b 为T2 序列;图3c 为T1ce 序列;图3d 为Flair 序列;图3e 为Groundtruth;图3f 为Brats2018 训练数据的存活周期分布直方图。

图3 Brats2018 训练数据样本
2.2 特征子集
表1 为通过以MI 为评价准则对原始特征集进行特征选择之后得到的特征子集,其中包括14 个T1 序列的纹理特征,11 个T2 序列的纹理特征,13 个Flair 序列的纹理特征,16 个T1ce 序列的纹理特征以及ED、ET、NCR 这3 个肿瘤子区域的16 个纹理特征,共70 种特征。表1 中特征名称的书写规则为:模态_纹理矩阵_矩阵属性。
2.3 实验结果及分析
为了对比本文提出的脑肿瘤的生存周期预测框架的表现能力,本文在进行相关调研的基础上设计了5 种对比实验,前3 种为Brats2018 Challenge的脑肿瘤患者存活周期分析赛道中取得前3 名算法:Xue[3]、Baid[5]、Weninger[7]。除 此之外,本文还通过对当前领域中常用的传统方法的进行了调研,设计了ICA[20]+LR、PCA[21]+RF 两种对比实验,其中ICA+LR 的计算原理是:提取与本文相同的特征,以ICA 为评价准则进行特征选择后用LR进行存活时间的预测。与ICA+LR 类似,PCA+RF的计算原理是:提取与本文相同的特征,以PCA 为评价准则进行特征选择后用RF 进行存活时间的预测。
本文在相同的实验环境下进行了以上对比方法的实验验证。为了直观反映本文提出的基于Adaboost 的脑肿瘤患者存活周期分析系统的泛化能力,本文按照Brats2018 的评测方法进行评测,即将回归问题转为分类问题进行评测。在本文提出的分析模型输出相关样本的存活周期预测结果之后,按照Brats2018 的方法将预测结果映射为3 个类别:长期、中期、短期,再以3 个存活类别的分析准确率作为评测结果。其中,长期为样本的存活周期大于15 个月,中期为存活周期大于10 个月小于15 个月,短期为存活周期小于15 个月。为了得到准确、可靠、有说服力的实验结果,本文对整个训练验证的过程进行5 折交叉验证,并以5 折交叉验证结果的均值作为最终输出结果,具体实验结果如表2 所示。通过表2 可以看出,本文提出的脑肿瘤患存活周期分析系统的预测准确率优于Xue、Baid 和Weninger 的方法,其中比Xue 的方法提高了5%,比Baid 提高了18.35%,比Weninger 提高了9.66%,比ICA+LR提高了10.625%,比PCA+RF 的方法提高了14.375%。从以上实验结果可以得出,对于基于MR 的脑肿瘤患者存活分析来说,显著特征的选择至关重要。本文在特征选择阶段选取了一个包含了70 种特征的特征子集,特征子集的个数既保证了特征中相对较低的冗余度,又最大程度地保留了显著特征。相较于Xue、Weninger 的方法,本文提取和选择的特征是以上两种方法的8~35 倍,提升了显著特征的范围,从而为提升脑肿瘤患者存活分析的准确率提供更多可学习特征。相较于Baid 的方法,本文通过特征选择得到的特征子集是其应用特征的1/7,降低了可训练特征的冗余度,减小了噪声特征的影响,从而为提升脑肿瘤患者存活分析的准确率提供了低冗余有益信息。基于传统降维的方法在处理大量特征的时候可能会产生部分显著特征丢失的问题,从而限制了传统机器学习分析方法的分析效果,所以本文也比基于降维的传统方法有实质提升。

表1 特征子集

表2 实验结果
3 结 束 语
为了提高脑肿瘤患者存活周期分析的准确性,本文提出一种基于Adaboost 的脑肿瘤患者存活周期预测系统,通过对脑肿瘤患者多序列MR 进行纹理特征提取、特征选择以及分析模型训练等一系列工作,完成了对脑肿瘤患者存活周期的分析。通过对Brsts2018 训练数据的交叉验证实验表明,本文提出的分析系统的分析准确率优于当前领域的一些典型算法。
