龙头鱼ISSR-PCR多态性引物最佳退火温度的筛选
2020-06-01林彦均杨天燕朱岚倩郑亦佳王莹莹斯舒谨郭泽豪
林彦均 杨天燕 朱岚倩 郑亦佳 王莹莹 斯舒谨 郭泽豪

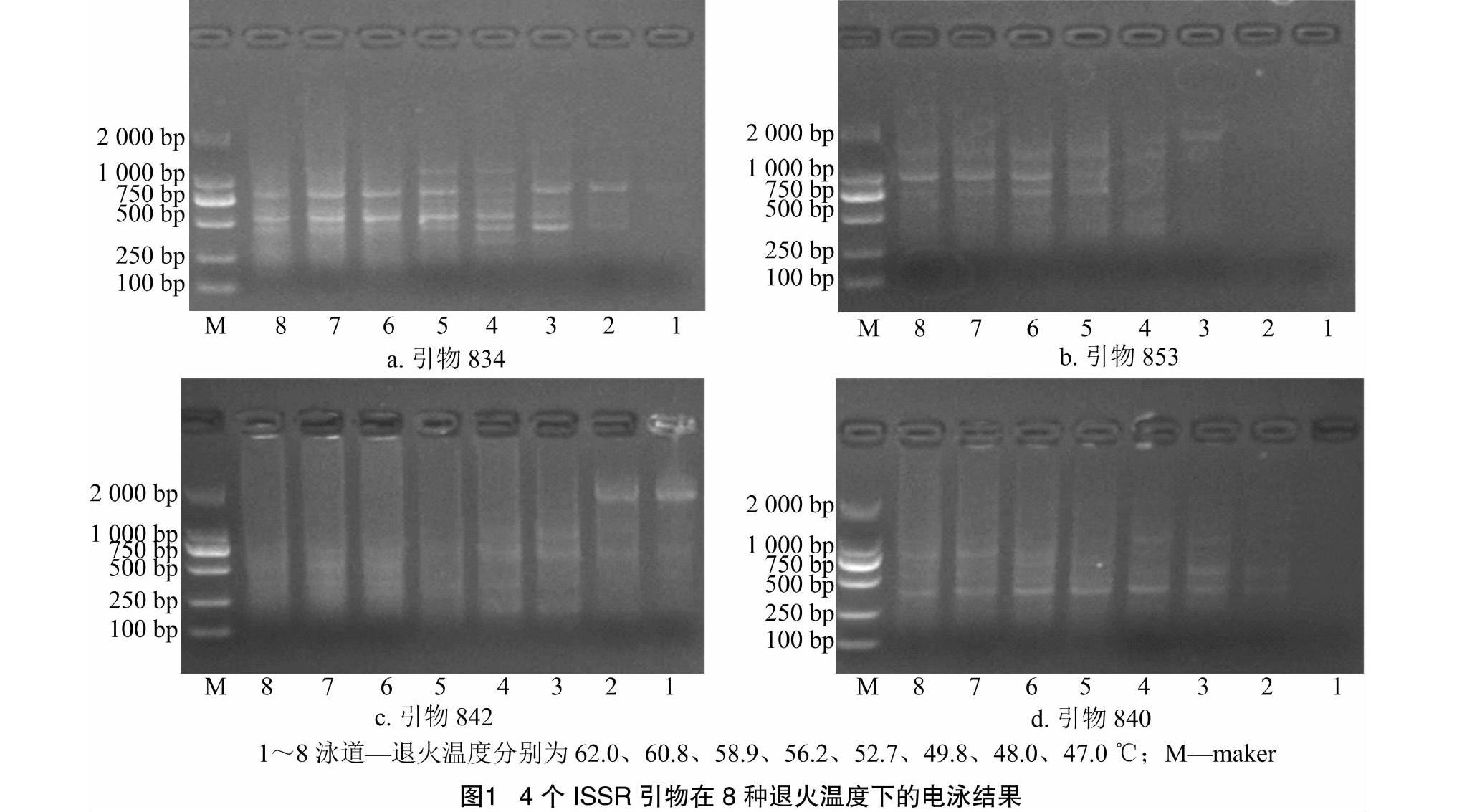
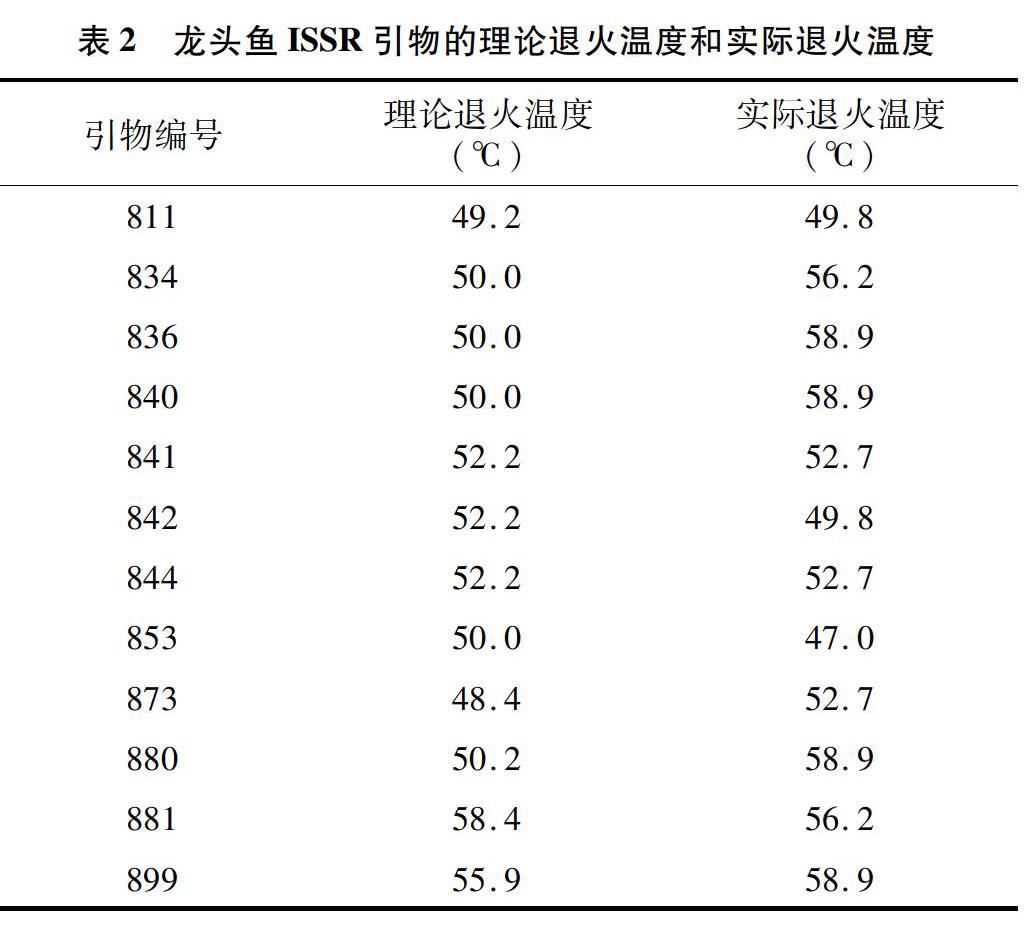
摘要:为了开展龙头鱼遗传多样性和近缘物种间的遗传变异分析,建立龙头鱼的简单序列重复区间标记(inter-simple sequence repeat,简称ISSR)-PCR反应体系,拟筛选龙头鱼ISSR-PCR多态性引物的最佳退火温度。参考加拿大哥伦比亚大学(UBC)公布的100条ISSR引物,采用温度梯度PCR的方法,得到适用于龙头鱼基因组DNA扩增的12条ISSR分子标记引物,分别为811、834、836、840、841、842、844、853、873、880、881、899。12条引物中,最高退火温度为58.9 ℃,最低退火温度为47 ℃,两者间的差异达到了11.9 ℃。12条引物的变异系数为7.71%,相关系数为0.23。结果表明,龙头鱼ISSR多态性引物的最佳退火温度与理论退火温度间没有显著相关性,同一种引物的退火温度在不同物种中也存在一定的差异,需要通过温度梯度试验进行具体分析。
关键词:龙头鱼;ISSR-PCR;退火温度;引物筛选
中图分类号: S917文献标志码: A
文章编号:1002-1302(2020)08-0070-04
收稿日期:2019-03-17
基金项目:2018年国家级大学生创新创业训练计划(编号:201810340010);浙江省大学生科技创新活动计划暨新苗人才计划(编号:2019R411013);浙江海洋大学人才引进科研基金(2016-2017);浙江省一流学科(A类)大学生创新性科研项目(编号:11034060216)。
作者简介:林彦均(1998—),女,福建宁德人,主要从事海洋资源与环境研究。E-mail:esperzione@163.com。
通信作者:杨天燕,博士,高级工程师,主要从事鱼类种群遗传学研究。E-mail:hellojelly1130@163.com。
龙头鱼[Harpadon nehereus (Hamilton,1822)]俗称狗母鱼、豆腐鱼、鼻涕鱼等,是海洋近岸或河口区常见的底层中小型经济鱼类,我国黄海南部、东海和南海均产之,龙头鱼尤其喜欢栖息于泥沙底质的环境中,具有较强的温度和盐度适应性[1]。龙头鱼肉质松软、营养丰富、鲜食美味,其干制品“龙头烤”更是倍受消费者喜爱。近年来,由于过度捕捞和环境污染等因素的影响,导致东海区资源结构及鱼类种群分布发生改变[2-3]。传统经济物种资源的衰退,使得龙头鱼等中小型经济鱼类的产量呈逐年上升的趋势,逐渐在我国东海和南海渔业中占据重要地位。目前,龙头鱼已经成为江浙沿海张网渔获物中数量较大的优势种类之一,多年来的渔业调查数据显示,龙头鱼资源数量稳定,具有较好的开发前景和潜力[4]。为了保护和合理开发利用这一重要渔业资源,避免出现种质资源的衰退和低龄化、小型化现象,需要对龙头鱼种群结构和遗传现状进行调查研究。目前,国内外针对龙头鱼遗传学方面的研究不多,主要集中在其线粒体DNA全序列的扩增测定、微卫星(microsatellite)和序列相关扩增多态性(sequence-related amplified polymorphism,简称SRAP)分子标记的开发和应用领域[5-7]。
简单序列重复区间标记(inter-simple sequence repeat,简称ISSR)是Zietkeiwitcz等于1994年在简单重复序列(simple sequence repeats,簡称SSR)基础上发展起来的一种新型分子标记技术,该技术是利用包含微卫星重复序列并在3′或5′端锚定的寡聚核苷酸引物对基因组DNA进行PCR扩增的标记系统[8]。ISSR标记技术具有成本较低、操作简单、快速灵敏、多态性丰富、稳定性较高等系列优点[9],被认为是集合了随机扩增多态性DNA标记(random amplified polymorphic DNA,简称RAPD)技术和SSR技术二者优势的一种新型分子标记技术。目前,ISSR己在多种动植物的种质鉴定、遗传多样性检测、基因定位、遗传多样性亲缘关系分析和遗传图谱构建等研究方面得到广泛应用[10-11]。
退火温度(annealing temperature)是PCR反应过程中不可缺少的组成部分,也是决定ISSR试验成功与否的重要因素之一[12-13]。一般而言,人工设计的低于20个碱基的ISSR-PCR引物,可以根据DNA熔解温度(Tm)计算公式Tm=4(G+C)+2(A+T)[14]计算得出最佳退火温度。但是在实际操作中,由于受到缓冲液、引物和DNA模板浓度等因素的影响,该温度往往不能保证PCR扩增的有效性。因此,合理设计ISSR-PCR退火温度,对于节约试验成本、提高试验效率具有重要意义。本研究开展了龙头鱼ISSR-PCR多态性引物退火温度的筛选,试验结果以期为进一步研究龙头鱼遗传多样性和近缘物种间的遗传变异分析奠定基础。
1 材料与方法
1.1 样本采集和DNA提取
本研究所用龙头鱼样本于2018年10月采集自浙江舟山沿海,剪取新鲜龙头鱼背部肌肉组织并将其浸泡于无水乙醇中,于-20 ℃冰箱冷冻保存。参考《分子克隆实验指南(第四版)》中的相关操作要求,采用传统的Tris平衡酚-三氯甲烷法提取基因组DNA[15],将自然风干的30 ng DNA溶解于 100 μL灭菌水中,并于4 ℃保存。使用分光光度计测定波长为260、280 nm处的吸光度比值D260 nm/280 nm以估算基因组DNA的纯度和浓度,并采用1%琼脂糖凝胶电泳(电压为8~10 V/cm)检测DNA的提取效果,综合选择提取效果较好的DNA作为PCR扩增的模板。
1.2 引物序列和退火温度的设定
依据加拿大哥伦比亚大学(University of British Columbia,简称UBC)公布的约100条ISSR引物,并参考海水鱼类中具有较好扩增多态性的引物,采用温度梯度PCR的方法,筛选出适合龙头鱼的、有较高分辨率和特异性的12条ISSR引物(表1),从而建立具有最佳退火温度的ISSR-PCR反应体系。
參考文献:
[1]陈素芝. 中国动物志 硬骨鱼纲:灯笼鱼目 鲸口鱼目 骨舌鱼目[M]. 北京:科学出版社,2002:83-85.
[2]刘 勇,程家骅. 东海、黄海秋季渔业生物群落结构及其平均营养级变化特征初步分析[J]. 水产学报,2015,39(5):691-700.
[3]林龙山,程家骅,凌建忠. 东海区底拖网渔业资源变动分析[J]. 海洋渔业,2007,29(4):371-374.
[4]陈 玲,水柏年,董文霞. 龙头鱼生长特征及资源的可持续利用[J]. 科技与管理,2012(3):68-70.
[5]Bo Z,Xu T J,Wang R X,et al. Complete mitochondrial genome of the Bombay duck Harpodon nehereus (Aulopiformes,Synodontidae)[J]. DNA Sequence,2013,24(6):660-662.
[6]Xu T J,Sun D Q,Li H Y,et al. Development and characterization of microsatellite markers for the lizardfish known as the Bombay duck,Harpadon nehereus (Synodontidae)[J]. Genetics & Molecular Research,2011,10(3):1701-1706.
[7]Zhu Z H,Li H Y,Qin Y,et al. Genetic diversity and population structure in Harpadon nehereus based on sequence-related amplified polymorphism markers[J]. Genetics & Molecular Research,2014,13(3):5974-5981.
[8]Zietkeiwitcz E,Rafalski J A,Labuda D. Genome fingerprinting by simple sequence repeat (SSR) anchored polymerase chain reaction amplification[J]. Genomics,1994,20(2):176-183.
[9]蒋彩虹,王元英,孙玉合. SSR和ISSR标记技术应用进展[J]. 中国烟草科学,2007,28(2):1-5.
[10]Reddy M P,Sarla N,Siddiq E A. Inter simple sequence repeat (ISSR) polymorphism and its application in plant breeding[J]. Euphytica,2002,128(1):9-17.
[11]Stolpovskiǐ I A,Lazebnyǐ O E,Stolpovskiǐ K,et al. The use of the ISSR-PCR method for identifying domesticated animal breeds and species,inferring their population structures,and assessing gene pool similarity[J]. Genetika,2010,46(6):825-833.
[12]Saiki R K. The design and optimization of the PCR[M]//Erlich H A. PCR technology. New York:Stockton Press,1989.
[13]Bartlett J M S,Stirling D. PCR protocols: volume 226[M]. 2nd ed. Totowa,NJ:Humana Press Inc,2003:89-100.
[14]Liu Y G,Chen S L,Li J,et al. Genetic diversity in three Japanese flounder (Paralichtlrys olivaceus) populations revealed by ISSR markers[J]. Aquaculture,2006,255(1/2/3/4):565-572.
[15]萨姆布鲁克 J,格林M R. 分子克隆实验指南[M]. 贺福初,陈 薇,杨晓明,译. 4版. 北京:科学出版社,2017.
[16]李爱丽,姜 涛,马峙英,等. 提高PCR产物的几种有效方法[J]. 生物技术通报,2003(2):33-35.
[17]常玉卉,张艾英,刘 盼,等. ISSR荧光标记毛细管电泳与谷子品种遗传多样性分析[J]. 分子植物育种,2017,15(8):3302-3308.
[18]王玉民,姜 昱,康岭生,等. 利用ISSR技术探讨大豆属植物的亲缘关系[J]. 吉林农业科学,2007,32(6):30-32,38.
[19]邹 奕,马龙彪,江 伟,等. ISSR分子标记技术在甜菜育种中的应用[J]. 中国糖料,2018,40(3):75-77,80.
[20]杨玉玲,马祥庆,张木清. ISSR分子标记及其在树木遗传育种研究中的应用[J]. 亚热带农业研究,2006,2(1):18-24.
[21]刘红艳,杨 东,张繁荣,等. 鱇浪白鱼ISSR-PCR最佳退火温度的筛选[J]. 江汉大学学报,2010,38(1):69-73.
[22]郁建锋,鲍 峰,韩晓磊,等. 松江鲈鱼ISSR-PCR反应体系的建立及优化研究[J]. 安徽农业科学,2008,36(33):14420-14421,14465.
[23]彭 海,张 静,张 梁,等. 长豇豆品种ISSR-PCR最佳退火温度的筛选[J]. 长江蔬菜,2007(9):49-51.
