利用mtCOI PCR-RFLP技术鉴别草地贪夜蛾与其他3种形态相近昆虫
2020-04-23孔子艺朱军生迟秀丽孔宝玉
孔子艺, 王 鹏, 朱军生, 国 栋, 迟秀丽, 孔宝玉, 胡 磊, 褚 栋*
1山东省植物病虫害综合防控重点实验室,青岛农业大学植物医学学院,山东 青岛 266109; 2山东省植物保护总站,山东 济南 250100; 3胶州市农业局植保站,山东 胶州 266300
草地贪夜蛾Spodopterafrugiperda(Smith),又称秋粘虫,属鳞翅目Lepidoptera夜蛾科Noctuidae,原产于美洲热带和亚热带地区(Nagoshietal.,2017b),近年来成为一种重大入侵农业害虫。受国际交流、进出口贸易和季风气候的影响,该虫于2016年首次在非洲出现,后扩散到撒哈拉以南的大部分国家以及亚洲的印度、也门、缅甸等国家(郭井菲等,2018; Goergenetal.,2016)。2018年年底该虫零星传入我国云南(吴秋琳等,2019),随后在国内扩散蔓延。截至2019年7月5日,已入侵我国22个省的1128个县区,危害面积达55.6万hm2(王磊等,2019)。草地贪夜蛾具有杂食性、迁飞性、暴发性以及繁殖力强等特点,能危害多种植物,尤其导致玉米等禾本科作物严重减产(郭井菲等,2019a),对我国玉米等作物安全生产构成严重威胁。当前,全国各地广泛开展了草地贪夜蛾的田间调查与监测工作,预防其进一步的扩散蔓延。
最近国内调查发现,草地贪夜蛾与甜菜夜蛾SpodopteraexiguaHübner、斜纹夜蛾Spodopteralitura(Fabricius)、粘虫Mythimnaseparata(Walker)常混合发生,形态极为相似,而传统的形态学鉴定方法受到多种因素的影响,在调查时易将这4种害虫混淆,不能准确快速地识别(郭井菲等,2019b)。因此,该虫的田间调查、监测亟需快速准确的识别技术。目前,物种分子鉴定包括扩增片段长度多态性(amplified fragment length polymorphisms, AFLP)(杨兆富和张雅林,2005)、磷酸甘油醛异构酶基因(triosephosphate isomerase, Tpi)(Nagoshietal., 2012)以及线粒体细胞色素氧化酶亚基I基因(mitochondrial cytochrome oxidase subunit I, mtCOI)(马英等,2018; Meagher & Gallo-Meagher,2003)测序等方法。近年来,PCR-RFLP(polymerase chain reaction-restriction fragment length polymorphism)(秦丽等,2013)、SCAR(sequence characterized amplified region)(刘循等,2009)等方法也常用于种间快速鉴别。
本研究首先下载并比对了草地贪夜蛾与甜菜夜蛾、斜纹夜蛾、粘虫的mtCOI基因序列(截至2019年6月19日),其次分析了草地贪夜蛾mtCOI基因序列的酶切位点,筛选确定了能区分该虫与其他3种害虫的候选酶切位点,然后根据mtCOI基因目的片段设计出新的上游引物并进行PCR-RFLP验证。结果表明构建的mtCOI PCR-RFLP方法可快速将草地贪夜蛾从其他3个形态相近昆虫中鉴别出。本研究结果为近期传入我国的草地贪夜蛾的快速鉴别提供了方法。
1 材料与方法
1.1 mtCOI基因序列下载与比对及酶切位点分析
从美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)下载草地贪夜蛾(362条)和甜菜夜蛾(387条)、斜纹夜蛾(116条)、粘虫(200条)的对应mtCOI基因序列(截至2019年6月19日),去除存在插入缺失等问题的低质量序列,删除两端多余的序列,利用软件DNAsp5分别将4种昆虫的mtCOI序列归为不同的单倍型并编号。利用软件MEGA 5.05将下载整理后的序列以邻接法(neighbor joining, NJ)建树,各分支置信度均进行1000次重复检验,并通过分析序列间的遗传距离确定为4种不同种的昆虫,个体数等于序列数。将序列分析结果分别显示为草地贪夜蛾与甜菜夜蛾、斜纹夜蛾、粘虫4种昆虫的所有mtCOI基因序列利用MAGE 5.05进行比对分析,通过酶切位点汇总在线工具(http:∥www.novopro.cn/tools/rest_summary.html)对草地贪夜蛾mtCOI基因序列进行限制性内切酶位点分析,进一步筛选可以区分草地贪夜蛾与其他3种形态相近昆虫的酶切位点。
1.2 mtCOI基因序列目的片段引物设计
参照NCBI中下载整理后所分析试虫的mtCOI基因序列,以包含Sbf I酶切位点的序列为模板,利用网站(http:∥primer3plus.com/cgi-bin/dev/primer3plus.cgi)对目的片段设计引物,其中上游简并引物为5′-ACCWTTAATATTAGGAGCYCCWGA-3′;因酶切位点与所分析mtCOI基因序列末端距离仅36 bp,无法设计便于实验和观察结果的下游引物,因此下游引物直接利用Nagoshietal. (2017a)实验中所设计的下游引物(5′-GATGTAAAATATGCTCGTGT-3′)。扩增产物预期长度为672 bp。经PCR验证,此对引物可以分别对草地贪夜蛾和甜菜夜蛾、斜纹夜蛾、粘虫的mtCOI基因序列进行有效扩增。
1.3 样本采集及基因组DNA提取
所有样品均采于2019年。包括草地贪夜蛾青岛种群、草地贪夜蛾玉溪种群、草地贪夜蛾日照种群、甜菜夜蛾青岛种群、斜纹夜蛾广东种群、斜纹夜蛾济南种群、粘虫济南种群和粘虫青岛种群共8个种群。从每个种群中随机选取3头,提取单头虫体腿部DNA。样品DNA的提取采用TIANamp Genomic DNA Kit试剂盒,单头虫体腿部用液氮研磨后,按照说明书进行标准操作提取DNA。
1.4 目的片段的mtCOI PCR-RFLP
对mtCOI目的片段采用TaKaRa Taq酶进行PCR扩增,PCR扩增的体系如下(13 μL):DNA模板2 μL,上下游引物各0.26 μL,dNTP 0.26 μL,TaKaRa Taq 酶0.13 μL,10×PCR Buffer (Mg2+)1.3 μL,ddH2O 8.79 μL。PCR反应程序:94 ℃预变性5 min,94 ℃变性1 min,50 ℃退火1 min,72 ℃延伸1 min,72 ℃延伸7 min,共35个循环,扩增片段为672 bp。每个个体的mtCOI基因序列PCR扩增产物共24个样本交由北京擎科生物科技有限公司测序,结果用于物种鉴定和序列分析。相关PCR扩增产物进一步用限制性内切酶Sbf I进行酶切,后使用2.0%琼脂糖凝胶电泳检测。观察并分析凝胶电泳结果。
2 结果与分析
2.1 mtCOI基因序列分析
从NCBI数据库下载于2019年6月19日前发布的草地贪夜蛾和甜菜夜蛾、斜纹夜蛾、粘虫的对应mtCOI基因序列。因草地贪夜蛾与其他3种形态相近鳞翅目昆虫均具有部分未知序列,已知mtCOI基因序列经对齐筛选整理后仅可保留分析538 bp,例如草地贪夜蛾mtCOI基因序列GU439148.12其中的538 bp,如下:TATTGTAACAGCCCATGCTTTTATTATAATTTTTTTTATAGTTATACCAATTATAATTGGAGGATTTGGAAATTGACTTGTACCTTTAATATTAGGAGCCCTGATATAGCTTTCCCACGTATAAATAATATAAGTTTTTGACTTTTACCCCCATCTTTAACTTTATTAATTTCTAGTAGCATTGTAGAAAATGGAGCAGGAACTGGATGAACAGTTTACCCCCCCCTCTCCTCTAATATTGCTCATGGGGGTAGTTCAGTAGATTTAGCTATTTTCTCACTTCATTTAGCTGGAATTTCATCTATTTTAGGAGCTATTAACTTTATTACCACTATTATTAATATACGATTAAATAATTTATCATTTGATCAAATACCTTTATTTATTTGAGCTGTAGGTATTACCGCATTTTTATTATTATTATCTTTACCTGNTTTAGCTGGAGCTATTACTATATTACTTACTGATCGAAATCTAAATACATCATTTTTCGATCCTGCAGGAGGAGGNGATCCTATTCTTTATCAACATTTATTT。
利用软件DNAsp5分别将4种昆虫的mtCOI序列归为不同的单倍型并编号,对整理后mtCOI基因序列利用软件MEGA 5.05进行系统树发育分析判断不同物种(图1),分别得到草地贪夜蛾、甜菜夜蛾、斜纹夜蛾、粘虫的mtCOI基因序列117、281、101、56条。
2.2 mtCOI酶切位点分析
通过酶切位点汇总在线工具(http:∥www.novopro.cn/tools/rest_summary.html)对比寻找得出:Nagoshietal. (2017a)上下游引物获得811 bp的mtCOI片段(图2),所有草地贪夜蛾个体在该片段的556~561 bp处均存在Sbf I内切酶酶切位点(表1),因有些单倍型中所包含的mtCOI基因序列过多,此处仅列举具有代表性的序列;其他3种形态相近昆虫均无Sbf I酶切位点(图2)。草地贪夜蛾PCR产物经过Sbf I内切酶酶切,可出现420 bp左右的特征带;斜纹夜蛾、甜菜夜蛾、粘虫种群均不能被Sbf I内切酶酶切。
2.3 mtCOI基因序列分析
从每种昆虫采集样品的每个种群中随机选取3头进行mtCOI扩增、测序,共得到24条序列,系统发育分析确定分别为9个草地贪夜蛾个体、3个甜菜夜蛾个体、6个斜纹夜蛾个体及6个粘虫个体。对样品的mtCOI基因序列对比分析发现,Nagoshietal. (2017a)上下游引物获得811 bp的mtCOI片段556~561 bp处,所有草地贪夜蛾个体(9个个体)的mtCOI基因序列均存在Sbf I酶切位点(且561~811 bp间均不存在此酶切位点),其余3个物种所有个体(15个个体)在此处均无Sbf I酶切位点(图2)。
2.4 基于mtCOI PCR-RFLP鉴定及分析
新设计的上游引物与下游引物结合可分别对草地贪夜蛾与甜菜夜蛾、斜纹夜蛾、粘虫进行有效扩增(图3)。图4表明,将草地贪夜蛾的PCR扩增产物酶切后电泳,结果中出现一条420 bp左右的特征带;其他3种形态相近昆虫的PCR扩增产物酶切后电泳均不出现此特征带。因此,mtCOI PCR-RFL技术可以区分草地贪夜蛾与其他3种形态相近昆虫,若出现此420 bp左右的特征带,即为草地贪夜蛾;反之,则为其他3种形态相近昆虫。

图1 基于mtCOI对草地贪夜蛾与其他3种形态相近昆虫的系统发育分析
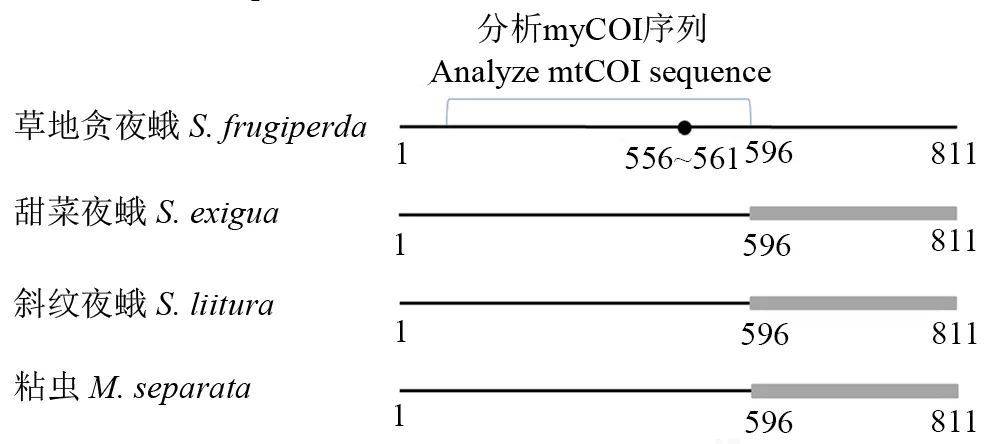
图2 Sbf I酶切位点的分布

表1 草地贪夜蛾与其他3种形态相近昆虫mtCOI基因序列代表Sbf I酶切位点

图3 新设计上游引物位置

图4 草地贪夜蛾与其他3种形态相近昆虫mtCOI PCR-RFLP电泳图谱
3 讨论
草地贪夜蛾的毁灭性极强,是威胁世界粮食作物生产的跨国界迁飞性重大害虫。因为该虫刚侵入我国不久,且与甜菜夜蛾、斜纹夜蛾、粘虫的体长、体色、翅展长度等形态特征相近(马丽等,2016; Yin,2010; 赵胜园等,2019),田间为害特点相似(郭井菲等,2019b),传统的形态学鉴定方法受到虫态等限制,很容易与这3种鳞翅目害虫混淆,不能对其进行准确识别。因此,对草地贪夜蛾迅速准确地进行物种识别,快速鉴定当地是否有草地贪夜蛾入侵,是对该虫进行监测、预警和防治的关键,具有重要理论意义和现实意义。
目前,可应用于物种鉴定的分子生物学技术方法很多,如AFLP(杨兆富和张雅林,2005)、RAPD(Qiuetal.,2003)、SSR(丁思敏等,2018)等,但是这些技术操作复杂、成本高、速度相对较慢或其他一些因素限制其大规模应用,而PCR-RFLP技术具有快速、灵敏、简便、稳定等特性(周新改等,2012),是国内外广泛采用的一种快速高效物种鉴定分子生物学手段。本研究对草地贪夜蛾及其他3种形态相近鳞翅目昆虫的mtCOI基因序列分析发现,所有草地贪夜蛾在556~561 bp处均存在Sbf I酶切位点,而其他3种害虫均无此酶切位点,实验也证实所选取的上下游引物通用性强,在草地贪夜蛾及甜菜夜蛾、斜纹夜蛾、粘虫中均能进行有效扩增,利用Sbf I作为内切酶的PCR-RFLP方法可以鉴别草地贪夜蛾与其他3种形态相近鳞翅目昆虫。
本研究中,由于甜菜夜蛾、斜纹夜蛾、粘虫mtCOI在所用片段的596 bp后未知,若在此后存在酶切位点,电泳结果也有可能出现1条>457 bp和1条<216 bp的2条带,在进行物种判断的时候需要注意。此外,本研究仅对草地贪夜蛾及甜菜夜蛾、斜纹夜蛾、粘虫的mtCOI基因已知序列进行了对比分析,如果存在其他物种,也可能影响该技术的应用,尚需对其他物种进行验证。
致谢:感谢为本研究提供斜纹夜蛾样品的仲恺农业工程学院农业与生物学院申建梅博士,以及参与样品采集的所有人员。
