基于CNCPS-S模型对全混合日粮蛋白质在绵羊瘤胃内有效降解率的预测研究
2020-04-17张晓娟高新梅贺刚刚
■张晓娟 程 曾 高新梅 贺刚刚 高 巍
(石河子大学动物科技学院,新疆石河子832003)
瘤胃蛋白质(CP)降解率是评定反刍动物饲料营养价值和确定反刍动物蛋白质需要量的重要参数,对其准确预测有助于生产者获取瘤胃微生物蛋白质合成所需氮源及小肠可代谢蛋白质需要量,从而更好的配制日粮[1]。利用数学模型进行预测已被证明是一种既能提高动物生产性能又能指导科学饲养管理简单而有力的工具[2]。在众多模型体系当中,康奈尔净碳水化合物-净蛋白质体系(Cornell net carbohydrate and pro⁃tein system,CNCPS)[3]应用较为广泛,可预测奶牛和肉牛的营养需求、生产性能以及养分排泄等[4-5]。该体系瘤胃降解模型综合考虑了饲料食糜外流速度(Kp)、各蛋白质组分的含量及其瘤胃降解速率(Kd)等因素,使分析过程更加科学,操作简便且不需要瘘管动物[6]。Shannak 等[7]对饲料CNCPS 各蛋白组分含量和瘤胃可降解蛋白质含量进行了相关分析,证实CNPCS各蛋白质组分含量能准确预测出饲料瘤胃非降解饲料蛋白质含量。Polat等[8]研究表明,利用CNCPS可以准确预测土耳其阉羊对谷物类饲料各蛋白组分的有效降解率。在CNCPS牛用体系基础上Cannas等又开发了CNCPS绵羊体系(CNCPS for sheep,CNCPS-S)[9]并更名为小反刍动物营养系统(Small ruminant nutrition system,SRNS)[10]。由于动物饲养环境各异、饲草料种类也不同,国内对CNCPS-S体系下各种饲料原料化学组分及生物学指标亟待建立和填补。与此同时,对该模型预测饲料蛋白质在绵羊瘤胃CP降解的研究也较少,因此验证该预测模型的可行性是十分必要的。
全混合日粮饲喂技术已在绵羊生产中广泛应用[11],对其蛋白质消化利用研究具有重要的现实意义和生产指导作用。由于缺乏各种单一饲料原料的化学组分和生物学价值等参数信息,我们设计了三种不同精粗比的全混合日粮(TMR)作为评价对象,来探讨模型对TMR 的CP 降解预测能力,同时也避免了各种饲料原料间的组合效应干扰。先应用CNCPS-S 预测TMR 的CP 在绵羊瘤胃的降解率,再与尼龙袋法实测CP 瘤胃的有效降解率进行对比验证,分析预测模型的准确性与适用性。以期为进一步准确预测绵羊的CP 肠道消化率提供模型参数,同时为CNCPS-S 在我国的推广及合理有效利用TMR提供一定参考依据。
1 材料与方法
1.1 试验动物与饲养管理
选取3只40 kg左右的哈萨克公羊作为试验动物,代谢笼内单笼饲养,检疫驱虫后手术安装永久性的瘤胃瘘管。使用配制好的TMR1作为试验的基础饲粮,每天09:00、21:00分别等量饲喂日粮,自由饮水。
1.2 试验日粮
从新疆当地采集饲料样品(小麦秸、棉籽壳、苜蓿干草、精料补充料)制备成三种不同精粗比(分别为30∶70、35∶65、40∶60)的全混合日粮(TMR1、TMR2、TMR3)(见表1)。
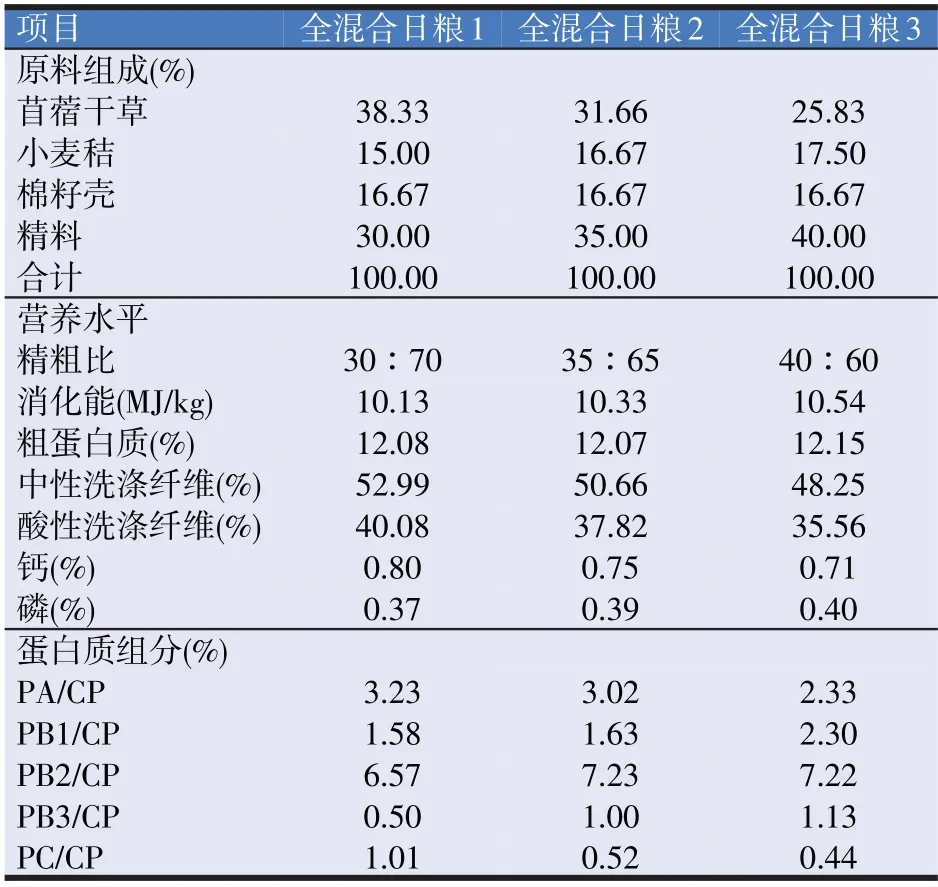
表1 TMR的组成及营养水平(风干基础)
粗蛋白质(CP)参照《饲料分析及饲料质量检测技术》进行测定。中性洗涤纤维(NDF)和酸性洗涤纤维(ADF)参照Van Soest 等[12]的方法测定。可溶性粗蛋白质(SOLP)、非蛋白氮(NPN)、中性洗涤不溶蛋白质(NDFIP)和酸性洗涤不溶蛋白质(ADFIP)参照Lici⁃tra 等[13]的方法测定。CNCPS 模型将粗蛋白质分为PA、PB1、PB2、PB3 和PC 五个部分,各蛋白质组分含量根据Polat等[14]的方法进行计算。
1.3 试验方案
参照高新梅等[15]的方法测定瘤胃食糜外流速度(Kp)。按唐福[16]方法进行尼龙袋试验,测定瘤胃动态降解参数。CNCPS 的各蛋白组分按照Polat 等[14]的方法进行计算。
1.4 计算与分析
按照Ørskov等[17]的方法计算瘤胃降解参数:

式中:t——营养成分在瘤胃内的停留时间(h);
P——t时刻营养成分的动态瘤胃降解率(%);
a——营养成分的快速降解组分(%);
b——营养成分的慢速降解组分(%);
c——b的降解速率(%/h)。下式同。
瘤胃食糜的外流速度(Kp)的计算公式:

式中:Yb——t时刻瘤胃内容物中镱的浓度(mg/kg);
C0——零时刻瘤胃内容物中镱的浓度(mg/kg);
t——停止灌注后瘤胃内容物的采样时间(h)。
采用SAS 统计软件的NLIN 过程错位法(DUD)拟合求出Kp。

康奈尔模型[18]的瘤胃降解规律方程:
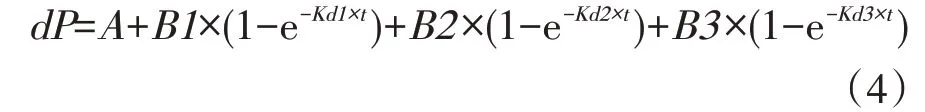
将CP 动态瘤胃降解率(dP)及饲料A,B1、B2 和B3 组分占CP 的比例代入方程,利用SAS 统计软件包的NLIN 程序拟和出饲料B1、B2 和B3 组分的瘤胃降解速率Kd1、Kd2、Kd3。
参照Cannas 等[19]的公式预测粗饲料食糜外流速度(Kpf)和精饲料食糜外流速度(Kpc):
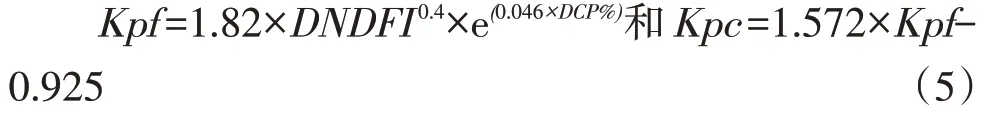
式中:DNDFI——中性洗涤纤维占日粮摄入量的比例(以FBW的百分比表示,%);
DCP——CP占日粮的比例(%)。
按照TMR 饲料精粗比例分别计算得3 种TMR的Kp。
瘤胃可降解蛋白数量[14]的预测公式:

式中:RDPA、RDPBj——分别为PA、PB1、PB2、PB3 组分的瘤胃降解量(g/d);
RDPEP——瘤胃可降解蛋白质的总量(g/d);
I——日粮的采食量(g/d);
Kpj——预测食糜外流速度(%/h)。下式同。
CP瘤胃降解率的预测(RD):
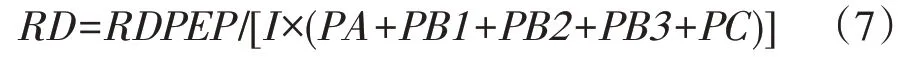
式中:I×(PA+PB1+PB2+PB3+PC)——饲料组分中蛋白质的总摄入量(g/d)。
1.5 数据统计分析
用Excel 2007 录入三种TMR 的蛋白质组分基础数据,采用SAS 9.4 软件包的非线性回归程序计算出各时间点的动态降解率和有效降解率,再进行单因素方差分析(One-way Anova),Duncan's方法进行多重比较。实测值和预测值的对比分析先用Graphpad Prism 6软件进行配对样本t检验,比较两个样本均数之间差异,绘制散点图,再运用线性回归对回归参数(相关系数、截距、斜率、决定系数和均方根误差)进行分析。
2 结果
2.1 DM和CP的瘤胃动态降解特性
TMR 精粗比水平的不同对DM 降解率产生了极显著影响(P<0.01)(图1)。TMR1、TMR2 和TMR3 的DM 降解率在48 h 后趋于平缓,TMR3 在各个时间点的DM降解率都分别高于另外两组,尤其是在16、60、72 h差异较大(P<0.05)。随着底物在瘤胃中反应的进行,TMR1 的CP 降解率在36 h 以前有明显的上升趋势,36 h以后CP降解率达到平台期;TMR2在60、72 h的CP 降解率显著高于另外两组(P<0.05);TMR3 在12、16 h的CP降解率均高于另外两组(P<0.05)。本试验DM和CP的瘤胃降解程度取决于饲料精料比例,饲料发酵的难易程度和饲料在瘤胃内的滞留时间等多种因素,伴随着CP降解率的升高同种饲料DM降解率也会升高,这与张颖等[20]报道的DM降解率和CP降解率呈正相关的研究结果是相似的。
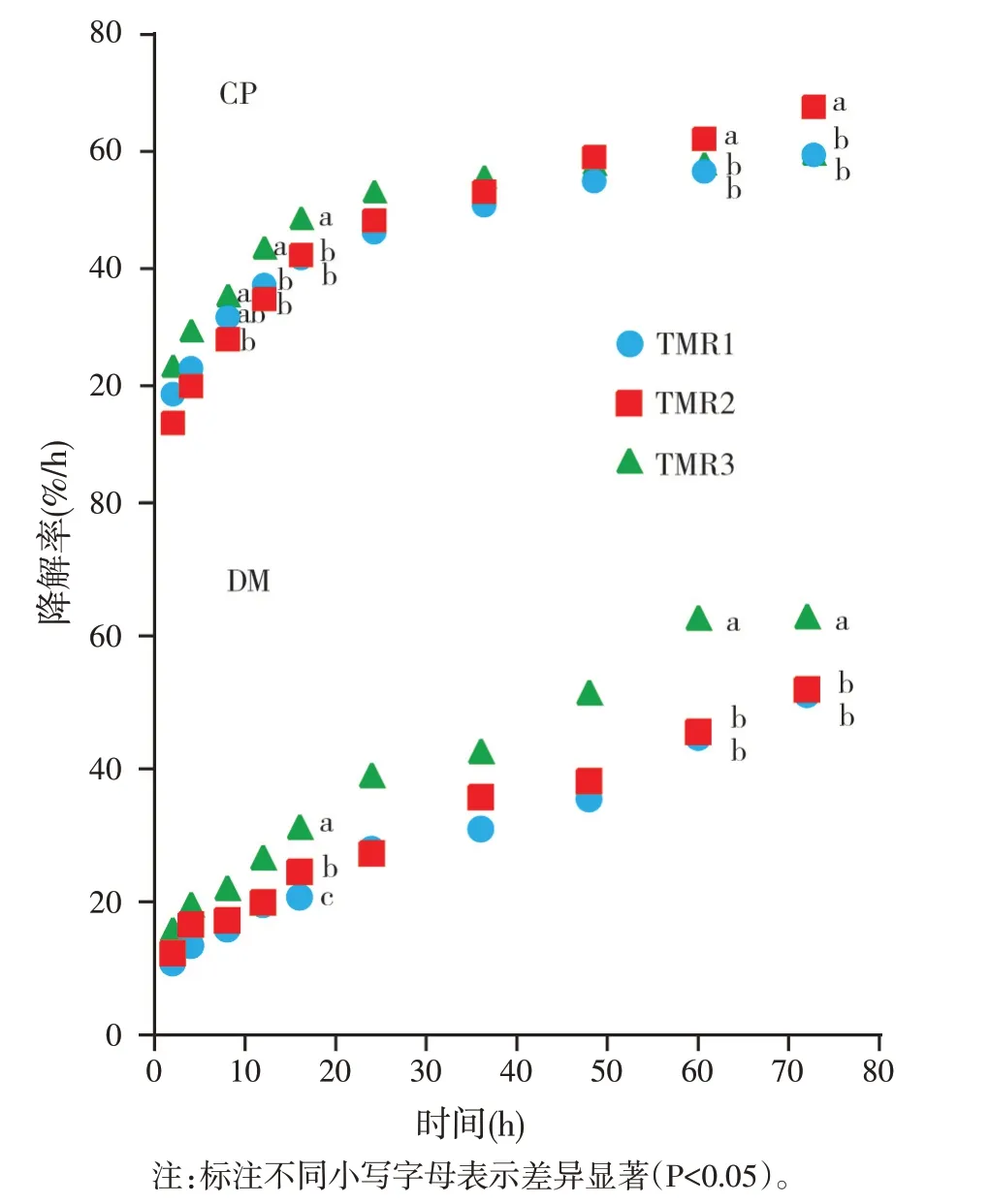
图1 3种TMR的DM和CP在瘤胃中的动态降解率
2.2 不同TMR的瘤胃食糜外流速度
根据不同时间点固相食糜中的Yb 浓度,采用SAS 统计软件的NLIN 过程拟合了Kp(实测值),并应用CNCPS-S模型得到了Kp的预测值(见图2)。TMR1、TMR2 和TMR3 实测Kp 平均值分别为3.58、3.83 和4.05;TMR1、TMR2 和TMR3 预测Kp 平均值分别为1.62、1.63和1.64。可以看出Kp预测值之间的差值很小,三者Kp的预测值均显著低于实测值(P<0.05)。

图2 三种TMR的瘤胃食糜外流速度
2.3 蛋白质组分瘤胃降解速率的估算
按CNCPS体系对饲料蛋白质组分分类,A组分在瘤胃中迅速降解,C 组分不能被瘤胃微生物降解,用最小二乘数据拟合估计了三种TMR 的B1、B2、B3 蛋白组分的瘤胃降解速率,TMR1、TMR2和TMR3的Kd1平 均 值 分 别 为140.44%/h、155.65%/h、185.92%/h;TMR1、TMR2和TMR3的Kd2平均值分别为16.29%/h、14.33%/h、13.29%/h;TMR1、TMR2 和TMR3 的Kd3 平均值分别为3.77%/h、1.47%/h、1.39%/h。
2.4 三种TMR的DM、CP在瘤胃的有效降解率

表2 3种TMR中DM、CP在瘤胃的有效降解率(%)
由表2可以看出,TMR3的DM有效降解率显著高于另外两组TMR(P<0.05),然而CP 有效降解率未表现出显著差异(P>0.05)。
2.5 CNCPS-S对CP瘤胃降解率的预测
由于预测Kp值偏低(见图2),所以本试验采用两种方式对CP降解率进行预测,方式一为代入预测Kp计算CP降解率预测值,方式二为代入实测Kp计算CP降解率预测值(见表3)。RDPB2 在TMR1、TMR2 和TMR3 之间未表现出差异(P>0.05),但RDPA、RDPB1、RDPB3、RDPEP在三种TMR之间均存在显著差异(P<0.05);还可看出蛋白质瘤胃降解率预测值在三种TMR 之间存在极显著差异(P<0.01),表现为TMR1>TMR3>TMR2。(方式一和方式二结果相似)。

表3 预测蛋白质在瘤胃中的降解量和降解率
2.6 CP瘤胃降解率预测值与实测值之间的比较
对两种方式得到的CP瘤胃降解率预测值分别与实测值进行了配对样本t检验(见表4)。方式一中可以看出,三种TMR的预测值均极显著高于实测值(P<0.01);平均数偏差(预测值减去实测值)分别为(38.93±1.80)%、(17.99±2.08)%和(22.64±4.96)%,均为正值;说明模型高估了瘤胃降解率,预测的准确性较差。方式二的结果显示三种TMR的预测值和实测值之间均未表现出显著差异(P>0.05);平均数偏差(预测值减去实测值)较方式一有所降低,分别为(33.25±0.47)%、(11.16±0.45)%、(11.03±0.80)%,说明方式二预测效果更好。
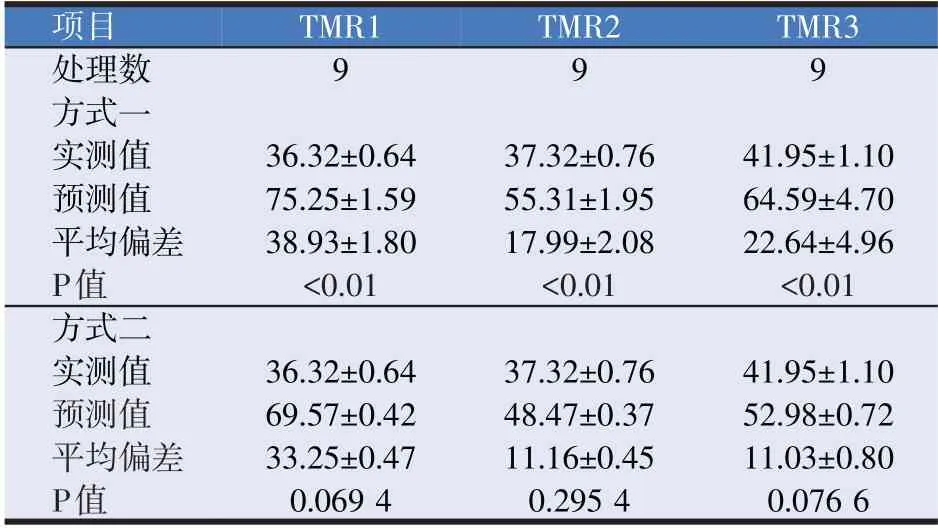
表4 瘤胃降解率预测值与实测值之间的比较(%)
图3 显示了3 种TMR 蛋白质瘤胃降解率预测值(X)与实测值(Y)的对比情况,可以看出所有点均落在Y=X线的下方,表明CNCPS-S 高估了CP瘤胃降解率。方式二TMR2和TMR3的点分布范围较方式一更集中于Y=X线的附近,表明通过方式二预测蛋白质瘤胃降解率准确性更好。
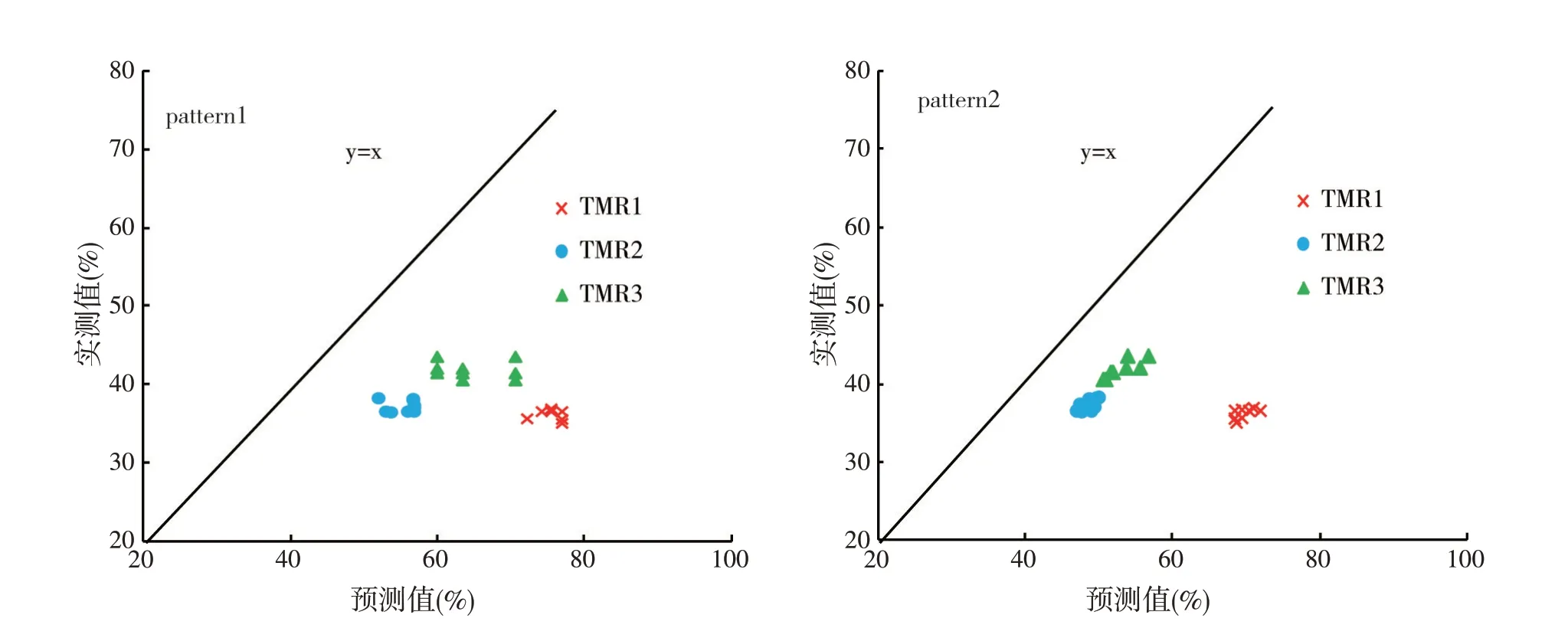
图3 瘤胃降解率预测值与实测值的比较
对两种方式得到的CP瘤胃降解率预测值分别与实测值进行线性回归(见表5)。方式一中TMR1、TMR2 和TMR3 实测值和预测值的相关系数均较低(分别为0.14、0.02、0.12),说明模型预测能力较差。方式二中预测值和实测值相关系数变化范围较大,TMR1、TMR2 表现为中等程度相关(分别为0.55 和0.66),TMR3的相关系数较高(0.84),表明预测值和实测值进行相关分析是较为可信的。
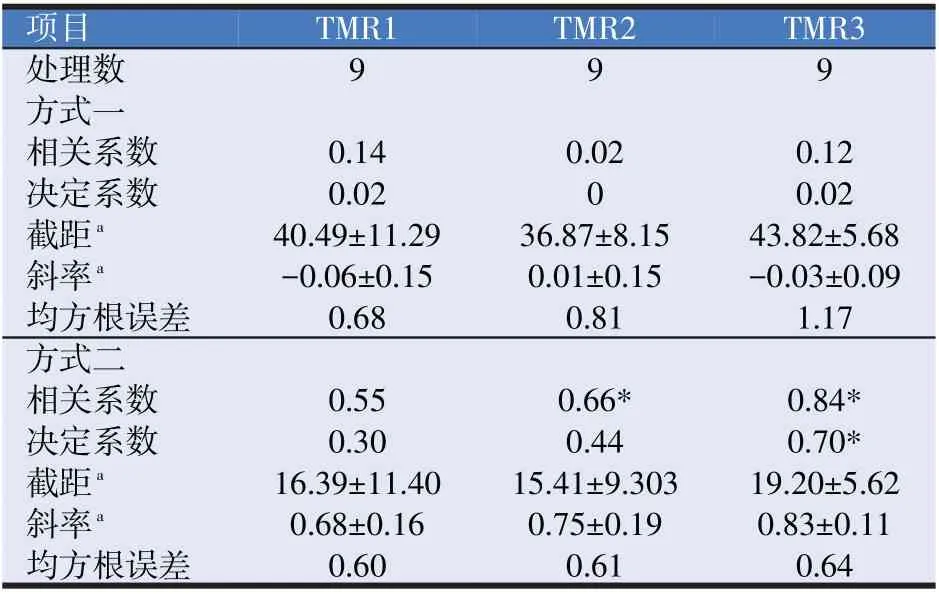
表5 瘤胃降解率预测值和实测值的回归参数
方式一中三种TMR的R2分布在0到0.02之间,说明回归方程可靠性很低。方式二中TMR1和TMR2的R2较低(分别为0.30 和0.44),但TMR3 的R2较高(0.70),说明模型对TMR3有较好的预测能力。
方式一的TMR1 和TMR3 的斜率为负值,表明预测值与实测值呈负相关关系,建立的回归方程无意义。方式二的三种TMR 斜率分别为0.68±0.16、0.75±0.19、0.83±0.11,均接近于1,说明有较好预测能力。
方式一和方式二的RMSE均较小,范围在0.60%~1.17%之间,低的RMSE 说明CNCPS-S 对蛋白质降解率的预测比较准确。
3 讨论
3.1 对食糜外流速度影响因素的研究
瘤胃食糜外流速度是计算饲料营养物质有效降解率的关键参数。前人研究表明,日粮精粗比[21]、饲料种类[22]、标记物的种类[15]和粗饲料的长短[23]等因素都能影响Kp值的大小。Colucci等[24]研究发现Kp值随粗料比例的增加而增加,这与本试验结论相反,本试验的Kp值随精料比例的增加而增加。高新梅[25]研究表明,不同精粗比的TMR 在等能等氮条件下,Kp 值随着NDF水平的降低而升高,这与本试验的研究结果一致。
准确预测蛋白质瘤胃降解率的关键因素在于对各蛋白组分Kd 和Kp 的估计[26]。显然本试验中CNCPS-S 低估了Kp(图2),而Kp 预测值偏低可能的原因在于:①动物的种类[27-28]。Kp 模型最初[29]是在CNCPS牛用体系中被提出,后来Cannas[30]报道当牛用体系的Kp 预测模型应用在小反刍动物上时,与实测值相比Kp值被低估了。②模型不具有针对性。Can⁃nas 和Van Soest 是基于不同动物类型(奶牛、水牛、绵羊、山羊)的数据而建立新的模型来预测小反刍动物的Kp[19],不仅仅是基于绵羊的数据而建立,可能会造成本试验预测值偏低。③CNCPS-S 建立了两种方程分别预测Kpf和Kpc,本试验采用的是全混合日粮,既包括精料也包括粗料,将Kpf和Kpc分别算出后,再乘以各自所占日粮的比例才能整合出总的Kp 值,可能导致预测效果较差。④物理有效纤维。在牛用体系中,Kp 方程使用物理有效纤维作为日粮颗粒大小的调节因子,这也是唯一能够解释颗粒大小对Kp 影响的因素。Van Soest 等[31]报道称在小型反刍动物和大型反刍动物之间可能有所不同。然而,羊用模型沿用了牛用模型的公式也可能会造成本试验Kp预测值偏低。显然,需要在这一领域进行更多的研究。
3.2 蛋白质组分瘤胃降解速率的研究
CNCPS v6.0 数据表[32]报道的饲料库Kd1、Kd2、Kd3 的 分 布 范 围 分 别 在130~300%/h、3~20%/h 和0.05~2%/h 之间,目前还没有全混合日粮各蛋白组分Kd 的评估数据。本试验三种TMR 的Kd1 值在CNCPS v6.0数据表范围之内,但均在200%/h以下,测定结果可能偏低。郑焕玲[33]应用CNCPS 预测奶牛常用单一饲料的Kd 值,拟合所得Kd1 和Kd2 与CNCPS同类饲料的相比符合良好,但Kd3差别较大。孙旭[34]对单一饲料拟合所得Kd3与CNCPS给出数据范围相比偏高,与本试验TMR1 所测Kd3(2%/h)高于CNCPS v6.0数据表报道的范围的结果一致,可能因为TMR 有些细小颗粒是难以消化的但从尼龙袋中溢出而假定被降解。后来Van Amburgh 等[32]又报道了CNCPS v6.1饲料数据库的更新情况并且总结出各个蛋白质组分的最新划分以及各组分的瘤胃降解速率,新的蛋白质组分[35-36]划分为PA1、PA2、PB1、PB2、PC,降解速率分别为200%/h、10~40%/h、3~20%/h、4~9%/h、0%/h。若将本试验中所得的蛋白质组分瘤胃降解速率与CNCPS v6.1的范围比较,可以看出本试验所测Kd1(140.44~185.92%/h)远高于CNCPS 给定的范围(3~20%/h),而且本试验所测Kd2(13.29~16.29%/h)也高于CNCPS给定的范围(4~9%/h),因此今后的研究工作中有必要使用新的蛋白质组分划分法进行蛋白质瘤胃降解速率测定。
3.3 蛋白质组分的瘤胃降解预测模型的准确性分析
理想的预测模型[37]需要满足3 个条件:①决定系数(R2)大于0.6;②截距接近于0;③斜率接近于1。R2的取值范围在0~1之间,R2越接近1,说明回归直线对实测值的拟合程度越好,用来衡量模型的预期值和实测值的差距。均方根误差(RMSE)也是用来衡量预测值和实测值偏差的一个指标,RMSE 越低表明预测效果越好。
本研究CP瘤胃降解率预测值和实测值的平均偏差较高;相关系数和决定系数均较低(范围在0~0.14之间);TMR1和TMR3的斜率为负值,截距远大于零;3 种TMR 蛋白质瘤胃降解率的点落在Y=X 回归线的下方(图3);以上结果均表明CNCPS-S 高估了CP 瘤胃降解率,模型预测能力较差(方式一)。因此又对预测模型进行改良(方式二),代入实测Kp重新预测瘤胃降解率(表3、表4、表5)。对比分析结果显示预测值和实测值平均偏差较低;相关系数范围在0.55~0.84 之间;决定系数在0.30~0.70之间;斜率接近1以及RMSE较低,表明预测模型基本可以接受但存在一定偏差。
最后,必须认识到蛋白质瘤胃降解率的预测并非易事,因为诸多因素的相互作用(饲料蛋白质组成和特性,抗营养因子,动物健康状况等)都会对预测模型准确性产生影响[38],目前关于CP 瘤胃预测模型的研究甚少,无法与前人相关成果对比分析,仍然需要我们进行大量的研究才能使该模型更好地在我国得到广泛应用。
4 结论
本研究对蛋白质瘤胃降解率的预测模型进行改良后显示出模型有较好预测能力,但仍未达到满意的程度。尚需进一步增加平行样本量以期提高预测模型准确性。
