利用RIL群体创制低山嵛酸花生新种质
2020-04-16郭建斌罗怀勇周小静陈伟刚淮东欣任小平姜慧芳
郭建斌 黄 莉 刘 念 罗怀勇 周小静 陈伟刚 吴 贝 淮东欣 任小平 姜慧芳
利用RIL群体创制低山嵛酸花生新种质
郭建斌 黄 莉 刘 念 罗怀勇 周小静 陈伟刚 吴 贝 淮东欣 任小平 姜慧芳*
中国农业科学院油料作物研究所/ 农业农村部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
花生是我国食用植物油的重要来源, 改善花生油脂品质是我国花生育种的重要目标。利用不同遗传背景的亲本(中花10号×ICG12625)杂交构建含有140个家系的重组自交系群体(RIL), 对超长链饱和脂肪酸(花生酸、山嵛酸和二十四碳烷酸)含量测定表明, RIL家系超长链饱和脂肪酸含量的变异范围为4.27%~7.05%, 均值为5.54%, 最低值分别比父本和母本低1.46和1.63个百分点, 降低率达25.27%和27.62%; 山嵛酸含量的变异范围为1.86%~3.37%, 均值为2.53%, 3份家系的山嵛酸含量稳定低于2% (QT0002、QT0075和QT0120), 比低值亲本中花10号分别低0.57、0.51和0.51个百分点, 降低率分别为23.45%、20.98%和20.98%。利用前期构建的遗传图谱, 通过WinQTLcart软件检测到一个与山嵛酸相关的稳定的QTL, 位于B04染色体13.31~16.34 M内, 该区间内含有131个预测基因。本结果为高油酸花生品种的山嵛酸遗传改良奠定了基础。
花生; 重组自交系; 脂肪酸; 山嵛酸
花生是我国主要的油料作物和经济作物, 花生籽仁中含有46%~58%的脂肪和22%~32%的蛋白质[1], 具有很高的营养价值和经济价值。随着人民生活水平的不断提高, 对花生生产提出了更多和更高的要求, 因此花生育种目标越来越高也越复杂。花生不同品种脂肪酸组分存在很大差异, 在花生脂肪酸组分中, 不饱和脂肪酸约占80%, 饱和脂肪酸约占20%[2], 对人体健康有利, 但与优质植物油[3](茶籽油饱和脂肪酸含量为7.5%~18.8%, 橄榄油饱和脂肪酸含量为15%)相比, 其饱和脂肪酸含量偏高。在花生油脂中, 亚油酸和花生烯酸为人体必需脂肪酸。亚油酸具有降低人体内血液胆固醇和甘油三酯的功能, 并能软化血管和阻止血栓的形成, 但亚油酸含量过高会降低油脂的耐储存性[4-7]; 油酸和花生烯酸能选择性地降低人体血液中有害的低密度胆固醇[8]。棕榈酸含量过高会导致心血管疾病的发生[9-10];超长链饱和脂肪酸(花生酸、山嵛酸和二十四碳烷酸)碳链长、饱和程度高、不易氧化分解, 摄入过量会对人体造成病变[11], 而山嵛酸在超长链饱和脂肪酸中占比最大, 因此花生油脂脂肪酸品质改良的重点是提高油酸和花生烯酸含量、减少棕榈酸和山嵛酸等超长链饱和脂肪酸含量。油酸含量和花生烯酸呈极显著正相关, 与棕榈酸呈极显著负相关, 但与山嵛酸无显著相关性[12-13]。由此可见, 油酸含量的提高不仅能增加花生烯酸含量还可降低棕榈酸的含量。近年来, 我国高油酸花生品种的选育速度加快, 培育出了如花育32、花育917、开农61、开农176、开农H03-3、冀花11、冀花13、中花24、豫花37等品种[14]。但是, 在这些高油酸花生品种中, 其山嵛酸含量没有明显改善, 因此高油酸花生品种的山嵛酸还有待进一步降低。目前关于低山嵛酸含量品种的选育还未见报道。郭建斌等[12]对黄淮及长江流域产区主推的60份花生品种测试表明, 山嵛酸的变异范围为1.98%~3.38%; Worthington等[15]对不同生态地理来源的82个基因型鉴定表明, 山嵛酸的变异幅度为1.3%~5.1%, 是花生育种工作中遗传变异最大的资源。所以降低山嵛酸含量的潜力是很大的。本研究选用普通油酸花生RIL群体(中花10×ICG12625), 对群体脂肪酸进行变异分析, 在群体中挖掘新的种质资源, 进而为培育出低山嵛酸新品种提供条件。
1 材料与方法
1.1 试验材料
以中花10号为母本、ICG12625为父本进行杂交, 采用单粒传法构建包含140个家系的重组自交系群体(RIL)。其中, 中花10号籽仁较大, 粉色种皮; ICG12625籽仁较小, 黑色种皮。
1.2 试验设计
于2015—2017年在中国农业科学院油料作物研究所试验田(湖北武汉)连续种植RIL群体和亲本材料, 每份材料1行, 行长2.50 m, 行距0.30 m, 完全随机区组设计, 2次重复, 常规田间管理。
1.3 脂肪酸测定及数据处理
根据国标法(GB/T 5510-2011)处理花生样品, 用Agilent 7890B型气相色谱仪分析花生脂肪酸甲脂,采用Microsoft Excel统计与计算数据, SPSS软件进行方差分析。
2 结果与分析
2.1 亲本及RIL群体中超长链饱和脂肪酸含量差异
中花10号和ICG12625在超长链饱和脂肪酸(VLCSFA)上无显著差异, 3年平均值分别为5.90%和5.73%, 在花生酸(C20:0)和山嵛酸(C22:0)上存在差异(图1)。140份RIL家系连续3年种子中超长链饱和脂肪酸均值的变异范围为4.27%~7.05%, 平均值为5.54%, 变异系数为9.84%。在RIL群体中出现了超长链饱和脂肪酸含量为4.27%的家系, 分别比父本和母本低1.46和1.63个百分点, 降低率达25.27%和27.62%, 群体中含量低于5%的家系有21份, 占比15%。RIL各家系超长链饱和脂肪酸含量呈正态分布, 偏度和峰度分别为0.49和0.33。相关性分析表明(图2), 超长链饱和脂肪酸与花生酸、山嵛酸和二十四碳烷酸(C24:0)均呈极显著正相关, 相关系数分别为0.75、0.92和0.69; 山嵛酸与花生酸和二十四碳烷酸也呈极显著正相关, 相关系数分别为0.52和0.64。超长链饱和脂肪酸中山嵛酸含量最高, 且与花生酸和二十四碳烷酸呈极显著正相关,所以降低山嵛酸含量是降低超长链饱和脂肪酸的关键。

图2 超长链饱和脂肪酸相关性分析
2.2 亲本及RIL群体山嵛酸含量差异
从表1可看出, 父本ICG12625连续3年山嵛酸含量均高于母本中花10号。在RIL群体中, 山嵛酸的变异均超出了亲本间差异, 表现出超亲优势。利用SPSS软件对RIL群体山嵛酸3年表型值方差分析表明, RIL群体差异达到极显著水平(=5.79,<0.0001)。2015年山嵛酸的变异范围为1.80%~3.51%, 平均值为2.53%, 高于双亲均值(2.44%), 家系之间山嵛酸含量相差1.71个百分点, 而两亲本相差0.53个百分点, 群体中山嵛酸含量低于2%的家系有4份; 2016年RIL群体中山嵛酸含量低于2%的家系有4份, 2017年山嵛酸含量低于2%的家系有14份, 3年平均值低于2%的家系有3份, 分别为QT0020 (1.86%)、QT0075 (1.92%)和QT0120 (1.92%), 所以通过RIL群体可以创制出山嵛酸含量低的优良后代材料。这3份家系及亲本其他脂肪酸含量见表2, 可看出山嵛酸含量低的家系其花生酸和二十四碳烷酸含量也低, 故通过降低山嵛酸含量可达到降低超长链饱和脂肪酸的目的。对比分析3个环境下RIL群体山嵛酸含量结果, 2016 (2.66%)> 2015 (2.53%)>2017 (2.40%)。RIL群体中山嵛酸含量表现出丰富的变异, 3年变异系数分别为12.39%、13.96%和11.99%。在3个环境下均呈连续分布, 其偏度和丰度绝对值小于0.48, 接近正态分布, 是典型的数量性状特征(图3)。综合分析3年的检测结果, 140份家系3年山嵛酸均值的变异范围为1.86%~3.37%, 均值为2.53%。3份低山嵛酸家系的山嵛酸含量比低值亲本中花10号分别低0.57、0.51和0.51个百分点, 降低率为23.45%、20.98%和20.98%。
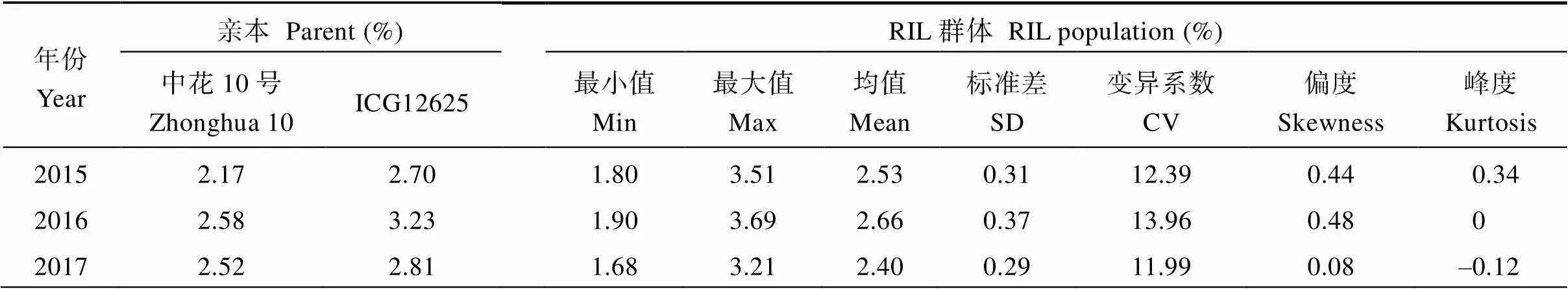
表1 亲本及RIL群体中山嵛酸表型统计分析

表2 3份低山嵛酸家系其他脂肪酸表现
2.3 山嵛酸含量标记分析
结合本实验室前期通过该群体构建的遗传连锁图[16]和3年山嵛酸的表型值, 通过WinQTLCart2.5进行QTL定位分析, 在标记区间GM2246-2– AGGS1236内检测到稳定的控制山嵛酸含量的QTL (), 解释表型变异的9.13%~12.90%。在单一环境及环境均值下, QTL加性效应均为负, 增效等位基因来自于ICG12625。对该QTL的侧翼标记在RIL群体中进行分析, 标记GM2246-2和AGGS1236扩增出AA的家系与扩增出aa的家系在山嵛酸含量上存在极显著差异(表3), 在多个环境中表现一致。超长链饱和脂肪酸中山嵛酸含量占比最大, 两者相关系数为0.92。因此用标记GM2246-2和AGGS1236在RIL群体中对超长链饱和脂肪酸进行鉴定分析, 基因型为AA的家系超长链饱和脂肪酸含量低于基因型为aa的家系, 且差异达到了极显著水平(图4)。所以, 此标记可用于山嵛酸和超长链饱和脂肪酸含量的鉴定并且为其精细定位提供基础。

图3 RIL群体山嵛酸性状在3年中的分布

图4 标记在RIL群体中超长链饱和脂肪酸的表型效应
VLCSFA: very long-chain saturated fatty acid; a: GM2246-2; b: AGGS1236.
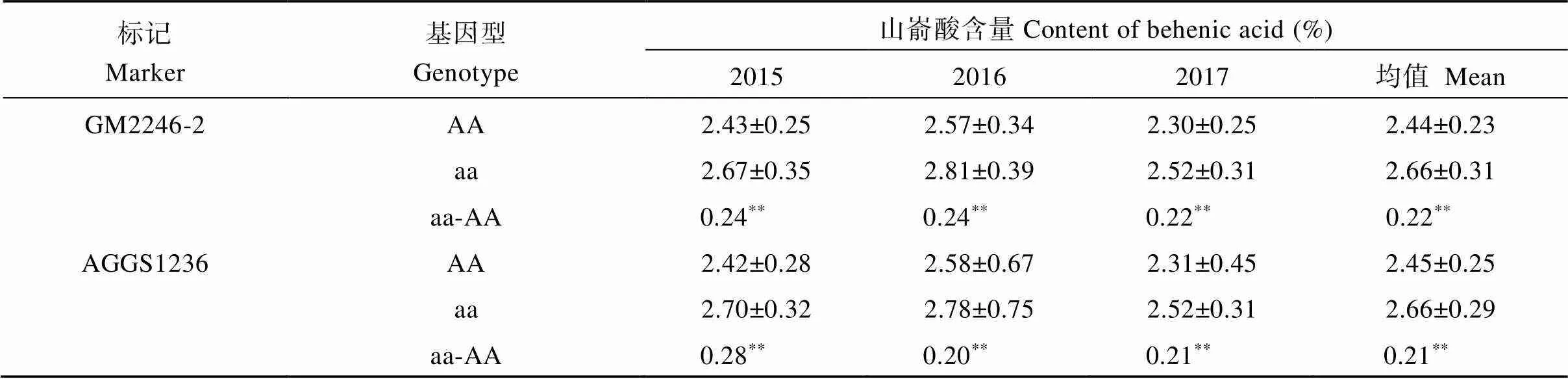
表3 标记GM2246-2和AGGS1236在RIL群体中山嵛酸含量的表型效应
AA代表等位基因与中花10一致的RIL株系, aa代表等位基因与ICG12625一致的RIL株系;**表示在0.01水平上的极显著差异。
AA represent RIL line with allele consistent with Zhonghua 10 and aa represent RIL line with allele consistent with ICG12625.**represent significant difference at the 0.01 probability level.

图5 GO富集分析
位于B04染色体64.24~68.79 cM区间内, 通过侧翼标记序列与物理图谱[17]比对, 该区间位于B04染色体13.31~16.43 M内, 在这3.12 M区间内含有131个预测基因, 其中11个基因未被注释, 编码未知蛋白。为了进一步分析这些基因的功能分类, 本研究对该区间内131个基因进行GO (Gene Ontology)分析。如图5所示, 分子功能分类中有4个GO条目显著富集, 共有20个基因隶属于水解酶活性和核苷酸结合等相关功能显著富集; 在生物学过程分类中富集到了9个GO条目, 29个基因参与生物/细胞刺激反应、信号转导、防御/压力反应和碳水化合物代谢过程等; 而细胞组分这一分类的GO条目并没有显著富集。
3 讨论
本研究对RIL群体F8~F10代超长链饱和脂肪酸含量测试表明, 超长链饱和脂肪酸和山嵛酸含量变异广泛, 呈正态分布, 属数量性状。多年变异系数均值分别为9.84%和12.78%, 说明花生在超长链饱和脂肪酸和山嵛酸含量上存在很大的遗传分化。超长链饱和脂肪酸和山嵛酸存在超亲现象, 在RIL群体中出现了超长链饱和脂肪酸含量为4.27%的家系, 山嵛酸含量为1.86%的家系, 因此, 利用不同遗传背景的品种杂交可以创制优良种质。廖伯寿等[18-19]通过重组自交系群体的构建, 在后代中发现了18份抗黄曲霉或抗青枯病的高油家系, 其中抗黄曲霉兼抗青枯病高油新种质1份, 高油抗青枯病农艺性状优良的种质3份。郭建斌等[20]通过RIL群体后代表型的鉴定, 获得了高油种质1份, 高油酸低棕榈酸的种质2份, 这将为以后培育高油、高油酸、低棕榈酸的新品种奠定基础。而本研究获得了超长链饱和脂肪酸含量和山嵛酸含量低的超亲重组后代, 超长链饱和脂肪酸含量低于5%的种质有21份, 山嵛酸含量低于2%的种质3份。因此, 通过杂交构建重组自交系群体以及在性状基本稳定的后代中鉴定选择, 是选育优良花生品种的有效途径。目前推广品种的山嵛酸含量在2.53%左右[12], 培育低山嵛酸含量的品种十分有必要。
超长链脂肪酸的生物合成分为2个阶段。第一阶段为脂肪酸的从头合成, 由脂肪酸合酶催化完成; 第二个阶段为碳链的延伸, 由脂肪酸碳链延长酶催化完成。在碳链延伸过程中, 碳链延长酶以C20为底物, 生成C22, 继续以C22为底物, 生成C24。花生酸、山嵛酸和二十四碳烷酸在脂肪酸生物合成途径上存在底物与产物的关系, 所以3种超长链饱和脂肪酸存在相关性。
本研究在染色体B04上检测到一个稳定的控制山嵛酸的QTL (), 在不同环境下分别解释表型变异的9.13%~12.90%。郭建斌等[20]通过RIL群体在A05染色体上检测到一个与山嵛酸相关的QTL, 解释表型变异的3.99%; Wang等[11]利用2个群体在A10、A09、A07和B08上均检测到与山嵛酸相关的QTL, 解释表型变异的4.89%~9.44%。与之相比, 本研究发掘的QTL是新的。不同研究者利用不同遗传背景的材料, 获得的结果不一样, QTL重复性差, 说明不同亲本在基因位点的差异不同。将QTL侧翼标记GM2246-2和AGGS1236的序列与物理图谱比对, 发现该QTL位于B04染色体13.31~16.43 M内, 在这3.12 M区间内含有131个预测基因, GO分析表明只有49个基因被显著富集, 其中有20个基因显著富集在分子功能分类中, 有29个基因显著富集在生物学过程分类中。该QTL侧翼标记将为改良花生山嵛酸性状提供重要参考以及为山嵛酸的精细定位奠定基础。
4 结论
获得了3份山嵛酸含量低于2%的花生新种质, 分别为QT0002 (1.86%)、QT0075 (1.92%)和QT0120 (1.92%); 1个稳定的与山嵛酸相关的QTL (), 解释表型变异的9.13%~12.90%。
[1] 廖伯寿. 花生(中国种植业优质高产技术丛书). 武汉: 湖北科学技术出版社, 2003. pp 5–6. Liao B S. Peanut (China’s Planting Industry High-quality and High-yield Technology Series). Wuhan: Hubei Scientific and Technical Publishers, 2003. pp 5–6 (in Chinese).
[2] 顾黎. 花生油中脂肪酸组成的气相色谱——质谱分析. 林区教学, 2007, (2): 124–125. Gu L. Gas chromatography: mass spectrometry analysis of fatty acid composition in peanut oil., 2007, (2): 124–125 (in Chinese).
[3] 熊秋芳, 张效明, 文静, 李兴华, 傅廷栋, 沈金雄. 菜籽油与不同食用植物油营养品质的比较——兼论油菜品质的遗传改良. 中国粮油学报, 2014, 29(6): 122–128. Xiong Q F, Zhang X M, Wen J, Li X H, Fu T D, Shen J X. Comparison of nutritional values between rapeseed oil and several other edible vegetable oils: discussion of rapeseed quality genetic improvement., 2014, 29(6): 122–128 (in Chinese with English abstract).
[4] 郑畅, 杨湄, 周琦, 黄凤洪, 邓乾春, 郭萍梅, 刘昌盛. 高油酸花生油与普通油酸花生油的脂肪酸、微量成分含量和氧化稳定性. 中国油脂, 2014, 39(11): 40–43. Zheng C, Yang M, Zhou Q, Huang F H, Deng Q C, Guo P M, Liu C S. Contents of fatty acid and minor component and oxidative stability of high oleic peanut oil and normal oleic peanut oil., 2014, 39(11): 40–43 (in Chinese with English abstract).
[5] Braddock J C, Sims C A, O’Keefe S F. Flavor and oxidative stability of roasted high oleic acid peanuts., 1995, 60: 489–493.
[6] Bolton G E, Sanders T H. Effect of roasting oil composition on the stability of roasted high-oleic peanuts., 2002, 79: 129–132.
[7] Talcott S T, Passeretti S, Duncan C E, Gorbet D W. Polyphenolic content and sensory properties of normal and high oleic acid peanuts., 2005, 90: 379–388.
[8] 苏宜香, 郭艳. 膳食脂肪酸构成及适宜推荐比值的研究概况. 中国油脂, 2003, 28(1): 31–34. Su Y X, Guo Y. A review of dietary fatty acid composition and recommended optimal ratio., 2003, 28(1): 31–34 (in Chinese with English abstract).
[9] 田永全. 脂肪酸的营养功能. 中国食物与营养, 2007, (8): 51–52. Tian Y Q. Nutritional function of fatty acids., 2007, (8): 51–52 (in Chinese).
[10] 姚云游. 花生油与橄榄油营养价值的比较. 中国油脂, 2005, 30(4): 66–68. Yao Y Y. Comparison of peanut oil and olive oil in nutritional value., 2005, 30(4): 66–68 (in Chinese with English abstract).
[11] Wang M L, Khera P, Pandey M K, Wang H, Qiao L, Feng S, Tonnis B, Barkley N A, Pinnow D, Holbrook C C, Clubreath A K, Varshney R K, Guo B. Genetic mapping of QTLs controlling fatty acids provided insights into the genetic control of fatty acid synthesis pathway in peanut (L.)., 2015, 10: e0119454.
[12] 郭建斌, 吴贝, 陈伟刚, 黄莉, 陈玉宁, 周小静, 罗怀勇, 刘念, 任小平, 姜慧芳. 花生品种主要脂肪酸含量在不同生态区的稳定性. 作物学报, 2019, 45: 676–682. Guo J B, Wu B, Chen W G, Huang L, Chen Y N, Zhou X J, Luo H Y, Liu N, Ren X P, Jiang H F. Stability of major fatty acids of peanut varieties grown in different ecological regions., 2019, 45: 676–682 (in Chinese with English abstract).
[13] 李丹阳, 刘凯歌, 卢济明, 杨鑫雷, 崔顺立, 穆国俊, 陈焕英, 刘立峰. 花生SSR标记与品质性状的相关分析. 分子植物育种, 2017, 15(7): 226–232. Li D Y, Liu K G, Lu J M, Yang X L, Cui S L, Mu G J, Chen H Y, Liu L F. Correlation analysis of SSR markers and quality traits in peanut (L.)., 2017, 15(7): 226–232 (in Chinese with English abstract).
[14] 刘芳, 王积军, 汤松. 我国高油酸花生品种选育与推广应用. 中国农技推广, 2017, 33(1): 14–15. Liu F, Wang J J, Tang S. Breeding, extension and application of high oleic peanut varieties in China., 2017, 33(1): 14–15 (in Chinese).
[15] Worthington R E, Hammons R O, Allison J R. Varietal differences and seasonal effects on fatty acid composition and stability of oil from 82 peanut genotypes., 1972, 20: 729–730.
[16] Huang L, Ren X P, Wu B, Li X P, Chen W G, Zhou X J, Chen Y N, Pandey M K, Jiao Y Q, Luo H Y, Lei Y, Varsheny R K, Liao B S, Jiang H F. Development and deployment of a high-density linkage map identified quantitative trait loci for plant height in peanut (L.)., 2016, 6: 39478.
[17] Bertioli D J, Cannon S B, Froenicke L, Huang G, Farmer A D, Cannon E K, Liu X, Gao Y D, Clevenger J, Dash S, Ren L H, Moretzsohn M C, Shirasawa K, Huang W, Vidigal B, Abernathy B, Chu Y, Niederhuth C E, Umale P, Araújo A C G, Kozik A, Kim K D, Burow M D, Varshney R K, Wang X J, Zhang X Y, Barkley N, Guimarães P M, Isobe S, Guo B Z, Liao B S, Stalker H T, Schmitz R J, Scheffler B E, Leal-Bertioli S C M, Xun X, Jackson S A, Michelmore R, Ozias-Akins P. The genome sequences ofand, the diploid ancestors of cultivated peanut., 2016, 48: 438–446.
[18] 廖伯寿, 雷永, 王圣玉, 李栋, 黄家权, 姜慧芳, 任小平. 花生重组近交系群体的遗传变异与高油种质的创新. 作物学报, 2008, 34: 999–1004. Liao B S, Lei Y, Wang S Y, Li D, Huang J Q, Jiang H F, Ren X P. Genetic diversity of peanut RILs and enhancement for high oil genotypes., 2008, 34: 999–1004 (in Chinese with English abstract).
[19] 廖伯寿, 雷永, 李栋, 王圣玉, 黄家权, 任小平, 姜慧芳, 晏立英. 利用RIL群体创造抗黄曲霉兼抗青枯病的高油花生新种质. 作物学报, 2010, 36: 1296–1301. Liao B S, Lei Y, Li D, Wang S Y, Huang J Q, Ren X P, Jiang H F, Yan L Y. Novel high oil germplasm with resistance toand bacterial wilt developed from recombinant inbred lines., 2010, 36: 1296–1301 (in Chinese with English abstract).
[20] 郭建斌. 花生含油量及脂肪酸组成的QTL分析. 华中农业大学硕士学位论文, 湖北武汉, 2016. Guo J B. QTL Analysis for Oil Content and Fatty Ocid Traits in Peanut (L.). MS Thesis of Huazhong Agricultural University, Wuhan, Hubei, China, 2016 (in Chinese with English abstract).
Novel peanut genotype with low behenic acid developed from recombinant inbred lines
GUO Jian-Bin, HUANG Li, LIU Nian, LUO Huai-Yong, ZHOU Xiao-Jing, CHEN Wei-Gang, WU Bei, HUAI Dong-Xin, REN Xiao-Ping, and JIANG Hui-Fang*
Oil Crops Research Institute, China Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, the Ministry of Agriculture and Rural Affairs, Wuhan 430062, Hubei, China
Peanut is an important source of edible vegetable oil in China, and improving the quality of peanut oil is an important goal in peanut breeding. In this study, the contents of very long-chain saturated fatty acids (arachidic acid, behenic acid, and tetracosanoic acid) were determined by using RIL population containing 140 lines derived from the cross between Zhonghua 10 and ICG12625 with different genetic backgrounds. The content of very long- chain saturated fatty acid in the RILs was 4.27%–7.05%, with an average of 5.54%, the minimum value was 1.46 and 1.63 percentage points lower than that of male and female parents, with reduction rates of 25.27% and 27.62%, respectively. The behenic acid content ranged from 1.86% to 3.37%, with an average of 2.53%. Three lines of QT0002, QT0075, and QT0120 had behenic acid content lower than 2%, which was 0.57, 0.51, and 0.51 percentage points lower than that of Zhonghua10 with the reduction rate of 23.45%, 20.98%, and 20.98%, respectively. A stable QTL located in 13.31–16.34 M of B04 was detected using a linkage map and WinQTLcart software. In this region of 13.31–16.34 M contained 131 prediction genes. The results lay a foundation for the genetic improvement of low behenic acid and high-oleic peanut varieties.
peanut; recombined inbreed lines (RILs); fatty acid; behenic acid
10.3724/SP.J.1006.2020.94119
本研究由国家自然科学基金项目(31571713, 31761143005, 31801403, 31871666), 农作物种质资源保护项目(2017NWB033), 国家农作物种质资源共享服务平台(NICGR2017-36)和国家现代农业产业技术体系建设专项(CARS-13-花生种质资源评价)资助。
This study was supported by the National Natural Science Foundation of China (31571713, 31761143005, 31801403, 31871666), the Crop Germplasm Resources Protection Project (2017NWB033), the Plant Germplasm Resources Sharing Platform (NICGR2017-36), and the China Agricultural Research System (CARS-13-Germplasm Resource Evaluation for Peanut).
姜慧芳, E-mail: peanutlab@oilcrops.cn, Tel: 027-86711550
E-mail: guojianbin1990@163.com
2019-08-14;
2019-12-26;
2020-01-16.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200115.1355.027.html
