黑龙江省稻瘟病菌无毒基因AVR-Pita及其同源基因的检测与分析
2020-03-31孟峰张亚玲靳学慧
孟峰 张亚玲 靳学慧
黑龙江省稻瘟病菌无毒基因及其同源基因的检测与分析
孟峰 张亚玲*靳学慧
(黑龙江八一农垦大学, 黑龙江 大庆 163319; *通信联系人, E-mail: byndzyl@163.com)
【】为了检测黑龙江省稻瘟病菌无毒基因及其同源基因分布情况与变异机制,了解其变异类型的致病表型。采用3个无毒基因、和的特异性引物,对202个采自黑龙江省各稻区的稻瘟病菌单孢分离菌株的DNA进行PCR扩增,通过琼脂糖凝胶电泳检测分析,挑选不同带型和不同地区代表菌株的PCR产物进行测序。测序结果与相应无毒基因序列进行碱基与氨基酸序列的比较分析,并利用水稻抗性单基因系,对不同变异类型的稻瘟病菌株进行功能验证。的出现频率为36.14%,出现频率为59.41%。在黑龙江省202个菌株DNA中未扩增出目的条带。对和的部分PCR产物进行序列分析,检测出有5种变异类型,它们是、、、和。经功能验证,、、和无毒功能丧失。而无毒基因未检测出变异菌株。变异能力较强,导致大多数菌株无毒功能丧失,需与其他抗性基因搭配使用。在黑龙江省稻瘟病菌生理小种中未发现基因。基因序列在菌株中比较稳定。
稻瘟病菌;及其同源基因;功能验证
稻瘟病是由稻瘟病菌引起的,是世界上最重要的水稻病害之一,严重威胁水稻高产稳产,每年因该病害引起的水稻产量损失高达10%~30%,严重时颗粒无收[1]。实践证明,利用品种抗性是防治稻瘟病最经济、安全、有效的途径。目前定位到的稻瘟病主效抗性基因已达100多个,其中,已克隆的有36个[2]。稻瘟病菌与水稻之间的互作符合经典的基因对基因学说,即致病菌中的无毒()基因在功能上与水稻中的特定抗病基因()相对应。水稻品种的抗性基因与稻瘟病菌的无毒基因相互作用时,表现出抗瘟性[3-4]。目前已报道的无毒基因有70多个,其中,12个被克隆,包括、和等[5-14]。Orbach等[7]成功克隆了稻瘟病菌无毒基因。与水稻抗性基因互作的无毒基因是一个编码含223个氨基酸的分泌蛋白,含有保守的金属蛋白酶结构域,是首个被证实编码蛋白能与相对应的抗病基因产物直接互作的无毒基因。Khang等[15]发现了的两个新同源体,命名为和,并将重新命名为。甘玉姿等[16]在菲律宾稻瘟病菌生理小种中发现了的新同源序列,将其命名为。其中无毒基因和基因是有功能的,与抗性基因相互作用,而和不具有无毒功能[16,17]。刘殿宇等[18]研究发现2014年和2015年黑龙江省出现频率分别为23.0%和22.9%;李思博等[19]在2014和2015年辽宁省检测到的出现频率为89.91%和76.00%;朱名海等[20]在南繁区检测到的出现频率为93.33%。
本研究结合无毒基因家族的基因扩增、测序结果和变异类型的功能验证,对采集自黑龙江省不同稻区的稻瘟病菌进行分析,从而明确黑龙江省稻瘟病菌基因家族的分布情况及变异类型,以期为黑龙江省抗瘟品种的合理布局与稻瘟病的有效防控提供依据。
1 材料与方法
1.1 供试菌株
2017年在黑龙江省7个市15个县水稻种植区内采集水稻穗颈瘟标样,经分离获得单孢菌株202个,采用滤纸片保存法[21]保存备用。
1.2 供试材料
供试水稻品种为国际水稻研究所选育的含有基因的丽江新团黑谷抗稻瘟病近等基因系IRBtLa-kl,感病对照为丽江新团黑谷。
1.3 稻瘟病菌基因组DNA提取
将分离纯化的稻瘟病菌单孢菌株在PDA固体培养基上活化培养,挑取适量菌丝块接到酵母液体培养基中,于28℃、120 r/min摇床振荡培养3~5 d,收集菌丝体,分装于1.5 mL离心管中,–20℃下保存备用。使用真菌DNA提取试剂盒(D3390-01 OmegaBio-Tek公司)提取稻瘟病菌基因组DNA,用微量分光光度计测定DNA浓度,并将DNA原液稀释成60 ng/µL的工作液备用。原液–20℃下保存。
1.4 引物设计
根据文献中已克隆的于NCBI上查找其基因序列,利用Primer Premier 5.0设计一对特异性引物,和的特异性引物参照甘玉姿等[16]序列。所有引物均委托上海生工生物工程技术有限公司合成,引物序列见表1。
1.5 PCR扩增及测序分析
PCR体系(20 µL)包括r酶0.1 µL,10×缓冲液(Mg+) 2.0 µL,dNTP混合液1.6 µL,正反向引物各0.3 µL,DNA模板 1 µL,加dd H2O补足20 µL。扩增程序:94℃下预变性4 min;94℃下变性45 s,55℃下退火45 s,72℃下延伸60 s,30个循环;72℃下延伸10 min。扩增产物在1%的琼脂糖凝胶中电泳检测并在凝胶电泳成像系统下观察并拍照,统计无毒基因扩增频率。
从202个稻瘟病菌菌株中挑选带型不同和不同地区来源的部分菌株送上海生工生物工程技术有限公司测序,测序结果采用Lasergene 7.0的Seq Man软件进行比对与拼接;并采用DNAMAN软件对有差异的核苷酸序列进行比较分析。
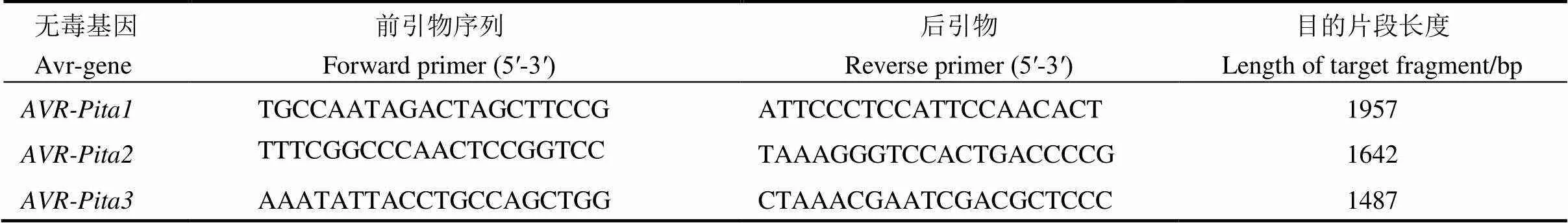
表1 用于扩增稻瘟病菌无毒基因AVR-Pita的引物
1.6 水稻稻瘟病接种与调查
水稻3叶1心时,用无菌水洗下孢子,双层纱布过滤,调节孢子量为1×105个/mL,将5 mL菌悬液加入5 mL明胶溶液摇匀后喷洒秧苗,25℃下遮光保湿培养24 h后自然条件下培养,接种后5 d调查稻瘟病发病情况,调查标准参照文献[22]。
2 结果与分析
2.1 AVR-Pita及其同源基因PCR产物扩增检测
以202个稻瘟病菌菌株的DNA为模板,根据无毒基因及其同源基因序列设计的引物进行PCR扩增(图1),电泳结果显示共有5种带型:无带、高带(正常带型)、低带、双带和三带。双带为非目的条带[23],三带为多位点现象。引物在供试的202个菌株中,73个菌株能扩增出目的条带,出现频率为36.1%(表2),其中大庆杜蒙、肇源,哈尔滨方正、尚志出现频率为0%(表2);引物在所有供试菌株DNA中均未扩增出目的条带;引物电泳结果显示120个菌株DNA中扩增出目的条带,出现频率为59.4%。供试202个菌株中54个菌株DNA同时扩增出和。

A−AVR-Pita1的部分扩增结果; B−AVR-Pita3的部分扩增结果。M1−分子标记DL2000;+:存在;-:缺失;1:低带;2:双带;3:三带。
Fig. 1. PCR detection ofand its homologous genesamplification to the tested strains.

表2 2017年黑龙江省稻瘟病菌无毒基因AVR-Pita1及其同源基因分布情况
2.2 AVR-Pita及其同源基因的序列分析
2.2.1 无毒基因的序列分析
从73个扩增出目的条带的菌株中挑选不同地区不同带型的菌株20个进行测序。将测序得到的基因序列与参考序列(AF207841.1)比对。结果显示(图2),20个菌株的无毒基因菌株序列可分为5类,一类为存在基因序列1250 bp的缺失,从基因上游1004 bp至下游246 bp处。此变异类型与余欢等[24]研究结果一致,我们将其命名为。其余四类为单碱基的替换、缺失与插入(图2),我们分别将其命名为和。与Dai等[25]报道的结果序列一致。
的4类碱基序列所翻译氨基酸序列与已克隆的氨基酸序列比对结果见图3,除312(T/G)为同义突变外,其余碱基的突变均会导致氨基酸的错义翻译。氨基酸序列不仅在6L(Insert)、88(R/K)和104(K/N)存在错义突变,而且在174位处发生移码突变导致后面氨基酸发生变化。氨基酸序列存在8处错义突变:6L(Insert)、82(N/S)、83(D/N)、88(R/K)、104(K/N)、136(G/E)、174(V/I)和192(Y/C),的氨基酸序列存在7处错义突变:6 L(Insert)、82(N/S)、83(D/N)、88(R/K)、104(K/N)、174(V/I)和192(Y/C)。的氨基酸序列不同于其他4类,它与氨基酸序列比对一致性为25.45%(图4),氨基酸的缺失与错义翻译较多,将进行单独分析。
20个测序的菌株中,包含2个菌株,占测序菌株的1/10;包含4个菌株,占测序菌株的1/5;类型较多,包含10个菌株,占测序菌株的1/2;包含3个菌株,占测序菌株的3/20;包含1个菌株,占测序总菌株的1/20(图5)。
2.2.2 无毒基因的序列分析
从120个含有目的条带的菌株中挑选不同地区的菌株20个进行测序,将测序得到的基因序列与已克隆的基因序列比对后发现,20个菌株的基因序列比对结果完全一致。说明在这20个菌株中没有出现变异情况。
2.3 AVR-Pita1致病表型分析
对无毒基因型的5种突变类型进行功能验证,如图6所示,以丽江新团黑谷(LTH)为对照。结果显示,、-、和不能被识别表现有毒性,无毒功能丧失。C可以被识别而表现为无毒性。
3 讨论
了解家族成员在稻瘟病菌中的分布与变异情况,不仅对研究稻瘟病菌的遗传多样性具有重要价值而且可为抗病品种的布局及病情的分子预警提供参考依据。2017年,从黑龙江省各稻区水稻穗颈瘟标样中分离获得单孢菌株202个,利用202个单孢菌株对及其同源基因进行PCR扩增检测,结果发现出现频率最高,为59.41%,且分布范围最广,各稻区均有分布。出现频率为36.14%,其中,大庆杜蒙、肇源,哈尔滨方正、尚志出现频率为0。在54个菌株的DNA中同时扩增出和,与序列之间存在71%~72%一致性,验证了这2个基因可能起源于同一个原始基因的复制事件。在供试202个菌株的DNA中均未扩增出目的条带,与甘玉姿等[16]对菲律宾稻瘟病菌生理小种研究结果一致。本研究未检测出目的基因,推测2017年黑龙江省稻瘟病菌生理小种中可能不含或含有少量基因。本研究由于田间分离的菌株已经经过了水稻品种的筛选,不能代表实际的田间群体,所以不能很好地反映田间实际菌株无毒基因的分布情况。

图2 稻瘟病菌供试菌株AVR-Pita1-A、AVR-Pita1-B、AVR-Pita1-C和AVR-Pita1-D碱基序列比对
Fig. 2. Base sequence comparison ofandof the tested strains.
图3 稻瘟病菌和氨基酸序列对比分析
Fig. 3. Comparison and analysis of amino acid sequence ofandgenotypes

图4 稻瘟病菌AVR-Pita1-D 氨基酸序列对比分析
Fig. 4. Comparison and analysis of amino acid sequence ofgenotypes of
图5 稻瘟病菌变异类型比例
Fig. 5. Proportion ofvariant types

图6 稻瘟病菌供试菌株AVR-Pita1-1、AVR-Pita1-A、AVR-Pita1-B、AVR-Pita1-C和AVR-Pita1-D致病症状
Fig. 6. Symptoms after infected by the strains with,,and
研究者们认为,导致无毒基因不稳定的重要因素为所处的周边基因环境,和均处于染色体的亚端粒区[15],并且在稻瘟病菌1、3、4、5、6、7等染色体上均有发现[26],具有高度可变性。存在插入、缺失与点突变等多种变异类型。Dai等[25]检测到27种突变类型,Zhou等[23]在毒性菌株B2中发现了完整的转座子Pot3,但在黑龙江省202个菌株的DNA中未检测到Pot3。与和不同,大多数菌株的以比较稳定的形式存在于稻瘟病菌基因组第7染色体上,本研究测序的20个菌株中均未发现变异情况,与前人研究结果相符。
本研究发现存在5种变异类型,其中4种无毒功能丧失,仅可以被识别而表现为无毒性。但-C类型仅占测序菌株数的3/20,大多数菌株已丧失无毒功能。于连鹏[27]在黑龙江省48个水稻品种中检测到35个品种含有基因,占供试水稻品种的72.92%。周江鸿等[28]报道,在我国稻瘟病菌群体中抗性基因表现为中等至强毒力水平。所以在黑龙江省稻瘟病的抗病育种中不宜单独使用,需要与含有其他抗性基因的品种搭配使用,才能达到抗病效果。变异能力较强,应同时加强对的田间监测,及时了解的变化规律,以有效地控制病害发生。
[1] Couch B C, Kohn L M. A multilocus gene genealogy concordant with host preference indicates segregation of a new species,, from[J]., 2002, 94(4): 683-693.
[2] 曹妮, 陈渊, 季芝娟, 曾宇翔, 杨长登, 梁燕. 水稻抗稻瘟病分子机制研究进展[J]. 中国水稻科学, 2019, 33(6): 489-498.
Cao N, Chen Y, Ji Z J, Zeng Y X, Yang C D, Liang Y. Recent progress in molecular mechanism of rice blast resistance[J]., 2019, 33(6): 489-498. (in Chinese with English abstract)
[3] Marcel S, Sawers R, Oakeley E, Angliker H,Paszkowski U. Tissue-adapted invasion strategies of the rice blast fungus[J]., 2010, 22(9):3177-3187.
[4] Liu W, Zhou X, Li G, Li L, Kong L, Wang C, Zhang H, Xu J. Multiple plant surface signals are sensed by different mechanisms in the rice blast fungus for appressorium formation [J]., 2011, 7(1): e1001261.
[5] Sweigard J A,Carroll A M, Kang S, Farrall L, Chumley F G, Valent B. Identification, cloning, and character- ization of, a gene for host species specificity in the rice blast fungus[J]., 1995, 7(8):1221-1233.
[6] Kang S, Sweigard J A, Valent B. Thehost specificity gene familyin the blast fungus[J]., 1995, 8(6):939-948.
[7] Orbach M J, Farrall L, Sweigard J A,Chumley F G, Valent B. A telomeric avirulence gene determines efficacy for the rice blast resistance gene[J]., 2000, 12(11):2019-2032.
[8] Collemare J, Pianfetti M, Houlle AE,Morin D, Camborde L, Gagey M J, Barbisan C, Fudal I, Lebrun M H, BőHnert H U.avirulence genebelongs to an infection-specific gene cluster involved in secondary metabolism[J]., 2010, 179(1):196-208.
[9] Farman M L, Leong S A. Chromosome walking to theavirulence gene ofgrisea: Discrepancy between the physical and genetic maps[J]., 1998, 150(3):1049-1058.
[10] Li W, Wang B, Wu J, Lu G,Hu Y, Zhang X, Zhang Z G, Zhao Q, Zhang H Y, Wang Z Y, Wang G L, Han B, Wang Z H, Zhou B.Theavirulence geneencodes a predicted secreted protein that triggers the immunity in rice mediated by the blast resistance gene[J]., 2009, 22(4):411-420.
[11] Yoshida K, Saitoh H, Fujisawa S, Kanzaki H, Matsumura H, Yoshida K, Tosa Y, Chuma I, Takano Y, Win J, Kamoun S, Terauchi R. Association genetics reveals three novel avirulence genes from the rice blast fungal pathogen[J]., 2009, 21(5):1573-1591.
[12] Wu J, Kou Y, Bao J,Li Y, Tang M, Zhu X, Ponaya A, Xiao G, Li J, Li C, Song M, Cumagun C J, Deng Q, Lu G, Jeon J, Naqvi N I, Zhou B. Comparative genomics identifies theavirulence effectorthat triggers-mediated blast resistance in rice[J]., 2015, 206(4):1463-1475.
[13] Schneider D R, Saraiva A M, Azzoni A R, Miranda H R, Toledo M A, Pelloso A C, Souza A P. Overexpression and purification of, a mutant of the effector proteinfrom[J]., 2010, 74(1):24-31.
[14] Zhang S, Wang L, Wu W, He L,Yang X, Pan X. Function and evolution ofavirulence generesponding to the rice blast resistance gene[J]., 2015, 5:11642.
[15] Khang C H, Berruyer R, Giraldo M C,Kankanala P, Park S Y, Czymmek K, Kang S, Valent B. Translocation ofeffectors into rice cells and their subsequent cell-to-cell movement[J]., 2010, 22(4):1388-1403.
[16] 甘玉姿, 肖贵, 邓启云, 吴俊, 柏斌, 卢向阳, 周波. 菲律宾稻瘟病菌生理小种中及其同源基因的序列与功能分析[J].中国生物防治学报,2018,34(3):488-498.
Gan Y Z, Xiao G, Deng Q Y, Wu J, Bai B, Lu X Y, Zhou B. Sequence and functional analysis ofand its homologous genes in physiological races ofin the Philippines[J]., 2018,34(3):488-498. (in Chinese with English abstract)
[17] Chuma I, Isobe C, Hotta Y, Ibaragi K, Futamata N, Kusaba M, Yoshida K, Terauchi R, Fujita Y, Nakayashiki H, Valent B, Tose Y. Multiple translocation of theeffector gene among chromosomes of the rice blast fungusand related species[J]., 2011, 7(7): 395-396.
[18] 刘殿宇. 黑龙江省稻瘟病菌致病性与无毒基因检测及遗传多样性分析[D]. 大庆: 黑龙江八一农垦大学, 2017.
Liu D Y. Pathogenicity avirulent gene detection and genetic diversity analysis ofin Heilongjiang Province[D]. Daqing: Heilongjiang Bayi Agricultural University, 2017. (in Chinese with English abstract)
[19] 李思博. 辽宁省稻瘟菌无毒基因及水稻抗瘟基因鉴定[D]. 沈阳:沈阳农业大学, 2018.
Li S B. Identification of avirulent genes and resistance genes ofin Liaoning Province[D]. Shenyang: Shenyang Agricultural University, 2018. (in Chinese with English abstract)
[20] 朱名海, 赵美, 舒灿伟, 周而勋. 南繁区稻瘟病菌无毒基因的检测[J]. 华中农业大学学报,2017,36(4):21-25.
Zhu M H,Zhao M,Shu C W,Zhou E X. Detection of avirulence genes infrom South China Crop Breeding Area[J]., 2017,36(4):21-25.(in Chinese with English abstract)
[21] 蒋金芬, 韩红萍, 梁友方. 滤纸片法低温冷冻保存菌种的实验室应用[J]. 中国公共卫生, 2006, 22(3):310.
Jiang J F, Han H P, Liang Y F. Laboratory application of filter paper method for cryopreservation[J]., 2006, 22(3):310-310. (in Chinese)
[22] 靳学慧. 农业植物病理学[M]. 赤峰: 内蒙古科学技术出版社,1999.
Jin X H. Agricultural Plant Pathology[M]. Chifeng: Inner Mongolia Science and Technology Press, 1999. (in Chinese)
[23] Zhou E, Jia Y, Singh P, Correll J C, Lee F N. Instability of theavirulence genealters virulence[J]., 2007, 44(10):1034.
[24] 余欢, 姜华, 王艳丽, 孙国昌. 无毒基因在不同寄主梨孢菌中的变异研究[J]. 浙江农业学报, 2015, 27(8):1414-1421.
Yu H, Jiang H, Wang Y L, Sun G C. Study on the variation of avirulence genes in different host Phasporium[J]., 2015, 27(8): 1414-1421.(in Chinese with English abstract)
[25] Dai Y, Jia Y, Correll J, Wang X, Wang Y. Diversification and evolution of the avirulence genein field isolates of[J]., 2010, 47(12):973-980.
[26] Chuma I, Isobe C, Hotta Y, Ibaragi K, Kusaba M, Yoshida K, Terauchi R, Fujita Y, Nakayashiki H, Valent B, Tosa Y. Multiple Translocation of theeffector gene among chromosomes of the rice blast fungusand related species[J]., 2011, 7(7):395-396.
[27] 于连鹏. 黑龙江省主栽水稻品种、和抗瘟基因检测和抗性评价[D]. 大庆: 黑龙江八一农垦大学,2017.
Yu L P.,andgenes detection and blast resistance evaluation of main rice varieties in Heilongjiang Province[D]. Daqing: Heilongjiang Bayi Agricultural University,2017.(in Chinese with English abstract)
[28] 周江鸿, 王久林, 蒋琬如, 雷财林, 凌忠专.我国稻瘟病菌毒力基因的组成及其地理分布[J]. 作物学报, 2003(5):646-651.
Zhou J H, Wang J L, Jiang W R, Lei C L, Ling Z Z. Composition and geographical distribution of virulence genes of rice blast fungus in China[J]., 2003(5):646-651.(in Chinese with English abstract)
Detection and Analysis ofAvirulent Geneand Its Homologous Genes in Heilongjiang Province
MENG Feng, ZHANG Yaling*, JIN Xuehui
(Heilongjiang Bayi Agricultural University, Daqing 163319, China;*Corresponding author, E-mail: byndzyl@163.com)
【】We aimto investigate the distribution and variation of avirulentfamily offrom Heilongjiang Province, and to understand the pathogenic phenotypes ofavirulencealleles.【】Specific primers of the three avirulent genes,andwere used. 202single-spore isolates were obtained from different regions of Heilongjiang Province, their DNA were PCR-amplified by using avirulent genes primers in 2017. PCR products of representative strains falling into different types and from different aresa were selected and sequenced by using agarose gel electrophoresis detection. The base and amino acid sequences were compared with those of the corresponding avirulent genes, and the rice resistant single gene line was used to verify the function of different strains of.【】The frequency ofis 36.14%, and that ofis 59.41%. The target band ofcould not be amplified from the DNA of202 strains from Heilongjiang Province. Sequence analysis of some PCR products ofandwere carried out. Five variation types ofwere detected; they were-,-,,and. Functional verification showed that,,and-lost their function. No mutant strain was detected in the virulent gene. 【】had strong mutation ability, which led to the loss of avirulent function of most strains, this should be used in conjunction with other resistant genes.was not found inHeilongjiang Province. The sequence ofgene was stable in the strain.
;gene family; functional verification
S435.111.4+1
A
1001-7216(2020)02-0143-07
10.16819/j.1001-7216.2020.9085
2019-07-21;
2019-11-21。
黑龙江省自然科学基金资助项目(QC2011C046);黑龙江省农垦总局科技攻关计划资助项目(HNK125A-08-06, HNKXIV-01-04-02, HKKYZD190205);黑龙江省教育厅项目(12521376);黑龙江八一农垦大学学成、引进人才科研启动计划资助项目(XDB201605, XDB201802)。
