利用剩余杂合体衍生的近等基因系精细定位水稻粒长微效QTL qGL1.1
2020-03-31李盼盼朱玉君郭梁庄杰云樊叶杨
李盼盼 朱玉君 郭梁 庄杰云 樊叶杨,*
利用剩余杂合体衍生的近等基因系精细定位水稻粒长微效QTL
李盼盼1朱玉君1郭梁2庄杰云1樊叶杨1,*
(1中国水稻研究所 水稻生物学国家重点实验室/国家水稻改良中心, 杭州 310006;2袁隆平农业高科技股份有限公司, 长沙 410001;*通信联系人, E-mail: fanyeyang@caas.cn)
【】本研究旨在对前期在水稻第1染色体长臂521.8 kb的区间内定位到的进行精细定位。从和所在区间分别呈杂合的2个BC2F9单株配组衍生的F4群体中,筛选到Wn28826-RM1231区间内杂合片段呈梯系排列的3个单株,构建了3套F5:6近等基因系。2017年种植于浙江杭州,考查千粒重、粒长和粒宽。利用SAS软件的GLM程序进行双因素方差分析,对的效应进行了验证。在此基础上,筛选出杂合片段更小且呈交迭排列的6个剩余杂合体,发展了6套F8:9近等基因系,2018年种植于海南陵水。对每套近等基因系中双亲基因型株系的表型差异进行双因素方差分析。在2个试验中对粒长和千粒重均呈极显著差异,效应方向一致且大小稳定。密阳46等位基因能分别增加粒长0.027 mm和提高千粒重0.17 g,贡献率分别达到27.12%和19.09%。鉴于在前后试验中对粒长影响最为显著,而对粒宽作用不显著,故将重新命名为。通过比较各套近等基因系的分离区间的基因组位置,最终将定位于Wn29077和Wn29154之间约76.8 kb的区间内。
水稻;近等基因系;微效作用;粒长;精细定位
水稻(L.)是保障粮食安全的重要作物之一,其单产的提高对粮食安全发挥了重要的作用。千粒重是水稻产量构成因子的三要素之一,主要由籽粒大小和充实度控制,而籽粒大小一般以粒长、粒宽和粒厚等来衡量,此外粒长和粒宽还与稻米外观品质息息相关[1]。因此,分离和克隆籽粒大小相关的基因可为水稻高产优质育种奠定重要的理论基础并提供可用的基因资源。
籽粒大小属于典型的数量性状,由大量的数量性状座位(Quantitative Trait Loci,QTL)控制。迄今已有17个QTL获得克隆,它们分布于除水稻第1、10、11和12染色体外的8条染色体上。其中,/、、/、、/、//、、、和等10个QTL主要控制粒长和千粒重[2-16];、、和等4个QTL主要控制粒宽和千粒重[17-21];对粒长和粒宽的效应大小相近且方向相同,故最终控制千粒重[22];/和虽然也对粒长和粒宽兼具效应,但其效应方向相反,故对千粒重并不表现出显著效应[23-25]。这些QTL主要涉及植物激素、泛素-蛋白酶体通路和G蛋白信号等分子途径[26-27]。这些研究丰富了我们对水稻籽粒大小的分子调控机制的了解,但其调控通路网络尚不清楚。
主效QTL和微效QTL均是解析复杂性状遗传机制的重要组成部分[28-29]。主效QTL因其效应较大,能在不同遗传背景和环境中稳定检测到,故而是精细定位和克隆的首选;相对地,微效QTL效应较小,更易受遗传背景和环境的干扰,故而在初级定位中较难稳定检测到[30],目前尚未有籽粒大小微效QTL克隆的报道。因此,籽粒大小微效QTL的发掘、精细定位和克隆,对全面揭示籽粒大小调控机制具有重要意义。
在前期研究中,我们应用珍汕97///珍汕97//珍汕97/密阳46衍生的高代回交群体,在水稻第1染色体长臂将千粒重QTL分解为和,其中,被界定于RM11554-RM11569约521.8 kb的区间内,密阳46等位基因显著增加粒长,提高粒重[31]。本研究针对,新构建了分离区间呈梯系排列的3套近等基因系(Near Isogenic Lines, NILs),对其效应进行验证,并进一步发展了分离区间更为精细且交迭排列的6套近等基因系,开展精细定位。
1 材料与方法
1.1 水稻材料
本研究所用定位群体为9套近等基因系,其中3套F5:6近等基因系用于效应验证,6套F8:9近等基因系用于精细定位,材料构建过程(图1)如下。
利用珍汕97///珍汕97//珍汕97/密阳46组合衍生的1个BC2F9群体,从中挑选在区间(RM11437-RM11615)和区间(RM11615-RM11800)[32]分别呈杂合的2个单株,配组衍生1个F4群体。经分离区间标记检测,筛选到Wn28826-RM1231区间内杂合片段呈梯系排列的3个单株,自交形成3个NIL-F2型的F5群体,分别从每个群体中挑选分离区间呈母本纯合型(即珍汕97纯合型)和父本纯合型(即密阳46纯合型)的单株,自交后构建了3套F5:6近等基因系,命名为Y1、Y2和Y3。Y1包含珍汕97纯合型株系30个和密阳46纯合型株系29个,Y2和Y3分别包含珍汕97纯合型和密阳46纯合型株系各30个。3套近等基因系的分离区间分别为Wn28826-RM1231、Wn28990-RM1231和Wn29154-RM1231(图2-A)。
Fig. 1. Procedure for developing the nine sets of near isogenic lines (NILs).
同时,为进一步筛选杂合区间更为精细的梯系剩余杂合体材料,从衍生出Y1的F5群体中挑选整个分离区间(Wn28826-RM1231)呈杂合的2个单株,经过“剩余杂合体-NIL-F2型群体-剩余杂合体-NIL-F2型群体”2次标记筛选,获得了杂合片段更小且呈交叠排列的6个F7单株,自交形成6个NIL-F2型F8群体。从中挑选珍汕97纯合型和密阳46纯合型单株,自交后构建了6套F8:9近等基因系,分别命名为LP1、LP2、LP3、LP4、LP5和LP6,每套近等基因系分别包含珍汕97纯合型株系和密阳46纯合型株系各30个。LP3和LP5来源于同一个F5单株,LP1、LP2、LP4和LP6则来源于另一个F5单株。6套近等基因系的分离区间分别为Wn28826-Wn28893、Wn28826-Wn28990、Wn28990-Wn29048、Wn29048-Wn29125、Wn29125-RM1231和Wn29154-RM1231(图2-B)。
1.2 田间试验及性状考查
3套F5:6近等基因系于2017年5月种植在浙江省杭州市中国水稻研究所试验基地(以下简称杭州试验)。6套F8:9近等基因系于2018年12月种植在海南省陵水市中国水稻研究所试验基地(简称陵水试验)。采用完全随机区组设计,2次重复,每株系种植1行,每行8株,行株距16.7 cm×26.7 cm,正常田间管理。每株系混收中间4株。根据Zhang等[31]方法挑选饱谷600粒,采用万深SC-G自动种子考种分析及千粒重仪测定千粒重、粒长和粒宽。
1.3 标记检测
采用Zheng等[33]的方法提取微量DNA,其中NIL-F2型群体分单株提取DNA,近等基因系每株系取中间6株的叶片混提DNA。在RM11554-RM11569区间共开发了8对特异分子标记,包括2对InDel、2对CAPS和4对dCAPS标记(表1)。按常规方法进行PCR扩增[34],采用2%琼脂糖凝胶电泳或6%~10%的非变性聚丙烯酰胺凝胶电泳检测PCR扩增产物和酶切产物。
1.4 数据分析
采用Microsoft Excel计算2个试验9套近等基因系千粒重、粒长和粒宽的基本统计数据,包括平均值、标准差、变异系数、变异范围、偏斜度和峰度。应用SAS软件的一般线性模型(Proc GLM)[35]对每套近等基因系中珍汕97纯合型株系和密阳46纯合型株系的表型差异进行双因素方差分析。当检测到显著的(<0.05)基因型差异时,则计算加性效应和贡献率[36]。

A–用于验证的3套F5:6近等基因系; B–用于精细定位的6套F8:9近等基因系。
Fig. 2. Genotypic compositions of the nine NILs sets in the target region.
2 结果与分析
2.1 性状分布与表型变异
在千粒重、粒长和粒宽这3个性状上,3套F5:6近等基因系在杭州试验以及6套F8:9近等基因系在陵水试验的性状均呈连续分布,且偏斜度和峰度较小,表现出数量性状的特点(表2)。
从平均值以及变异范围来看,各套近等基因系的粒长均值和变异范围在两地试验中相差不大,但各套近等基因系的千粒重和粒宽均值和变异范围在杭州试验中均比陵水试验中小,表明粒长受地域环境的影响比千粒重和粒宽小。
比较同一世代中各套近等基因系在同一个性状上的平均值,发现杭州试验中Y1和Y3同一性状的平均值较为接近,而Y2均值最大;陵水试验中6套近等基因系的平均值呈线性连续分布,且LP4在3个性状上的平均值都高于其他5套近等基因系。表明同一世代各套近等基因系的遗传背景还存在控制这3个性状的微效QTL差异。

表1 本研究新开发的DNA标记

表2 9套近等基因系千粒重、粒长和粒宽的表型变异
按2种基因型划分,比较各套近等基因系同一性状双亲基因型分布,发现千粒重和粒长存在明显的群体间差异(图3)。Y1和LP4中2种基因型株系在千粒重和粒长性状上,表现出珍汕97纯合型株系趋向于低值区而密阳46纯合型株系趋向于高值区的特点,特别是粒长,呈现出孟德尔式分离。表明Y1和LP4的分离区间可能存在控制粒长和千粒重的QTL。

图3 9套近等基因系千粒重和粒长分布
Fig. 3. Distributions of 1000-grain weight and grain length in nine NILs sets.
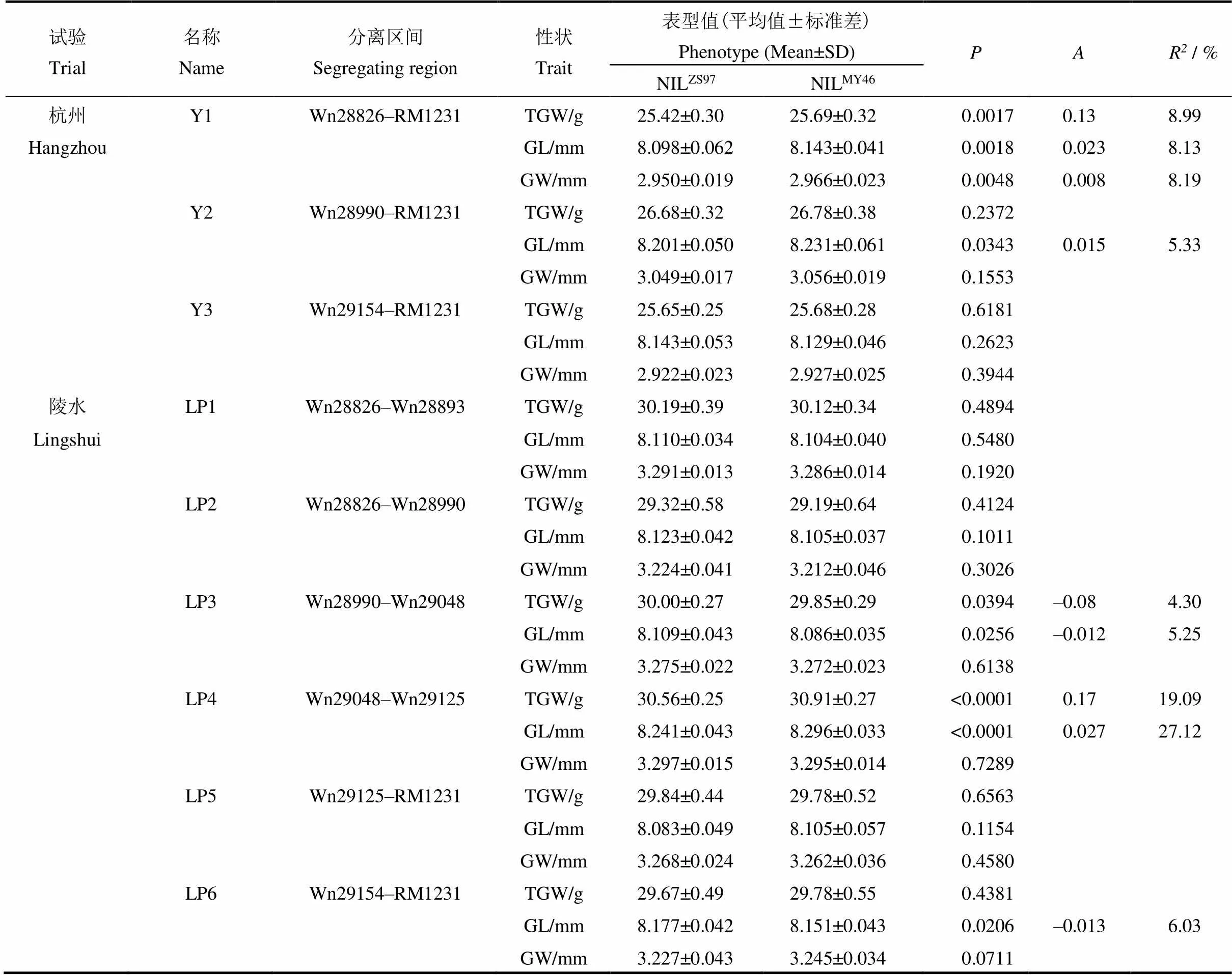
表3 9套近等基因系千粒重、粒长和粒宽的QTL检测结果
−密阳46等位基因取代珍汕97等位基因所产生的遗传效应。R−效应对表型方差的贡献率。
,Additive effect of replacing a Zhenshan 97 allele with a Milyang 46 allele.R,Proportion of phenotypic variance explained by the QTL effect.
2.2 QTL效应验证
前期将定位于RM11554-RM11569约521.8 kb的区间,对千粒重和粒长表现显著效应,其加性效应大小分别为0.06 g和0.017 mm,增效等位基因均来自于密阳46[31]。杭州试验中3套近等基因系千粒重、粒长和粒宽的双因素方差分析表明。在分离区间为Wn28826-RM1231的Y1中,3个性状均在双亲纯合基因型株系间检测到极显著差异(<0.01),加性效应分别为0.13 g、0.023 mm和0.008 mm,增效等位基因均来自密阳46;在分离区间为Wn28990-RM1231的Y2中,仅粒长在双亲纯合基因型株系间分别检测到显著差异(<0.05),加性效应大小为0.015 mm;而在分离区间为Wn29154-RM1231的Y3中,3个性状在双亲纯合基因型株系间的差异均未达到显著水平(表3)。比较本研究中QTL对粒长的效应大小和方向,发现其与效应大小相近,方向一致,表明Y1和Y2很可能存在的分离,而Y3则不存在的分离。比较3套近等基因系的分离区间(图3-A),将初步界定于Wn28944-Wn29154之间约210.2 kb的范围内。
2.3 精细定位
Wn29048-Wn29125区间呈分离的LP4中,千粒重和粒长双亲纯合基因型株系间均检测到极显著差异(<0.0001)。加性效应大小分别为0.17 g和0.027 mm,增效等位基因均来自密阳46(图4),贡献率达到19.09%和27.12%,粒宽则未在双亲纯合基因型株系间检测到显著差异,表明LP4存在的分离(表3)。

图4 携带ZS97纯合型和MY46纯合型qGL1.1等位基因的近等基因系的籽粒(标尺:10 mm)
Fig. 4. Grains of NILZS97and NILMY46for(Scale bar: 10 mm).
分离区间为Wn28990-Wn29048的LP3中也检测到千粒重和粒长的显著差异,但值仅为0.0394,其加性效应大小分别为0.08 g和0.012 mm,但其增效等位基因来自珍汕97,贡献率为4.30%和5.25%。鉴于LP3中检测到的千粒重和粒长QTL效应大小仅为效应值的一半左右,更重要的是效应方向相反。因此,该QTL很可能不是,而是一个新的控制千粒重和粒长的QTL。
分离区间为Wn29154-RM1231的LP6中仅检测到粒长的显著差异(< 0.05),其加性效应大小和方向与LP3类似:加性效应大小为0.013 mm,珍汕97增效等位基因增加粒长,贡献率为6.03%。鉴于LP3与LP6的分离区间相互独立,我们推测LP6可能还存在另外一个控制粒长的QTL。
而分离区间为Wn28826-Wn28893、Wn28826-Wn28990和Wn29125-RM1231的LP1、LP2和LP5中则均未检测到3个性状在双亲纯合基因型株系间的显著差异。有意思的是,LP5是除LP4外唯一1套粒长性状上密阳46纯合型均值大于珍汕97纯合型均值的近等基因系,一个密阳46等位基因取代珍汕97等位基因所产生的粒长遗传效应为(8.105–8.083) / 2 = 0.011 mm,该值正好约等于LP4和LP6的粒长加性效应值之和。因此我们推测,LP5粒长的表现很可能是和LP6所检测到的QTL共同作用的结果。
综上所述,最终将定位于LP4和LP5两套近等基因系共有分离标记Wn29125及其两侧重组区间,即Wn29077-Wn29154,大小约76.8 kb。鉴于在前期试验和本研究中对粒长表现最为显著,且对粒宽作用不显著,故将重新命名为。
3 讨论
微效QTL由于效应较小,很容易被遗传背景和环境的效应掩盖,所以较难在初定位中稳定检测到。而构建遗传背景高度纯合的近等基因系,可以有效消除遗传背景的干扰,进而对效应较小的微效QTL 进行验证和分解, 乃至精细定位和克隆[37]。近年来,本实验室利用剩余杂合体构建近等基因系,通过多轮“梯系剩余杂合体筛选-近等基因系构建- QTL分析”,在水稻第1染色体长臂定位和分解出6个微效QTL,其中、、和,分别界定在120.4 kb、77.5 kb、35.2 Mb和125.5 kb的染色体区间内[31,38-40]。本研究将()定位至76.8 kb的范围内,进一步佐证了该方法和策略在微效QTL精细定位中的有效性和高效性。
在F5:6近等基因系Y1中,粒长和千粒重的加性效应分别为0.023 mm和0.13 g,值和贡献率分别为0.0018和0.0017、8.13%和8.09%;而在F8:9近等基因系LP4中,粒长和千粒重的加性效应分别为0.027 mm和0.17 g,值均小于0.0001,贡献率分别为27.12%和19.09%。这表明随着自交的连续,遗传背景更加同质,在加性效应基本不变的情况下,显著性水平和贡献率均得到了大幅提升。此外,不同世代的近等基因系虽然分别种植于浙江杭州和海南陵水,但表现一致,表明在不同环境中能稳定发挥作用,在高产育种中具有重要的应用潜力和价值。
LP5中双亲纯合基因型株系间尽管没有检测到粒长的显著差异,但从粒长的加性效应大小来看,恰好近似于LP4和LP6的粒长加性效应值之和,效应的方向也与效应较大的LP4中检测到的QTL,即的效应方向一致。基于此,我们推测LP5同时包含了以及LP6中检测到的另一个微效粒长QTL。因此,我们将定位于LP4和LP5共有分离标记Wn29125及其两侧重组区间,即Wn29077-Wn29154区间内。
经Rice Genome Annotation Project Database (http://rice.plantbiology.msu.edu/)检索,在Wn29077-Wn29154区间76.8 kb的范围内预测有13个注释基因。其中5个编码已知功能域蛋白,8个是编码未知功能蛋白。5个注释基因分别是LOC_Os01g 50680(编码OsSub2-假定同源类枯草杆菌蛋白酶)、LOC_Os01g50690(编码包含G蛋白-β亚基WD重复结构域的蛋白),LOC_Os01g50700(编码脱水家族蛋白),LOC_Os01g50720(MYB家族转录因子)和LOC_Os01g50750(编码C3H4型结构域的锌指蛋白)。8个未知功能的注释基因包括4个编码逆转座子:LOC_Os01g50640、LOC_Os01g50650、LOC_ Os01g50660和LOC_Os01g50670,4个编码未知功能的表达蛋白或假设蛋白:LOC_Os01 g50730、LOC_Os01g50740、LOC_Os01g50630和LOC_ Os01g50710。在已克隆的籽粒大小相关QTL中,编码的G 蛋白γ亚基功能缺失时,促进了细胞的伸长,进而产生长粒的表型,此外,G蛋白的α亚基和β亚基也分别报道与籽粒大小有关[27]。因此,编码包含G蛋白-β亚基WD重复结构域蛋白的LOC_Os01g50690可能是的候选基因。
LP3和LP6也均检测到粒长的显著效应,与相比,不但效应较小,而且方向相反。考虑到这两套近等基因系的分离区间相互独立,我们推测在的上下游还各存在1个与相斥的粒长QTL,其效应尚需进一步验证。
[1] Xing Y Z, Zhang Q F. Genetic and molecular bases of rice yield[J]., 2010, 61: 421-442.
[2] Duan P G, Ni S, Wang J M, Zhang B L, Xu R, Wang Y X, Chen H Q, Zhu X D, Li Y H.Regulation ofby OsmiR396 controls grain size and yield in rice[J]., 2016, 2: 15203.
[3] Hu J, Wang Y X, Fang Y X, Zeng L J, Xu J, Yu H P, Shi Z Y, Pan J J, Zhang D, Kang S J, Zhu L, Dong G J, Guo L B, Zeng D L, Zhang G H, Xie L H, Xiong G S, Li J Y, Qian Q. A rare allele ofenhances grain size and grain yield in rice[J]., 2015, 8: 1455-1465.
[4] Yu J P, Xiong H Y, Zhu X Y,Zhang H L, Li H H, Miao J L, Wang W S, Tang Z S, Zhang Z Y, Yao G X, Zhang Q, Pan Y H, Wang X, Rashid M A R, Li J J, Gao Y M, Li Z K, Yang W C, Fu X D, Li Z C.contributing to rice grain length and yield was mined by Ho-LAMap[J]., 2017, 15: 28.
[5] Yu J P, Miao J L, Zhang Z Y, Xiong H Y, Zhu X Y, Sun X M, Pan Y H, Liang Y T, Zhang Q, Abdul Rehman R M, Li J J, Zhang H L, Li Z C. Alternative splicing ofcontrols grain length and yield inrice[J]., 2018, 16: 1667-1678.
[6] Liu Q, Han R X, Wu K, Zhang J Q, Ye Y F, Wang S S, Chen J F, Pan Y J, Li Q, Xu X P, Zhou J W, Tao D Y, Wu Y J, Fu X D. G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]., 2018, 9: 852.
[7] Fan C C, Xing Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]., 2006, 112: 1164-1171.
[8] Qi P, Lin Y S, Song X J, Shen J B, Huang W, Shan J X, Zhu M Z, Jiang L W, Gao P J, Lin H X. The novel quantitative trait locuscontrols rice grain size and yield by regulating Cyclin-T1;3[J]., 2012, 22: 1666-1680.
[9] Zhang X J, Wang J F, Huang J, Lan H X, Wang C L, Yin C F, Wu Y Y, Tang H J, Qian Q, Li J Y, Zhang H S. Rare allele ofassociated with grain length causes extra-large grain and a significant yield increase in rice[J]., 2012, 109(52): 21534-21539.
[10] Hu Z J, Lu S J, Wang M J, He H H, Sun L, Wang H R, Liu X H, Jiang L, Sun J L, Xin X Y, Kong W, Chu C C, Xue H W, Yang J S, Luo X J, Liu J X. A novel QTLencodes the GSK3/SHAGGY-like kinase OsGSK5/OsSK41 that interacts with OsARF4 to negatively regulate grain size and weight in rice[J]., 2018, 11:736-749.
[11] Xia D, Zhou H, Liu R j, Dan W H, Li P B, Wu B, Chen J X, Wang L Q, Gao G J, Zhang Q L, He Y Q., a novel QTL encoding a GSK3/SHAGGY-like kinase, epistatically interacts withto produce extra-long grains in rice[J]., 2018, 11: 754-756.
[12] Ying J Z, Ma M, Bai C, Huang X H, Liu J L, Fan Y Y, Song X J., a major QTL that negatively modulates grain length and weight in rice[J]., 2018, 11: 750-753.
[13] Wu W G, Liu X Y, Wang M H, Meyer R S, Luo X J, Ndjiondjop M N, Tan L B, Zhang J W, Wu J Z, Cai H W, Sun C Q, Wang X K, Wing R A, Zhu Z F. A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication[J]., 2017, 3: 17064.
[14] Wang A H, Hou Q Q, Si L Z,Huang X H, Luo J H, Lu D F, Zhu J J, Shangguan Y Y, Miao J S, Xie Y F, Wang Y C, Zhao Q, Feng Q, Zhou C C, Li Y, Fan D L, Lu Y Q, Tian Q L, Wang Z X, Han B.The PLATZ transcription factor GL6 affects grain length and number in rice[J]., 2019, 180 : 2077-2090.
[15] Ishimaru K, Hirotsu N, Madoka Y, Murakami N, Hara N, Onodera H, Kashiwagi T, Ujiie K, Shimizu B I, Onishi A, Miyagawa H, Katoh E. Loss of function of the IAA-glucose hydrolase geneenhances rice grain weight and increases yield[J]., 2013, 45(6): 707-711.
[16] Si L Z, Chen J Y, Huang X H, Gong H, Luo J H, Hou Q Q, Zhou T Y, Lu T T, Zhu J J, Shangguan Y Y, Chen E W, Gong C X, Zhao Q, Jing Y F, Zhao Y, Li Y, Cui L L, Fan D L, Lu Y Q, Weng Q J, Wang Y C, Zhan Q L, Liu K Y, Wei X H, An K, An G, Han B.controls grain size in cultivated rice[J]., 2016, 48(4): 447-456.
[17] Song X J, Huang W, Shi M, Zhu M Z, Lin H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]., 2007, 39(5): 623-630.
[18] Li Y B, Fan C C, Xing Y Z, Jiang Y H, Luo L J, Sun L, Shao D, Xu C J, Li X H, Xiao J H, He Y Q, Zhang Q F. Natural variation inplays an important role in regulating grain size and yield in rice[J].2011, 43(12): 1266-1269.
[19] Duan P G, Xu J S, Zeng D L, Zhang B L, Geng M F, Zhang G Z, Huang K, Huang L J, Xu R, Ge S, Qian Q, Li Y H. Natural variation in the promoter ofcontributes to grain size diversity in rice[J]., 2017, 10: 685-694.
[20] Liu J F, Chen J, Zheng X M, Wu F Q, Lin Q B, Heng Y Q, Tian P, Cheng Z J, Yu X W, Zhou K N, Zhang X, Guo X P, Wang J L, Wang H Y, Wan J M.acts in the brassinosteroid signaling pathway to regulate grain width and weight in rice[J].2017, 3: 17043.
[21] Wang S K, Wu K, Yuan Q B, Liu X Y, Liu Z B, Lin X Y, Zeng R Z, Zhu H T, Dong G J, Qian Q, Zhang G Q, Fu X D. Control of grain size, shape and quality byin rice[J].2012, 44(8): 950-954.
[22] Song X J, Kuroha T, Ayano M, Furuta T, Nagai K, Komeda N, Segami S, Miura K, Ogawa D, Kamura T, Suzuki T, Higashiyama T, Yamasaki M, Mori H, Inukai Y, Wu J Z, Kitano H, Sakakibara H, Jacobsen S E, Ashikari M. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice[J]., 2015, 112(1): 76-81.
[23] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E B, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice[J].2015, 47(8): 944-948.
[24] Wang S K, Li S, Liu Q, Wu K, Zhang J Q, Wang S S, Wang Y, Chen X B, Zhang Y, Gao C X, Wang F, Huang H X, Fu X D. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]., 2015, 47(8): 949-954.
[25] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H, Liu Q Q.acts as a transcriptional activator to regulate rice grain shape and appearance quality[J]., 2018, 9: 1240.
[26] Li N, Xu R, Duan P G, Li Y H. Control of grain size in rice[J]., 2018, 31: 237-251.
[27] 刘喜,牟昌铃,周春雷,程治军,江玲,万建民. 水稻粒型基因克隆和调控机制研究进展[J]. 中国水稻科学,2018, 32(1): 1-11.
Liu X, Mou C L, Zhou C L, Cheng Z J, Jiang L, Wang J M. Research progress on cloning and regulation mechanism of rice grain shape genes[J]., 2018, 32(1): 1-11. (in Chinese with English abstract)
[28] Mackay T F C, Stone E A, Ayroles J F. The genetics of quantitative traits: challenges and prospects[J].2009, 10: 565-577.
[29] Kumar J, Gupta D S, Gupta S, Dubey S, Gupta P, Kumar S. Quantitative trait loci from identification to exploitation for crop improvement[J].2017, 36: 1187-1213.
[30] Yamamoto T, Yonemaru J, Yano M. Towards the understanding of complex traits in rice: substantially or superficially[J]., 2009, 16: 141-154.
[31] Zhang H W, Fan Y Y, Zhu Y J, Chen J Y, Yu S B, Zhuang J Y. Dissection of theregion into two tightly-linked minor QTLs having stable effects for grain weight in rice[J]., 2016, 17: 98.
[32] Guo L, Wang K, Chen J Y, Huang D R, Fan Y Y, Zhuang J Y. Dissection of two quantitative trait loci for grain weight linked in repulsion on the long arm of chromosome 1 of rice (L.) [J]2013, 1: 70-76.
[33] Zheng K L, Huang N, Bennett J, Khush G S. PCR-based marker-assisted selection in rice breeding//IRRI Discussion Paper Series No. 12. Los Banos: International Rice Research Institute, 1995.
[34] Chen X, Temnykh S, Xu Y, Cho Y G, McCouch S R. Development of a microsatellite framework map providing genome-wide coverage in rice(L.) [J].1997, 95: 553-567.
[35] SAS Institute Inc. SAS/STAT User’s Guide[M]. Cary, NC: SAS Institute, 1999.
[36] Dai W M, Zhang K Q, Wu J R, Wang L, Duan B W, Zheng K L, Cai R, Zhuang J Y. Validating a segment on the short arm of chromosome 6 responsible for genetic variation in the hull silicon content and yield traits of rice[J].2008, 160: 317-324.
[37] Abiola O, Angel J M, Avner P, Bachmanov A A, Belknap J K, Bennett B, Blankenhorn E P, Blizard D A, Bolivar V, Brockmann G A, Buck K J, Bureau J F, Casley W L, Chesler E J, Cheverud J M, Churchill G A, Cook M, Crabbe J C, Crusio W E, Darvasi A, Haan G D, Dermant P, Doerge R W, Elliot R W, Farber C R, Flaherty L, Flint J, Gershenfeld H, Gibson J P, Gu J, Gu W, Himmelbauer H, Hitzemann R, Hsu H C, Hunter K, Iraqi F F, Jansen R C, Johnson T E, Jones B C, Kempermann G, Lammert F, Lu L, Manly K F, Matthews D B, Medrano J F, Mehrabian M, Mittlemann G, Mock B A, Mogil J S, Montagutelli X, Morahan G, Mountz J D, Nagase H, Nowakowski R S, O'Hara B F, Osadchuk A V, Paigen B, Palmer A A, Peirce J L, Pomp D, Rosemann M, Rosen G D, Schalkwyk L C, Seltzer Z, Settle S, Shimomura K, Shou S, Sikela J M, Siracusa L D, Spearow J L, Teuscher C, Threadgill D W, Toth L A, Toye A A, Vadasz C, Van Zant G, Wakeland E, Williams R W, Zhang H G, Zou F; Complex Trait Consortium. The nature and identification of quantitative trait loci: A community’s view[J].2003, 4(11): 911-916.
[38] Wang L L, Chen Y Y, Guo L, Zhang H W, Fan Y Y, Zhuang J Y. Dissection ofto three QTLs for grain weight and grain size in rice (L.) [J]., 2015, 202: 119-127.
[39] Dong Q, Zhang Z H, Wang L L,Zhu Y J, Fan Y Y, Mou T M, Ma L Y, Zhuang J Y. Dissection and fine-mapping of two QTL for grain size linked in a 460-kb region on chromosome 1 of rice[J]., 2018, 11: 44.
[40] Wang W H, Wang L L, Zhu Y J, Fan Y Y, Zhuang J Y. Fine-mapping of, a quantitative trait locus for 1000-grain weight in rice[J]., 2019, 26(4): 220-228.
Fine Mapping of, a Minor QTL for Grain Length, Using Near Isogenic Lines Derived from Residual Heterozygotes in Rice
LI Panpan1, ZHU Yujun1, GUO Liang2, ZHUANG Jieyun1, FAN Yeyang1,*
(1State Key Laboratory of Rice Biology and Chinese National Center for Rice Improvement, China National Rice Research Institute, Hangzhou 310006, China;2Yuan Longping High-tech Agriculture Co, Changsha 410001, China;*Corresponding author, E-mail: fanyeyang@caas.cn)
The objective of this study is to fine-mapwhich was previously mapped in a 521.8-kb region on the long arm of rice chromosome 1.In this study, two BC2F9plants carryingand,respectively, was crossed and produced an F4population. Three plants carrying sequential heterozygous segments in the interval Wn28826-RM1231 were selected and selfed to develop three sets of near isogenic lines (NILs) comprising the two homozygous genotypes. The NILs in F5:6were planted in Hangzhou in 2017 and the 1000-grain weight, grain length and grain width were measured. The effect ofwas validated by two-way analysis of variance (ANOVA) using SAS program. Then six new sequential residual heterozygotes carrying smaller heterozygous segments overlapped in the interval Wn28826-RM1231 were identified. Six sets of pairwise NILs in F8:9were planted in Lingshui in 2018 and the three traits were measured. Phenotypic differences between the two homozygous genotypic groups were analyzed using ANOVA.The allelic direction ofremained consistent and the genetic effects were stable in the two trials. The Milyang 46 allele could significantly increase grain length and 1000-grain weight by 0.027 mm and 0.17 g with the contribution up to 27.12% and 19.09%, respectively.Considering thatstably showed significant effect to grain length but not to grain width in both previous and this study,was renamed asAs a result,was delimited into a 76.8-kb region flanked by Wn29077 and Wn29154 based on comparing of the segregating regions among the six sets of NILs.
rice; near isogenic line; minor effect; grain length; fine mapping
Q943.2; S511.03
A
1001-7216(2020)02-0125-10
10.16819/j.1001-7216.2020.9125
2019-11-21;
2019-12-28。
国家重点研发计划资助项目(2017YFD0100305);中央公益性科研院所基本科研业务费专项(2017RG001-2)。
